Method Article
Utilisation de méthodes peu invasives pour évaluer la consommation de carburant et les rythmes circadiens chez les personnes âgées
Dans cet article
Résumé
Un protocole novateur et peu invasif est présenté pour évaluer l’impact synergique de l’utilisation de carburant et des rythmes circadiens sur les individus vieillissants, en utilisant des cellules mononucléées du sang périphérique.
Résumé
Le vieillissement est associé à de multiples changements physiologiques qui contribuent de manière synergique et indépendante à l’incapacité physique et au risque de maladies chroniques. Bien que l’étiologie de l’incapacité physique liée à l’âge soit complexe et multifactorielle, le déclin de la fonction mitochondriale semble coïncider avec la progression du déclin fonctionnel chez de nombreuses personnes âgées. La raison pour laquelle il y a une diminution de la fonction mitochondriale avec le vieillissement reste insaisissable, mais la science émergente indique que le métabolisme des carburants et les rythmes circadiens peuvent influencer la fonction mitochondriale.
Des études récentes ont établi que les rythmes circadiens sont perturbés avec le vieillissement et que les rythmes circadiens perturbés ont des conséquences pathologiques qui ont un impact sur la fonction mitochondriale et se chevauchent avec de nombreuses maladies chroniques associées à l’âge. Les méthodes quantitatives actuelles d’évaluation directe de la fonction mitochondriale sont invasives et nécessitent généralement une biopsie musculaire, ce qui peut poser des difficultés pour le recrutement des participants et l’adhésion à l’étude, compte tenu des niveaux perçus de douleur et de risque potentiels. Ainsi, un protocole innovant et relativement non invasif pour évaluer les changements dans la fonction mitochondriale au niveau cellulaire et les schémas circadiens chez les personnes âgées a été adapté. Plus précisément, un analyseur de flux métabolique en temps réel est utilisé pour évaluer la fonction bioénergétique mitochondriale des globules blancs sous la disponibilité différentielle du substrat.
L’expression des gènes de l’horloge circadienne dans les globules blancs pour établir une corrélation croisée avec la bioénergétique mitochondriale et les résultats du rythme circadien est également analysée. On croit que ces approches méthodologiques novatrices faciliteront les futurs essais cliniques en fournissant des méthodes peu invasives pour étudier la préférence du substrat mitochondrial et les rythmes circadiens chez les personnes âgées.
Introduction
Les progrès réalisés au cours du siècle dernier ont entraîné une augmentation de l’espérance de vie et du vieillissement de la population adulte. À l’avenir, le pourcentage d’adultes âgés de 65 ans et plus devrait augmenter de 5 % entre 2020 et 2050 aux États-Unis1. Cette augmentation de l’espérance de vie n’implique pas une augmentation de l’espérance de vie - la période de la vie associée au fonctionnement indépendant. La réalité est que le vieillissement s’accompagne d’innombrables changements biologiques qui affectent le métabolisme cellulaire et la physiologie, produisant un déclin progressif du fonctionnement cognitif et physique 2,3. Alors que l’espérance de vie humaine continue d’augmenter, il est de plus en plus nécessaire de préserver la capacité fonctionnelle et l’indépendance à l’âge de4 ans.
On sait depuis longtemps que le déclin de la fonction physique et de l’indépendance avec l’âge est multifactoriel, bien qu’il soit fréquemment associé à l’apparition de maladies chroniques et d’événements déclencheurs aigus5. À l’inverse, il a été démontré que ces baisses de performance physique et de caractéristiques musculaires sont associées au développement d’un handicap avec l’âge, sans lien clair avec une seule maladie6. Compte tenu des difficultés à connaître l’étiologie exacte des maladies chroniques et de l’incapacité physique, on a pensé que les déficiences de la fonction mitochondriale coïncidaient avec l’apparition et la progression de la maladie chronique et la perte de la fonction physique chez les adultes vieillissants 7,8.
Les mitochondries fournissent la majorité de l’adénosine triphosphate (ATP), nécessaire à de nombreux processus cellulaires9. Les tissus hautement oxydatifs dépendent des mitochondries pour une production d’énergie adéquate ; avec le vieillissement, la capacité oxydative et la synthèse mitochondriale d’ATP diminuent. Ce déclin est dû en partie aux dommages oxydatifs de l’ADN mitochondrial (ADNmt), qui entraînent une accumulation progressive de mutations et de délétions de l’ADNmt10. L’accumulation de mutations et de délétions de l’ADNmt entraîne une diminution de la formation de protéines fonctionnelles de la chaîne de transport d’électrons, entraînant ainsi une réduction de la capacité des cellules à produire de l’ATP. Le déclin de la fonction mitochondriale associé à l’âge est plus notable dans les tissus hautement oxydatifs, tels que le cœur et les muscles squelettiques11. Des études ont démontré que les mitochondries du muscle gastrocnémien dans les échantillons de rats plus âgés présentent une réduction d’environ 50 % de la production et de la teneur en ATP par rapport aux échantillons plus jeunes12. De plus, il a été démontré que la capacité de production d’ATP mitochondrial dans le muscle squelettique humain diminue d’environ 8 % par décennie de lavie13. Ces résultats suggèrent que le déclin de la fonction mitochondriale lié à l’âge peut contribuer à une diminution de la production d’énergie dans les organismes.
On pense qu’un régulateur clé de l’activité mitochondriale est le coactivateur-1 du récepteur activé par les proliférateurs de peroxysomes (PPARγ) γ (PGC-1α)14. La détérioration de l’activité de PGC-1α ou une baisse de son abondance entraîne une réduction de l’activité oxydative mitochondriale et, par conséquent, une altération de la production d’énergie. De plus, une baisse de la qualité mitochondriale peut affecter la qualité des muscles squelettiques et entraîner par la suite le développement ou l’exacerbation de la sarcopénie, de la dynapénie et du déclin de la capacité fonctionnelle15,16. Les preuves du déclin concomitant de la fonction mitochondriale et de la qualité des muscles squelettiques lié à l’âge suggèrent un lien entre l’altération mitochondriale et la pathogenèse du déclin fonctionnel17. Récemment, cela a été confirmé chez des personnes âgées fonctionnelles vivant dans la communauté, montrant que la réduction du métabolisme des mitochondries des muscles squelettiques prédit le déclin de la mobilité dans cette population18. Bien que le mécanisme exact conduisant au déclin mitochondrial avec l’âge ne soit pas clair, des preuves récentes ont mis en évidence une interaction réciproque entre l’horloge circadienne et la fonction mitochondriale, avec des conséquences sur l’utilisation du carburant mitochondrial et la biogenèse19.
Consommation de carburant
La fonction mitochondriale semble être influencée par le métabolisme du carburant et le type de carburant utilisé au niveau cellulaire dans le tissu musculaire squelettique11. Pendant les périodes d’épuisement du carburant, en particulier l’épuisement des glucides chez l’homme, la préférence pour la production d’énergie (mitochondriale) change. À de faibles niveaux de glucose, la préférence de carburant s’éloigne du glucose au profit des acides gras et des corps cétoniques dérivés de l’acide. Ce changement métabolique est marqué par la régulation à la hausse du métabolisme des lipides dans les adipocytes suivie d’une libération accrue de cétones dans le sang4. Le passage de l’utilisation du glucose aux cétones avec un régime cétogène semble avoir un effet bénéfique sur la production d’espèces réactives de l’oxygène mitochondriales, la défense antioxydante, la synthèse de l’ATP et la biogenèse20.
Le passage métabolique du métabolisme des glucides au métabolisme des lipides se produit pendant les périodes de faible disponibilité des nutriments environnementaux et lorsque les réserves de glycogène ont été épuisées. Lorsque ce changement est initié, les triglycérides stockés sont décomposés en glycérol, un substrat pour la gluconéogenèse, et en acides gras libres, qui sont transportés vers le foie pour être oxydés par β-oxydation en acétyl coenzyme A (acétyl CoA). Les corps cétoniques sont synthétisés, principalement dans le foie, par une condensation en deux étapes de trois molécules d’acétyl-CoA en β-hydroxy-β-méthylglutaryl-CoA, qui sont ensuite transformées en corps cétoniques, y compris l’acétoacétate et le 3-βeta hydroxybutyrate21. Ces corps cétoniques sont distribués aux tissus de tout le corps, la consommation la plus élevée se produisant dans le cœur, le cerveau et les muscles squelettiques21. Avec le vieillissement, l’oxydation des acides gras mitochondriaux est altérée, ce qui a un impact sur le commutateur métabolique22. Il a été proposé que les déficiences de l’utilisation du carburant mitochondrial entraînent un dysfonctionnement mitochondrial supplémentaire, ce qui contribue à son tour aux maladies liées à l’âge et au déclin fonctionnel23.
Les modifications de la consommation d’oxygène mitochondrial des cellules mononucléées du sang périphérique (PBMC) ont été étudiées pour évaluer les modèles associés au dysfonctionnement et à la vascularisation. Hartman et al. ont mené une étude visant à déterminer la corrélation entre la consommation d’oxygène et la dilatation à médiation diverse, qui a suggéré un lien entre le dysfonctionnement mitochondrial et le dysfonctionnement des cellules musculaires lisses vasculaires24. En ce qui concerne d’autres organes, les PBMC ont été corrélées à un fonctionnement cognitif et cérébral supérieur, tel que déterminé par la respirométrie25. Ainsi, la bioénergétique et la capacité respiratoire des PBMC peuvent servir de biomarqueurs potentiels pour évaluer la capacité fonctionnelle des organes ou des tissus dans tout le corps.
Rythme circadien
Un autre facteur important affectant la santé mitochondriale est le rythme circadien. Les rythmes circadiens sont des oscillations de ~24 h dans le comportement et la physiologie qui se produisent en l’absence de signaux environnementaux26. Ces rythmes fonctionnent de manière prédictive pour soutenir l’homéostasie du système et des tissus. Le mécanisme qui sous-tend les rythmes circadiens est une boucle de rétroaction transcription-traduction appelée horloge circadienne27. Il a été démontré au cours des 15 dernières années que le mécanisme de l’horloge circadienne existe dans pratiquement toutes les cellules du corps28. En plus de garder l’heure, le mécanisme de l’horloge moléculaire contribue également à un programme quotidien d’expression des gènes, appelé sortie de l’horloge circadienne29. Les gènes de sortie de l’horloge sont uniques à chaque type de tissu et sont fonctionnellement associés à des voies importantes pour le métabolisme cellulaire, l’autophagie, la réparation et l’homéostasie. Des preuves récentes ont montré que la santé mitochondriale dépend de la fonction de l’horloge circadienne et influence la fonction mitochondriale, y compris la biogenèse mitochondriale, l’utilisation de carburant et la mitophagie30.
De nouvelles preuves dans des études précliniques et cliniques ont démontré que tout au long du vieillissement, il existe des perturbations des rythmes circadiens31. Il s’agit notamment de perturbations des cycles normaux de sommeil et d’éveil, d’une diminution de l’amplitude des rythmes de la température corporelle centrale et d’une capacité retardée à s’adapter aux changements de phase31. Une étude, par exemple, a mis à l’épreuve le système circadien de souris adultes et âgées (20+ mois) en décalant l’horaire d’éclairage de 6 heures. Il a été constaté que les vieilles souris mettaient plus de temps à réentraîner leurs schémas d’activité au nouveau programme de lumière32. Conformément aux changements dans le comportement circadien, l’analyse des horloges tissulaires a révélé que les horloges tissulaires centrales et périphériques étaient altérées dans la cohorte vieillissante.
Plus récemment, plusieurs groupes ont effectué une analyse transcriptomique de l’horloge circadienne et de la sortie de l’horloge dans différents tissus à l’âge de33 ans. Les résultats de ces études mettent en évidence qu’il y a une reprogrammation à grande échelle de la sortie de l’horloge circadienne avec l’âge. Cela signifie que même si l’horloge centrale maintient une fonction de synchronisation, les gènes ciblés pour l’expression quotidienne sont largement différents. Par exemple, deux études ont recueilli des biopsies musculaires de sujets humains toutes les 4 h pendant 24 h, les résultats déterminant que le pic et le creux de l’expression des gènes de l’horloge sont inversés entre les rongeurs nocturnes et les humains diurnes 34,35,36. Cela indique que lorsque l’expression du gène de l’horloge est comparée uniquement sur la phase active par rapport à la phase de repos (et non sur la lumière par rapport à l’obscurité), les modèles d’expression du gène de l’horloge dans les muscles sont pratiquement les mêmes entre les espèces. Il est proposé que ce changement de la production de l’horloge associé à l’âge entraîne des altérations de la régulation des voies qui incluent les caractéristiques connues du vieillissement, telles que la fonction mitochondriale, les dommages et la réparation de l’ADN et l’autophagie37.
Justification de l’étude
Le lien entre la fonction mitochondriale et le déclin de la fonction physique est bien établi. Cependant, la cause sous-jacente du dysfonctionnement mitochondrial reste un sujet de débat. Des recherches récentes suggèrent que l’utilisation du carburant cellulaire et les rythmes circadiens peuvent jouer un rôle dans ce processus. Les méthodes traditionnelles d’évaluation de la fonction mitochondriale, telles que la mesure de la consommation d’oxygène mitochondriale dans un échantillon de biopsie musculaire, sont souvent perçues comme douloureuses et invasives, ce qui peut décourager la participation, en particulier dans les populations à faible masse musculaire, telles que les adultes fragiles et sarcopéniques38.
Compte tenu de ces limites, il est nécessaire de disposer d’une méthode moins invasive pour évaluer les changements dans l’utilisation du carburant cellulaire et le rythme circadien chez les personnes âgées. Cette étude vise à évaluer un nouveau protocole peu invasif qui peut être utilisé pour évaluer le métabolisme des carburants et le rythme circadien dans cette population. Les résultats de cette étude contribueront à une meilleure compréhension des changements liés à l’âge et de la réponse aux interventions médicales ou comportementales, servant de modèle pour de futures études dans ce domaine.
Protocole
Les procédures impliquant des participants humains ont été approuvées par le comité d’éthique de la recherche (Florida Ethics Policy 1.0104) et le conseil d’examen institutionnel de l’Université de Floride.
1. Fonction mitochondriale
- Isolement des cellules mononucléées du sang périphérique (PBMC)
- Prélever les PBMC à l’aide de tubes de prélèvement sanguin spéciaux de 8 ml (16 mm x 125 mm, contenant soit 0,1 M d’anticoagulant à base de citrate de sodium, soit d’anticoagulant à base d’héparine sodique) (voir le tableau des matières).
REMARQUE : Les tubes de prélèvement sanguin contiennent un milieu de séparation du sang composé d’un gel de polyester thixotrope et d’un milieu dégradé (voir le tableau des matériaux) pour améliorer la séparation des globules blancs. - Traitez les tubes de préparation cellulaire de 8 mL dans les 2 h suivant le prélèvement pour obtenir des résultats viables.
- Mélangez les tubes de prélèvement sanguin après les avoir remplis de sang (soit par inversion douce, soit sur un mélangeur) et conservez-les à température ambiante (RT) pendant un maximum de 2 h.
- Centrifuger les tubes de prélèvement sanguin dans un rotor pivotant adapté à 2 000 × g pendant 15 min (ou l’équivalent de 30 000 G-min ; ne pas dépasser 2 000 × g) à RT.
REMARQUE : Ces tubes sont plus hauts qu’un tube à centrifuger ordinaire de 15 ml ; Par conséquent, une attention supplémentaire doit être accordée à l’utilisation du bon rotor. - Aspirer et jeter dans un contenant de déchets biologiques dangereux ~80 % de la couche de plasma, et prélever la couche cellulaire du tube de prélèvement sanguin (à l’aide d’une pipette de transfert) dans un tube à centrifuger de 15 ml.
REMARQUE : Lorsque vous utilisez un tube de 15 ml, ajoutez 7 ml de couche cellulaire et remplissez à 14 ml avec une solution saline tamponnée au phosphate (PBS) stérile. Le rapport entre les cellules et le PBS doit être de 1:1. Utilisez plusieurs tubes si nécessaire. Mélangez doucement en inversant. - Centrifugeuse dans un rotor pivotant à 900 × g pendant 5 min (ou l’équivalent de 4 500 G-min) à RT.
- Sans déranger la pastille de cellule, aspirez la solution PBS et jetez-la dans un conteneur de déchets biologiques dangereux.
- Remettre la pastille en suspension dans 1 mL de milieu basal (BM ; voir le tableau des matériaux) en triturant doucement. Ajouter le BM à un total de 10 ml et mélanger délicatement en l’inversant. Effectuez un comptage cellulaire.
- Centrifuger la suspension de la cellule dans un rotor pivotant à 900 × g pendant 5 min à RT.
REMARQUE : Lorsque les cellules sont utilisées le même jour pour l’évaluation bioénergétique, passez à l’étape 1.1.10 ; Si les cellules sont congelées pour être stockées, passez à l’étape 1.1.13. - Calculer le volume de milieu de dosage (AM ; voir Tableau des matériaux) pour la remise en suspension de la pastille cellulaire suivante afin d’atteindre la concentration cellulaire souhaitée.
REMARQUE : Par exemple, pour une densité d’ensemencement de 150 000 cellules/puits dans 50 μL, la concentration souhaitée de la suspension cellulaire est de 3 millions de cellules/mL. - Sans perturber la pastille de cellule, aspirez le surnageant et jetez-le dans un conteneur de déchets biologiques dangereux.
- Remettez doucement la pastille en suspension dans 1 mL ou moins le matin. Ajoutez AM au volume total calculé pour la concentration cellulaire souhaitée. Les cellules sont maintenant prêtes à être ensemencées dans une plaque de culture cellulaire revêtue (voir la table des matériaux).
REMARQUE : Les étapes suivantes concernent la congélation des cellules pour l’entreposage (1.1.13-1.1.16) et ne s’appliquent pas à la procédure du jour même. - Pour congeler les cellules, aspirez le surnageant sans déranger la pastille cellulaire et jetez-la dans un conteneur de déchets biologiques dangereux.
- Remettre doucement les cellules en suspension dans un milieu de congélation (voir tableau des matériaux) à la concentration souhaitée (5-10 millions de cellules/mL). Aliquote des volumes souhaités dans des cryoflacons étiquetés.
- Congeler lentement la suspension cellulaire à l’aide d’un récipient de congélation (voir le tableau des matériaux) pendant au moins 4 h ou toute la nuit à -80 °C.
- Après 4 heures, ou le lendemain matin, transférez les cryoflacons dans un stockage d’azote liquide, où ils devraient résider en phase vapeur.
- La veille de la mesure, enduire le fond d’une microplaque de culture cellulaire (voir Tableau des matériaux) avec de la colle cellulaire (voir Tableau des matériaux). Préparez l’adhésif selon les instructions du fabricant, à une concentration recommandée de 22,4 μg/μL, en diluant le volume approprié de solution mère adhésive dans du bicarbonate de sodium 0,1 M (pH 8,0). Appliquez 25 μL d’adhésif dans chaque puits de la microplaque de culture cellulaire.
- Après 20 min, siphonnez le liquide restant de chaque puits et lavez les puits deux fois avec 200 μL d’eau stérile et purifiée. Laissez sécher la plaque dans l’enceinte de biosécurité (pendant ~2 h).
- Enveloppez la plaque dans du papier d’aluminium ou placez-la dans un récipient secondaire fermé et conservez-la au réfrigérateur à 4 °C.
- Prélever les PBMC à l’aide de tubes de prélèvement sanguin spéciaux de 8 ml (16 mm x 125 mm, contenant soit 0,1 M d’anticoagulant à base de citrate de sodium, soit d’anticoagulant à base d’héparine sodique) (voir le tableau des matières).
- Évaluation bioénergétique des PBMC avec un analyseur de flux métabolique en temps réel
REMARQUE : À l’aide d’un analyseur de flux et du lecteur multimode (voir Table des matériaux), une densité d’ensemencement optimale de 150 000 PBMC par puits, une concentration finale de cyanure de carbonyle-4 (trifluorométhoxy) phénylhydrazone (FCCP) de 2 μM et une concentration finale de Hoechst 33342 de 4 μM (avec un temps d’incubation d’au moins 20 min après l’injection) ont été déterminées.- Pour hydrater les cartouches de capteur avec hydrobooster, retirez la cartouche de capteur avec la plaque utilitaire de la boîte et placez la cartouche « capteur » vers le haut sur le banc. Pipetez 200 μL d’étalon (voir tableau des matériaux) dans chaque puits de la plaque utilitaire et placez fermement l’hydrobooster sur la plaque utilitaire, puis la cartouche du capteur. Inspectez et éliminez toutes les bulles d’air emprisonnées, et placez-les dans un incubateur sans CO2 à 37 °C pendant la nuit.
- Allumez l’analyseur de flux et ouvrez ensuite le logiciel d’analyse de dosage (voir Tableau des matériaux). Réglez la température à 37 °C. Laissez l’instrument allumé toute la nuit pour permettre la stabilisation de la température réglée.
REMARQUE : Les étapes de programmation suivantes (1.2.4-1.2.10) peuvent être effectuées la veille ou le jour de l’expérience - Pour programmer le fichier du logiciel d’analyse de dosage (voir Table des matériaux), ouvrez le modèle pour le test de résistance à l’oxydation du substrat. Allez dans Définitions de groupe, ouvrez l’onglet Stratégies d’injection , modifiez le test de stress Inhibiteur + Sous-bœuf en Test de stress Etomoxir + Sous-Buffle, puis ajoutez deux autres stratégies d’injection : UK5099 + Test de stress Sub-Ox et BTPES + Test de stress Sub Ox. Dans chaque stratégie d’injection, dans la fenêtre Condition d’injection , cliquez sur A pour l’orifice A et entrez la concentration finale de l’inhibiteur respectif (Étomoxir : 4 μM ; UK5099 : 2 μM ; BTPES : 3 μM).
- Cliquez sur les ports B, C et D, puis entrez les concentrations finales respectives (port B : concentration d’oligomycine [1,50 μM], port C : FCCP [2,0 μM] ; port D : Roténone + Antimycine A [0,5 μM]). Ajoutez Hoechst 33342, 4 μM à la fenêtre des composés pour le port D.
- Ouvrez l’onglet Prétraitements et choisissez Prétraitements, le cas échéant. Par exemple : Contrôle, Expérimental ou habituel (par exemple, Base de référence et suivi). Ajoutez des descriptions dans la fenêtre ci-dessous si nécessaire.
- Ouvrez l’onglet Milieux de dosage et, dans le menu déroulant Milieux de dosage, choisissez Milieu RPMI, pH 7,4, entrez le numéro de lot, le personnel préparant le milieu, le moment de la préparation et les suppléments ajoutés au milieu (10 mM de glucose, 2 mM de glutamine, 1 mM de pyruvate ; voir la table des matériaux).
- Ouvrez l’onglet Type de cellule et entrez PBMC-Subject ID comme Nom, PBMC comme Type de cellule, 150000 comme Densité d’ensemencement, ID Sujet comme Source, le personnel qui a préparé les cellules, Date de préparation et Jour de dégel de la cellule, le cas échéant.
- Cliquez sur Générer des groupes, allez dans la carte des plaques et attribuez les puits de la plaque de 96 puits aux groupes respectifs. Conservez les puits A1, A12, H1 et H12 en tant que puits d’arrière-plan.
- Allez dans Protocole, cochez la case Equilibrate (paramètre par défaut) et entrez les informations suivantes : Ligne de base : cinq cycles de mesure : 3 min de mixage, 0 min d’attente et 3 min de mesure ; Milieu ou inhibiteur (port A) : six cycles de mesure : 3 min de mélange, 0 min d’attente et 3 min de mesure ; Oligomycine (port B) : trois cycles de mesure : 3 min de mélange, 0 min d’attente et 3 min de mesure ; FCCP (port C) : trois cycles de mesure : 3 min de mixage, 0 min d’attente et 3 min de mesure ; Roténone + Antimycine A + Hoechst 33342 (Port D) : trois cycles de mesure : 3 min de mélange, 0 min d’attente et 3 min de mesure.
- Le jour de l’évaluation bioénergétique, sortez la microplaque de culture cellulaire enrobée du réfrigérateur et laissez-la se réchauffer jusqu’à la RT dans l’enceinte de biosécurité.
- Préparez le milieu d’essai (AM) en mélangeant 97 mL de BM préchauffé et des suppléments : 1 mL de pyruvate (concentration finale : 1 mM), 1 mL de glucose (concentration finale 10 mM) et 1 mL de glutamine (concentration finale : 2 mM). Placez le AM à 37 °C dans un incubateur sans CO2 jusqu’à utilisation.
- Pour ensemencer les cellules, ajoutez 50 μL de suspension cellulaire (à partir de 1.1.12) contenant 150 000 PBMC dans chaque puits, à l’exception de A1, H1, A12 et H12 (désignés comme puits de fond).
CRITIQUE : La densité de semis optimale a été déterminée au préalable. - Couvrez la plaque cellulaire avec son couvercle et laissez reposer les cellules pendant 60 minutes à la RT dans l’enceinte de biosécurité pour faciliter la distribution uniforme des cellules.
CRITIQUE : Observez les cellules au microscope pour assurer une distribution homogène des cellules dans le puits. - Pour l’imagerie cellulaire, allumez le lecteur multimode (voir la Table des matières) quelques heures avant la première incubation cellulaire ; Attendez que le protocole d’initiation du lecteur soit terminé avant d’ouvrir le logiciel d’imagerie (voir la Table des matériaux). Ouvrez le logiciel d’imagerie et réglez la température de préchauffage sur 37 °C.
- Pour préparer les réactifs de dosage, suivez les instructions du kit de dosage pour reconstituer les réactifs, avec de légères modifications comme indiqué dans le tableau 1.
CRITIQUE : Les concentrations finales optimales de FCCP et de Hoechst33342 ont été déterminées précédemment. - Après 60 minutes de repos cellulaire, inspectez les puits avec un microscope de culture cellulaire et notez toute anomalie visuelle, telle que des agrégations de cellules.
- Centrifuger la plaque de culture cellulaire à RT dans une centrifugeuse avec un rotor pivotant pour microplaques à 60 × g pendant 1 min, avec une accélération minimale et une rupture minimale ; Retournez la plaque et centrifugez à nouveau à 40 × g pendant 1 min. Réglez l’accélération et la pause au minimum. Incuber les cellules pendant 25-30 min à 37 °C dans un incubateur sans CO2 .
REMARQUE : Lorsque l’imagerie cellulaire est effectuée, la plaque cellulaire est incubée dans le lecteur multimode préchauffé à la place (voir le tableau des matériaux), et une image en fond clair est prise de chaque puits pendant cette période d’incubation (première incubation cellulaire). - Scannez le code-barres de la plaque de culture cellulaire à l’aide du lecteur de codes-barres (voir Table des matériaux), sélectionnez Démarrer le balayage en fond clair, placez la plaque de culture cellulaire sur le plateau de la plaque et lancez la rétraction du plateau. Sélectionnez les puits contenant des cellules pour l’analyse et lancez l’analyse.
- Lors de la première incubation cellulaire, chargez les ports de la cartouche du capteur avec les réactifs, comme indiqué dans le tableau 1 : port (A) : 20 μL de AM (témoins), d’étomoxir (inhibiteur du transport des acides gras à longue chaîne dans les mitochondries), UK 5099 (acide 2-cyano-3-(1-phényl-1H-indol-3-yl)-2-propénoïque ; inhibiteur du transporteur mitochondrial du pyruvate), ou BPTES (Bis-2-(5-phénylacétamido-1, sulfure de 3,4-thiadiazol-2-yl)éthyle ; inhibiteur de la conversion de la glutamine en glutamate) ; port (B) : 22 μL d’oligomycine (inhibiteur de l’ATP synthase) ; orifice (C) : 25 μL de FCCP (découplement du cyanure de carbonyle-4 (trifluorométhoxy) phénylhydrazone) ; port (D) : 27 μL de roténone/antimycine A/Hoechst 33342 (inhibiteurs des complexes mitochondriaux I et III, respectivement, et colorant nucléaire). Remettez la cartouche du capteur dans l’incubateur sans CO2 à 37 °C pendant 5 minutes supplémentaires.
REMARQUE : Suivez les instructions du fabricant pour le chargement du port. - Sortez la plaque de culture cellulaire de l’incubateur ou du lecteur de plaques après la première incubation cellulaire et une fois l’imagerie cellulaire terminée (le cas échéant), et ajoutez du AM chaud dans chaque puits jusqu’à un volume final de 180 μL par puits. Placez la plaque cellulaire à 37 °C dans un incubateur sans CO2 pendant encore 15 à 25 minutes (deuxième incubation cellulaire).
- Lancez le test métabolique en commençant l’étalonnage du capteur pendant la deuxième incubation cellulaire. Cliquez sur Exécuter le test lorsque le test est prêt à être lancé avec l’étalonnage de la cartouche du capteur. Lorsque vous y êtes invité, transférez la cartouche du capteur de l’incubateur sans CO2 à 37 °C vers le plateau de l’analyseur de flux et démarrez l’étalonnage. Suivez l’invite du logiciel d’analyse de test et remplacez la plaque utilitaire de la plaque cellulaire une fois l’étalonnage terminé.
REMARQUE : L’étalonnage prend environ 20 min. - Après le test métabolique, suivez l’invite du logiciel et retirez la cartouche du capteur et la plaque cellulaire de l’instrument. Retirez la cartouche du capteur de la plaque de cellule et couvrez la plaque de cellule avec son couvercle.
- Une fois que le temps d’incubation du colorant Hoechst a été déterminé dans les expériences préliminaires, scannez le code-barres de la plaque et suivez les instructions du logiciel pour acquérir une image fluorescente de chaque puits contenant une cellule avec le lecteur multimode.
REMARQUE : Le logiciel d’imagerie et d’analyse de test est interfacé, ce qui permet d’importer les données de comptage cellulaire dans le fichier de données d’analyse de test pour la normalisation des données en numération cellulaire.
2. Expression des gènes de l’horloge circadienne
REMARQUE : L’expression des gènes de l’horloge des PBMC par les participants sera examinée en isolant l’ARN à l’aide du kit de sang d’ARN (voir la table des matériaux).
- Prélever 3 mL du sang du participant directement dans un tube d’ARN (voir le tableau des matériaux) avec 6 mL de réactifs stabilisants. Vortex pendant 10 s pour bien mélanger.
REMARQUE : Si les réactifs stabilisants ne sont pas soigneusement mélangés à l’échantillon de sang du participant, il y a un risque accru d’erreur d’étude. - Transférez 1,0 mL de l’échantillon de sang stabilisé dans un tube de 15 mL, avec 5 mL de tampon de lyse érythrocytaire. Incuber sur glace pendant 10-15 min. Vortex le tube 2x pendant l’incubation. Centrifuger le tube à 4 °C à 400 × g pendant 10 min.
- Identifiez la pastille et versez soigneusement le surnageant contenant des globules rouges lysés. Ajouter 2 mL de tampon de lyse érythrocytaire (voir le tableau des matières), remettre les cellules en suspension, agiter brièvement et centrifuger à 400 × g pendant 10 min à 4 °C.
- Videz le surnageant et laissez le tube inversé sur du papier absorbant pendant 1 à 2 min. Épongez tout liquide autour du bord du tube avec du papier propre avant l’étape suivante.
- Ajouter délicatement 600 μL de tampon de lyse de l’ARN (voir le tableau des matériaux). En suivant le protocole du fabricant, ajoutez du β-mercaptoéthanol et remettez le granulé en suspension.
- Pipeter le lysat directement dans un homogénéisateur de lysat cellulaire jetable (voir le tableau des matériaux), une colonne de centrifugation placée dans un tube de collecte de 2 mL et centrifuger pendant 2 min à vitesse maximale pour homogénéiser. Jetez la colonne de centrifugation de l’homogénéisateur de lysat cellulaire jetable et conservez le lysat homogénéisé.
- Ajouter 1 volume (600 μL) d’éthanol à 70 % dans le lysat homogénéisé et mélanger par pipetage. Pipeter soigneusement l’échantillon, y compris tout précipité qui aurait pu se former, dans une nouvelle colonne de centrifugation dans un tube de collecte de 2 ml, mais ne pas humidifier le bord. Centrifugeuse pendant 15 s à >8 000 × g.
- Transférez la colonne d’essorage (voir le tableau des matériaux) dans un nouveau tube de prélèvement de 2 ml. Appliquez 700 μL de tampon de lavage rigoureux (voir le tableau des matériaux) sur la colonne d’essorage et centrifugez pendant 15 s à >8 000 × g pour le lavage. Jetez le flux continu.
- Ouvrez soigneusement la colonne d’essorage et ajoutez 500 μL de tampon de lavage doux (voir tableau des matériaux). Fermez le bouchon et centrifugez à pleine vitesse (20 000 × g) pendant 3 min.
- Transférez la colonne de centrifugation dans un tube de microcentrifugation de 1,5 mL et pipetez 30 à 50 μL d’eau sans RNase directement sur la membrane de silice. Centrifuger pendant 1 min à >8 000 × g pour éluer l’ARN. Répétez l’opération une fois de plus.
REMARQUE : L’ARN peut être stocké à -80 °C. - Effectuer le traitement de la DNase sur une colonne à l’aide du kit de DNase sans RNase (voir Tableau des matériaux), selon le protocole du fabricant.
- Générer de l’ADNc à l’aide de 500 ng d’ARN total et d’un système de réaction en chaîne par polymérase (PCR) en temps réel (voir le tableau des matériaux), selon le protocole du fabricant. Diluez tous les échantillons d’ADNc à l’échelle 1:25 dans de l’eau exempte de RNase et utilisez 4 mL pour effectuer une PCR quantitative par transcription inverse (qRT-PCR).
- Utilisez une méthode d’ajout d’amorces (voir le tableau des matières) pour compléter la qRT-PCR avec 10 mM de chacune des amorces illustrées au tableau 2. Effectuez une qRT-PCR à l’aide d’un système en temps réel (voir le tableau des matériaux).
- Normalisez les niveaux d’ARNm des gènes choisis à l’aide des niveaux d’ARNm Rpl26 . Utilisez la méthode 2-DDCt pour calculer la quantification relative.
- Pour déterminer si l’expression d’un ARNm donné présentait une oscillation circadienne, utilisez un test de vraisemblance LR_rhythmicity39 (en utilisant p≤ 0,01) pour détecter la rythmicité circadienne dans une condition expérimentale40.
3. Plan d’analyse des données
REMARQUE : Un inventaire médical sera utilisé pour classer les participants en fonction de l’utilisation des médicaments43.
- Utilisez un modèle linéaire à effets mixtes, dans lequel l’âge, le sexe, le poids, la taille, la pression artérielle, la fréquence cardiaque et d’autres facteurs de risque doivent être inclus comme covariables.
- Un terme à effet aléatoire doit être inclus pour expliquer la corrélation des données à l’intérieur du sujet.
- Pour l’ajustement du modèle, une sélection de variable à rebours avec le principe hiérarchique doit être implémentée.
- À partir du modèle ajusté, mesurez les changements du point temporel 1 au point temporel 2, compte tenu de toutes les covariables du modèle ajusté.
Résultats
Le protocole proposé comprend des données préliminaires qui servent de validation pour la méthodologie. Le protocole intègre un analyseur de flux métabolique en temps réel pour examiner la fonction mitochondriale et l’utilisation du carburant cellulaire, ainsi que l’extraction d’ARN et la qRT-PCR pour analyser les gènes du rythme circadien (par exemple, BMAL1, CLOCK, Nfil2, Nr1d1, Dbp, Cry1, Per2).
La figure 1 présente le taux de consommation d’oxygène (OCR) de PBMC humains isolés chez cinq participants témoins, 10 jours après une analyse initiale. Les données sont utilisées pour comparer les valeurs avant et après et montrent les valeurs moyennes de la respiration basale, de la réponse aiguë, de la respiration maximale et de la capacité de réserve après l’injection d’un témoin, d’un étomoxir, d’un UK5099 et d’un BPTES. Notamment, la figure 1C montre une réponse aiguë négative significative après l’injection d’étomoxir, mais aucun effet significatif n’a été observé pour la respiration basale, la respiration maximale ou la capacité de réserve.
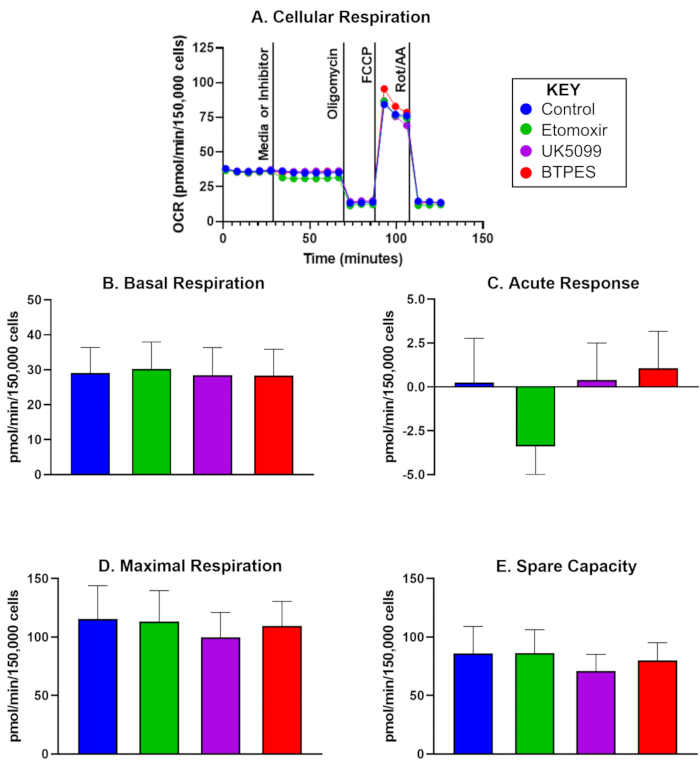
Figure 1 : Consommation d’oxygène de cellules mononucléées isolées du sang périphérique humain (PBMC) (A) Taux de consommation d’oxygène en temps réel (OCR ; pmol/(min∙150 000 cellules) de PBMC isolés d’un sujet témoin, mesuré à l’aide d’un analyseur de flux et évalué à l’aide du test d’oxydation du substrat. Les cellules ont été ensemencées à une densité de 150 000 cellules/puits. La première injection était soit un milieu (contrôle), soit un inhibiteur (etomoxir, UK5099 ou BPTES ; voir le texte pour plus de détails) et s’est produite après la mesure de la fréquence respiratoire cellulaire basale. La réponse aiguë à la limitation du substrat mitochondrial a été déterminée comme la différence d’OCR basale avant et après l’injection de l’inhibiteur. L’oligomycine, l’inhibiteur de l’ATP synthase, inhibe la respiration couplée à la production d’ATP et produit la respiration par fuite de protons. FCCP, le découpleur, induit une respiration maximale et découplée ; La roténone et l’antimycine A (inhibiteurs des complexes I et III, respectivement) inhibent toutes les respirations, sauf la respiration non mitochondriale (voir le texte pour plus de détails). (B-E) Quantification de la respiration cellulaire (n = 5 ; les données sont présentées sous forme de moyenne ± ET). (B) OCR basal avant l’injection de l’inhibiteur, (C) réponse aiguë à l’inhibiteur (variation de l’OCR par rapport au débit basal avant l’injection de l’inhibiteur), (D) OCR maximal et (E) capacité de réserve (différence entre l’OCR maximal et l’OCR basal après la première injection). La réponse aiguë (C) à l’injection d’étomoxir pourrait suggérer une dépendance plus élevée de l’OCR à l’égard des acides gras en tant que substrat énergétique dans des conditions basales par rapport aux autres groupes de substrats, sans effet notable sur l’OCR lors d’une demande énergétique élevée (D). Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Composé | AM (μL) ajouté au composé | Crosse (μM) | stock (μL) pour le stock de travail | AM (μL) pour le matériel de travail | Stock de travail (μM) | Matériel de travail (μL) [port] | Conc. final (μM) |
| Étomoxir | 700 | 160 | 500 | 1500 | 40 | 20 [A] | 4 |
| Royaume-Uni5099 | 700 | 80 | 500 | 1500 | 20 | 20 [A] | 2 |
| BPTES | 700 | 120 | 500 | 1500 | 30 | 20 [A] | 3 |
| oligo | 420 | 150 | 300 | 2700 | 15 | 22 [B] | 1.5 |
| FCCP | 720 | 100 | 600 | 2400 | 20 | 25 [C] | 2 |
| Rot/AA/H | 540 | 50 | 300 | 2700 | 5 | 27 [D] | 0.5 |
Tableau 1 : Préparation des réactifs pour l’essai d’oxydation du substrat et concentrations des solutions mères, de travail et finales. Tous les réactifs font partie du test de stress cellulaire mito ou des kits de test de stress d’oxydation du substrat. Abréviations : oligo = oligomycine ; FCCP = découpleur de cyanure de carbonyle-4 (trifluorométhoxy) phénylhydrazone ; Rot/AA/H = roténone/antimycine A/Hoechst 33342. Etomoxir, UK5099, BTPES : inhibiteurs de l’oxydation des acides gras, du glucose et de la glutamine, respectivement.
| Bmal1 | Vers l’avant – GCACGACGTTCTTTTTTGT |
| Revers – GCAGAAGCTTTTTCGATCTGCTTTT | |
| Horloge | À l’avant – CGTCTCAGACCCTCTCTCAAC |
| Inverser – GTAAATGCTGCCTGGGTGGA | |
| Pleur1 | Vers l’avant – ACTGCTATTGCCCTGTTGGT |
| Inverser – GACAGGCAAATAACGCCTGA | |
| Par 1 | À l’avant – ATTCGGGTTACGAAGCTCCC |
| Revers – GGCAGCCCTTTCATCCACAT | |
| Per2 | Vers l’avant – CATGTGCAGTGGAGCAGATTC |
| Revers – GGGGTGGTAGCGGATTTCAT | |
| Rev-erb α | À l’avant – ACAGATGTCAGCAATGTCGC |
| Revers – CGACCAAACCGAACAGCATC |
Tableau 2 : Amorces du gène de l’horloge circadienne.
Discussion
Le déclin de la fonction mitochondriale et la régulation du rythme circadien avec l’âge sont de plus en plus considérés comme des facteurs contribuant aux maladies liées à l’âge. La modification des rythmes circadiens par des modifications du mode de vie, comme l’alimentation et l’activité physique, représente une stratégie potentielle pour promouvoir un vieillissement en santé et réduire le déclin de la mobilité associé au vieillissement. Cependant, les méthodes actuelles d’évaluation directe de la fonction mitochondriale sont invasives et nécessitent souvent une biopsie musculaire, ce qui peut poser des problèmes de recrutement et de rétention des participants en raison de la douleur et des risques perçus.
L’évaluation des marqueurs de la santé circadienne et métabolique à l’aide de méthodes moins invasives, telles que la collecte de sang, fournirait des résultats précieux pour l’exploration et la mise à l’essai de cibles thérapeutiques dans le cadre d’études futures. Ces méthodes peu invasives ont le potentiel de faire progresser considérablement le domaine en fournissant de nouvelles informations sur l’interaction complexe entre le rythme circadien et la santé métabolique et leur impact sur la fonction. Le but de cette étude est d’évaluer la relation entre le métabolisme énergétique cellulaire et le rythme circadien. En particulier, l’analyse du flux bioénergétique est utilisée pour évaluer la fonction mitochondriale dans diverses conditions de disponibilité du substrat, ainsi que la surveillance de l’expression génique d’un groupe de gènes circadiens dans les globules blancs des participants. En utilisant les deux bras de l’analyse, la bioénergie et l’expression des gènes, une compréhension complète de la relation entre ces deux processus fondamentaux peut être obtenue.
L’analyse statistique de ces données de séries chronologiques d’un point de vue circadien donne un aperçu de la force, de l’amplitude et du moment des rythmes circadiens. En conclusion, l’intégration de l’analyse de l’expression génique, de la bioénergétique cellulaire et des mesures métaboliques au niveau de l’organisme constitue une approche nouvelle et innovante qui mettra en lumière l’interaction entre le métabolisme énergétique et les rythmes circadiens chez l’homme.
Dans une étude pilote, nous avons détecté une réponse aiguë dans l’OCR des PBMC à la limitation de l’utilisation des acides gras (suite à l’injection d’étomoxir, un inhibiteur de la carnitine palmitoyl transférase 1a). Cette découverte suggère que chez les PBMC de ce groupe particulier de participants, il pourrait y avoir une dépendance aux acides gras comme substrat énergétique pendant la respiration basale. Cependant, la respiration maximale n’a pas été affectée, ce qui suggère que d’autres sources d’énergie, telles que le glucose et la glutamine, peuvent compenser l’utilisation réduite des acides gras lors d’une forte demande énergétique. Des études futures devraient déterminer si a) la bioénergétique des PBMC reflète l’énergie du corps entier et b) si des interventions telles que l’alimentation limitée dans le temps pourraient affecter les préférences en matière de substrat énergétique.
L’analyse des flux des PBMC comporte plusieurs étapes critiques. Tout d’abord, avant d’évaluer les échantillons expérimentaux, la densité d’ensemencement cellulaire (cellules par puits) doit être optimisée en s’assurant qu’il y a une distribution uniforme continue des cellules dans chaque puits et sur chaque plaque, la concentration finale de FCCP doit être optimisée en effectuant des tests de concentrations en utilisant les concentrations 0, 0,125, 0,25, 0,5, 1,0 et 2,0 μM, et, le cas échéant, la coloration Hoechst 33342 doit être optimisée en suivant les instructions du fabricant. Deuxièmement, la normalisation des données métaboliques en fonction des paramètres cellulaires est essentielle pour la comparabilité des données entre les expériences. Dans le présent protocole, le comptage des cellules après la conclusion du test de l’analyseur de flux utilisant des cellules colorées au Hoechst 33342 et un dispositif d’imagerie cellulaire est décrit. Si un dispositif approprié n’est pas disponible, d’autres méthodes de normalisation peuvent être appliquées, telles que la teneur en protéines cellulaires totales ou en ADN nucléaire par puits. Il y a une modification notable qui peut être utilisée dans le protocole, par rapport à celles qui ont été proposées. Plus précisément, le protocole peut être complété à l’aide d’un kit de dosage individuel pour chacun des trois inhibiteurs, par rapport aux deux kits proposés ici (voir le tableau des matériaux).
L’utilisation des PBMC comme substitut pour étudier l’interaction entre le métabolisme énergétique et les rythmes circadiens chez les personnes âgées est limitée par l’hypothèse que leur réponse au traitement peut refléter avec précision la réponse dans d’autres tissus et organes. Bien que cette approche soit nouvelle et peu invasive, il est important de reconnaître que différents tissus et organes, tels que le cerveau, le foie et les muscles squelettiques, peuvent réagir différemment dans diverses conditions. Une étude préclinique a démontré que l’expression du gène de l’horloge était altérée chez les souris nourries et à jeun, conduisant à la régulation positive partielle des gènes cibles de BMAL1 dans les tissus hépatiques et musculaires, mais à la régulation négative d’autres41. Ces tissus et organes périphériques sont très représentatifs des processus métaboliques et peuvent être influencés par des signaux environnementaux qui ont un impact sur les mécanismes d’expression des gènes de l’horloge42. Des recherches supplémentaires sont nécessaires pour bien comprendre la relation entre les tissus périphériques, les organes et l’horloge circadienne centrale.
Une autre limitation est que les participants ne sont pas disqualifiés pour prendre une ordonnance, ce qui pourrait poser des limites à l’analyse statistique. Pour contrer cette limitation, dans les recherches futures, un inventaire médical sera utilisé, qui a été validé dans des populations de personnes âgées qui prennent des médicaments43. Les participants seront classés en fonction des médicaments recommandés enregistrés dans la section d’examen des données du protocole. Il existe un total de trois catégories, en termes de médicaments, dont il a été démontré qu’ils 1) accélèrent le déclin fonctionnel, 2) ralentissent le déclin fonctionnel et 3) influencent la fonction des muscles squelettiques.
Enfin, la capacité oxydative mitochondriale du muscle squelettique humain présente un rythme jour-nuit, culminant entre 18h00 et 23h00 et diminuant entre 08h00 et 11h0044. Il n’est pas encore clair si cela vaut pour la capacité oxydative mitochondriale des PBMC. Cependant, des données préliminaires suggèrent que les PBMC et le métabolisme mitochondrial sont liés45. Étant donné que l’information sur les biopsies musculaires et les altérations des PBMC n’est pas aussi claire, il faut faire preuve de prudence lors de l’analyse des résultats. Compte tenu de cette limite, il est important de garder cette information à l’esprit lors de l’évaluation et de l’élaboration d’un protocole, car elle peut fournir un contexte et des informations précieux qui pourraient aider à assurer la validité et l’efficacité du protocole.
À notre connaissance, aucune étude antérieure n’a évalué les modèles d’utilisation du carburant ou les rythmes circadiens à l’aide des méthodes proposées dans ce projet. Notre objectif est d’examiner la réactivité des marqueurs de l’utilisation du carburant mitochondrial et de la santé circadienne aux changements. Cette étude présente une méthode peu invasive pour mesurer un biomarqueur très sensible, qui peut servir d’alternative dans de futures études interventionnelles où la biopsie musculaire n’est pas réalisable.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Cette étude a été financée par le Older American’s Independence Center (NIH/NIA P30AG028740), avec l’aide du Clinical and Translational Science Institute (NIH/NCRR UL1TR000064).
matériels
| Name | Company | Catalog Number | Comments |
| Agilent Technologies Cell Imaging | Agilent Technologies | Cell image software | |
| Agilent/Seahorse Wave desktop program | Agilent Technologies | 5994-0039EN | Software used to analyse data from the celluar analyser and stress test assay |
| Agilent/Seahorse XFe96 Flux Analyzer | Agilent Technologies | S7800B | Real-time cellular flux analyzer; flux analyser |
| Bar Code Reader | Agilent Technologies | G2615-90007 | |
| Seahorse Wave Desktop Software | Agilent technologies | Data acquisition software; assay analysis; wave program | |
| Seahorse XF 1.0 M Glucose solution | Agilent Technologies | 103577-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 100 mM Pyruvate solution | Agilent Technologies | 103578-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 200 mM Glutamine solution | Agilent Technologies | 103579-100 | Supplement to basal medium to make assay medium |
| Seahorse XF Cell Mito Stress Test | Agilent Technologies | 103015-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF Mito Fuel Flex Test | Agilent Technologies | 103260-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF RPMI Medium | Agilent Technologies | 103576-100 | Basal medium for PBMCs |
| Seahorse XFe96 FluxPak mini | Agilent Technologies | 102601-100 | Sensor cartridges and cell culture microplates |
| Cytation 1 Cell Imaging Multi-Mode Reader | Agilent/BioTek | Multimode reader to image cells | |
| CPT Sodium Heparin Tube, 16 x 125 mm x 8.0 mL | Becton Dickinson | 362753 | Blood collection tubes for isolation of peripheral blood mononuclear cells |
| CellTak Cell and Tissue Adhesive | Corning | 354240 | Cell adherent to coat cell culture microplate |
| Phosphate Buffered Saline | Corning | 21-040-CV | Buffer to wash blood cells |
| Ficoll Paque Plus | Cytiva | GE17-1440-02 | Gradient medium |
| Lunar Prodigy DXA scanner | General Electric | EN 60601-2-7 5.1 | Whole body lean mass and fat/lean tissue mass ratio |
| Freezing container, Nalgene Mr. Frosty | MilliporeSigma | C1562 | Freezing container used to slow-freeze cell suspension |
| Buffer EL. | Qiagen | 79217 | Erythrocyte lysis buffer |
| Buffer RLT | Qiagen | 79216 | RNA lysis buffer |
| Buffer RPE | Qiagen | 1018013 | Mild washing buffer |
| Buffer RW1 | Qiagen | 1053394 | Stringent washing buffer |
| QIAamp DNA Micro Kit | Qiagen | 56304 | DNA preps: QIAamp MinElute Columns, Proteinase K, Carrier RNA, Buffers, Collection Tubes (2 ml) |
| QIAamp RNA Blood Mini Kit | Qiagen | 52304 | RNA blood kit; Used to isolate RNA |
| QIAshredder | Qiagen | 79656 | disposable cell-lysate homogenizers |
| RNase-Free DNase Set | Qiagen | 79254 | Used to perform DNA digest |
| 2-Mercaptoethanol (Reagent) | Thermo Fisher Scientific | MFCD00004890 | |
| 2-mL collection tubes, 100 count | Thermo Fisher Scientific | AM12480 | |
| Fast SYBR Green Master Mix | Thermo Fisher Scientific | 4385612 | Primers are added to this and used to carry out qRT-PCR |
| Microcentrifuge Tubes, 1.5 mL | Thermo Fisher Scientific | 69715 | Used to hold RNA purification filter during RNA purification |
| Narrow p1000 pipette tips | Thermo Fisher Scientific | 02-707-402 | |
| QuantStudio 3 Real-Time PCR System, MiniAmp Plus Thermal Cycler, and 96-Well Plates Package | Thermo Fisher Scientific | A40393 | |
| Tempus Blood RNA Tube | Thermo Fisher Scientific | 4342792 | RNA Tube |
| Tempus Spin RNA Isolation kit | Thermo Fisher Scientific | 4380204 | RNA extraction and isolation |
Références
- UN Department of Economic and Social Affairs. Population Division 2019, World Population Prospects. UN Department of Economic and Social Affairs. , (2019).
- Anton, S., Leeuwenburgh, C. Fasting or caloric restriction for healthy aging. Experimental Gerontology. 48 (10), 1003-1005 (2013).
- Dziechciaż, M., Filip, R. Biological psychological and social determinants of old age: Bio-psycho-social aspects of human aging. Annals of Agricultural and Environmental Medicine. 21 (4), 835-838 (2014).
- Anton, S. D., et al. Flipping the metabolic switch: understanding and applying the health benefits of fasting. Obesity. 26 (2), 254-268 (2018).
- Fried, L. P., Guralnik, J. M. Disability in older adults: evidence regarding significance, etiology, and risk. Journal of the American Geriatrics Society. 45 (1), 92-100 (1997).
- Manini, T. Development of physical disability in older adults. Current Aging Science. 4 (3), 184-191 (2011).
- Chung, H. Y., et al. Molecular inflammation: underpinnings of aging and age-related diseases. Ageing Research Reviews. 8 (1), 18-30 (2009).
- Sun, N., Youle, R. J., Finkel, T. The mitochondrial basis of aging. Molecular Cell. 61 (5), 654-666 (2016).
- Tarasov, A. I., Griffiths, E. J., Rutter, G. A. Regulation of ATP production by mitochondrial Ca2. Cell Calcium. 52 (1), 28-35 (2012).
- Chistiakov, D. A., Sobenin, I. A., Revin, V. V., Orekhov, A. N., Bobryshev, Y. V. Mitochondrial aging and age-related dysfunction of mitochondria. Biomed Research International. 2014, 238463 (2014).
- Boengler, K., Kosiol, M., Mayr, M., Schulz, R., Rohrbach, S. Mitochondria and ageing: role in heart, skeletal muscle and adipose tissue. Journal of Cachexia, Sarcopenia, and Muscle. 8 (3), 349-369 (2017).
- Drew, B., et al. Effects of aging and caloric restriction on mitochondrial energy production in gastrocnemius muscle and heart. American Journal of Physiology Regulatory, Integrative and Comparative Physiology. 284 (2), R474-R480 (2003).
- Short, K. R., et al. Decline in skeletal muscle mitochondrial function with aging in humans. Proceedings of the National Academy of Sciences. 102 (15), 5618-5623 (2005).
- Musci, R. V., Hamilton, K. L., Miller, B. F. Targeting mitochondrial function and proteostasis to mitigate dynapenia. European Journal of Applied Physiology. 118 (1), 1-9 (2018).
- Picca, A., et al. Targeting mitochondrial quality control for treating sarcopenia: lessons from physical exercise. Expert Opinion on Therapeutic Targets. 23 (2), 153-160 (2019).
- Fernandez-Marcos, P. J., Auwerx, J. Regulation of PGC-1α, a nodal regulator of mitochondrial biogenesis. The American Journal of Clinical Nutrition. 93 (4), 884-890 (2011).
- Kim, Y., Triolo, M., Hood, D. A. Impact of aging and exercise on mitochondrial quality control in skeletal muscle. Oxidative Medicine and Cellular Longevity. 2017, 3165396 (2017).
- Wang, H., Hiatt, W. R., Barstow, T. J., Brass, E. P. Relationships between muscle mitochondrial DNA content, mitochondrial enzyme activity and oxidative capacity in man: alterations with disease. European Journal of Applied Physiology and Occupational Physiology. 80 (1), 22-27 (1999).
- Tian, Q., et al. Muscle mitochondrial energetics predicts mobility decline in well-functioning older adults: The baltimore longitudinal study of aging. Aging Cell. 21 (2), e13552 (2022).
- Sardon Puig, L., Valera-Alberni, M., Cantó, C., Pillon, N. J. Circadian rhythms and mitochondria: connecting the dots. Frontiers in Genetics. 9, 452 (2018).
- Gano, L. B., Patel, M., Rho, J. M. Ketogenic diets, mitochondria, and neurological diseases. Journal of Lipid Research. 55 (11), 2211-2228 (2014).
- Liesa, M., Shirihai, O. S. Mitochondrial dynamics in the regulation of nutrient utilization and energy expenditure. Cell Metabolism. 17 (4), 491-506 (2013).
- Lesnefsky, E. J., Chen, Q., Hoppel, C. L. Mitochondrial metabolism in aging heart. Circulation Research. 118 (10), 1593-1611 (2016).
- Hartman, M. L., et al. Relation of mitochondrial oxygen consumption in peripheral blood mononuclear cells to vascular function in type 2 diabetes mellitus. Vascular Medicine. 19 (1), 67-74 (2014).
- Mahapatra, G., et al. Blood-based bioenergetic profiling is related to differences in brain morphology in African Americans with Type 2 diabetes. Clinical Science. 132 (23), 2509-2518 (2018).
- Moore-Ede, M. C. Physiology of the circadian timing system: predictive versus reactive homeostasis. The American Journal of Physiology. 250 (5), R737-R752 (1986).
- Young, M. W. Life's 24-hour clock: molecular control of circadian rhythms in animal cells. Trends in Biochemical Sciences. 25 (12), 601-606 (2000).
- Yoo, S. H., et al. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proceedings of the National Academy of Sciences. 101 (15), 5339-5346 (2004).
- Zhang, R., Lahens, N. F., Ballance, H. I., Hughes, M. E., Hogenesch, J. B. A circadian gene expression atlas in mammals: implications for biology and medicine. Proceedings of the National Academy of Sciences. 111 (45), 16219-16224 (2014).
- de Goede, P., Wefers, J., Brombacher, E. C., Schrauwen, P., Kalsbeek, A. Circadian rhythms in mitochondrial respiration. Journal of Molecular Endocrinology. 60 (3), R115-R130 (2018).
- Hood, S., Amir, S. The aging clock: circadian rhythms and later life. The Journal of Clinical Investigation. 127 (2), 437-446 (2017).
- Sellix, M. T., et al. Aging differentially affects the re-entrainment response of central and peripheral circadian oscillators. The Journal of Neuroscience. 32 (46), 16193-16202 (2012).
- Sato, S., et al. Circadian reprogramming in the liver identifies metabolic pathways of aging. Cell. 170 (4), 664-677 (2017).
- Lundell, L. S., et al. et al. feeding alters lipid and amino acid metabolite rhythmicity without perturbing clock gene expression. Nature Communications. 11 (1), 4643 (2020).
- Perrin, L., et al. Transcriptomic analyses reveal rhythmic and CLOCK-driven pathways in human skeletal muscle. eLife. 7, e34114 (2018).
- Gutierrez-Monreal, M. A., Harmsen, J. -. F., Schrauwen, P., Esser, K. A. Ticking for metabolic health: the skeletal-muscle clocks. Obesity. 28, S46-S54 (2020).
- Wolff, C. A., et al. Defining the age-dependent and tissue-specific circadian transcriptome in male mice. bioRxiv. 42 (1), 111982 (2023).
- Wilson, D., Breen, L., Lord, J. M., Sapey, E. The challenges of muscle biopsy in a community based geriatric population. BMC Research Notes. 11 (1), 830 (2018).
- Ding, H., et al. Likelihood-based tests for detecting circadian rhythmicity and differential circadian patterns in transcriptomic applications. Briefings in Bioinformatics. 22 (6), 224 (2021).
- Ding, Z., Lamb, T. M., Boukhris, A., Porter, R., Bell-Pedersen, D. Circadian clock control of translation initiation factor eIF2α activity requires eIF2γ-dependent recruitment of rhythmic PPP-1 phosphatase in Neurospora crassa. mBio. 12 (3), e00871 (2021).
- Di Francesco, A., Di Germanio, C., Bernier, M., de Cabo, R. A time to fast. Science. 362 (6416), 770-775 (2018).
- Kalfalah, F., et al. Crosstalk of clock gene expression and autophagy in aging. Aging. 8 (9), 1876-1895 (2016).
- Psaty, B. M., et al. Assessing the use of medications in the elderly: methods and initial experience in the cardiovascular health study. Journal of Clinical Epidemiology. 45 (6), 683-692 (1992).
- van Moorsel, D., et al. Demonstration of a day-night rhythm in human skeletal muscle oxidative capacity. Molecular Metabolism. 5 (8), 635-645 (2016).
- Janssen, J. J. E., et al. Extracellular flux analyses reveal differences in mitochondrial PBMC metabolism between high-fit and low-fit females. American Journal of Physiology. Endocrinology and Metabolism. 322 (2), E141-E153 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon