Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Méthodes optimisées pour l’immobilisation de surface des collagènes et les dosages de liaison du collagène
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce travail présente un protocole optimisé pour immobiliser et quantifier de manière reproductible le collagène de type I et III sur des microplaques, suivi d’un protocole de test de liaison in vitro amélioré pour étudier les interactions collagène-composé à l’aide d’une méthode de fluorescence résolue en temps. L’analyse et l’interprétation des données sont fournies étape par étape.
Résumé
La fibrose se produit dans divers tissus en réponse réparatrice à une blessure ou à un dommage. Cependant, si elle est excessive, la fibrose peut entraîner une cicatrisation des tissus et une défaillance des organes, ce qui est associé à une morbidité et une mortalité élevées. Le collagène est un facteur clé de la fibrose, le collagène de type I et de type III étant les principaux types impliqués dans de nombreuses maladies fibrotiques. Contrairement aux protocoles conventionnels utilisés pour immobiliser d’autres protéines (par exemple, l’élastine, l’albumine, la fibronectine, etc.), il n’est pas facilement disponible des protocoles complets permettant d’immobiliser de manière reproductible différents types de collagènes afin de produire des revêtements stables. L’immobilisation du collagène est étonnamment difficile car de multiples conditions expérimentales peuvent affecter l’efficacité de l’immobilisation, notamment le type de collagène, le pH, la température et le type de microplaque utilisé. Ici, un protocole détaillé pour immobiliser et quantifier de manière reproductible les collagènes de type I et III, ce qui permet d’obtenir des gels/films stables et reproductibles, est fourni. De plus, ce travail démontre comment réaliser, analyser et interpréter in vitro des études de liaison à la fluorescence résolues dans le temps pour étudier les interactions entre les collagènes et les composés candidats de liaison au collagène (par exemple, un peptide conjugué à un chélate métallique portant, par exemple, de l’europium [Eu(III)]). Une telle approche peut être universellement appliquée à diverses applications biomédicales, y compris le domaine de l’imagerie moléculaire pour développer des sondes d’imagerie ciblées, le développement de médicaments, les études de toxicité cellulaire, les études de prolifération cellulaire et les immunoessais.
Introduction
L’accumulation de tissu conjonctif fibreux dans le cadre du processus naturel de cicatrisation des plaies après une lésion tissulaire est connue sous le nom de fibrose. Cependant, si le dépôt de tissu fibreux ne se termine pas et se poursuit au-delà de ce qui est nécessaire pour la réparation tissulaire, la fibrose devient excessive 1,2. Une fibrose excessive altère la physiologie et la fonction des organes et pourrait entraîner des lésions organiques et potentiellement une défaillance des organes3,4,5. Les deux principaux facteurs de fibrose sont les protéines de la matrice extracellulaire (MEC), le collagène de type I et de type III,6. Le collagène est une protéine structurelle présente dans divers organes qui représente environ un tiers de la teneur totale en protéines du corps humain1. Il existe 28 types différents de collagènes identifiés par le séquençage du génome humain, et les plus abondants sont les collagènes fibrillaires7. Le collagène fibrillaire primaire est le collagène de type I, qui confère à l’ECM une résistance à la traction et une résistance à la déformation8. Le collagène de type III est un composant structurel qui apporte de l’élasticité et se colocalise avec le collagène de type I. Il s’exprime au cours de l’embryogenèse et se trouve naturellement en petites quantités dans la peau, les muscles et les vaisseaux sanguins des adultes9.
La synthèse in vivo du collagène commence par un processus intracellulaire dans lequel l’ARNm est transcrit dans le noyau, puis se déplace vers le cytoplasme, où il est traduit. Après la traduction, la chaîne formée subit une modification post-traductionnelle dans le réticulum endoplasmique, où se forme le pro-collagène (le précurseur du collagène). Le pro-collagène se rend ensuite dans l’appareil de Golgi pour une modification finale avant d’être excrété dans l’espace extracellulaire10. Par clivage protéolytique, le pro-collagène se transforme en tropocollagène. Celle-ci est ensuite réticulée soit par une voie de réticulation médiée par l’enzyme catalysée par l’enzyme lysyl oxydase (LOX), soit par une voie de réticulation non enzymatique impliquant la réaction de Maillard11. Les protocoles in vitro d’immobilisation du collagène reposent principalement sur la capacité du collagène à s’auto-assembler. Le collagène est extrait des tissus en fonction de sa solubilité, qui dépend en grande partie de l’étendue de la réticulation des fibrilles de collagène individuelles7. Le collagène fibrillaire est dissous dans l’acide acétique, et les fibrilles peuvent se reformer lorsque le pH et la température sont ajustésà 12. In vitro, la fibrillogenèse du collagène peut être considérée comme un processus en deux étapes7. La première étape est la phase de nucléation, où les fibres de collagène forment des fibrilles dimères et trimères avant d’être réarrangées pour former une structure hélicoïdale triple. La deuxième phase est la phase de croissance, où les fibrilles commencent à se développer latéralement et entraînent la formation caractéristique de la bande D, qui est généralement observée par des changements de turbidité7. Des études de microscopie à force atomique (AFM) ont également révélé que le collagène de type I et de type III ont des caractéristiques différentes (Tableau 1)13.
Pour étudier les interactions de liaison entre le collagène et d’autres composés, le collagène doit être immobilisé de manière reproductible dans les puits de microplaques. Il existe différents protocoles pour immobiliser le collagène soluble 14,15,16. Les microplaques disponibles dans le commerce qui sont pré-recouvertes de collagène sont généralement utilisées pour la culture cellulaire. Cependant, les microplaques pré-enrobées ont une très fine couche d’une quantité inconnue de collagène enrobée sur les puits, ce qui les rend inadaptées aux tests de liaison in vitro. Il y a plusieurs défis lors de l’immobilisation du collagène sur les puits de la plaque. L’un des principaux défis est de choisir un type de microplaque approprié, car les différents types de collagènes (par exemple, les types I et III) ont des propriétés chimiques différentes et, par conséquent, s’immobilisent de manière plus stable et plus efficace en fonction du matériau de la microplaque. Un autre défi est les conditions expérimentales du protocole d’immobilisation, car le processus de fibrillogenèse dépend de plusieurs facteurs, notamment la température, le pH, la concentration du stock de collagène et la concentration ionique du tampon7.
Pour étudier les interactions entre le collagène (la cible) et d’autres composés (c’est-à-dire un peptide cible), il est également nécessaire de développer un test de criblage robuste pour étudier la spécificité et la sélectivité du composé vis-à-vis de la cible en mesurant la constante de dissociation, Kd. La position de l’équilibre de formation d’un complexe bimoléculaire entre une protéine (collagène) et un ligand est exprimée en fonction de la constante d’association Ka, dont l’amplitude est proportionnelle à l’affinité de liaison. Cependant, le plus souvent, les biochimistes expriment les relations d’affinité en termes de constante de dissociation à l’équilibre, Kd, du complexe bimoléculaire, qui est défini comme Kd = 1/Ka (Kd et est l’inverse de Ka). Plus la valeur Kd est faible, plus la force de liaison entre la protéine et le ligand est forte. L’avantage d’utiliser Kd pour comparer l’affinité de liaison de différents ligands pour la même protéine (et inversement) est lié au fait que les unités de Kd pour un complexe bimoléculaire sont mol/L (c’est-à-dire l’unité de concentration). Dans la plupart des conditions expérimentales, la valeurK d correspond à la concentration du ligand qui conduit à une saturation de 50 % des sites de liaison disponibles sur la cible à l’équilibre17,18. La constante de dissociation est généralement extraite en analysant l’occupation fractionnée du récepteur (FO), qui est définie comme le rapport entre les sites de liaison occupés et le total des sites de liaison disponibles, en fonction de la concentration du ligand. Cela peut être fait à condition qu’un test analytique capable de distinguer et de mesurer la quantité de ligand lié soit disponible.
Les tests de liaison de ligands in vitro peuvent être réalisés à l’aide de diverses méthodes bioanalytiques, notamment la photométrie optique, les méthodes de radioligands, la spectrométrie de masse à plasma à couplage inductif (ICP-MS) et la résonance plasmonique de surface (SPR). Parmi les méthodes photométriques, celles basées sur l’émission de fluorescence nécessitent généralement le marquage de ligands ou de protéines avec des fluorophores pour augmenter la sensibilité et améliorer la limite de détection du dosage. Les chélatés de certains ions lanthanide(III), tels que Eu(III), sont très attrayants en tant que fluorophores car ils ont de grands décalages de Stokes, des bandes d’émission étroites (offrant un bon rapport signal/bruit), un photoblanchiment limité et de longues durées de vie d’émission. Il est important de noter que cette dernière propriété permet d’utiliser la fluorescence résolue en temps (TRF) à partir de fluorophores Eu(III) pour abolir l’autofluorescencede fond 19. Dans la version DELFIA (Dissociation-enhanced lanthanide fluorescent immunoassay) du test TRF basé sur Eu(III), des ligands marqués avec un chélate Eu(III) non luminescent sont incubés avec le récepteur immobilisé sur des microplaques. Le complexe ligand/récepteur marqué est séparé du ligand non lié, et la fluorescence Eu(III) est activée par dissociation du complexe Eu(III) à un pH acide, suivie d’une recomplexation avec un chélateur améliorant la fluorescence pour former un complexe Eu(III)20 hautement fluorescent intégré dans la micelle.
L’étape de décomplexation peut être raisonnablement réalisée avec des chélateurs, tels que le pentaacétate de diéthylènetriamine (DTPA), qui présentent une cinétique de décomplexation rapide. Cependant, les complexes Eu(III) avec certains chélateurs macrocycliques, tels que DOTA (acide 1,4,7,10-tétraazacyclododécane1,4,7,10-tétraacétique) et ses dérivés monoamides (DO3AAm), présentent une grande stabilité thermodynamique et une inertie cinétique très élevée. Dans ce cas, les étapes de décomplexation doivent être optimisées avec précision pour obtenir une activation suffisante et reproductible du TRF21 à base d’Eu(III). Il convient de noter que les complexes de lanthanides (Ln(III))-DOTA et Ln(III)-DO3AAm sont ceux qui sont les plus couramment utilisés comme agents de contraste pour l’imagerie moléculaire in vivo par les techniques d’imagerie par résonance magnétique (IRM)22. Ainsi, le test TRF basé sur Ln(III) est l’outil de choix pour étudier in vitro l’affinité de liaison des sondes moléculaires IRM avec leurs cibles biologiques prévues. À l’heure actuelle, il n’existe pas de protocoles complets et reproductibles pour l’immobilisation du collagène de type I et de type III, ni d’un pipeline reproductible pour réaliser des expériences de liaison in vitro Eu(III) TRF. Pour surmonter ces limitations, des méthodes reproductibles ont été développées pour auto-assembler et immobiliser le collagène de type I et de type III et générer des gels et des films stables, respectivement, avec la concentration suffisante de collagène requise pour les essais de liaison in vitro . Un protocole optimisé pour Eu(III) TRF de complexes hautement inertes à base d’Eu(III)-DO3Aam est présenté. Enfin, un test TRF in vitro optimisé sur microplaque Eu(III) pour mesurer le Kd des ligands marqués à Eu(III) vers le collagène de type I et de type III immobilisé est démontré (Figure 1).
Protocole
REMARQUE : Toutes les informations sur le produit utilisées pour ce travail sont présentées dans la table des matériaux.
1. Immobilisation du collagène
REMARQUE : Assurez-vous que chaque puits de la microplaque utilisé pendant l’essai de liaison a des puits adjacents libres pour éviter la fluorescence croisée. Effectuez cette partie du protocole sur de la glace car le collagène s’auto-assemble à des températures et des niveaux de pH élevés. Effectuez cette procédure dans une hotte de culture tissulaire et dans des conditions stériles, car les microplaques sont ensuite incubées dans un incubateur de culture tissulaire (CT).
- Immobilisation du collagène de type I sur les microplaques à 96 puits (Figure 2)
Jour 1- Préparez un plateau en silicone avec de la glace. Placez le flacon contenant du collagène de type I, la solution saline froide tamponnée au phosphate (PBS) 10x et les microplaques sur de la glace, et vaporisez le tout avec de l’éthanol à 70 %. Placez le matériau sous le capot TC.
- Neutralisez le collagène en utilisant des volumes égaux de collagène de type I et 10x PBS (pH 7,4).
- Retournez la solution plusieurs fois, en vous assurant qu’aucune bulle ne se forme.
- Ajoutez 100 μL de collagène neutralisé dans tous les autres puits et toutes les autres rangées de la microplaque, et incubez à 37 °C pendant 18 à 20 h pour évaporer le collagène jusqu’à ce qu’il soit sec.
Jour 2 - Lavez les microplaques avec 100 μL de 1x PBS, pH 7,4, deux fois pour éliminer tout collagène non lié.
- Transférez les microplaques dans l’incubateur à 37 °C pendant encore 2 h pour qu’elles sèchent avant de les utiliser pour d’autres expériences de liaison.
- Immobilisation du collagène de type III sur les microplaques à 96 puits (Figure 3)
Jour 1- Préparez un plateau en silicone avec de la glace. Placez le flacon contenant du collagène de type III, le PBS froid 10x et les microplaques sur de la glace, et vaporisez le tout avec de l’éthanol à 70 %. Placez le matériau sous le capot TC.
- Neutralisez le collagène en utilisant des volumes égaux de collagène de type III et 10x PBS (pH 7,4).
- Ajouter 70 μL de collagène neutralisé dans un puits sur deux et sur une rangée sur deux de la microplaque, et incuber à 37 °C pendant 2 h en plaçant la microplaque sous le capot de culture tissulaire pour évaporer le collagène jusqu’à ce qu’il soit sec.
Jour 2 - Lavez deux fois les microplaques avec 70 μL de 1x PBS, pH 7,4 pour éliminer tout collagène non lié.
- Transférez les microplaques dans l’incubateur pendant 1 h à 37 °C, puis transférez les microplaques sur la paillasse et laissez sécher 1 h avant de les utiliser dans d’autres expériences de liaison.
2. Évaluation de la stabilité des gels/films de collagène immobilisés
- Incubation avec PBS pendant 1 h
REMARQUE : Au cours de l’expérience de liaison, incubez le collagène immobilisé avec le composé d’intérêt. Il est important d’étudier la stabilité du gel ou du film de collagène qui en résulte. Pour ce faire, mesurez la stabilité de trois conditions : pas de lavage = mesure le collagène immobilisé directement après l’incubation ; lavage = mesure le collagène immobilisé après avoir lavé la plaque deux fois avec 100 μL de PBS ; et 1 h PBS mimic & wash = mesure le collagène immobilisé après incubation pendant 1 h avec PBS suivi de deux lavages avec PBS. Ci-dessous, la méthode d’incubation PBS est expliquée.- Ajouter 70 μL de PBS (1x) dans chacun des puits enrobés de collagène et incuber la microplaque à température ambiante pendant 1 h.
- Aspirez l’excès de liquide de chaque puits à l’aide d’une pipette et lavez-le deux fois avec du PBS (1x) avant d’effectuer le test de quantification des protéines décrit ci-dessous.
- Quantification de la quantité de collagène immobilisé à l’aide d’un dosage de l’acide bicinchoninique (BCA)
REMARQUE : Utilisez le kit de dosage des protéines Pierce BCA (table des matériaux) en suivant les instructions du fabricant. Établissez des normes de collagène respectives pour ce test. La plage de concentration du collagène I est de 0 à 3 000 μg/mL et celle du collagène III de 0 à 750 μg/mL. Au total, faites 11 standards par collagène.- Préparez le volume total de réactif de travail (WR) nécessaire en suivant les instructions du fabricant.
- Ajouter 25 μL de chacun des étalons de collagène dans les puits des microplaques (en double). Ces solutions sont utilisées pour dessiner la courbe standard.
- Ajouter 200 μL de solution réactive de travail dans chacun des puits contenant les étalons et les puits recouverts de concentrations inconnues de collagène.
- Placez la microplaque sur un agitateur de plaque pendant 30 s. Couvrez les microplaques et laissez incuber à 37 °C pendant 30 min.
- Retirez les microplaques et laissez-les refroidir à température ambiante. Mesurez l’absorbance à 560 nm à l’aide d’un lecteur de plaques.
- Tracez une courbe d’étalonnage en traçant l’A560 (AU) en fonction de la concentration (μg/mL) des 11 solutions étalons, et utilisez la courbe d’étalonnage pour calculer la quantité de collagène.
3. Essai de liaison du ligand TRF d’Europium(III) (Figure 1)
REMARQUE : Le composé utilisé est un peptide candidat de liaison au collagène (CBP) marqué avec un seul complexe Eu(III)-DO3AAm, appelé Eu(III)-DO3AAm-CBP (Figure 4).
- Incubation des plaques recouvertes de collagène avec le composé Eu(III)-DO3AAm-CBP
- Préparez des solutions du composé Eu(III)-DO3AAm-CBP avec des concentrations comprises entre 0,1 et 15 μM (0,1 μM, 0,5 μM, 1 μM, 3 μM, 5 μM, 7 μM, 10 μM et 15 μM) dans 1x PBS.
- Ajouter 75 μL de chaque concentration de composé dans les puits enrobés de collagène (planche A). Effectuez l’expérience en trois exemplaires pour calculer la quantité de composé qui se lie au collagène.
- À l’aide d’une deuxième plaque non revêtue (plaque B) et ajoutez 75 μL de chaque composé dans les puits vides pour calculer la liaison non spécifique du composé à la plaque. Utilisez des triplets pour chaque concentration.
- Incuber les microplaques pendant 1 h à température ambiante.
- À l’aide d’une pipette, aspirez et jetez l’excès de solution de chaque puits, et lavez les puits avec 1x PBS deux fois pour éliminer l’excès de composé non lié. Effectuez cette étape à l’aide des microplaques enrobées de collagène et non enrobées.
- À une troisième plaque non revêtue (plaque C), ajouter 10 μL de la même gamme de concentrations Eu(III)-DO3AAm-CBP (en double). Utilisez la lecture de fluorescence de la solution Eu(III)-DO3AAm-CBP pour faire une courbe d’étalonnage.
REMARQUE : Ne lavez pas et n’aspirez pas la solution de cette plaque.
- Extraction acide des lectures d’europium(III) et de fluorescence résolue dans le temps (TRF)
REMARQUE : Veuillez vous référer aux informations supplémentaires sur la préparation et l’étalonnage des volumes de la solution acide (AS) et de la solution tampon (BS). Les volumes de SA et de BS nécessaires pour atteindre de manière reproductible un pH optimal étaient respectivement de 54 μL et 46 μL dans ce travail. Effectuez l’opération suivante sur la plaque A, la plaque B et la plaque C.- Ajouter 54 μL de solution acide (SA) dans chaque puits et placer la plaque dans l’incubateur à 37 °C pendant 90 min, en recouvrant les microplaques d’une feuille d’aluminium pour éviter l’évaporation. La température et le temps d’incubation doivent être soigneusement contrôlés pour obtenir une décomplexation reproductible.
- Ajouter 46 μL de solution tampon (BS) dans chaque puits et agiter doucement la plaque pendant 30 s.
- Ajouter 100 μL de solution d’enrichissement (ES) et agiter la plaque pendant 30 s.
- Attendez 30 min avant de lire la plaque à l’aide d’un lecteur de plaque TRF. Utilisez les paramètres répertoriés dans le Tableau 2.
4. Analyse des données
- Quantification de la concentration de collagène immobilisé sur les puits
- Obtenir l’équation de la courbe d’étalonnage de l’A560 (AU) en fonction de la concentration (μg/mL) des 11 solutions étalons.
- Utilisez les lectures d’absorbance acquises à partir des puits contenant les étalons de collagène.
- Calculez les valeurs moyennes des puits dupliqués et tracez l’absorbance moyenne en fonction des concentrations connues de protéines (collagène) (μg/mL) pour obtenir l’équation de la courbe standard.
- Utilisez les valeurs d’absorbance pour calculer la masse (μg) et la concentration (M) du collagène immobilisé.
- Calculez les valeurs moyennes d’absorbance dans les trois puits qui contenaient du collagène immobilisé et enregistrez l’écart-type.
- Utilisez l’équation de la courbe standard obtenue à partir de la courbe standard du collagène (étape 2.2.6) pour convertir l’absorbance mesurée à partir des puits recouverts de collagène en concertation. À partir de là, calculez la concentration de collagène qui a été immobilisée dans les puits expérimentaux en μg/mL.
- Convertir la concentration calculée à l’étape 4.1.2.2 (μg/mL) en grammes/litre, puis, en fonction du poids moléculaire du collagène, en molaire (M).
- Enfin, calculez la masse de collagène immobilisée dans chaque puits en divisant la concentration par le volume de collagène ajouté au puits (100 μL pour le collagène de type I et 70 μL pour le collagène de type III).
- Obtenir l’équation de la courbe d’étalonnage de l’A560 (AU) en fonction de la concentration (μg/mL) des 11 solutions étalons.
- Calcul de la constante de dissociation (Kd) (Graphique 4)
- Extrayez les lectures de fluorescence.
- Exportez les lectures de fluorescence du lecteur de plaques vers une feuille de calcul.
REMARQUE : Dans les essais de liaison, il est important de tenir compte de la liaison non spécifique potentielle d’un composé à la surface plastique des plaques. - Calculer les valeurs moyennes des mesures en trois exemplaires à partir de chaque concentration de composé pour les trois différentes plaques : les lectures de liaison spécifique des puits revêtus (planche A), la liaison non spécifique des puits non revêtus (planche B) et la totalité de l’Eu(III)-DO3AAm-CBP en solution dans les puits non revêtus (planche C).
- Déterminez les valeurs de fluorescence du composé lié en soustrayant les lectures de fluorescence des puits non revêtus (planche B) de celles des puits revêtus (planche A).
Équation 1 : Détermination de la fluorescence liée17 :
Fluorescence liée = Spécifique (puits enrobés) - Non spécifique (puits non enrobés) - Générer une courbe d’étalonnage à l’aide des lectures du composé marqué à l’Eu(III) en solution (planche C). Tracez les lectures de fluorescence obtenues en fonction de la concentration du composé marqué à l’Eu(III). Effectuez un ajustement de régression linéaire.
- Exportez les lectures de fluorescence du lecteur de plaques vers une feuille de calcul.
- Convertissez les lectures de fluorescence en concentrations.
- Convertir les lectures de la fluorescence liée (étape 4.2.3) en concentration à l’aide de la courbe de fluorescence standard à partir des données générées à l’aide des concentrations de composés en solution (étape 4.2.1.4).
REMARQUE : Lorsque l’on compare les propriétés de liaison d’un composé à différentes protéines cibles qui s’immobilisent à différentes concentrations, ces dernières devront être prises en compte lors du calcul de la quantité de composé liée à la cible (c’est-à-dire composé lié/protéine). - Divisez la concentration du composé lié par la concentration de la protéine immobilisée dans le puits.
REMARQUE : Pour ce calcul, utilisez la concentration de collagène immobilisé qui a été calculée après l’incubation des puits avec du PBS pendant 1 h (expérience dite d’imitation PBS ; section 2.1 ci-dessus). Il s’agit de tenir compte des pertes potentielles de collagène pendant l’étape d’incubation et l’étape de lavage qui ne contribueront pas au signal de fluorescence final. - Tracez les données à l’aide d’un nuage de points qui a les concentrations du composé sur l’axe des x (μM) et du composé/protéine lié sur l’axe des y.
- Convertir les lectures de la fluorescence liée (étape 4.2.3) en concentration à l’aide de la courbe de fluorescence standard à partir des données générées à l’aide des concentrations de composés en solution (étape 4.2.1.4).
- Obtenez les valeursK d .
- Ajustez les données acquises à l’étape 4.2.2.3 à l’aide de deux modèles cinétiques de liaison possibles : la liaison sur un site et la liaison sur un site avec une pente de colline. Les équations de chaque modèle sont illustrées à la figure 6.
- Choisissez le modèle qui offre un ajustement non ambigu avec la valeur R au carré la plus élevée lors de l’ajustement des données.
- Exclure les valeurs aberrantes pour chaque ensemble de lectures de fluorescence par concentration et par plaque.
- Calculez la valeur finale de Kd et présentez les données sous forme de moyenne ± d’écart-type d’expériences indépendantes.
REMARQUE : Pour des résultats robustes, effectuez des mesures en trois exemplaires dans chaque plaque et au moins trois expériences indépendantes avec des microplaques différentes.
- Calculez l’occupation fractionnaire (FO).
REMARQUE : D’après l’équation 2, la concentration de la cible est inconnue et, par conséquent, en utilisant l’algèbre et le Kd, de l’équation 3, une équation réalisable pour calculer l’occupation fractionnaire apparaît sous la forme de l’équation 4.
Équation 2 : Définition de l’occupation fractionnée17 :
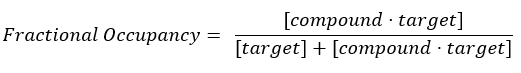
Équation 3 : La constante de dissociation, Kd, qui est la concentration à laquelle le composé occupe 50 % de la cible à l’équilibre17 :
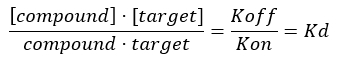
Équation 4 : Équation réarrangée pour calculer l’équation FO17 :
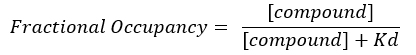
- Calculer le FO à l’aide des valeurs indépendantesde K d obtenues pour chaque plaque. Tracez les résultats, la moyenne et les écarts-types du FO en fonction de la concentration du composé.
- Déclarez le FO avec des valeurs allant de 0 à 1 ou en pourcentage avec des valeurs allant de 0 % à 100 %.
- Extrayez les lectures de fluorescence.
Résultats
Évaluation de la stabilité et de la concentration du collagène de type I et de type III immobilisé dans des gels/films
La quantification de la concentration de collagène immobilisé par puits a été effectuée à l’aide de trois conditions différentes : a) dans des puits sans lavage avec PBS après immobilisation des protéines (pas de lavage) ; b) dans les puits avec une étape de lavage (deux fois avec PBS) après immobilisation pour éliminer toute protéi...
Discussion
Ce travail présente une méthode reproductible d’immobilisation du collagène de type I et de type III. Il fait également la démonstration d’un protocole d’acquisition, d’analyse et d’interprétation in vitro des données de liaison TRF Eu(III) afin de caractériser les propriétés de liaison d’un ligand candidat envers le collagène de type I et III. Les protocoles d’immobilisation du collagène de type I et de type III présentés ici ont été développés e...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous sommes reconnaissants aux bailleurs de fonds suivants de soutenir ce travail : (1) le Conseil de la recherche médicale du Royaume-Uni (MR/N013700/1) et le King’s College de Londres, membres du Partenariat de formation doctorale en sciences biomédicales du MRC ; (2) subvention du programme BHF RG/20/1/34802 ; (3) subvention de projet BHF PG/2019/34897 ; (4) Subvention du Centre d’excellence en recherche de la BHF de King’s RE/18/2/34213 ; (5) le programme ANID Millennium Science Initiative - ICN2021_004 ; et (6) la subvention de base de l’ANID FB210024.
matériels
| Name | Company | Catalog Number | Comments |
| 10x PBS | Gibco | 14200075 | Use this to make 1x PBS by diluting in water (1:10) |
| 10x PBS | Gibco | 14200075 | Use this to make 1x PBS by diluting in water (1:10) |
| 2M HCL | Made in house and details are in the supporting document | ||
| 2M HCL | Made in house and details are in the supporting document | ||
| 2M Sodium hydroxide +2M Glycine | Made in house and details are in the supporting document | ||
| 2M Sodium hydroxide +2M Glycine | Made in house and details are in the supporting document | ||
| Cell-star 96 well microplate | Greiner Bio-One | 655 160 | |
| Cell-star 96 well microplate | Greiner Bio-One | 655 160 | |
| DELFIA enhacement solution | Perkin Elmer | 1244-104 | |
| DELFIA enhacement solution | Perkin Elmer | 1244-104 | |
| Ice | |||
| Ice | |||
| Infinite 200 PRO NanoQuant microplate reader | TECAN | ||
| Infinite 200 PRO NanoQuant microplate reader | TECAN | ||
| Non-binding (NBS) 96 well microplates | Corning | 3641 | |
| Non-binding (NBS) 96 well microplates | Corning | 3641 | |
| pH electrode Inlab Routine | Mettler Toledo | 51343050 | |
| pH electrode Inlab Routine | Mettler Toledo | 51343050 | |
| pH meter (sevenCompact) | Mettler Toledo | ||
| pH meter (sevenCompact) | Mettler Toledo | ||
| Pierce BCA protein assay kit | Thermofisher | 23227 | |
| Pierce BCA protein assay kit | Thermofisher | 23227 | |
| Tissue culture incubator (37 °C, 5% CO2) | |||
| Type I bovine collagen, 3 mg/mL | Corning | 354231 | |
| Type III human placenta collagen, 0.99 mg/mL | Advanced Biomatrix | 5021 |
Références
- Distler, J. H. W., et al. Review: Frontiers of antifibrotic therapy in systemic sclerosis. Arthritis and Rheumatology. 69 (2), 257-267 (2017).
- Wynn, T. A. Fibrotic disease and the TH1/TH2 paradigm. Nature Reviews Immunology. 4 (8), 583-594 (2004).
- Saha, P., et al. Magnetic resonance T1 relaxation time of venous thrombus is determined by iron processing and predicts susceptibility to lysis. Circulation. 128 (7), 729-736 (2013).
- Mirshahi, M., et al. Defective thrombolysis due to collagen incorporation in fibrin clots. Thrombosis Research. 8, 73-80 (1988).
- Comerota, A. J. The ATTRACT trial: Rationale for early intervention for iliofemoral DVT. Perspectives in Vascular Surgery and Endovascular Therapy. 21 (4), 221-225 (2009).
- Bateman, E. D., Turner-Warwick, M., Adelmann-Grill, B. C. Immunohistochemical study of collagen types in human foetal lung and fibrotic lung disease. Thorax. 36 (9), 645-653 (1981).
- Pawelec, K. M., Best, S. M., Cameron, R. E. Collagen: A network for regenerative medicine. Journal of Materials Chemistry B. 4 (40), 6484-6496 (2016).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Copes, F., Pien, N., Van Vlierberghe, S., Boccafoschi, F., Mantovani, D. Collagen-based tissue engineering strategies for vascular medicine. Frontiers in Bioengineering and Biotechnology. 7, 166 (2019).
- Veis, A. The biochemistry of collagen. Annals of Clinical and Laboratory Science. 5 (2), 123-131 (1975).
- Bielajew, B. K., Hu, J. C., Athanasiou, K. A. Collagen: Quantification, biomechanics, and role of minor subtypes in cartilage. Nature Reviews Materials. 5, 730-747 (2020).
- Zhao, Z., et al. Structural and functional plasticity of collagen fibrils. DNA and Cell Biology. 38 (4), 367-373 (2019).
- Eryilmaz, E., Teizer, W., Hwang, W. In vitro analysis of the co-assembly of type-I and type-III collagen. Cellular and Molecular Bioengineering. 10 (1), 41-53 (2017).
- Jagnow, J., Clegg, S. Klebsiella pneumoniae MrkD-mediated biofilm formation on extracellular matrix- and collagen-coated surfaces. Microbiology. 149 (9), 2397-2405 (2003).
- O'Sullivan, D., O'Neill, L., Bourke, P. Direct plasma deposition of collagen on 96-well polystyrene plates for cell culture. ACS Omega. 5 (39), 25069-25076 (2020).
- Caravan, P., et al. Collagen-targeted MRI contrast agent for molecular imaging of fibrosis. Angewandte Chemie - International Edition. 46 (43), 8171-8173 (2007).
- Copeland, R. A. . Enzymes: A Practical Introduction to Structure, Mechanism, and Data Analysis. , (2000).
- Salahudeen, M. S., Nishtala, P. S. An overview of pharmacodynamic modelling, ligand-binding approach and its application in clinical practice. Saudi Pharmaceutical Journal. 25 (2), 165-175 (2017).
- Bünzli, J. C. G., Piguet, C. Taking advantage of luminescent lanthanide ions. Chemical Society Reviews. 34 (12), 1048-1077 (2005).
- Hemmilii, I. Luminescent lanthanide chelates - A way to more sensitive diagnostic methods. Journal of Alloys and Compounds. 225 (1-2), 480-485 (1995).
- De Silva, C. R., Vagner, J., Lynch, R., Gillies, R. J., Hruby, V. J. Optimization of time-resolved fluorescence assay for detection of europium-tetraazacyclododecyltetraacetic acid-labeled ligand-receptor interactions. Analytical Biochemistry. 398 (1), 15-23 (2010).
- Digilio, G., Lacerda, S., Lavin Plaza, B., Phinikaridou, A. Extracellular matrix targeted MRI probes. Analysis & Sensing. 3 (1), (2022).
- Phinikaridou, A., et al. Tropoelastin: A novel marker for plaque progression and instability. Circulation. Cardiovascular imaging. 11 (8), 007303 (2018).
- Guzaeva, T. V., et al. Protein A used in DELFIA for the determination of specific antibodies. Immunology Letters. 35 (3), 285-289 (1993).
- Nasiri, A. H., Nasiri, H. R. Polymerase assays for lead discovery: An overall review of methodologies and approaches. Analytical Biochemistry. 563, 40-50 (2018).
- Capuana, F., et al. Imaging of dysfunctional elastogenesis in atherosclerosis using an improved gadolinium-based tetrameric MRI probe targeted to tropoelastin. Journal of Medicinal Chemistry. 64 (20), 15250-15261 (2021).
- Drescher, D. G., Drescher, M. J., Ramakrishnan, N. A. Surface plasmon resonance (SPR) analysis of binding interactions of proteins in inner-ear sensory epithelia. Methods in Molecular Biology. 493, 323-343 (2009).
- Murali, S., Rustandi, R. R., Zheng, X., Payne, A., Shang, L. Applications of surface plasmon resonance and biolayer interferometry for virus-ligand binding. Viruses. 14 (4), 717 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon