In vivo Imagerie du cerveau entier de larves de poisson-zèbre à l’aide de la microscopie à fluorescence tridimensionnelle
Dans cet article
Résumé
Un protocole d’imagerie in vivo du cerveau entier de larves de poisson-zèbre à l’aide de la microscopie à fluorescence tridimensionnelle est présenté ici. La procédure expérimentale comprend la préparation des échantillons, l’acquisition d’images et la visualisation.
Résumé
En tant qu’animal modèle vertébré, le poisson-zèbre larvaire est largement utilisé en neurosciences et offre une occasion unique de surveiller l’activité du cerveau entier à la résolution cellulaire. Ici, nous fournissons un protocole optimisé pour effectuer l’imagerie du cerveau entier des larves de poisson-zèbre à l’aide de la microscopie à fluorescence tridimensionnelle, y compris la préparation et l’immobilisation des échantillons, l’incorporation des échantillons, l’acquisition d’images et la visualisation après imagerie. Le protocole actuel permet l’imagerie in vivo de la structure et de l’activité neuronale d’un cerveau larvaire de poisson-zèbre à une résolution cellulaire pendant plus de 1 h en utilisant la microscopie confocale et la microscopie à fluorescence conçue sur mesure. Les étapes critiques du protocole sont également discutées, y compris le montage et le positionnement de l’échantillon, la prévention de la formation de bulles et de poussière dans le gel d’agarose, et l’évitement des mouvements dans les images causés par la solidification incomplète du gel d’agarose et la paralysie du poisson. Le protocole a été validé et confirmé dans plusieurs contextes. Ce protocole peut être facilement adapté pour l’imagerie d’autres organes d’une larve de poisson zèbre.
Introduction
Le poisson-zèbre (Danio rerio) a été largement adopté comme animal vertébré modèle en neurosciences, en raison de sa transparence optique au stade larvaire, de son développement rapide, de son faible coût d’entretien et de la disponibilité de divers outils génétiques 1,2,3,4. En particulier, la transparence optique des larves combinée à des rapporteurs fluorescents génétiquement codés d’événements biologiques 5,6,7,8,9 offre une occasion unique d’imager à la fois l’activité neuronale et la structure au niveau du cerveau entier 10,11,12,13,14 . Cependant, même avec un microscope qui prend en charge la résolution cellulaire, les images acquises ne retiennent pas nécessairement l’information au niveau de la cellule unique; La qualité de l’image optique peut être dégradée en raison de l’aberration introduite par le gel d’agarose utilisé pour le montage de l’échantillon, le poisson peut être monté à un angle, de sorte que les régions d’intérêt ne sont pas entièrement contenues dans le champ de vision du microscope, et le poisson peut se déplacer pendant l’enregistrement, provoquant des artefacts de mouvement dans les images ou empêchant l’extraction précise du signal des images.
Ainsi, un protocole efficace et reproductible est nécessaire pour acquérir des données d’image de haute qualité avec un minimum de bruit et de mouvement. Malheureusement, les protocoles accessibles au public pour l’imagerie d’un cerveau entier de larves de poisson-zèbre in vivo 15,16,17,18,19 ne décrivent que brièvement la procédure, laissant à chaque expérimentateur le soin de régler des parties substantielles des détails, tels que la solidification de l’agarose, les techniques de montage précises et le positionnement des échantillons à l’aide de pinces. De plus, les incohérences dans les méthodes de concentration et d’immobilisationde l’agarose 10,11,14,15,16,17,18,19 peuvent entraîner des défis découlant du déplacement des poissons pendant le processus d’imagerie.
Ici, un protocole détaillé pour l’imagerie du cerveau entier des larves de poisson-zèbre à l’aide de la microscopie à fluorescence tridimensionnelle est fourni. La figure 1 fournit une vue d’ensemble graphique du protocole : préparation et immobilisation des échantillons, incorporation des échantillons, acquisition d’images et visualisation après imagerie. L’imagerie structurelle et fonctionnelle du cerveau larvaire du poisson zèbre in vivo est démontrée à l’aide d’un microscope confocal commercial et d’un microscope à fluorescence conçu sur mesure. Ce protocole peut être adapté par les praticiens pour l’imagerie cérébrale avec certains stimuli sensoriels ou contextes comportementaux en fonction des besoins expérimentaux et de la conception.
Protocole
Toutes les expériences sur le poisson zèbre ont été approuvées par le Comité institutionnel de soin et d’utilisation des animaux (IACUC) du KAIST (KA2021-125). Un total de 12 poissons-zèbres adultes avec expression panneuronale de l’indicateur de calcium GCaMP7a [Tg(huc:GAL4); Tg(UAS:GCaMP7a)] sur un fond de casper [mitfa(w2/w2);mpv17(a9/a9)] ont été utilisés pour la reproduction. Ce groupe était composé de huit femmes et quatre hommes, âgés de 3 à 12 mois. Des expériences d’imagerie ont été réalisées sur des larves de poisson-zèbre 3-4 jours après la fécondation (d.p.f.), un stade au cours duquel leur sexe ne peut pas être déterminé.
1. Préparation des échantillons de poisson zèbre
- Recueillir des embryons après avoir élevé des poissons adultes d’une lignée transgénique souhaitée, tels que Tg (huc: GAL4); Tg(UAS:GCaMP7a)20,21,22, dans une boîte de Petri remplie d’eau d’œuf (voir le tableau des matières). Placer les embryons dans un incubateur à 28 °C et les élever à 3-4 d.p.f. larves23,24,25.
- Si le fond de poisson zèbre n’est pas albinos, pour empêcher la formation de pigmentation, transférer les embryons 24 h après la fécondation (h.p.f.) dans la boîte de Petri remplie d’eau d’œuf contenant 200 μM de 1-phényle 2-thiourée (PTU; voir le tableau des matériaux)25,26. Toutes les 24 h, transférer le poisson dans un nouveau plat avec de l’eau d’œuf fraîche contenant 200 μM de PTU.
NOTE: Les poissons-zèbres sont maintenus dans des conditions normales à 28 ° C et un cycle lumière:obscurité de 14h10. Le traitement PTU est connu pour affecter le comportement et la fonction thyroïdienne des larves de poisson-zèbre27,28. Par conséquent, il est important d’utiliser le PTU avec prudence et de contrôler soigneusement les facteurs de confusion potentiels dans toute expérience.
- Si le fond de poisson zèbre n’est pas albinos, pour empêcher la formation de pigmentation, transférer les embryons 24 h après la fécondation (h.p.f.) dans la boîte de Petri remplie d’eau d’œuf contenant 200 μM de 1-phényle 2-thiourée (PTU; voir le tableau des matériaux)25,26. Toutes les 24 h, transférer le poisson dans un nouveau plat avec de l’eau d’œuf fraîche contenant 200 μM de PTU.
- Pour trouver l’échantillon exprimant des protéines fluorescentes d’intérêt (p. ex., GCaMP7a panneuronal), filtrez l’échantillon au microscope à épifluorescence et sélectionnez un échantillon avec une expression brillante.
- Préparer l’échantillon sélectionné de poisson-zèbre 3-4 d.p.f. dans la boîte de Petri remplie d’eau d’œuf (figure 2A).
REMARQUE: Pour enregistrer l’activité neuronale spontanée, il est recommandé d’utiliser des échantillons âgés de 80 à 100 h.p.f., car le niveau d’activité spontanée est faible avant 80 h.p.f. et le développement de la pigmentation, même avec un fond casper, peut dégrader la qualité de l’image après 100 h.p.f. - Préparer une solution de bromure de pancuronium 14,19,29,30,31 à 0,25 mg/mL en ajoutant 1 mL d’une solution mère de 2,5 mg/mL (voir le tableau des matières) à10 mL d’eau d’œuf. Aliquote de la solution de bromure de pancuronium dans des tubes microcentrifugés de 1,5 mL.
- Transférer l’échantillon présélectionné dans la boîte de Petri à l’aide d’une pipette de transfert.
REMARQUE: Essayez de transporter un volume minimum d’eau d’œuf avec l’échantillon. - Transférer 0,1 mL de la solution de bromure de pancuronium dans la boîte de Petri pour la paralysie.
REMARQUE : Le bromure de pancuronium a un effet modérateur potentiel sur l’activité neuronale chez les larves de poisson-zèbre32. Il est essentiel d’examiner attentivement la concentration et la durée de l’exposition au bromure de pancuronium.
2. Préparation de gel d’agarose à 2 % (poids/volume)
- Allumez un bloc de chaleur et réglez la température cible à 37 °C. Attendez que l’appareil chauffe et s’équilibre à la température réglée.
- Dissoudre 0,2 g de poudre d’agarose à bas point de fusion (voir le tableau des matières) dans 10 ml d’eau d’œuf.
- Chauffer la solution d’agarose au micro-ondes et la mélanger en agitant et en tourbillonnant jusqu’à ce que l’agarose soit complètement dissoute.
- Aliquote du gel d’agarose dans des tubes microcentrifugeuses de 1,5 mL et stocker les tubes de microcentrifugeuses sur le bloc thermique (figure 2B).
REMARQUE: Vérifiez si les bulles dans le gel d’agarose ont disparu.
3. Montage et positionnement de l’échantillon
- Vérifiez l’échantillon au stéréomicroscope pour vérifier que le mouvement des larves s’est arrêté et pour évaluer visuellement la santé de l’échantillon en vérifiant son rythme cardiaque (figure 2C). Si le rythme cardiaque est trop lent, jetez l’échantillon.
REMARQUE : Si le rythme cardiaque du poisson photographié est trop lent (p. ex., moins de 60 battements par minute), il peut ne pas être possible d’effectuer une imagerie à long terme. Pour évaluer le bien-être du poisson, la fréquence cardiaque peut être vérifiée visuellement en la comparant à d’autres poissons dans la même boîte de Pétri. Cela permettra de s’assurer que le poisson photographié est en bonne santé et suffisamment stable pour la procédure d’imagerie. - À l’aide de la pipette de transfert, placer une seule larve de poisson-zèbre dans le gel d’agarose dans le tube de microcentrifugation de 1,5 ml (figure 2D).
REMARQUE : Assurez-vous de jeter la pipette après avoir transféré l’échantillon. - Versez le gel d’agarose dans la boîte de Petri pour faire une couche de 1-2 mm. Transférer l’échantillon dans les tubes microcentrifugeuses dans la boîte de Petri à l’aide de la pipette de transfert, de sorte que la larve soit placée au centre de la boîte.
REMARQUE: S’il y a de la poussière et des bulles dans le gel d’agarose, retirez-les à l’aide de la pipette de transfert. - Utilisez une pince pour positionner l’échantillon dans l’orientation souhaitée de manière à ce que la tête et la queue soient plates (figure 2E).
- Faire pivoter l’échantillon à l’aide d’une pince de manière à ce que les deux yeux soient à niveau (figure 2F).
REMARQUE : Les procédures d’alignement (position et rotation) doivent être terminées avant que le gel d’agarose ne commence à se solidifier. - Après l’alignement, attendez que le gel d’agarose se soit solidifié (Figure 2G,H).
REMARQUE: Le temps d’attente pour que le gel d’agarose se solidifie peut varier de 5 à 10 minutes, selon le volume et la taille du gel. - Après solidification du gel d’agarose, remplir la boîte de Petri avec de l’eau d’œuf et placer la boîte de Petri avec l’échantillon incorporé sur la platine du microscope (Figure 2I).
4. Acquisition d’images
- Allumez le système de microscope (par exemple, lasers, contrôleurs confocaux, microscope et ordinateur; voir Tableau des matériaux) et vérifiez que l’ensemble du système fonctionne.
- Sélectionnez un objectif à faible grossissement et localisez l’échantillon au centre du champ de vision.
- Choisissez un objectif à immersion dans l’eau ou plongeant dans l’eau avec un grossissement approprié (p. ex., objectif plongeur d’eau à ouverture numérique (NA) de 16 x 0,8; voir le tableau des matériaux). Effectuez des ajustements fins au champ de vision.
- Réglez les paramètres d’imagerie (par exemple, la taille de l’image, la puissance laser, le temps d’exposition, le nombre d’images) à l’aide d’un logiciel d’acquisition d’images.
REMARQUE: Définissez les paramètres d’imagerie pour obtenir les meilleurs résultats possibles pour des besoins spécifiques (voir la configuration pour l’acquisition d’images dans la section des résultats représentatifs). Si l’image est saturée, réduisez la puissance laser. - Trouvez le cerveau de l’échantillon en déplaçant la scène et déterminez son épaisseur à l’aide du mode d’affichage en direct dans le logiciel en modifiant les plans focaux manuellement de haut en bas. Définissez les limites inférieure et supérieure du volume.
REMARQUE: Assurez-vous que tout le cerveau est contenu dans le champ de vision le long des directions latérale et axiale. - Définissez une taille de pas z en tenant compte de la résolution axiale du microscope.
REMARQUE: La taille optimale du pas z pour l’imagerie du cerveau larvaire du poisson zèbre dépend de la modalité d’imagerie et de la résolution du microscope. À titre d’exemple, une taille de pas en z de 5 μm a été utilisée compte tenu de l’épaisseur de la feuille de lumière et du diamètre moyen des corps cellulaires10. - Procédez à l’acquisition de l’image pour le champ de vision défini.
- Pour l’imagerie structurelle volumétrique, acquérir une image 3D (x, y, z) de l’ensemble du cerveau en modifiant les plans focaux et en obtenant des images 2D de chaque plan z séquentiellement.
- Pour l’imagerie fonctionnelle d’un seul plan z, acquérir des images de séries temporelles (x, y, t) de l’activité neuronale du cerveau à une certaine profondeur.
- Pour l’imagerie fonctionnelle volumétrique, acquérir une image 4D (x, y, z, t) de l’activité neuronale dans l’ensemble du cerveau en obtenant des images 3D séquentiellement.
Remarque : Définissez le nombre de trames en tenant compte de la taille de mémoire disponible de l’ordinateur. Un temps d’acquisition inférieur à 1 h est recommandé en raison de la durée de l’effet du bromure de pancuronium.
- Après avoir acquis des images, enregistrez les résultats et enregistrez les paramètres d’imagerie (par exemple, la taille des pixels, la taille du pas z, la fréquence d’images, la puissance laser) pour l’analyse de l’image.
- Exportez les images dans un format approprié pour le rendu des données et l’analyse des images.
REMARQUE: L’exportation d’images au format de fichier d’image balisé (TIF) est recommandée. TIF prend en charge la compression d’image sans perte et est compatible avec la plupart des logiciels de traitement d’image et des langages de programmation. En outre, le format TIF prend en charge l’inclusion de métadonnées, telles que les paramètres d’acquisition, la résolution d’image et d’autres informations pertinentes qui peuvent aider à l’interprétation et à la reproductibilité des données.
5. Configuration des visualisations à l’aide de napari
REMARQUE: napari est une visionneuse d’images multidimensionnelle open-source dans un environnement Python avec un rendu basé sur une unité de traitement graphique (GPU)33. Le plugin napari-animation fournit une création programmatique de films. L’utilisation de Fidji, un programme de traitement d’images open source, est recommandée pour le traitement d’images à usage général, tel que le filtrage et la transformation géométrique (voir Tableau des matériaux). Le code source utilisé pour la visualisation à l’aide de napari est disponible sur GitHub (https://github.com/NICALab/Zebrafish-brain-visualization).
- Installez napari et napari-animation en utilisant pip ou conda. Après l’installation, créez un nouveau fichier de bloc-notes jupyter.
REMARQUE: L’exécution avec jupyter notebook, qui est un outil interactif pour Python, est recommandée par rapport au script Python. - Importer napari et napari-animation.
6. Visualisation des structures à l’aide de napari
- Pour visualiser des images et créer des films du cerveau du poisson zèbre rendu, chargez l’image 3D (x, y, z) et ouvrez la fenêtre napari. Connectez le plug-in napari-animation (Figure 3A).
- Définissez des paramètres tels que la taille du voxel, la carte de couleurs et les limites de contraste dans les contrôles de calque.
- Ajustez les paramètres de la visionneuse (par exemple, perspective, angles) dans la zone de travail.
- Pour capturer l’image rendue, appuyez sur le bouton de capture dans l’assistant d’animation.
- Pour générer des images du volume rendu, modifiez les paramètres de la visionneuse et ajoutez des images clés (Figure 3B).
- Après avoir ajouté des images clés, définissez le nombre d’images (étapes) entre les images clés dans l’assistant d’animation. Enregistrez l’animation rendue.
7. Traitement d’images et visualisation de l’activité neuronale à l’aide de napari
NOTE : Pour visualiser les images de séries chronologiques de l’activité neuronale en tant qu’images superposées d’un arrière-plan statique et d’une activité, un algorithme de décomposition doit être appliqué aux images brutes. Utilisez une implémentation MATLAB d’un algorithme de décomposition appelé BEAR24. La version MATLAB de BEAR est disponible sur GitHub (https://github.com/NICALab/BEAR).
- Pour décomposer l’arrière-plan statique et l’activité neuronale, appliquez BEAR aux images brutes de séries chronologiques (x, y, t ou x, y, z, t). Après la décomposition, enregistrez les images de l’arrière-plan et de l’activité neuronale sous forme de fichiers TIF (Figure 3C).
- Chargez les images d’arrière-plan et d’activité neuronale et ouvrez la fenêtre napari. Connectez le plugin napari-animation.
- Utilisez une carte de couleurs grises pour les images d’arrière-plan et une carte de couleurs chaudes pour les images d’activité neuronale (Figure 3C).
- Définissez des paramètres tels que les limites d’opacité et de contraste dans les contrôles de calque.
- Pour générer des films d’activité neuronale, modifiez les paramètres de la visionneuse et ajoutez des images clés pour rendre l’animation.
- Après avoir ajouté des images clés, définissez le nombre d’images (étapes) entre les images clés dans l’assistant d’animation. Enregistrez l’animation rendue.
Résultats Représentatifs
La structure et l’activité neuronale des cerveaux larvaires de poisson-zèbre exprimant l’indicateur de calcium pan-neuronal GCaMP7a (Tg(huc:GAL4); Tg(UAS:GCaMP7a))20,21,22 avec un fond casper (mitfa(w2/w2);mpv17(a9/a9))34 a été imagé à 3-4 d.p.f. en suivant le protocole décrit.
Pour l’imagerie structurelle volumétrique, l’échantillon a été imagé à l’aide d’un système commercial de microscopie confocale à balayage ponctuel équipé d’une lentille d’objectif plongeant dans l’eau 16x 0,8 NA. Un laser d’excitation de 488 nm a été utilisé pour l’imagerie structurelle et fonctionnelle. La fréquence d’images, la résolution de l’image, la taille des pixels et la taille des pas axiaux étaient respectivement de 0,25 Hz, 2048 x 2048, 0,34 μm et 1,225 μm. L’acquisition de l’image a pris environ 1 h et 20 min. Le champ de vision volumétrique de l’image acquise couvrait les régions cérébrales du cerveau antérieur, du mésencéphale et du cerveau postérieur (Figure 4A). Les corps cellulaires neuronaux du bulbe rachidien, de la plaque cérébelleuse, du tectum optique, de l’habenula, et du télencéphale dorsal dans tout le cerveau de 4 d.p.f. larves de poisson-zèbre étaient clairement visibles sur les images de microscopie confocale (Figure 4B). Le rendu 3D des images de microscopie confocale a été réalisé à l’aide de napari27 en suivant le protocole susmentionné (Figure 4C et Vidéo 1).
Pour l’imagerie fonctionnelle en 2D, l’échantillon a été imagé à l’aide du même système de microscopie confocale, équipé d’une lentille d’objectif plongeant dans l’eau 16x 0,8 NA. La fréquence d’images, la résolution de l’image et la taille des pixels étaient respectivement de 2 Hz, 512 x 256 et 1,5 μm. Les corps cellulaires neuronaux étaient clairement visibles à la fois en arrière-plan et dans l’activité neuronale superposée (Figure 5A,B).
Pour l’imagerie fonctionnelle 3D, un système de microscopie 3D conçu sur mesure18 a été utilisé, capable d’imager in vivo l’activité neuronale d’un cerveau entier de poisson-zèbre larvaire avec un champ de vision de 1 040 μm × 400 μm × 235 μm et des résolutions latérales et axiales de 1,7 μm et 5,4 μm, respectivement. Le taux d’imagerie était jusqu’à 4,2 volumes par seconde. Semblable au rendu des données d’imagerie structurelle, napari a été utilisé pour le rendu 3D des données d’imagerie calcique du cerveau entier (Figure 5C et Vidéo 2).
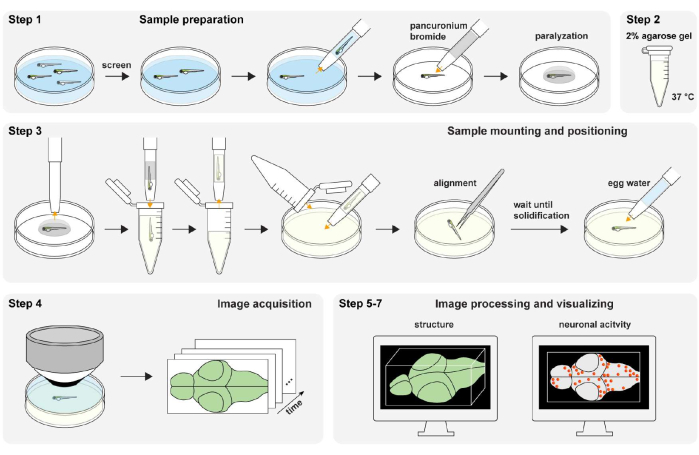
Figure 1 : Vue d’ensemble de la procédure expérimentale. Préparation de l’échantillon de poisson zèbre et paralysie (étape 1). La préparation de gel d’agarose à 2% (poids / volume) (étape 2). Montage et positionnement de l’échantillon (étape 3). Acquisition d’images (étape 4). Traitement d’images et visualisation de la structure et de l’activité neuronale (étape 5-7). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Procédure expérimentale de préparation de l’imagerie du cerveau entier. (A) Échantillon de poisson-zèbre filtré exprimant GCaMP7a panneuronal dans une boîte de Petri remplie d’eau d’œuf. (B) Équipement et matériel nécessaires au montage et au positionnement des échantillons. (1) bloc thermique à 37 °C; (2) gel d’agarose à 2 % (poids/volume); 3° tube microcentrifuge de 1,5 mL; 4° l’eau des œufs; (5) solution de bromure de pancuronium à 0,25 mg/mL; (6) Boîte de Pétri; 7° forceps; (8) pipette de transfert. (C) Image au stéréomicroscope de l’échantillon paralysé. La flèche noire pointe vers le cœur de l’échantillon. (D) L’échantillon dans le tube de microcentrifugation de 1,5 mL est transféré à l’aide d’une pipette. L’encart montre une vue agrandie de la zone encadrée. (E) L’échantillon (flèche noire) est placé au centre de la boîte de Petri à l’aide d’une pince. (F) L’échantillon est aligné à l’aide de forceps. (G) Un exemple d’échantillon incorporé mal aligné. (H) Un exemple d’échantillon incorporé aligné. (I) L’échantillon est placé sur une platine de microscope sous la lentille de l’objectif pour l’acquisition d’images. Barre d’échelle: 500 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
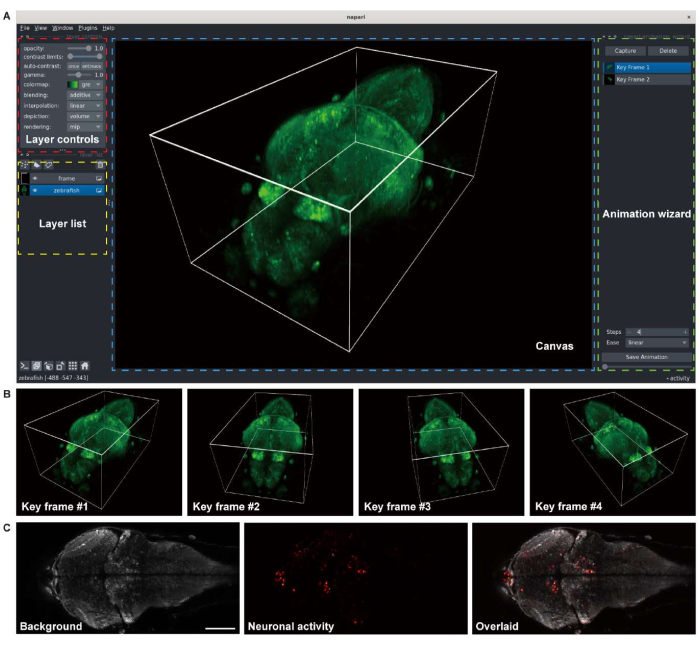
Figure 3 : Visualisation d’images cérébrales larvaires de poissons-zèbres. (A) Le rendu 3D d’une image de microscopie confocale d’un cerveau larvaire de poisson zèbre (4 d.p.f) exprimant GCaMP7a panneuronal. napari, une visionneuse d’images multidimensionnelle open-source dans un environnement Python, a été utilisée pour le rendu. La fenêtre napari comprend des contrôles de calque (boîte rouge), une liste de calques (boîte jaune), un canevas (boîte bleue) et un assistant d’animation (boîte verte). (B) Des images clés avec plusieurs paramètres de visionneuse sont ajoutées pour le rendu d’une animation. (C) Image de microscopie confocale de l’imagerie accélérée 2D de l’activité neuronale dans le cerveau larvaire du poisson zèbre. L’image est décomposée en arrière-plan (à gauche) et en activité neuronale (au milieu), puis superposée (à droite). Barre d’échelle: 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
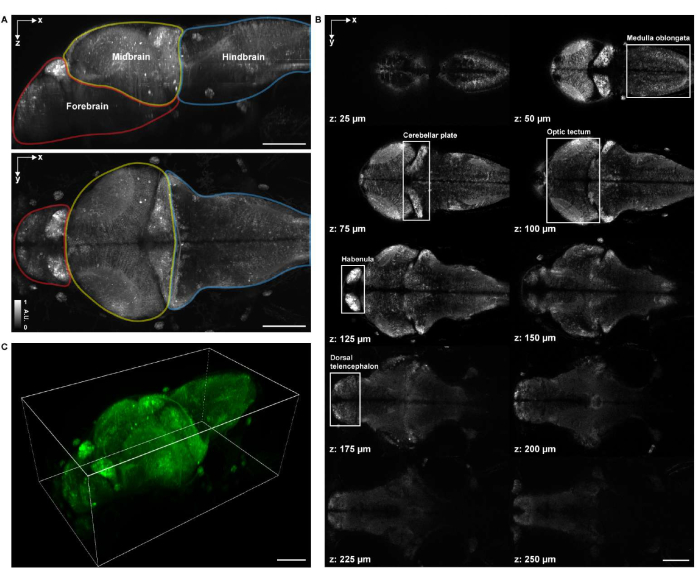
Figure 4 : Structure d’imagerie d’un cerveau larvaire de poisson-zèbre. (A) Projection d’intensité maximale (MIP) d’une image de microscopie confocale d’un cerveau larvaire de poisson zèbre (4 d.p.f.) exprimant GCaMP7a panneuronal. En haut : MIP latéral. En bas : MIP axial. Chaque limite contient le cerveau antérieur (rouge), le mésencéphale (jaune) et le cerveau postérieur (bleu). (B) Un total de 10 tranches axiales à plusieurs profondeurs à partir d’une image volumétrique du cerveau (à z = 25 μm, 50 μm, 75 μm, 100 μm, 125 μm, 150 μm, 175 μm, 200 μm, 225 μm, 250 μm, comptées de haut en bas; z = 0 μm indique la surface supérieure du cerveau). Chaque case blanche représente la région du cerveau (bulbe rachidien, plaque cérébelleuse, tectum optique, habenula, télencéphale dorsal). (C) Le cerveau entier rendu à l’aide de napari. Barre d’échelle: 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
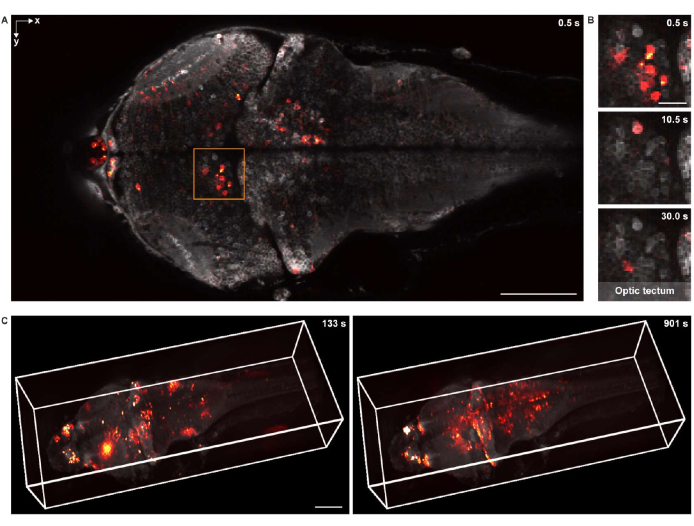
Figure 5 : Imagerie de l’activité neuronale d’une larve de cerveau de poisson-zèbre. (A) Image de microscopie confocale de l’activité neuronale dans un cerveau larvaire de poisson-zèbre (4 d.p.f.) exprimant la GCaMP7a panneuronale. Barre d’échelle: 100 μm. (B) Vue agrandie de la zone encadrée en A montrant l’activité neuronale dans le tectum optique à plusieurs points temporels. Barre d’échelle: 20 μm. (C) Le rendu 3D de l’activité neuronale du cerveau entier dans le cerveau larvaire du poisson zèbre acquis à l’aide d’un microscope conçu sur mesure (gauche: t = 133 s; droite: t = 901 s). L’activité neuronale se superpose au fond statique. Barre d’échelle: 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
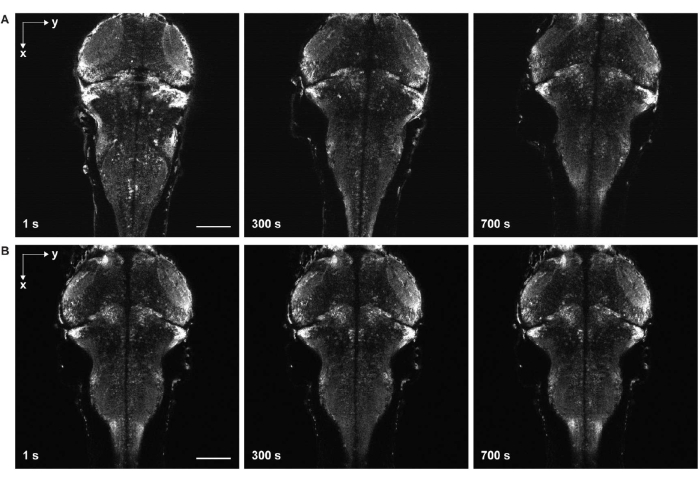
Figure 6 : Imagerie accélérée avec et sans dérive de l’échantillon. (A) Imagerie accélérée du cerveau larvaire du poisson zèbre exprimant GCaMP7a panneuronal avec dérive de l’échantillon. De l’eau d’œuf a été ajoutée à l’échantillon avant la solidification du gel d’agarose (étapes 3.6-3.7). Les poissons se déplaçaient dans les directions latérale et axiale. (B) Imagerie accélérée d’un cerveau larvaire de poisson-zèbre sans dérive de l’échantillon. De l’eau d’œuf a été ajoutée à l’échantillon après solidification du gel d’agarose (étapes 3.6-3.7). Barre d’échelle: 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Vidéo 1 : Rendu 3D de la structure cérébrale entière d’une larve de poisson-zèbre (4 d.p.f.) exprimant GCaMP7a panneuronal. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo 2 : Rendu 3D de l’activité neuronale du cerveau entier dans un cerveau larvaire de poisson-zèbre (4 d.p.f.) exprimant la GCaMP7a panneuronale. Veuillez cliquer ici pour télécharger cette vidéo.
Discussion
Le protocole actuel permet l’imagerie in vivo du cerveau entier des larves de poisson-zèbre sur une période prolongée (p. ex. plus de 1 h) et la visualisation des données d’imagerie structurelle et fonctionnelle acquises.
Les étapes les plus critiques sont le montage et le positionnement de l’échantillon. Lors de l’enrobage des échantillons, il est crucial d’empêcher la formation de bulles et d’éviter la poussière dans le gel d’agarose. Si le gel contient des bulles d’air et de la poussière, la qualité de l’image peut être gravement dégradée. Lors du positionnement de l’échantillon à l’aide d’une pince, il est important de s’assurer que l’échantillon est de niveau horizontale et verticale. Sinon, les régions d’intérêt peuvent ne pas être contenues dans le champ de vision du microscope. De plus, ce positionnement doit être effectué dans un court laps de temps avant la solidification du gel d’agarose pour éviter d’endommager sa surface, car de tels dommages compromettent la qualité de l’image.
Un autre défi majeur est d’éviter les mouvements dans les images provenant d’une solidification incomplète du gel d’agarose (Figure 6) et de la paralysie du poisson zèbre. Lorsque de l’eau d’œuf est ajoutée à l’échantillon avant la solidification complète du gel d’agorse, les poissons se déplacent lentement latéralement et axialement, se manifestant par une dérive de l’échantillon dans les images de séries chronologiques (figure 6A). Si la dose de paralytiques est insuffisante ou si la durée de l’effet prend fin, l’échantillon peut tenter de bouger, ce qui apparaît comme une contraction rapide dans les images accélérées.
Malgré l’importance des étapes susmentionnées pour l’acquisition d’images de haute qualité sans artefacts de mouvement, les protocoles 15,16,17,18,19 accessibles au public ne fournissent qu’un bref aperçu de la procédure expérimentale, sans ces détails. Par exemple, les images acquises sans protocoles d’immobilisation11 souffrent d’artefacts de mouvement substantiels qui rendent l’analyse d’image en aval difficile. En intégrant des composants essentiels, tels que la solidification du gel d’agarose et la paralysie des échantillons, notre protocole améliore considérablement la cohérence de la qualité des images acquises tout en minimisant les artefacts de mouvement.
En résumé, une procédure expérimentale optimisée et reproductible pour l’imagerie in vivo du cerveau larvaire du poisson zèbre est décrite. La validité et la reproductibilité de ce protocole pour l’imagerie in vivo de l’activité et de la structure cérébrales ont été confirmées dans de multiples contextes 14,18,29,30,31. Le présent flux de travail était axé sur l’imagerie du cerveau entier des larves de poisson-zèbre, mais il peut être facilement appliqué à l’imagerie d’autres organes de larves de poisson-zèbre35,36.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Les lignées de poisson zèbre utilisées pour l’imagerie calcique ont été fournies par le Zebrafish Center for Disease Modeling (ZCDM), Corée. Cette recherche a été soutenue par la Fondation nationale de recherche de Corée (2020R1C1C1009869, NRF2021R1A4A102159411, RS-2023-00209473).
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | SciLab | SL.Tub3513 | To aliquot agarose gel and pancuronium bromide solution |
| 15 mL Falcon tubes | Falcon | 352096 | To prepare agarose gel and pancuronium bromide solution |
| 16× 0.8NA water dipping objective lens | Nikon | CFI75 LWD 16×W | Objective lens for whole-brain imaging |
| 1-phenyl 2-thiourea (PTU) | Sigma-Aldrich | P7629-10G | 200 μM of 1-phenyl 2-thiourea (PTU) |
| 50 mL Falcon tubes | Falcon | 352070 | To prepare egg water |
| Disposable transfer pipette | SciLab | SL.Pip3032 | To transfer zebrafish larvae |
| Egg water | N/A | N/A | 0.6 g sea salt in 10 L deionized water |
| Forceps | Karl Hammacher GmbH | HSO 010-10 | Forceps used for sample positioning |
| Low melting point agarose | Thermo Scientific | R0801 | 2% (wt/vol) agarose gel |
| Napari | Napari | N/A | To visualize microscopy images in 3-D |
| NIS-Elements C | Nikon | N/A | Imaging software for confocal microscope |
| Pancuronium bromide | Sigma-Aldrich | P1918-10MG | 0.25 mg/mL of pancuronium bromide solution |
| Petri dish, 35 mm | SPL Life Sciences | 11035 | Petri dish used for sample embedding |
| Petri dish, 55 mm | SPL Life Sciences | 11050 | To prepare zebrafish larvae after screening |
| Point-scanning confocal microscopy system (C2 Plus) | Nikon | N/A | Confocal microscope for whole-brain imaging |
| Sea salt | Sigma-Aldrich | S9883-500G | Sea salt used for preparing egg water |
Références
- Choi, T. -. Y., Choi, T. -. I., Lee, Y. -. R., Choe, S. -. K., Kim, C. -. H. Zebrafish as an animal model for biomedical research. Experimental & Molecular Medicine. 53 (3), 310-317 (2021).
- Ahrens, M. B., et al. Brain-wide neuronal dynamics during motor adaptation in zebrafish. Nature. 485 (7399), 471-477 (2012).
- Panier, T., et al. Fast functional imaging of multiple brain regions in intact zebrafish larvae using selective plane illumination microscopy. Frontiers in Neural Circuits. 7, 65 (2013).
- Bianco, I. H., Kampff, A. R., Engert, F. Prey capture behavior evoked by simple visual stimuli in larval zebrafish. Frontiers in Systems Neuroscience. 5, 101 (2011).
- Akerboom, J., et al. Optimization of a GCaMP calcium indicator for neural activity imaging. Journal of Neuroscience. 32 (40), 13819-13840 (2012).
- Chen, T. -. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Piatkevich, K. D., et al. A robotic multidimensional directed evolution approach applied to fluorescent voltage reporters. Nature Chemical Biology. 14 (4), 352-360 (2018).
- Tian, L., et al. Imaging neural activity in worms, flies and mice with improved GCaMP calcium indicators. Nature Methods. 6 (12), 875-881 (2009).
- Looger, L., et al. Fast and sensitive GCaMP calcium indicators for imaging neural populations. Biorxiv. , (2021).
- Ahrens, M. B., Orger, M. B., Robson, D. N., Li, J. M., Keller, P. J. Whole-brain functional imaging at cellular resolution using light-sheet microscopy. Nature Methods. 10 (5), 413-420 (2013).
- Prevedel, R., et al. Simultaneous whole-animal 3D imaging of neuronal activity using light-field microscopy. Nature Methods. 11 (7), 727-730 (2014).
- Chhetri, R. K., et al. Whole-animal functional and developmental imaging with isotropic spatial resolution. Nature Methods. 12 (12), 1171-1178 (2015).
- Ahrens, M. B., Engert, F. Large-scale imaging in small brains. Current Opinion in Neurobiology. 32, 78-86 (2015).
- Yoon, Y. -. G., et al. Sparse decomposition light-field microscopy for high speed imaging of neuronal activity. Optica. 7 (10), 1457-1468 (2020).
- Randlett, O., et al. Whole-brain activity mapping onto a zebrafish brain atlas. Nature Methods. 12 (11), 1039-1046 (2015).
- Cong, L., et al. Rapid whole brain imaging of neural activity in freely behaving larval zebrafish (Danio rerio). eLife. 6, e28158 (2017).
- Bruzzone, M., et al. Whole brain functional recordings at cellular resolution in zebrafish larvae with 3D scanning multiphoton microscopy. Scientific Reports. 11 (1), 11048 (2021).
- Cho, E. -. S., Han, S., Lee, K. -. H., Kim, C. -. H., Yoon, Y. -. G. 3DM: deep decomposition and deconvolution microscopy for rapid neural activity imaging. Optics Express. 29 (20), 32700-32711 (2021).
- Zhang, Z., et al. Imaging volumetric dynamics at high speed in mouse and zebrafish brain with confocal light field microscopy. NatureBiotechnology. 39 (1), 74-83 (2021).
- Muto, A., Ohkura, M., Abe, G., Nakai, J., Kawakami, K. Real-time visualization of neuronal activity during perception. Current Biology. 23 (4), 307-311 (2013).
- Köster, R. W., Fraser, S. E. Tracing transgene expression in living zebrafish embryos. Developmental Biology. 233 (2), 329-346 (2001).
- Park, H. C., et al. Analysis of upstream elements in the HuC promoter leads to the establishment of transgenic zebrafish with fluorescent neurons. Developmental Biology. 227 (2), 279-293 (2000).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: an introduction. Journal of Visualized Experiments. (69), e4196 (2012).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). 4th edition. , (2000).
- Morsch, M., et al. Triggering cell stress and death using conventional UV laser confocal microscopy. Journal of Visualized Experiments. (120), e54983 (2017).
- Antinucci, P., Hindges, R. A crystal-clear zebrafish for in vivo imaging. Scientific Reports. 6, 29490 (2016).
- Parker, M. O., Brock, A. J., Millington, M. E., Brennan, C. H. Behavioral phenotyping of casper mutant and 1-pheny-2-thiourea treated adult zebrafish. Zebrafish. 10 (4), 466-471 (2013).
- Elsalini, O. A., Rohr, K. B. Phenylthiourea disrupts thyroid function in developing zebrafish. Development Genes and Evolution. 212 (12), 593-598 (2003).
- Han, S., Cho, E. -. S., Park, I., Shin, K., Yoon, Y. -. G. Efficient neural network approximation of robust PCA for automated analysis of calcium imaging data. Medical Image Computing and Computer Assisted Intervention. , 595-604 (2021).
- Shin, C., et al. Three-dimensional fluorescence microscopy through virtual refocusing using a recursive light propagation network. Medical Image Analysis. 82, 102600 (2022).
- Eom, M., et al. Statistically unbiased prediction enables accurate denoising of voltage imaging data. bioRxiv. , (2022).
- Johnston, L., et al. Electrophysiological recording in the brain of intact adult zebrafish. Journal of Visualized Experiments. (81), e51065 (2013).
- Sofroniew, N., et al. napari: a multi-dimensional image viewer for Python. Zenodo. , 3555620 (2022).
- White, R. M., et al. Transparent adult zebrafish as a tool for in vivo transplantation analysis. Cell Stem Cell. 2 (2), 183-189 (2008).
- Mickoleit, M., et al. High-resolution reconstruction of the beating zebrafish heart. Nature Methods. 11 (9), 919-922 (2014).
- Voleti, V., et al. Real-time volumetric microscopy of in vivo dynamics and large-scale samples with SCAPE 2.0. Nature Methods. 16 (10), 1054-1062 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon