Mesures de diffusion Raman améliorées en surface à molécule unique rendues possibles par des nanoantennes d’origami d’ADN plasmonique
Dans cet article
Résumé
Ce protocole démontre des mesures SERS (surface-enhanced Raman scattering) à molécule unique à l’aide d’une nanoantenne d’origami d’ADN (DONA) combinée à la microscopie à force atomique colocalisée (AFM) et aux mesures Raman.
Résumé
La diffusion Raman améliorée en surface (SERS) a la capacité de détecter des molécules uniques pour lesquelles une amélioration élevée du champ est requise. Le SERS à molécule unique (SM) est capable de fournir des informations spectroscopiques spécifiques aux molécules individuelles et fournit donc des informations chimiques plus détaillées que les autres techniques de détection SM. Dans le même temps, il est possible de démêler les informations des mesures SM qui restent cachées dans les mesures Raman de matériaux en vrac. Ce protocole décrit les mesures SM SERS à l’aide d’une nanoantenne d’origami d’ADN (DONA) en combinaison avec la microscopie à force atomique (AFM) et la spectroscopie Raman. Une structure de fourche d’origami d’ADN et deux nanoparticules d’or sont combinées pour former les DANA, avec un écart de 1,2 à 2,0 nm entre elles. Cela permet une amélioration du signal SERS jusqu’à10 fois 11, permettant des mesures de molécules uniques. Le protocole démontre en outre le placement d’une seule molécule d’analyte dans un point chaud SERS, le processus d’imagerie AFM et la superposition ultérieure de l’imagerie Raman pour mesurer un analyte dans une seule DONA.
Introduction
L’origami ADN est une technique nanotechnologique qui consiste à plier des brins d’ADN en formes et motifs spécifiques. La possibilité de créer des structures avec un contrôle précis à l’échelle nanométrique est l’un des principaux avantages de DNA origami1. La capacité de manipuler la matière à si petite échelle a le potentiel de révolutionner un large éventail de domaines, y compris la médecine, l’électronique et la science des matériaux2. Par exemple, des structures d’origami d’ADN ont été utilisées pour administrer des médicaments directement aux cellules cancéreuses 3,4, créer des capteurs à l’échelle nanométrique pour détecter des maladies5,6 et créer des motifs complexes sur les surfaces des matériaux 6,7. En outre, la possibilité d’utiliser l’origami d’ADN pour créer des structures complexes à l’échelle nanométrique a créé de nouvelles opportunités pour l’étude des processus biologiques fondamentaux à l’échelle nanométrique8.
La diffusion Raman améliorée en surface (SERS) est une technique analytique robuste qui détecte et identifie les molécules à des concentrations extrêmement faibles9. Il est basé sur l’effet Raman, qui est un changement dans la longueur d’onde de la lumière diffusée10. Le SERS nécessite un substrat métallique plasmonique pour améliorer le signal Raman des molécules adsorbées sur celui-ci. Cette amélioration peut être jusqu’à 10 11 fois supérieure au signal obtenu à partir de la même molécule mesurée par spectroscopie Raman conventionnelle, faisant du SERS une méthode très sensible pour analyser des traces de substances11.
L’amélioration du signal Raman des nanoparticules est principalement due à l’amélioration électromagnétique basée sur l’excitation de la résonance plasmonique de surface localisée (LSPR)12. Dans ce phénomène, les électrons à l’intérieur de la nanoparticule métallique oscillent collectivement autour de la surface de la nanoparticule métallique pendant l’incidence de la lumière. Il en résulte la création d’une onde stationnaire d’électrons connue sous le nom de plasmon de surface, qui peut entrer en résonance avec la lumière incidente. Le LSPR améliore considérablement le champ électrique près de la surface de la particule, et l’absorption optique de la particule est maximale à la fréquence de résonance plasmonique. L’énergie du plasmon de surface dépend de la forme et de la taille de la nanoparticule métallique, ainsi que des propriétés du milieu environnant13. Une amélioration plus importante pourrait être obtenue par des couplages plasmoniques, par exemple lorsque deux nanoparticules sont proches l’une de l’autre à une distance d’environ 2,5 fois la longueur du diamètre ou moins14. La proximité fait que le LSPR des deux nanoparticules interagit l’une avec l’autre, augmentant le champ électrique dans l’espace entre les particules de plusieurs ordres de grandeur, dépassant de loin l’amélioration d’une seule nanoparticule15,16,17. L’ampleur de l’amélioration est inversement proportionnelle à la distance entre les nanoparticules; à mesure que la distance diminue, un point critique apparaît où l’amélioration est suffisante pour détecter des molécules uniques (SM)18.
L’origami ADN représente une technologie clé qui permet d’organiser efficacement des nanoparticules plasmoniques pour exploiter et optimiser le couplage plasmonique19. Dans le même temps, les molécules d’intérêt peuvent être placées précisément à l’endroit où la détection optique est la plus efficace. Cela a été démontré avec des nanoantennes plasmoniques à base d’origami d’ADN pour la détection de fluorescence20. Contrairement à la détection de marqueurs fluorescents, le SERS offre la possibilité de détecter une empreinte chimique directe d’une molécule, ce qui rend le SERS monomoléculaire très attrayant pour la détection ainsi que pour la surveillance des réactions chimiques et des études mécanistes. L’origami d’ADN peut également être utilisé comme masque pour fabriquer des nanostructures plasmoniques aux formes bien définies21, bien que la possibilité de positionner précisément les molécules cibles dans le point chaud soit alors perdue.
En utilisant la microscopie à force atomique colocalisée (AFM) et les mesures Raman, nous pouvons obtenir les spectres Raman à partir d’une seule nanoantenne d’origami à ADN (DONA) et potentiellement d’une seule molécule si elle est placée à la position d’amélioration du signal la plus élevée. Le DONA comprend une fourche d’origami d’ADN et deux nanoparticules positionnées avec précision, entièrement recouvertes d’ADN complémentaire aux brins d’agrafes étendus attachés à l’origami d’ADN. Lors de l’hybridation de l’ADN, les nanoparticules sont liées à la fourche d’origami d’ADN avec un espace de 1,2 à 2,0 nm entre les surfaces des nanoparticules22. Un tel assemblage crée un point chaud entre les nanoparticules avec une augmentation du signal allant jusqu’à 1011 fois, calculée à partir de simulations de domaine temporel à différence finie (FDTD)22, permettant ainsi des mesures SM SERS. Cependant, le volume de l’amélioration la plus élevée est faible (dans la gamme 1-10 nm3 ), et par conséquent les molécules cibles doivent être positionnées précisément dans ce point chaud. La fourche d’origami d’ADN permet le positionnement d’une seule molécule entre les deux nanoparticules à l’aide d’un pont de fourche d’ADN et d’une chimie de couplage appropriée. Néanmoins, l’observation de tels SM dans les spectres Raman est très difficile22. Alternativement, les nanoparticules peuvent être entièrement recouvertes de la molécule cible, telle qu’un colorant TAMRA, pour permettre des mesures DONA uniques avec une intensité SERS21 plus élevée. Dans ce cas, le TAMRA est attaché de manière covalente au brin de revêtement d’ADN (Figure 1).
Protocole
1. Assemblage de fourche en origami ADN
- Auto-assemblez la structure de l’origami ADN dans un pot. Tout d’abord, ajouter 2,5 nM d’échafaudage circulaire M13mp18 (7 249 nucléotides, voir le tableau des matériaux) et 100 nM de 201 oligonucléotides courts (tableau 1) dans 1x TAE (tris[hydroxyméthyl]aminométhane [tris], acide acétique, acide éthylènediamine acétique [EDTA]) complété par 15 mM de tampon MgCl2 . Ensuite, réglez le volume total à 100 μL en utilisant de l’eau ultrapure.
- Recuit ensuite la solution par un gradient de température dans un thermocycleur, d’abord par chauffage rapide à 80 °C, puis par refroidissement de 80 °C à 20 °C à 1 °C/12 min, puis de 20 °C à 16 °C à 1 °C/6 min, suivi d’un refroidissement rapide de 16 °C à 8 °C.
- Utilisez des filtres Amicon de seuil de poids moléculaire de 100 kDa (MWCO) (voir le tableau des matériaux) pour purifier le mélange des agrafes en excès.
- Ajouter 100 μL de solution d’origami ADN aux filtres Amicon, ainsi que 400 μL d’eau ultrapure, puis centrifuger à 6 000 x g pendant 8 min à température ambiante (RT).
- Jetez le filtrat en retirant le filtre et en retournant le tube pour éliminer le lavage dans l’évier, puis ajoutez 400 μL d’eau ultrapure dans le filtre et centrifugez à nouveau. Répétez cette étape une fois de plus.
- Pour recueillir la solution de nanostructure purifiée, retourner le filtre dans un nouveau tube et centrifuger (1 000 x g, 2 min, RT) en suivant les instructions du fabricant (voir le tableau des matériaux). Cette solution peut être conservée au réfrigérateur à 8 °C jusqu’à 2 semaines.
REMARQUE: Utilisez 1x tampon TAE ou 1x TAE complété par un tampon MgCl2 de 15 mM au lieu d’eau ultrapure pour l’étape de purification du filtre Amicon, car l’eau diminue la stabilité et abaisse la concentration des fourches d’origami ADN obtenues. Pour une mesure de molécule de colorant unique, le mélange de brins d’agrafes d’ADN est remplacé par un mélange de brins modifié contenant un colorant TAMRA à l’extrémité 5'. Ce brin modifié se trouve au milieu du pont nanofork (tableau 2).
2. Revêtement de nanoparticules d’or (AuNP)
REMARQUE : Une version modifiée du protocole Liu et coll.23 a été utilisée pour enduire les AuNP, et le processus de revêtement a consisté à congeler la solution d’AuNP-ADN.
- Centrifuger (3 500 x g, 5 min, RT) 400 μL de solution AuNP de 60 nm de diamètre (obtenue commercialement; voir le tableau des matériaux) et, à l’aide d’une pipette, retirer le surnageant. Ensuite, résilibilisez la pastille dans 25 μL d’eau ultrapure. La concentration finale des AuNPs est de ~0,3 nM.
- Ajouter 1 μL de solution de tris-(2-carboxyéthyl)phosphine (TCEP) à 4 μL d’ADN modifié par le thiol (100 μM tel que fourni par le fabricant; voir le tableau des matériaux) et incuber pendant 10 minutes à TA.
- Après l’incubation, ajouter le mélange de 5 μL à la solution concentrée d’AuNP (étape 2.1), vortex pendant 5 s et congeler pendant au moins 2 h à -20 °C.
- Après décongélation à TA, centrifuger (3 500 x g, 5 min, RT) le mélange pour éliminer l’ADN de revêtement excessivement ajouté. À l’aide d’une pipette, retirer le surnageant et résorber la pastille dans 10 μL d’eau.
REMARQUE: Il y a deux brins de revêtement pour chacun des AuNP sur la nanofourche (tableau 2). Les étapes sont les mêmes à l’exception de la séquence du brin de revêtement ADN. Une fois les particules enrobées, elles sont très stables; Ils peuvent rester tels quels pendant des mois dans le réfrigérateur et peuvent même être congelés. Pour les AuNP entièrement enrobés, les brins d’ADN de revêtement ont un colorant TAMRA interne.
3. Assemblage DONA
- Ajouter la solution d’AuNP revêtue à la solution nanofork, avec un rapport molaire de concentration de 1,5:1.
- Ajouter MgCl2 à une concentration finale de 4 mMà l’aide d’une solution mère de MgCl2 de 50 mM. Régler le volume final à 20 μL avec de l’eau ultrapure.
- Hybrider les DONA à l’aide d’un gradient de température dans un thermocycleur. Tout d’abord, chauffer rapidement à 40 °C, puis refroidir de 40 °C à 20 °C à 1 °C/10 min, puis refroidir rapidement de 20 °C à 8 °C.
4. Électrophorèse sur gel
NOTE: Les nanoparticules non liées dans la solution DONA sont éliminées par électrophorèse sur gel d’agarose.
- Préparer le gel d’agarose à 1%. Pour cela, dissoudre 0,8 g d’agarose dans 80 mL de 1x TAE supplémenté de 5 mM de MgCl2.
- Ajouter 2,25 μL de tampon de charge (30 % de glycérol, 13 mM de MgCl2; voir tableau des matières) à 18 μL de solution de DONA pour atteindre une concentration finale de 5 mM MgCl2 à partir de 4 mMà l’étape 3.2. Le tampon de chargement garantit également que l’échantillon reste à l’intérieur de la poche du gel.
- Faites couler le gel pendant 60 min à 70 V dans un bain d’eau glacée. Le tampon courant est 1x TAE complété par 5 mM MgCl2.
- Découpez la bande d’intérêt et placez-la sur une lame de microscopie enveloppée de film plastique de paraffine, puis pressez la solution à l’aide d’une deuxième lame de microscopie enveloppée de film plastique de paraffine. À l’aide d’une pipette, recueillir le liquide pressé dans un tube de 500 μL.
5. Colocalisation des mesures AFM et Raman
- Traitement plasma d’une puce de silicium (voir tableau des matériaux) pendant 10 min, puis incuber 10 μL de la solution purifiée de DONA plus 10 μL de 100 mM MgCl2sur la puce pendant 3 h. Ensuite, lavez la puce deux fois avec un mélange 1:1 (en volume) d’éthanol et d’eau et séchez-la à l’air comprimé. Ensuite, collez la puce sur un disque magnétique et insérez-la dans l’instrument pour l’imagerie.
REMARQUE: Avant de commencer les mesures, la position du laser d’excitation Raman est ajustée pour être au-dessus de la pointe de la sonde AFM. Dans le présent travail, un microscope Raman HORIBA LabRAM HR Evolution couplé à un HORIBA Omegascope AFM est utilisé. L’instrument est contrôlé à l’aide des logiciels LabSpec et AIST. - Pour la mesure AFM, utilisez le mode de taraudage (mode AC) avec ACCESS-NC-A (fréquence de résonance: 300 kHz; constante du ressort: 45 N/m)pointes 24. Le mode AC contrôle automatiquement tous les paramètres à l’exception de la fréquence de balayage, qui est réglée sur 1 Hz.
REMARQUE: Après l’imagerie AFM, la pointe AFM est retirée afin qu’elle ne soit pas sur le chemin du laser Raman. Cela se fait à l’aide d’une fonction macro, « Probe away », programmée dans le système. De cette façon, pour tout point choisi dans l’image AFM, le laser sera dans la même position sans décalage par rapport à AFM. - Choisissez la longueur d’onde, la puissance et le temps d’accumulation du laser souhaités pour les mesures Raman, car tous ces paramètres dépendent de l’échantillon mesuré.
- Utilisez les paramètres mentionnés pour une mesure TAMRA AuNP entièrement revêtue: laser 633 nm, puissance de 100 μW et temps d’intégration de 1 s.
- Utilisez les paramètres mentionnés pour une seule mesure TAMRA : laser 633 nm, puissance de 400 μW et temps d’intégration de 4 s.
- Après avoir ajusté les paramètres, placez le curseur sur le DONA souhaité dans l’image AFM; La fonction « Déplacer le curseur » dans le logiciel le fait. Ensuite, démarrez la mesure Raman.
NOTE: Il existe différents modes d’acquisition des spectres: mesure en un seul point - cartographie temporelle à une dimension - où un seul point est mesuré au cours du temps25; et la cartographie de zone XY bidimensionnelle, où un étage motorisé est déplacé sous le laser pour acquérir des spectres à partir d’un réseau de points dans une grille XY26.
Résultats Représentatifs
En suivant le protocole, il faut s’assurer que la fourchette d’origami ADN est correctement assemblée; la méthode préférée pour examiner la structure des fourches est l’imagerie AFM. La plupart des fourches sont censées être solides, sans bras cassés. D’autre part, le pont entre les bras est difficile à imager en raison de son petit diamètre et de sa grande flexibilité; il nécessite également une pointe AFM très nette (Figure 2).
Le changement de couleur de la solution à chaque étape du processus de revêtement AuNP indique que tout fonctionne correctement. La couleur commence rouge foncé avec seulement les AuNPs, mais dès que l’ADN est ajouté, il passe au rouge violacé foncé. La congélation change la couleur en violet et, après décongélation, la ramène au rouge foncé (Figure 3A). La figure 3B illustre les spectres d’absorbance des AuNP nus et des AuNP recouverts d’ADN.
Après cela, dans les étapes d’assemblage DONA, la couleur reste rouge foncé tout au long du processus. Pendant la purification du gel d’agarose, une bande de dimères apparaît au-dessus de la bande libre AuNP, la bande la plus rapide de l’échantillon. Cette bande dimère correspond aux DONA et est ensuite découpée et pressée pour extraire l’échantillon (Figure 4).
Enfin, pour les mesures de colocalisation, l’imagerie AFM de l’échantillon est réalisée à la recherche de DONA (Figure 5). Ensuite, les spectres Raman de DONA uniques sont collectés et comparés pour s’assurer que les spectres obtenus proviennent de molécules TAMRA (Figure 6).

Figure 1 : Représentation schématique de la fourche d’origami d’ADN et de la DON entièrement assemblée. (A) Dimensions de la fourche d’origami d’ADN avec un pont de 90 nucléotides de long. (B) Vue latérale schématique d’une DON assemblée, avec deux AuNP et la fourche d’origami ADN entre les deux. C) Vue de dessus schématique de la DONA assemblée montrant la position de placement de la SM au milieu du pont. Veuillez cliquer ici pour voir une version agrandie de cette figure.
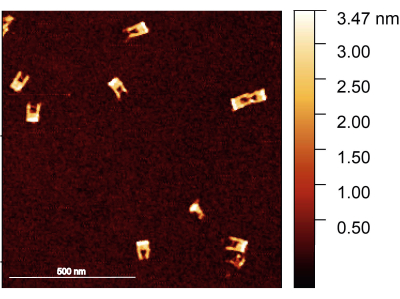
Figure 2 : Image AFM des fourches d’origami ADN après assemblage. Les fourches sont bien formées, avec le pont visible dans certaines fourches. Les fourches ont une hauteur comprise entre 1,5 et 2 nm. Barre d’échelle = 500 nm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
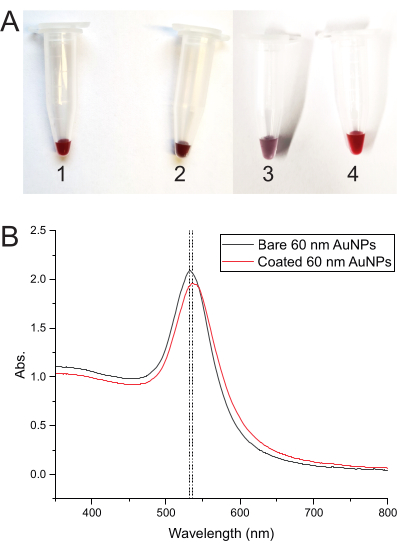
Figure 3 : Développement des couleurs et spectres d’absorbance des AuNP. (A) Tubes montrant la couleur de la solution d’AuNP en différentes étapes. (1) Couleur rouge foncé de la solution d’AuNP nue. (2) Rouge violacé foncé après l’ajout de l’ADN du revêtement aux AuNPs. (3) Couleur pourpre après congélation du mélange ADN-AuNP du revêtement. (4) La couleur revient au rouge foncé après décongélation du mélange. (B) Spectres d’absorbance des AuNP de 60 nm, montrant le déplacement du pic d’absorbance des AuNP nus (tube 1) aux AuNP recouverts d’ADN (tube 4). Veuillez cliquer ici pour voir une version agrandie de cette figure.
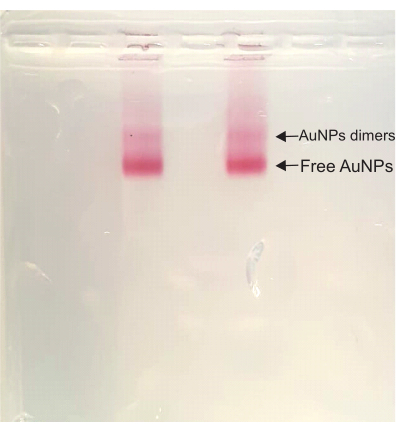
Figure 4 : Gel d’agarose de la solution DONA. Les deux voies ont le même échantillon, et la bande dimère (DONA) et la bande AuNP libre sont clairement visibles. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Image AFM des DANA. (A) L’image montre plusieurs structures de DONA; cette image AFM est utilisée pour les mesures de colocalisation. (B) Image agrandie de la DONA encerclée et de la coupe transversale verticale de la DONA. Veuillez cliquer ici pour voir une version agrandie de cette figure.
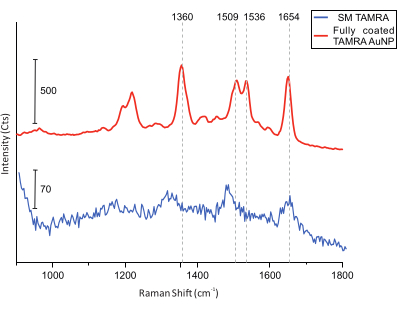
Figure 6 : Spectres SERS des DONA équipés d’AuNPs TAMRA entièrement enrobés et d’une seule molécule TAMRA. Le quadrillage vertical indique les principaux pics TAMRA. Les principaux pics TAMRA sont visibles dans le TAMRA AuNP entièrement revêtu. Bien que le rapport signal sur bruit pour les spectres SM TAMRA soit plus faible, les principaux pics sont identifiables : 1 360 cm-1 : étirement C-C ; 1 509 cm-1 : étirement C=C; 1 536 cm-1 : étirement C=C; 1 654 cm-1 : étirement C=O. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Liste des agrafes de fourche d’origami ADN. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 2 : Liste des brins d’ADN modifiés. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
SM SERS est un outil puissant qui permet aux chercheurs d’étudier le comportement et les interactions de molécules individuelles au sein d’un échantillon1. Une telle technique permet l’analyse de systèmes à un niveau de sensibilité sans précédent, fournissant de nouvelles informations sur le comportement fondamental de molécules individuelles et la distribution des propriétés chimiques ou physiques sur un ensemble de molécules, et aide à identifier les intermédiaires pertinents dans les processus chimiques. Cependant, placer une seule molécule dans le point chaud tout en s’assurant que le point chaud a suffisamment d’amélioration de surface peut être assez difficile27. Les DONA décrits dans ce protocole peuvent placer avec précision une seule molécule dans le point chaud entre deux nanoparticules d’or tout en assurant une amélioration de surface de 10à 11 fois.
L’écart entre les nanoparticules est essentiel pour que l’assemblage DONA atteigne l’amélioration de surface requise pour les études SM SERS. Les DONA sont optimisés pour les nanoparticules sphériques de tailles comprises entre 60 et 80 nm. De plus, la qualité des nanoparticules peut avoir un impact significatif sur l’hybridation des nanoparticules avec les fourches d’origami d’ADN ; Lorsque les nanoparticules utilisées dans l’étape du revêtement ont plus de 6 mois, l’efficacité de l’hybridation commence à diminuer.
Un autre aspect critique du protocole est que les étapes qui nécessitent un rapport exact entre les composants doivent être suivies avec précision, sinon les DONA ne seront pas formés correctement. La fourche en origami DNA est extrêmement sensible au protocole d’augmentation de la température, avec des changements affectant l’intégrité de la structure ou empêchant la formation de fourches.
La génération de carbone amorphe est un problème important lors des mesures SERS car ses pics sont généralement dans la même gamme que la zone d’empreinte digitale pour de nombreuses molécules (1 200-1 700 cm-1). Bien que la formation ne soit pas encore entièrement comprise, elle est généralement associée à une puissance laser élevée ou à de longs temps d’intégration28. Par mesure de précaution, la puissance laser la plus faible et le temps d’intégration le plus court possible doivent être utilisés. Cependant, cela n’est pas facile à accomplir car un équilibre doit être atteint entre l’obtention du signal SERS souhaité et la prévention de la génération de carbone amorphe.
Les DONA sont très polyvalents en tant que système SM en ce qui concerne les différents types et formes de nanoparticules qui pourraient être utilisés, tels que les sphères d’argent, les fleurs d’or ou les étoiles. De plus, la molécule étudiée peut être facilement remplacée en changeant uniquement le brin d’ADN au milieu du pont, sans modification de la procédure. Le passage à des protéines comme le cytochrome C pourrait se faire en ayant un brin de capture d’ADN modifié par la pyridine dans le pont, ce qui lierait le cytochrome C et garantirait qu’il est dans le point chaud pour les mesures SM SERS22. Cela se traduit également par un choix flexible du laser pour l’irradiation, en utilisant potentiellement un laser qui offre une amélioration maximale.
En résumé, cette méthode est fiable pour assembler des structures DONA et les utiliser pour des mesures de spectroscopie Raman à molécule unique améliorées en surface.
Déclarations de divulgation
Les auteurs n’ont pas d’intérêts financiers concurrents ou d’autres conflits d’intérêts.
Remerciements
Cette recherche a été soutenue par le Conseil européen de la recherche (ERC; subvention de consolidation n° 772752).
matériels
| Name | Company | Catalog Number | Comments |
| 100 kDa MWCO Amicon filters, 0.5 mL | Merck | UFC5100BK | |
| 10x TAE buffer (0.4 M Tris, 0.2 M acetic acid, 0.01 M EDTA) | SIGMA Aldrich | T9650 | |
| 60 nm goldspheres, bare (citrate) | NanoComposix | AUCN60 | |
| ACCESS-NC-A AFM probes | SCHAEFER-TEC | ||
| Agarose powder | SIGMA Aldrich | 9012-36-6 | |
| AuNP DNA coating strands | IDT | ||
| AuNP DNA coating strands (TAMRA) | SIGMA Aldrich | ||
| Glycerol | SIGMA Aldrich | 56-81-5 | |
| Heraeus Fresco 17 centrifuge | Thermo Fisher Scientific | ||
| HORIBA OmegaScope with a LabRAM HR evolution | HORIBA | ||
| Magnesium chloride | SIGMA Aldrich | 7786-30-3 | |
| Nanofork DNA bridge strand (TAMRA) | Metabion | ||
| Nanofork DNA staple strands | SIGMA Aldrich | ||
| ParafilmM | Carl Roth | CNP8.1 | |
| Primus 25 Thermocycler | Peqlab/VWR | ||
| Silicon wafer | Siegert wafer | BW14076 | |
| Single-stranded scaffold DNA, type p7249 (M13mp18) | Tilibit nanosystems | ||
| TCEP solution | SIGMA Aldrich | 51805-45-9 |
Références
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Placement of single proteins within the SERS hot spots of self-assembled silver nanolenses. Angewandte Chemie. 57 (25), 7444-7447 (2018).
- Dey, S., et al. DNA origami. Nature Reviews Methods Primers. 1 (1), 13 (2021).
- Tasciotti, E. Smart cancer therapy with DNA origami. Nature Biotechnology. 36 (3), 234-235 (2018).
- Ijäs, H., et al. Unraveling the interaction between doxorubicin and DNA origami nanostructures for customizable chemotherapeutic drug release. Nucleic Acids Research. 49 (6), 3048-3062 (2021).
- Wang, S., et al. DNA origami-enabled biosensors. Sensors. 20 (23), 6899 (2020).
- Kabusure, K. M., et al. Optical characterization of DNA origami-shaped silver nanoparticles created through biotemplated lithography. Nanoscale. 14 (27), 9648-9654 (2022).
- Kershner, R. J., et al. Placement and orientation of individual DNA shapes on lithographically patterned surfaces. Nature Nanotechnology. 4 (9), 557-561 (2009).
- Dutta, A., et al. Molecular states and spin crossover of hemin studied by DNA origami enabled single-molecule surface-enhanced Raman scattering. Nanoscale. 14 (44), 16467-16478 (2022).
- Langer, J., et al. Present and future of surface-enhanced Raman scattering. ACS Nano. 14 (1), 28-117 (2020).
- Jones, R. R., Hooper, D. C., Zhang, L., Wolverson, D., Valev, V. K. Raman techniques: fundamentals and frontiers. Nanoscale Research Letters. 14 (1), 231 (2019).
- Blackie, E. J., Le Ru, E. C., Etchegoin, P. G. Single-molecule surface-enhanced Raman spectroscopy of nonresonant molecules. Journal of the American Chemical Society. 131 (40), 14466-14472 (2009).
- Sepúlveda, B., Angelomé, P. C., Lechuga, L. M., Liz-Marzán, L. M. LSPR-based nanobiosensors. Nano Today. 4 (3), 244-251 (2009).
- Kelly, K. L., Coronado, E., Zhao, L. L., Schatz, G. C. The optical properties of metal nanoparticles: The influence of size, shape, and dielectric environment. The Journal of Physical Chemistry B. 107 (3), 668-677 (2003).
- Funston, A. M., Novo, C., Davis, T. J., Mulvaney, P. Plasmon coupling of gold nanorods at short distances and in different geometries. Nano Letters. 9 (4), 1651-1658 (2009).
- Niu, R., et al. DNA origami-based nanoprinting for the assembly of plasmonic nanostructures with single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 60 (21), 11695-11701 (2021).
- Fang, W., et al. Quantizing single-molecule surface-enhanced Raman scattering with DNA origami metamolecules. Science Advances. 5 (9), (2019).
- Zhan, P., et al. DNA origami directed assembly of gold bowtie nanoantennas for single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 57 (11), 2846-2850 (2018).
- Choi, H. K., et al. Single-molecule surface-enhanced Raman scattering as a probe of single-molecule surface reactions: promises and current challenges. Accounts of Chemical Research. 52 (11), 3008-3017 (2019).
- Liu, N., Liedl, T. DNA-assembled advanced plasmonic architectures. Chemical Reviews. 118 (6), 3032-3053 (2018).
- Acuna, G. P., et al. Fluorescence enhancement at docking sites of DNA-directed self-assembled nanoantennas. Science. 338 (6106), 506-510 (2012).
- Shen, B., Kostiainen, M. A., Linko, V. DNA origami nanophotonics and plasmonics at interfaces. Langmuir. 34 (49), 14911-14920 (2018).
- Tapio, K., et al. A versatile DNA origami-based plasmonic nanoantenna for label-free single-molecule surface-enhanced Raman spectroscopy. ACS Nano. 15 (4), 7065-7077 (2021).
- Liu, B., Liu, J. Freezing-driven DNA adsorption on gold nanoparticles: tolerating extremely low salt concentration but requiring high DNA concentration. Langmuir. 35 (19), 6476-6482 (2019).
- Aliano, A., et al. AFM, tapping mode. Encyclopedia of Nanotechnology. , 99 (2012).
- Recording Raman spectral images and profiles. Horiba Scientific Available from: https://www.horiba.com/int/scientific/technologies/raman-imaging-and-spcetroscopy/recording-spectral-images-and=profiles/ (2023)
- Raman Images Explained. Renishaw Available from: https://www.renishaw.com/en/raman-images-explained-25810 (2023)
- Kogikoski, S., Tapio, K., von Zander, R. E., Saalfrank, P., Bald, I. Raman enhancement of nanoparticle dimers self-assembled using DNA origami nanotriangles. Molecules. 26 (6), 1684 (2021).
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Amorphous carbon generation as a photocatalytic reaction on DNA-assembled gold and silver nanostructures. Molecules. 24 (12), 2324 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon