Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détection du virus SARS-CoV-2 par amplification isotherme médiée par boucle de transcription inverse
Dans cet article
Résumé
Nous fournissons ici un protocole complet pour normaliser et mettre en œuvre la méthode de détection du virus SARS-CoV-2 dans des échantillons humains par amplification isotherme médiée par boucle de transcription inverse (RT-LAMP). Cette méthode, réalisée en 60 min, pourrait être adaptée à n’importe quel laboratoire ou point de service à faible coût et à l’aide d’équipements peu coûteux.
Résumé
Le virus du coronavirus 2 du syndrome respiratoire aigu sévère (SRAS-CoV-2) a eu un impact considérable sur la santé humaine. Il continue d’être une menace pour la société moderne car de nombreuses personnes meurent des suites de l’infection. La maladie est diagnostiquée à l’aide de tests sérologiques et moléculaires, tels que l’amplification en chaîne par polymérase en temps réel (RT-PCR). Ce dernier présente plusieurs inconvénients car il nécessite une infrastructure spécialisée, un équipement coûteux et un personnel formé. Nous présentons ici un protocole décrivant les étapes nécessaires à la détection du virus SARS-CoV-2 à l’aide de l’amplification isotherme médiée par boucle de transcription inverse (RT-LAMP) dans des échantillons humains. Le protocole comprend des instructions pour la conception d’amorces in silico, la préparation des réactifs, l’amplification et la visualisation. Une fois standardisée, cette méthode peut être facilement mise en œuvre et adaptée à n’importe quel laboratoire ou point de service en 60 minutes à faible coût et en utilisant un équipement peu coûteux. Il est adaptable à la détection de différents agents pathogènes. Ainsi, il peut potentiellement être utilisé sur le terrain et dans les centres de santé pour effectuer une surveillance épidémiologique en temps opportun.
Introduction
Le coronavirus 2 du syndrome respiratoire aigu sévère (SRAS-CoV-2) est à l’origine de la maladie à coronavirus 2019 (COVID-19). L’Organisation mondiale de la santé a déclaré une urgence de santé publique de portée internationale le 30 janvier 2020 et une pandémie le 11 mars 2020. La pandémie a entraîné plus de 760 millions de cas et 6,87 millions de décès à la date de rédaction de cet article1.
L’impact de ce virus a mis en évidence la nécessité de disposer d’outils de surveillance meilleurs, plus précis, plus rapides et plus largement disponibles pour améliorer la détection et le contrôle des maladies infectieuses 2,3. Pendant la pandémie, les tests de diagnostic du SRAS-CoV-2 étaient basés sur la détection de l’acide nucléique, des anticorps et des protéines, mais la détection de l’acide nucléique par RT-PCR est l’étalon-or4. Cependant, la RT-PCR présente certaines limites ; Elle nécessite de l’équipement spécialisé, des infrastructures et du personnel formé en biologie moléculaire, ce qui limite son application aux laboratoires spécialisés. De plus, cela prend du temps (4 à 6 h), sans compter le temps de transport des échantillons au laboratoire, ce qui peut prendre5 jours. Ces contraintes empêchent le traitement efficace des échantillons et l’obtention de l’information nécessaire à la planification d’urgence et à la gestion épidémiologique.
L’amplification isotherme médiée par boucle de transcription inverse (RT-LAMP) présente plusieurs avantages par rapport à la RT-PCR, ce qui en fait une stratégie attrayante pour la conception de futurs tests de diagnostic sur le lieu de soins (POCT), en particulier dans les environnements aux ressources limitées6. Tout d’abord, il est très spécifique car il utilise entre quatre et six amorces qui reconnaissent six à huit zones de la séquence cible, qu’il s’agisse d’ADN ou d’ARN 7,8. Deuxièmement, parce qu’il fonctionne à une température constante, il n’a pas besoin d’équipement sophistiqué tel que des thermocycleurs en temps réel pour générer l’amplification, ni de personnel hautement qualifié pour le faire fonctionner. Troisièmement, le temps de réaction est très court (~60 min), et des réactifs peu spécialisés sont utilisés, ce qui en fait un outil rentable6. Compte tenu de ce qui précède et de l’urgence sanitaire causée par la pandémie de COVID-19, cette technique peut être considérée comme une méthode de diagnostic alternative, rapide, peu coûteuse et simple à mettre en œuvre dans n’importe quel laboratoire de recherche9.
Le protocole de normalisation et de mise en œuvre d’un RT-LAMP pour détecter le SARS-CoV-2 par des méthodes colorimétriques à l’aide d’un thermocycleur et d’un bain-marie est décrit dans cet article (Figure 1). Les points critiques, leurs limites et les alternatives pour les faire progresser sont discutés.
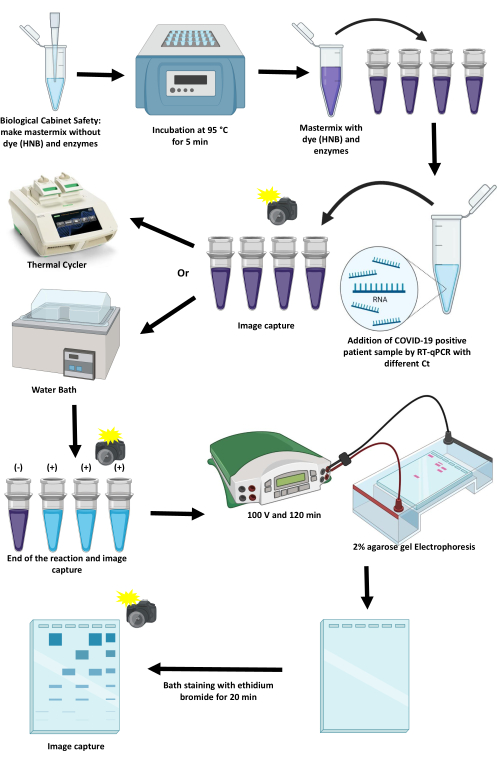
Figure 1 : Schéma du protocole d’amplification du SARS-CoV-2 à l’aide de la technique RT-LAMP. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
Les échantillons utilisés ont été fournis par le laboratoire clinique de l’hôpital universitaire Fundación Valle del Lili et correspondaient à l’ARN purifié de patients testés positifs au COVID-19 à l’aide de la technique RT-qPCR. Tous les patients ont fourni un consentement éclairé pour la recherche, et cette étude a été approuvée par le comité de bioéthique des études humaines de l’hôpital universitaire Fundación Valle del Lili.
1. Conception et préparation de l’apprêt RT-LAMP
REMARQUE : Les amorces LAMP peuvent être utilisées avec une variété de plateformes, y compris New England BioLabs (NEB) LAMP, Primer Explorer et LAMP assay versatile analysis (LAVA). Toutefois, pour ce protocole, l’outil LAMP de l’ONÉ a été utilisé. La conception des amorces peut être réalisée à l’aide des génomes du SRAS-CoV-2 obtenus à partir de la base de donnéesNextStrain 10. Le tableau 1 montre le jeu d’amorces utilisé dans ce protocole.
- Conception d’amorce pour LAMP
- Obtenir des séquences du génome viral.
- Effectuez des alignements de séquences pour obtenir la séquence consensuelle.
- Accédez à la plate-forme11 de l’outil de conception d’amorces NEB LAMP et suivez les instructions du guide rapide. Cet outil produit les mêmes résultats que le primer explorer V5, mais il est beaucoup plus convivial dans sa sortie. Utilisez les manuels d’utilisation de l’explorateur d’amorces comme guide pour la conception des amorces.
- Évaluation thermodynamique de l’ensemble des amorces
- À l’aide de l’outil Primer-Dimer12 , effectuez une analyse thermodynamique sur les amorces obtenues.
- Mettez les séquences d’amorçage dans l’outil. Ensuite, sélectionnez l’option Analyse multiplex et rapport de structure de dimères.
- Sélectionnez les jeux d’amorces dont ΔG n’est pas inférieur à -5.
- Évaluation de la spécificité des amorces conçues
- Utilisez la base de données de collecte de nucléotides (nt/nt) dans BLAST13 pour analyser chaque amorce.
- Pour effectuer la première analyse BLAST, sélectionnez la base de données Refseq_rna et filtrez la recherche avec le groupe de genres appartenant à la sous-famille des Orthocoronavirinae. Il s’agit de l’Alphacoronavirus (taxid :693996), du Gammacoronavirus (taxid :694013) et du Deltacoronavirus (taxid :1159901). De plus, évaluez la séquence par rapport à d’autres virus qui circulent en même temps que le sous-type H1N1 (taxid :114727), le virus de la grippe A (taxid :11320) et le virus de la grippe B (taxid :11520).
- Pour effectuer la deuxième analyse BLAST, sélectionnez le Betacoronavirus GenBank et filtrez la recherche avec Coronaviridae (taxid :11118) et SARS (taxid :694009). Ces groupes contiennent des séquences de tous les génomes identifiés du coronavirus du SRAS, y compris les génomes trouvés chez les chauves-souris, le bêtacoronavirus (taxid :694002).
- Pour ce protocole, assurez-vous que les amorces ne s’alignent pas avec des génomes autres que le génome cible, le SRAS-CoV-2.
- Préparation de l’apprêt
- Faites tourner les flacons contenant les amorces Iyophilisées à l’aide d’une microcentrifugeuse (10 000 x g, 1 min à température ambiante [RT]) pour éviter les pertes lors de l’ouverture du tube.
- Réhydrater la poudre d’Iyaphilise dans de l’eau de pyrocarbonate de diéthyle à 0,1 % (DEPC) ou de l’eau sans nucléase jusqu’à une concentration finale de 100 μM (tableau 2) et dissoudre complètement par pipetage de haut en bas. Ensuite, essorez à vitesse maximale (10 000 x g, 1 min à RT) dans une microcentrifugeuse pour recueillir toutes les solutions d’amorce au fond du tube.
- Préparez le mélange d’amorces 10x sous une enceinte de biosécurité avec l’amorce intérieure avant (FIP), l’amorce intérieure arrière (BIP), l’amorce extérieure avant (F3), l’amorce extérieure arrière (B3), l’amorce bouclée arrière (LB) et les amorces bouclées avant (LF), comme indiqué dans le tableau 2. Pour éviter les pertes, pipetez ou agitez doucement la solution d’amorce avant d’effectuer un essorage rapide (10 000 x g, 1 min à RT) avec une microcentrifugeuse.
- Conservez le mélange d’apprêt 10x à −20 °C pour un stockage à long terme ; Cependant, préparez-vous suffisamment pour un maximum de cinq expériences, indépendamment de plusieurs échantillons, afin d’éviter trop de cycles de gel-dégel.
REMARQUE Si un volume plus petit du mélange d’apprêt est nécessaire, ajustez les valeurs en calculant les nouveaux volumes (Tableau 2). De plus, les ensembles RdRp et RdRp/Hel n’incluent pas l’amorce LF car les amorces en boucle ne sont pas nécessaires pour les réactions RT-LAMP. Par conséquent, remplacez le volume de l’apprêt LF par de l’eau sans nucléase ou de l’eau à 0,1 % de DEPC.
2. Réaction RT-LAMP
- Allumez l’armoire à flux laminaire conformément aux instructions du fabricant et attendez au moins 3 minutes pour que le flux d’air se stabilise.
- Une fois que le flux d’air est stable, nettoyez et désinfectez les surfaces internes de l’armoire à l’aide d’une technique aseptique. Pour ce faire, utilisez les désinfectants suivants dans cet ordre : 1000 ppm d’ammonium quaternaire (chlorure de benzalkonium), 2 % d’hypochlorite, 3 % de peroxyde d’hydrogène et 70 % d’éthanol.
REMARQUE : Dans ce cas, la technique aseptique consiste à appliquer le désinfectant et à le retirer avec des serviettes de l’intérieur de la cabine vers l’extérieur sans passer sur les surfaces préalablement nettoyées. - À l’aide des désinfectants de l’étape 2.2, nettoyez les matériaux qui entreront dans la cabine dans le même ordre.
REMARQUE : Il faut apporter dans l’armoire des micropipettes, des boîtes d’embouts de filtre, des flacons avec des tubes de 1,5 ml et 0,6 ml, des tubes PCR de 0,2 ml, des supports et un bécher de 400 ml. - Apportez des serviettes et des gants en nitrile dans la cabine. Après cela, éteignez l’armoire et exposez-la à la lumière ultraviolette (UV) pendant 15 min.
ATTENTION : Pour éviter d’endommager les tissus et l’ADN d’une exposition prolongée aux rayonnements, évitez la lumière UV jusqu’à l’expiration du temps défini à l’étape 2.4.
REMARQUE Effectuez l’assemblage illustré à la figure 2 avant de commencer le protocole et commencez le bain-marie après avoir terminé l’étape 2.4. Il est crucial de remplir le récipient métallique presque à ras bord d’eau potable et de régler la température de la plaque chauffante de laboratoire en fer à 90 °C, car cela se traduira par une température de ~66,3 °C dans le système, qui est surveillée par le thermomètre à mercure. - Une fois la période d’irradiation terminée, redémarrez l’armoire et suivez les recommandations de l’étape 1.1.
- Placez les réactifs (tableau 3, tableau 4 et tableau 5) dans une glacière remplie de glace ou un petit réfrigérateur en polystyrène. Mettez le récipient dans l’armoire après l’avoir nettoyé avec de l’éthanol à 70%.
- Dans un tube de microcentrifugation de 0,6 mL, préparer le mélange LAMP du gène à amplifier (RdRp, N-A et RdRp/Hel), en ajoutant uniquement les composants suivants : 10x tampon, MgSO4, dNTPs, 1x mélange d’amorces et de l’eau sans nucléases ou 0,1% d’eau DEPC ; Bien mélanger par pipetage pour homogénéiser.
ATTENTION : En raison d’une manipulation et d’un comportement inappropriés à l’intérieur de l’armoire, il existe un risque élevé de contamination par les réactifs. Les règles suivantes doivent être suivies pour atténuer ce problème : (i) utiliser des embouts stériles et des pointes filtrantes ; (ii) utiliser une pointe pour chaque réactif ; (iii) se déplacer lentement et prudemment pour éviter de perturber l’écoulement laminaire ; (iv) maintenir l’ordre et utiliser le moins de matériaux possible ; et (v) utiliser des gants différents pour préparer le mélange et ajouter le matériel génétique.
REMARQUE : Conservez tous les réactifs, en particulier les enzymes, sur de la glace, car les changements de température peuvent les dénaturer et altérer l’activité de la polymérase. - Placez le(s) tube(s) de 0,6 mL avec le capuchon fermé dans un bloc chauffant et incubez à 95 °C pendant 5 min.
REMARQUE : Allumez le bloc chauffant pour les tubes de 1,5 à 2,0 ml situés à l’extérieur de l’armoire pendant au moins 30 minutes avant de commencer la préparation du mélange LAMP et surveillez la température (95 °C) avec un thermomètre à mercure ou à alcool. - Une fois l’incubation terminée, placez les tubes dans une glacière en polystyrène remplie de glace pendant 5 min.
- Remettez les tubes dans l’armoire à flux laminaire et terminez la préparation du mélange LAMP en ajoutant les enzymes ADN polymérase (Bst 3.0), transcriptase inverse et ADN polymérase haute fidélité (tableau 3, tableau 4 et tableau 5). Dans le cas de l’utilisation de la détection colorimétrique, ajoutez le colorant bleu d’hydroxinaphtol (HNB).
- Après avoir ajouté ces réactifs, mélangez très bien les réactifs LAMP en les pipetant pour solubiliser les enzymes et le colorant.
- Remplissez chaque tube PCR avec 22,0 μL du mélange, en faisant attention de ne pas créer de bulles. Ensuite, ajoutez 3,0 μL d’eau à 0,1 % de DEPC ou d’eau exempte de nucléases dans le témoin négatif ou le tube sans matrice (NTC) et mettez de côté le ou les tubes restants pour l’ajout (matériel génétique).
REMARQUE : Conservez les tubes PCR dans une glacière remplie de glace jusqu’à ce que l’échantillon soit ajouté afin d’éviter d’activer l’enzyme Bst 3.0 et de démarrer prématurément la réaction. - Retirez tous les matériaux de l’armoire et utilisez de l’éthanol à 70 % pour nettoyer les surfaces. Ensuite, éteignez-le en suivant les instructions du fabricant.
- Dans une zone séparée, ajoutez 3 μL de l’échantillon dans chaque tube PCR et homogénéisez-le soigneusement. Pour ce faire, utilisez une micropipette de 20 μL et des embouts filtrants.
ATTENTION : La micropipette utilisée pour ajouter le matériel génétique doit être utilisée exclusivement à cet effet et ne peut pas être utilisée pour préparer le mélange. De cette façon, la contamination des réactifs est évitée. De plus, gardez les échantillons d’ARN sur la glace en tout temps pour réduire le risque de dégradation de l’ARN. Utilisez l’équipement de protection individuelle (EPI) suivant pour l’ajout d’échantillons : blouse jetable, casquette, masque N95, leggings, lunettes de laboratoire et gants en nitrile. - Avant d’effectuer la réaction colorimétrique, prenez des photos des tubes PCR avec un appareil photo de haute qualité. La couleur de départ avec HNB est le violet.
- Effectuer la réaction dans le système ou l’équipement suivant : (i) thermocycleur et (ii) bain-marie.
- Thermocycleur : Déposer les tubes dans le bloc de réaction et mettre en place le thermoprofil (voir tableau 6) sur l’équipement.
- Bain-marie : Déposez les tubes dans des récipients circulaires et ajustez-les très bien pour éviter qu’ils ne sortent. Après cela, placez les récipients dans le bain-marie (Figure 2A, B) à la température indiquée dans le tableau 6.
- Dans le cas du bain-marie, une fois que les tubes sont à l’intérieur du système, démarrez la minuterie pendant 60 min (tableau 6).
- Retirez les tubes du thermocycleur ou du bain-marie après le temps de réaction et rangez-les à 4 °C pour le fonctionnement électrophorétique ou à -20 °C jusqu’à l’utilisation.
- Si une réaction colorimétrique a été effectuée, prenez des photos des tubes PCR à l’aide d’un appareil photo de haute qualité. La couleur finale avec HNB est le bleu ciel.
3. Analyse des produits d’amplification dans le gel d’agarose
REMARQUE : Ces étapes sont suggérées comme vérifications supplémentaires de la réaction colorimétrique ou contrôle des performances au cours de l’étape de normalisation. En effet, la technique pourrait présenter un énorme risque de contamination pour le laboratoire qui effectue ces tests.
- Placez le lit à l’intérieur de la chambre d’électrophorèse de manière à ce que les caoutchoucs de bord touchent les parois, créant ainsi un espace scellé pour l’ajout d’agarose (chambre interne) (Figure 3A, B).
- Après avoir terminé l’étape 3.1, peser la quantité nécessaire d’agarose dans un bécher de 500 ml pour obtenir un gel à 2 %. Après cela, ajoutez le volume requis de tampon 0,5x Tris-acétate EDTA (TAE) et passez au micro-ondes pendant 1 à 2 min.
REMARQUE : L’agarose est complètement fondue lorsqu’elle est translucide et sans grumeaux lorsqu’elle est retirée du four. Si cela n’est pas confirmé, des régions mal gélifiées peuvent subsister, ce qui entraîne une altération du fonctionnement électrophorétique et de la visualisation des produits d’amplification. - Sortez le bécher du four et versez l’agarose dans la chambre interne créée à l’étape 3.1 (Figure 3C). Par la suite, vérifiez qu’il n’y a pas de bulles, et s’il y en a, retirez-les à l’aide d’une pointe de micropipette.
- Disposez le peigne pour former les puits et laissez l’agarose gélifier pendant environ 30 min à température ambiante (RT).
- Après ce temps, ajoutez 5 ml de tampon TAE 0,5x pour faciliter le retrait des rayons et du lit contenant le gel. Positionnez ensuite le gel de manière à ce que les puits soient dans l’anode (Figure 3D).
- Remplissez la chambre d’électrophorèse avec un tampon TAE 0,5x à la capacité spécifiée par le fabricant, en vous assurant que les électrodes sont en contact avec le tampon.
- Ajoutez 3 μL de marqueur de poids moléculaire dans le premier puits du gel et ajoutez 9 μL de NTC et chaque échantillon dans les puits suivants. Faites-les en combinant 7 μL de produit d’amplification avec 3 μL de tampon de chargement ; puis chargez 9 μL de ce mélange dans les puits du gel.
- Couvrez la chambre d’électrophorèse avec le couvercle et connectez les câbles aux ports d’alimentation dans le motif de couleur. Réglez la source d’alimentation sur les paramètres suivants : 100 V et ampérage constant pendant 120 min.
- Une fois l’électrophorèse terminée, placez le gel dans le récipient avec la solution de coloration (bromure d’éthidium) et incubez pendant 30 min.
- Après l’incubation, retirez le gel de la solution de coloration et placez-le dans un sac à fermeture éclair. Cela permet d’éviter la contamination de l’équipement qui sera utilisé pour visualiser les amplicons.
- Visualisez le gel sur un transilluminateur ou un imageur comme l’Amersham Imager 600.
Résultats
La mise en œuvre du protocole commence par la conception de l’ensemble des amorces pour chaque gène cible selon le protocole décrit ci-dessus. En juin 2020, 5 000 génomes du SARS-CoV-2 ont été obtenus à partir de la base de données NextStrain avec une représentativité de 10 % des génomes colombiens. Ces séquences ont été alignées pour obtenir la séquence consensuelle qui a été utilisée dans le processus de conception de l’amorce. Le tableau 1 montre le jeu d’amorces choisi pour l...
Discussion
Bien que le RT-LAMP soit considéré comme une méthodologie complémentaire pour effectuer des diagnostics moléculaires, il présente également certaines limites et étapes critiques qui doivent être prises en compte lors de la normalisation et de la mise en œuvre du protocole.
La normalisation LAMP pour la détection du SARS-CoV-2 a évalué les paramètres et composants suivants dans le mélange maître : (a) concentration et température d’alignement des amorces ; b) la concentration...
Déclarations de divulgation
Natalia Campillo-Pedroza est PDG de l’entreprise BioDx : Diagnóstico y Soluciones Biotecnológicas S.A.S. Les autres auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été financé par le Sistema General de Regalías de Colombie, numéro de subvention BPIN 2020000100092, et l’Universidad Icesi - Convocatoria Interna, numéro de subvention CA0413119. La MFVT a également été financée par les fonds de chaire adjointe de l’Universidad de los Andes. Les entités de financement n’ont pas participé à la conception, à l’exécution des activités, à la collecte des données, à l’analyse des données et à la préparation du manuscrit. Nous remercions l’hôpital universitaire Fundación Valle del Lili pour l’ARN viral des échantillons de Sars-CoV-2 et le Dr Alvaro Barrera-Ocampo pour les commentaires sur le manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| 1 kb DNA Ladder | SOLIS BIODYNE | 07-12-00050 | Store at -20 °C |
| 50x TAE Electrophoresis Buffer | ThermoScientific | B49 | Store at roome temperature |
| Accuris High Fidelity Polymerase | ACCURIS LIFE SCIENCE REAGENTS | PR1000-HF-200 | It can be used in case Q5 High-Fidelity DNA polymerase cannot be purchased. For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Agarose | PanReacAppliChem | A8963,0100 | N/A |
| Bst 3.0 DNA Polymerase 8000 IU/mL | New England BioLabs | M0374S/M0374L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Deoxynucleotide (dNTP) Solution Set | New England BioLabs | N0446S | Store at -20 °C |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220-25G | Handle it with caution under an extraction cabinet |
| GeneRuler 100 bp Plus DNA Ladder, ready-to-use | ThermoScientific | SM0322 | Store at -20 °C |
| Hydroxy naphthol blue disodium salt | Santa Cruz Biotechnology | sc-215156B | N/A |
| Q5 High-Fidelity DNA polymerase 2000 IU/mL | New England BioLabs | M0491S/M0491L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| WarmStart RTx Reverse Transcriptase 15000 IU/mL | New England BioLabs | M0380S/M0380L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
Références
- World Health Organization. . Who coronavirus (COVID-19) dashboard (no date). , (2023).
- Ibrahim, N. K. Epidemiologic surveillance for controlling Covid-19 pandemic: types, challenges and implications. Journal of Infection and Public Health. 13 (11), 1630-1638 (2020).
- Rojas-Gallardo, D. M., et al. COVID-19 in Latin America: Contrasting phylodynamic inference with epidemiological surveillance. (Molecular epidemiology of COVID-19 in Latin America). medRxiv. , (2020).
- Liu, R., et al. Positive rate of RT-PCR detection of SARS-CoV-2 infection in 4880 cases from one hospital in Wuhan, China, from Jan to Feb 2020. Clinica Chimica Acta. 505, 172-175 (2020).
- Kevadiya, B. D., et al. Diagnostics for SARS-CoV-2 infections. Nature Materials. 20 (5), 593-605 (2021).
- Tomita, N., Mori, Y., Kanda, H., Notomi, T. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nature Protocols. 3 (5), 877-882 (2008).
- Li, Y., Fan, P., Zhou, S., Zhang, L. Loop-mediated isothermal amplification (LAMP): A novel rapid detection platform for pathogens. Microbial Pathogenesis. 107, 54-61 (2017).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): principle, features, and future prospects. Journal of Microbiology. 53 (1), 1-5 (2015).
- Augustine, R., et al. Loop-mediated isothermal amplification (LAMP): A rapid, sensitive, specific, and cost-effective point-of-care test for coronaviruses in the context of COVID-19 pandemic. Biology (Basel). 9 (8), 182 (2020).
- . Nextstrain Available from: https://nextstrain.org/ (2023)
- . Neb Lamp, NEB LAMP Available from: https://lamp.neb.com/ (2023)
- . Blast: Basic local alignment search tool (no date) Available from: https://blast.ncbi.nlm.nih.gov/ (2023)
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Lu, R., et al. Development of a novel reverse transcription loop-mediated isothermal amplification method for rapid detection of SARS-CoV-2. Virologica Sinica. 35 (3), 344-347 (2020).
- Najafov, A., Hoxhaj, G. . PCR Guru. , (2017).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. Biotechniques. 69 (3), 178-185 (2020).
- Ramírez-Chavarría, R. G., et al. Automatic analysis of isothermal amplification via impedance time-constant-domain spectroscopy: A SARS-CoV-2 case study. Chemosensors. 11 (4), 230 (2023).
- Haque, M. F. U., et al. A novel RdRp-based colorimetric RT-LAMP assay for rapid and sensitive detection of SARS-CoV-2 in clinical and sewage samples from Pakistan. Virus Research. 302, 198484 (2021).
- Donia, A., et al. Integration of RT-LAMP and microfluidic technology for detection of SARS-CoV-2 in wastewater as an advanced point-of-care platform. Food and Environmental Virology. 14, 364-373 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon