Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
BrainBeats en tant que plugin EEGLAB open-source pour analyser conjointement les signaux EEG et cardiovasculaires
Dans cet article
Résumé
La boîte à outils BrainBeats est un plugin EEGLAB open-source conçu pour analyser conjointement les signaux EEG et cardiovasculaires (ECG/PPG). Il comprend l’évaluation des potentiels évoqués par les battements cardiaques (HEP), l’analyse basée sur les caractéristiques et l’extraction d’artefacts cardiaques à partir de signaux EEG. Le protocole aidera à étudier l’interaction cerveau-cœur à travers deux lentilles (HEP et fonctionnalités), améliorant ainsi la reproductibilité et l’accessibilité.
Résumé
L’interaction entre le cerveau et le système cardiovasculaire suscite de plus en plus d’attention en raison de son potentiel à faire progresser notre compréhension de la physiologie humaine et à améliorer les résultats en matière de santé. Cependant, l’analyse multimodale de ces signaux est difficile en raison de l’absence de directives, d’outils statistiques et de traitement du signal standardisés, d’interfaces utilisateur graphiques (GUI) et d’automatisation pour le traitement de grands ensembles de données ou l’augmentation de la reproductibilité. Un autre vide existe dans les méthodes standardisées d’extraction des caractéristiques de l’EEG et de la variabilité de la fréquence cardiaque (HRV), ce qui compromet les diagnostics cliniques ou la robustesse des modèles d’apprentissage automatique (ML). En réponse à ces limitations, nous introduisons la boîte à outils BrainBeats. Mis en œuvre en tant que plugin EEGLAB open-source, BrainBeats intègre trois protocoles principaux : 1) Heartbeat-evoked potentials (HEP) et oscillations (HEO) pour évaluer l’interaction cerveau-cœur verrouillée dans le temps avec une précision de la milliseconde ; 2) l’extraction de caractéristiques EEG et HRV pour examiner les associations/différences entre diverses mesures cérébrales et cardiaques ou pour construire des modèles ML robustes basés sur les caractéristiques ; 3) Extraction automatisée d’artefacts cardiaques à partir de signaux EEG pour éliminer toute contamination cardiovasculaire potentielle lors de l’analyse EEG. Nous fournissons un tutoriel étape par étape pour appliquer ces trois méthodes à un ensemble de données open source contenant simultanément des signaux EEG, ECG et PPG à 64 canaux. Les utilisateurs peuvent facilement affiner les paramètres pour répondre à leurs besoins de recherche uniques à l’aide de l’interface utilisateur graphique (GUI) ou de la ligne de commande. BrainBeats devrait rendre la recherche sur l’interaction cerveau-cœur plus accessible et reproductible.
Introduction
Pendant longtemps, l’approche réductionniste a dominé la recherche scientifique en physiologie et cognition humaines. Cette approche consistait à disséquer des processus corporels et mentaux complexes en composants plus petits et plus gérables, ce qui permettait aux chercheurs de se concentrer sur des systèmes individuels de manière isolée. Cette stratégie est née en raison des défis liés à l’étude de la nature complexe et interconnectée du corps et de l’esprit humains1. Le réductionnisme a joué un rôle déterminant dans la compréhension isolée des sous-systèmes individuels, comme l’élucidation du rôle des canaux ioniques et des potentiels d’action pour la communication neuronale2 ou cardiaque3 . Cependant, une lacune importante subsiste dans notre compréhension de la façon dont ces systèmes isolés interagissent à une échelle spatiale et temporelle plus large. Le cadre multimodal (intégratif ou écologique) considère le corps humain comme un système multidimensionnel complexe, où l’esprit n’est pas considéré comme un produit du cerveau mais comme une activité de l’être vivant, une activité qui intègre le cerveau dans les fonctions quotidiennes du corps humain4. Les approches multimodales et réductionnistes ne sont pas exclusives, tout comme nous ne pouvons pas étudier un neurone sans le cerveau entier ou le cerveau entier sans comprendre les propriétés individuelles des neurones. Ensemble, ils ouvrent la voie à une compréhension plus complète et synergique de la santé humaine, de la pathologie, de la cognition, de la psychologie et de la conscience. La présente méthode vise à faciliter l’investigation multimodale de l’interaction entre le cerveau et le cœur en fournissant une analyse conjointe de l’électroencéphalographie (EEG) et des signaux cardiovasculaires, à savoir l’électrocardiographie (ECG) et la photopléthysmographie (PPG). Cette boîte à outils, implémentée sous la forme d’un plugin EEGLAB dans MATLAB, répond aux limites méthodologiques existantes et est rendue open source pour faciliter l’accessibilité et la reproductibilité dans le domaine scientifique. Il met en œuvre les dernières directives et recommandations dans sa conception et ses paramètres par défaut pour encourager les utilisateurs à suivre les meilleures pratiques connues. La boîte à outils proposée devrait être une ressource précieuse pour les chercheurs et les cliniciens intéressés à 1) étudier les potentiels évoqués par les battements cardiaques, 2) extraire des caractéristiques des signaux EEG et ECG/PPG, ou 3) supprimer les artefacts cardiaques des signaux EEG.
Recherche cœur-cerveau
La relation entre le cœur et le cerveau a été historiquement étudiée via des méthodes de neuroimagerie telles que l’imagerie par résonance magnétique fonctionnelle (IRMf) et la tomographie par émission de positrons (TEP). À l’aide de ces outils, les chercheurs ont mis en évidence certaines régions du cerveau associées au contrôle cardiovasculaire (par exemple, la manipulation de la fréquence cardiaque et de la pression artérielle5), montré l’influence de la fréquence cardiaque sur le signal BOLD6, ou identifié des voies cerveau-corps potentielles contribuant aux maladies coronariennes (c’est-à-dire la pression artérielle évoquée par le stress7). Bien que ces études aient considérablement fait progresser notre compréhension de l’interaction complexe entre le système nerveux central (SNC) et la fonction cardiovasculaire, ces techniques de neuroimagerie sont coûteuses, ont une disponibilité limitée et sont confinées à des laboratoires contrôlés, ce qui limite leur praticité pour des applications réelles et à grande échelle.
En revanche, l’EEG et l’ECG/PPG sont des outils plus abordables et portables qui offrent la possibilité d’étudier les interactions cerveau-cœur dans des contextes et des populations plus diversifiés ou sur de plus longues périodes, offrant ainsi de nouvelles opportunités. L’ECG mesure les signaux électriques générés par chaque battement cardiaque lorsque le cœur se contracte et se détend via des électrodes placées sur la peau (généralement sur la poitrine ou les bras)8. La PPG mesure les changements de volume sanguin dans les tissus microvasculaires (c’est-à-dire le débit sanguin et le pouls) à l’aide d’une source lumineuse (par exemple, une LED) et d’un photodétecteur (généralement placé sur le bout d’un doigt, un poignet ou un front), en s’appuyant sur la façon dont le sang absorbe plus de lumière que les tissus environnants9. Les deux méthodes fournissent des informations précieuses sur la fonction cardiovasculaire, mais servent des objectifs différents et offrent des types de données distincts. Comme l’ECG, l’EEG enregistre les champs électriques générés par l’activité synchronisée de milliers de neurones corticaux qui se propagent à travers la matrice extracellulaire, les tissus, le crâne et le cuir chevelu jusqu’à ce qu’ils atteignent les électrodes placées à la surface du cuir chevelu10. En tant que telle, l’utilisation de l’EEG et de l’ECG/PPG est très prometteuse pour faire progresser notre compréhension des processus physiologiques, cognitifs et émotionnels sous-jacents aux interactions cerveau-cœur et de leurs implications pour la santé et le bien-être humains. Par conséquent, la capture de l’interaction cœur-cerveau à partir d’EEG, de signaux ECG/PPG avec la boîte à outils BrainBeats peut être particulièrement utile pour les domaines scientifiques suivants : diagnostic et prévisions cliniques, apprentissage automatique (ML) du big data, autosurveillance du monde réel11 et imagerie cérébrale/corporelle mobile (MoBI)12,13.
Deux approches pour l’analyse conjointe des signaux EEG et ECG
Il existe deux approches principales pour étudier les interactions entre l’EEG et les signaux cardiovasculaires :
Les potentiels évoqués par les battements de cœur (HEP) dans le domaine temporel : les potentiels liés aux événements (ERP), et les oscillations évoquées par les battements de cœur (HEO) dans le domaine temps-fréquence : perturbations spectrales liées aux événements (ERSP) et cohérence inter-essais (ITC). Cette approche examine comment le cerveau traite chaque battement de cœur. Avec une précision de l’ordre de la milliseconde (ms), cette méthode exige que les deux séries chronologiques soient parfaitement synchronisées et que les battements cardiaques soient marqués dans les signaux EEG. Cette approche a suscité de l’intérêt ces dernières années 14,15,16,17,18,19.
Approche basée sur les caractéristiques : cette approche extrait les caractéristiques de l’EEG et de la variabilité de la fréquence cardiaque (VFC) des signaux continus et examine les associations entre eux. Cela a été fait indépendamment pour l’EEG (souvent appelé EEG quantitatif ou qEEG20), l’ECG21, 22, 23 et le PPG24, 25, 26. Cette approche présente des applications prometteuses en capturant à la fois les variables liées à l’état et aux caractères. Notez que, pour les signaux EEG et cardiovasculaires, plus l’enregistrement est long, plus la variable de caractère 27,28,29 est dominante. Ainsi, les applications dépendent des paramètres d’enregistrement. Les analyses basées sur les caractéristiques suscitent un intérêt croissant, fournissant de nouvelles mesures quantitatives pour prévoir le développement des troubles mentaux et neurologiques, la réponse au traitement ou la rechute 30,31,32,33,34,35. Cette approche est particulièrement convaincante avec des ensembles de données volumineux et réels (p. ex., clinique, surveillance à distance), qui peuvent être obtenus plus facilement grâce aux récentes innovations en matière de neurotechnologie portable11. Une application moins explorée est l’identification d’associations entre des caractéristiques spécifiques du cerveau et du cœur, mettant en évidence la dynamique sous-jacente potentielle du système nerveux central. La variabilité de la fréquence cardiaque (VFC) peut être calculée à partir des signaux ECG et PPG. Il fournit des informations sur le système nerveux autonome (SNA) en mesurant les variations des intervalles de temps entre les battements cardiaques (c’est-à-dire les intervalles normaux à normaux)27. L’augmentation de l’activité sympathique (SNS) (par exemple, pendant le stress ou l’exercice) réduit généralement la VFC, tandis que l’activité parasympathique (PNS) (par exemple, pendant la relaxation) l’augmente. Un rythme respiratoire plus lent augmente généralement la VFC en raison de l’augmentation de l’activité du SNP, en particulier pour les enregistrements courts (<10 min)27. Des scores de VFC plus élevés suggèrent généralement un VNA plus résilient et adaptable, tandis qu’une VFC plus faible peut indiquer du stress, de la fatigue ou des problèmes de santé sous-jacents. Les enregistrements longs de la VFC (c’est-à-dire au moins 24 heures) fournissent un pronostic prédictif pour divers problèmes de santé, notamment les maladies cardiovasculaires, le stress, l’anxiété et certains troubles neurologiques27. Des mesures telles que la pression artérielle, la fréquence cardiaque ou le taux de cholestérol donnent des informations sur l’état du système cardiovasculaire. En revanche, la VFC ajoute un aspect dynamique, montrant comment le cœur réagit au stress et s’en remet .
Les avantages de BrainBeats par rapport aux méthodes existantes
Bien qu’il existe des outils, comme nous le verrons ci-dessous, pour traiter les signaux cardiovasculaires et EEG indépendamment les uns des autres, ils ne peuvent pas être analysés conjointement. De plus, la plupart des moyens disponibles pour traiter les signaux cardiovasculaires impliquent des licences coûteuses, ne permettent pas de traitement automatisé (particulièrement avantageux pour les grands ensembles de données), ont des algorithmes propriétaires qui empêchent la transparence et la reproductibilité, ou nécessitent des compétences de programmation avancées en ne fournissant pas d’interface utilisateur graphique (GUI)36. À notre connaissance, quatre boîtes à outils MATLAB open-source prennent en charge l’analyse HEP/HEO avec une interface graphique : la boîte à outils ecg-kit37, le pipeline BeMoBIL38, le plugin HEPLAB EEGLAB39 et la boîte à outils CARE-rCortex40. Bien que HEPLAB, BeMoBIL et ecg-kit facilitent l’analyse HEP en détectant les battements cardiaques et en les marquant dans les signaux EEG, ils ne fournissent pas d’analyse statistique ou sont limités au domaine temporel (c’est-à-dire HEP). Le plug-in CARE-rCortex a résolu ces problèmes en prenant en charge l’ECG et les signaux respiratoires, l’analyse du domaine temps-fréquence, les statistiques et les méthodes avancées de normalisation et de correction de base adaptées à l’analyse HEP/HEO. Cependant, il utilise la méthode Bonferroni pour la correction statistique de l’erreur de type 1 (c’est-à-dire les faux positifs), qui est trop conservatrice et non physiologiquement solide pour les applications EEG, ce qui entraîne une augmentation des erreurs de type II (c’est-à-dire les faux négatifs)41. De plus, la boîte à outils n’offre pas d’accès en ligne de commande pour l’automatisation. Enfin, des études récentes déconseillent les méthodes de correction de base 42,43,44, car elles réduisent le rapport signal/bruit (SNR) et sont statistiquement inutiles et indésirables.
Pour remédier à ces limitations, nous vous présentons la boîte à outils BrainBeats, actuellement implémentée en tant que plugin EEGLAB open-source dans l’environnement MATLAB. Il intègre les avantages suivants par rapport aux méthodes précédentes :
1) Une interface graphique facile à utiliser et des capacités en ligne de commande (pour les programmeurs visant à effectuer un traitement automatisé). 2) Des algorithmes, des paramètres et des lignes directrices validés pour le traitement des signaux cardiovasculaires, tels que la détection des pics R, l’interpolation des artefacts RR et le calcul des mesures de la VFC (p. ex., implanter des lignes directrices pour le fenêtrage, le rééchantillonnage, la normalisation, etc.27,45,46). Ceci est important car Vest et al. ont démontré comment des différences modestes dans ces étapes de traitement peuvent conduire à des résultats divergents, contribuant au manque de reproductibilité et d’applicabilité clinique des mesures de la VRH46. 3) Algorithmes validés, paramètres par défaut et directives pour le traitement des signaux EEG, y compris le filtrage et le fenêtrage44,47, le reréférencement48,49, la suppression des canaux anormaux et des artefacts 50,51,52, la décomposition ICA optimisée et la classification des composants indépendants 53,54,55,56. Les utilisateurs peuvent affiner tous les paramètres de prétraitement ou même prétraiter leurs données EEG avec la méthode de leur choix avant d’utiliser la boîte à outils en fonction de leurs besoins (par exemple, avec EEGLAB clean_rawdata plugin50,52, le pipeline BeMoBIL38, le pipeline PREP57, etc.). 4) Les potentiels évoqués par les battements cardiaques (HEP, c’est-à-dire le domaine temporel) et les oscillations (HEO ; perturbations spectrales liées aux événements avec des méthodes d’ondelettes ou de FFT, et la cohérence inter-essais sont disponibles via le logiciel standard EEGLAB) à partir des signaux ECG. Des statistiques paramétriques et non paramétriques avec corrections pour les erreurs de type 1 sont disponibles via le logiciel standard d’EEGLAB. Les statistiques non paramétriques comprennent les statistiques de permutation et les corrections spatio-temporelles pour les comparaisons multiples (par exemple, le clustering spatio-temporel ou l’amélioration du cluster sans seuil)58,59. Les utilisateurs peuvent utiliser le plug-in LIMO-EEG pour mettre en œuvre une modélisation linéaire hiérarchique, qui tient bien compte de la variance à l’intérieur et entre les sujets et met en œuvre une approche univariée de masse sans hypothèse avec un contrôle robuste des erreurs de type I et II60,61. Les analyses statistiques des données HEP/HEO peuvent être effectuées dans les domaines des canaux et des composants indépendants. 5) Analyse HEP/HEO et HRV à partir de signaux PPG (pour la première fois pour HEP/HEO). 6) Prend en charge l’extraction conjointe des caractéristiques EEG et HRV pour la première fois. 7) La boîte à outils fournit diverses visualisations de données pour inspecter les signaux à diverses étapes de traitement nécessaires et les sorties au niveau du sujet.
| Méthode | Détecter les pics R de l’ECG | Détection des ondes R de PPG | HEP/HEO | Caractéristiques de l’EEG et de la VFC | Supprimer les artefacts cardiaques de l’EEG | Interface graphique | Ligne de commande |
| Kit ECG | X | X | X | X | |||
| BeMoBIL | X | X | X | ||||
| HEPLAB | X | X | X | X | |||
| CARE-rCortex | X | X | X | X | |||
| BrainBeats | X | X | X | X | X | X | X |
TABLEAU 1 : Nouveautés apportées par BrainBeats par rapport à des méthodes similaires préexistantes.
Informations pour aider les lecteurs à décider si la méthode leur convient
Cette boîte à outils convient à tout chercheur ou clinicien disposant de données EEG et ECG/PPG. Le plugin ne prend pas encore en charge l’importation de signaux EEG et ECG/PPG à partir de fichiers séparés (bien que cette fonctionnalité soit bientôt disponible). La boîte à outils convient à toute personne souhaitant effectuer des analyses HEP/HEO, extraire des caractéristiques EEG et/ou HRV avec des méthodes standardisées, ou simplement supprimer des artefacts cardiaques des signaux EEG. Voir la figure 1 pour un schéma fonctionnel résumant le flux global et les méthodes de BrainBeats.
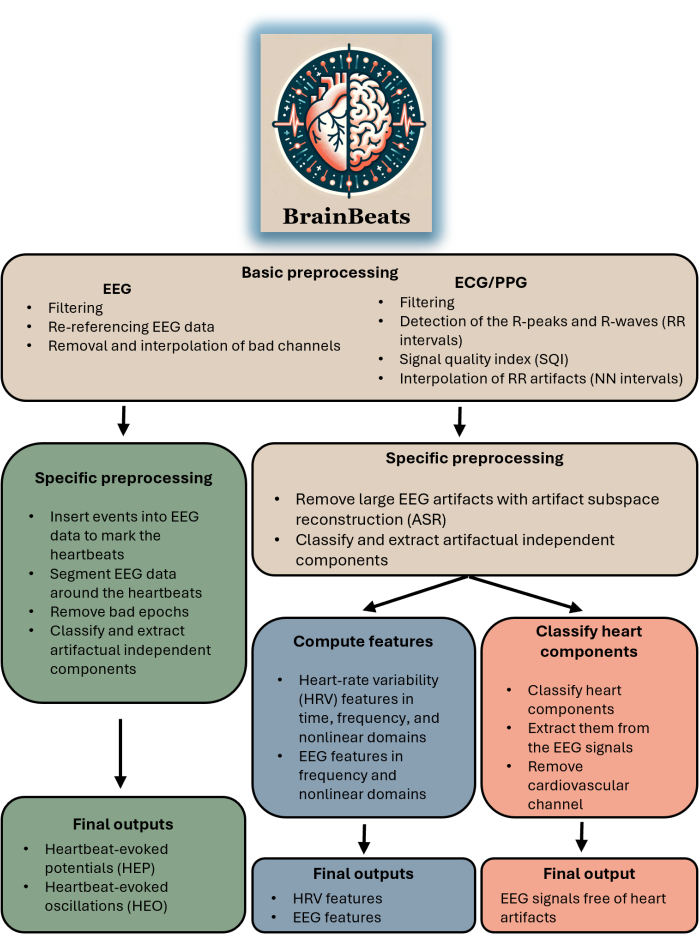
GRAPHIQUE 1. Schéma fonctionnel résumant l’architecture globale et le flux de BrainBeats. Les opérations communes aux trois méthodes sont brunes. Les opérations spécifiques aux potentiels évoqués par battement de cœur (HEP) et aux oscillations (HEO) sont vertes. Les opérations spécifiques à l’extraction des caractéristiques EEG et HRV sont bleues. Les opérations spécifiques à l’élimination des artefacts cardiaques des signaux EEG sont rouges. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
Le consentement éclairé de chaque participant a été obtenu, et le comité d’éthique de l’Université fédérale de l’Oural a approuvé le protocole expérimental.
1. Exigences de BrainBeats
- Installez MATLAB et EEGLAB sur l’ordinateur. EEGLAB peut être téléchargé à l’https://github.com/sccn/eeglab et décompressé (ou cloné pour les utilisateurs de Git) n’importe où sur l’ordinateur. Consultez la page GitHub pour plus de détails sur l’installation.
- Ajoutez le chemin d’accès au dossier EEGLAB dans le panneau d’accueil de MATLAB en cliquant sur le bouton Set Path . Sélectionnez le dossier eeglab avec le fichier décompressé et cliquez sur Enregistrer > Fermer.
- Lancez EEGLAB en tapant eeglab dans la fenêtre de commande de MATLAB.
- Installez le plugin BrainBeats en cliquant sur Fichier > Gérer les extensions EEGLAB. Tapez brainbeats dans la barre de recherche, sélectionnez le plugin BrainBeats dans la liste et cliquez sur Installer/Mettre à jour.
- Chargez l’exemple de jeu de données dans EEGLAB. Cliquez sur Fichier > Charger les données existantes. Naviguez dans le dossier EEGLAB, allez dans le dossier plugins, allez dans le dossier BrainBeats et ouvrez le dossier sample_data. Sélectionnez le fichier dataset.set.
REMARQUE : Cet ensemble de données correspond au sous-032 (état de repos avec les yeux ouverts) d’un ensemble de données multi-sujets open source62,63. Il a été choisi parce qu’il contient simultanément l’EEG (64 canaux), l’ECG (un canal) et le PPG (un canal), à une fréquence d’échantillonnage de 1000 Hz. Les signaux ECG et PPG ont été collectés à l’aide des entrées auxiliaires du système actiCHamp. Les données EEG ont été enregistrées avec des électrodes actives placées selon le système 10-20, avec FCz comme référence en ligne et Fpz comme électrode de terre, et avec l’impédance maintenue en dessous de 25 kOm. L’ECG a été enregistré à l’aide d’une électrode active placée sur le poignet droit, de l’électrode de référence sur le poignet gauche et du sol sur l’avant-bras intérieur gauche à 3 cm distalement du coude. PPG a été enregistré à partir de l’index gauche. Les données EEG, ECG et PPG sont synchronisées dans le temps puisqu’elles ont été enregistrées simultanément avec le même amplificateur. Voir les références62,63 pour plus de détails. - Cochez la case Enregistrer les sorties pour enregistrer tout ce qui se trouve dans le fichier .set correspondant au même emplacement que le fichier d’origine qui a été chargé dans EEGLAB.
REMARQUE : Pour ce tutoriel, nous avons fusionné les données EEG, ECG et PPG en un seul ensemble de données EEGLAB, chargé les coordonnées de l’électrode de la méthode des éléments de limite 3D (BEM) et sous-échantillonné le signal à 250 Hz pour réduire le poids du fichier (pour le téléchargement par l’utilisateur) et accélérer le temps de calcul. Étant donné que cet ensemble de données n’avait pas de canal EEG anormal, nous avons modifié artificiellement le canal TP9 pour illustrer l’algorithme de détection et de suppression des mauvais canaux de BrainBeats. De même, nous simulons artificiellement un grand artefact EEG au début du fichier et un artefact musculaire à haute fréquence dans les canaux temporaux de 3 à 6 s pour illustrer la fonction de suppression d’artefact de BrainBeats.
2. Potentiels évoqués par le battement de cœur (HEP) et oscillations (HEO)
- Ouvrez la première interface utilisateur générale (GUI) de BrainBeats pour sélectionner les principaux paramètres. Dans EEGLAB, cliquez sur Outils > BrainBeats > 1er niveau (niveau sujet). Sélectionnez Potentiels évoqués par battement de cœur HEP comme analyse à exécuter, ECG comme type de données cardiaques, et cliquez sur le bouton pour afficher la liste des canaux permettant de sélectionner le canal ECG étiqueté ECG (ou saisissez-le directement dans la zone de texte). Conservez les options Visualiser les sorties (recommandé) et Enregistrer les sorties sélectionnées, puis cliquez sur OK.
- Une deuxième fenêtre d’interface graphique s’affiche en fonction des choix précédents (c’est-à-dire l’analyse HEP avec les données ECG). Sélectionnez les paramètres de prétraitement pour les signaux EEG et ECG. Désactivez le prétraitement en décochant les cases Prétraiter l’ECG et Prétraiter l’EEG pour voir si les données ont déjà été prétraitées avant de lancer BrainBeats. Modifiez le bruit des lignes électriques à 50 Hz dans la section Prétraitement de l’EEG , car ces données EEG ont été enregistrées en Russie. Cliquez sur OK pour lancer.
- Un message d’avertissement s’affiche, demandant la confirmation de la suppression du canal PPG qui a été détecté. En effet, la boîte à outils n’est pas conçue pour analyser simultanément l’ECG et le PPG (ou d’autres canaux auxiliaires) et son maintien dans l’ensemble de données entraînera de graves erreurs (par exemple, suppression d’artefacts, mauvaise décomposition de l’ICA, etc.). Cliquez sur Oui. BrainBeats commence à effectuer des vérifications, à définir certains paramètres par défaut et sépare l’ECG des données EEG pour prétraiter le signal ECG et calculer les intervalles RR.
- Les séries chronologiques de l’ECG et du RR sont prétraitées à l’aide d’algorithmes validés de la boîte à outils Physionet Cardiovascular Signal46,64 adaptés au formatage des données de BrainBeats, à l’augmentation de la clarté, au réglage des paramètres et au temps de calcul (voir les références pour la validation des algorithmes). Le plugin émet les intervalles RR, les horodatages, le signal ECG filtré, les indices R-peaks et la fréquence cardiaque (HR). Réglez ces paramètres via l’interface graphique ou la ligne de commande.
REMARQUE : Le signal ECG est filtré par bande passante à l’aide d’un filtre personnalisé et validé (1-30 Hz) et scanne le signal pour identifier le complexe QRS et les pics R à l’aide de la méthode Pan-Tompkins (P&T)65, en mettant en œuvre certaines opérations de traitement du signal, notamment la différenciation, la quadrature, l’intégration et le lissage pour de meilleures performances. Le seuil d’énergie P&T est estimé sur la base de la fréquence d’échantillonnage et des valeurs ECG lissées afin d’éviter les perturbations dues à de grosses bosses. Si la variabilité de l’intervalle RR dépasse 1,5 fois la médiane, il recherche les pics manqués. Le signe moyen du pic R est calculé sur des segments de 30 s, et les points de crête sont affinés par un contrôle de la période réfractaire, gérant les conditions plates et assurant une détection cohérente. - Ensuite, BrainBeats identifie les intervalles RR anormaux ou les pics à l’intérieur des intervalles RR à l’aide d’une recherche avant-arrière et de seuils physiologiques. L’indice de qualité du signal (IQS) est calculéà 46 ; vérifiez cette option si le système affiche des avertissements où plus de 20 % de la série chronologique RR contient des artefacts RR (en dehors des limites physiologiques ou avec un SQI inférieur à 0,9). Un graphique affiche le signal ECG filtré, les pics R identifiés, les intervalles NN et les artefacts interpolés (voir Figure 2).
REMARQUE : Les artefacts RR sont interpolés par défaut à l’aide de la méthode cubique par morceaux préservant la forme pour obtenir les intervalles normaux à normaux (NN), mais les supprimer (non recommandé) ou utiliser une autre méthode d’interpolation (linéaire, cubique, voisin le plus proche, voisin précédent/suivant, spline, convolution cubique ou cubique Akima modifié) si nécessaire. Lorsque plusieurs canaux ECG sont présents, les intervalles RR sont estimés pour chacun d’entre eux, et le canal avec le moins d’artefacts RR est sélectionné pour les étapes suivantes. - Faites défiler les fenêtres zoomées de 30 s des pics R pour une inspection plus approfondie en appuyant sur les flèches droite/gauche. Si les données contiennent plusieurs canaux ECG/PPG, utilisez le canal avec le meilleur indice de qualité du signal pour les intervalles RR. BrainBeats ne prend pas en charge les signaux ECG et PPG simultanément pour le moment. À des fins de didacticiel, nous avons choisi un exemple de jeu de données qui contient les deux types de données.
- Une fois le signal ECG terminé, la bande passante BrainBeats filtre les données EEG à 1-40 Hz à l’aide d’un filtre FIR causal non linéaire à phase minimale par défaut pour réduire l’activité de frottis entre les périodes pré et post-heartbeat, préserver la causalité et éviter les retards de groupe indésirables44. Ceci est particulièrement important pour les utilisateurs qui examinent la période précédant le battement de cœur. Si le filtre passe-bas est réglé sur une valeur supérieure à la fréquence de la ligne d’alimentation (par exemple, passe-bas de 80 Hz avec une fréquence de ligne d’alimentation à 50 Hz), utilisez un filtre coupe-bande pointu pour supprimer l’artefact de bruit de ligne. Les données EEG sont ensuite référencées à l’infini à l’aide de l’algorithme REST (le mieux adapté à l’analyse HEP49), à moins que moins de 30 canaux ne soient détectés (auquel cas ils ne peuvent pas être référencés de manière fiable et un avertissement est généré pour informer les utilisateurs).
- BrainBeats détecte, supprime et interpole ensuite les canaux EEG anormaux (Figure 3). Vérifiez que les paramètres par défaut sont des lignes plates supérieures à 5 s (algorithme clean_flatlines), un écart-type maximal du bruit haute fréquence de 10, une longueur de fenêtre de 5 s (pour mieux capturer les artefacts de fréquence lente52), une corrélation minimale entre les canaux voisins de 0,65 et une portion maximale tolérée de 33 % (algorithme clean_channels).
REMARQUE : Le nombre d’échantillons RANSAC est fixé à 500 par défaut pour augmenter la convergence et la reproductibilité du rejet de mauvais canal (bien que cela augmente le temps de calcul). - Ensuite, insérez des pics R comme marqueurs d’événement dans les données EEG pour marquer chaque battement de cœur et segmentez les données autour de ces marqueurs sans suppression de la ligne de base (conformément aux directives43,66 ; Figure 4). Étant donné que les intervalles NN ont des longueurs différentes et que l’EEG doit être segmenté à une longueur constante, estimez la limite minimale de la taille de l’époque après les événements de pic R en utilisant le 5e centile des données de l’intervalle entre les battements (IBI) (c’est-à-dire la valeur en dessous de laquelle se situent 5 % des IBI les plus courts, affichée par une ligne rouge pointillée sur un histogramme ; voir la figure 5).
REMARQUE : Cette valeur du 5e centile est un bon compromis pour préserver autant d’époques que possible tout en veillant à ce qu’elles ne soient pas trop courtes, car la période d’intérêt pour l’analyse HEP/HEO est de 200 à 600 ms après le battement de cœur49,67. - Segmentez les données EEG de -300 ms avant les pics R à la valeur du 5ecentile après le pic R, avec le pic R à l’instant 0. Rejeter les époques inférieures à 550 ms ou contenant plus d’un pic R (ce qui biaiserait l’ERP/ERSP), conformément aux directives49,67. Les époques contenant de gros artefacts EEG sont détectées à l’aide de métriques RMS (Middle-Mean Square) et de rapport signal/bruit (SNR) et de la fonction isoutlier de MATLAB (Figure 6). Les époques artificielles sont supprimées.
- Effectuez une séparation de source à l’aveugle à l’aide de l’algorithme Infomax par défaut, en tenant compte du classement des données pour les meilleures performances53. Si vous le souhaitez, choisissez l’algorithme d’analyse en composantes indépendantes préconditionnées (PICARD) pour un calcul rapide54,54 en choisissant l’option Picard (rapide) ou l’algorithme Infomax modifié en choisissant l’option Infomax modifié (long mais reproductible) pour la méthode ICA (bien que cela implique des temps de calcul beaucoup plus longs). Utilisez le plugin ICLabel56 pour classer automatiquement les CI afin d’extraire les artefacts non cérébraux (les composants oculaires sont supprimés avec un niveau de confiance de 90 %, tandis que les bruits musculaires, linéaires et de canal sont supprimés avec un niveau de confiance de 99 % ; Figure 7).
- Conservez l’option Visualiser les sorties sélectionnée dans la première fenêtre de l’interface graphique pour visualiser la série chronologique EEG finale, le HEP moyen (Figure 8 en haut), le HEP pour chaque battement de cœur (Figure 8 en bas) et les oscillations évoquées par le battement de cœur (HEO ; Figure 9). Examiner les HEO en termes de perturbation spectrale liée à l’événement (ERSP, c’est-à-dire les changements de puissance EEG à travers les battements cardiaques ; Figure 9 en haut) et la cohérence de phase inter-essais (ITC, c’est-à-dire la cohérence de l’angle de phase entre les battements cardiaques ; Figure 9 en bas).
REMARQUE : L’ERSP est calculé à l’aide d’une ondelette à 3 cycles par défaut (avec une fenêtre conique de Hanning appliquée, rapport de tampon de 2) et avec le nombre de cycles dans les ondelettes utilisées pour les fréquences plus élevées augmentant lentement jusqu’à 20 % du nombre de cycles dans la fenêtre FFT équivalente à sa fréquence la plus élevée (1 moins 0,8). Cela contrôle les formes des fenêtres individuelles mesurées par la fonction et leurs formes dans les volets temps/fréquence résultants. Une ligne de base arbitraire est supprimée à des fins d’illustration, et ERSP est calculé pour les fréquences 7-25 Hz afin de capturer l’effet HEO typique décrit dans la littérature, à savoir 300-450 ms après le battement de cœur dans la bande alpha (8-13 Hz) sur les électrodes frontocentrales17,67. Les fréquences inférieures ne peuvent pas être estimées en raison de la courte taille d’époque définie par les intervalles de battement. Des statistiques non paramétriques (permutations) sont appliquées pour visualiser l’HEO pour une valeur p de 0,05, corrigée du taux de fausses découvertes (FDR, c’est-à-dire erreur de type 1 ou erreur familiale). Ces graphiques sont générés à des fins de tutoriel ou d’analyse d’essais uniques. - Des tracés de prétraitement sont générés pour visualiser les différentes étapes. Pour le désactiver, décochez la case Visualiser les prétraitements. Les données EEG finales (nettoyées et segmentées autour des pics R) n’incluent pas les données ECG car elles biaiseraient l’analyse ERP/ERSP. Pour conserver le canal du cœur dans la sortie finale, cochez la case Conserver le canal du cœur.
REMARQUE : Faites une pause ici si nécessaire avant de traiter le fichier suivant (condition ou participant suivant). - BrainBeats prend en charge la fonction d’historique d’EEGLAB. À la fin de toutes les opérations, tapez eegh dans la fenêtre de commande de MATLAB pour afficher la ligne de commande afin de répéter toutes les étapes ci-dessus via une seule ligne de commande, avec les paramètres sélectionnés manuellement dans l’interface graphique, ce qui permet une automatisation facile. Trouvez les sorties de prétraitement (par exemple, l’indice de qualité du signal de la série chronologique cardiovasculaire, les intervalles NN, les canaux EEG supprimés, les segments et les composants, etc.) dans la structure EEGLAB : EEG.brainbeats.preprocessings. Tous les paramètres sont également exportés dans EEG.brainbeats.parameters.
- Pour les utilisateurs avancés, effectuez toutes les étapes ci-dessus avec les paramètres par défaut avec les lignes de commande suivantes (voir le script du tutoriel dans le référentiel BrainBeats pour plus d’options) :
eeglab ; fermer; % Lancer EEGLAB sans l’interface graphique
main_path = fileparts(which('eegplugin_BrainBeats.m')) ; CD(main_path) ;
EEG = pop_loadset('filename','dataset.set',
'filepath',fullfile(main_path, 'sample_data')) ; %Load l’exemple de jeu de données
EEG = brainbeats_process(EEG,'analyse',
'hep', 'heart_signal', 'ECG', 'heart_channels,
{'ECG'},'clean_eeg',true) ; % Lancez BrainBeats 1er niveau pour traiter le fichier pour l’analyse HEP avec les paramètres par défaut - Les étapes ci-dessus ont effectué HEP/HEO à partir du signal ECG. Procédez comme suit pour le signal PPG.
- Dans les étapes suivantes, effectuez les mêmes opérations mais à l’aide d’un signal PPG. Chargez à nouveau le même ensemble de données (voir étape 1.5) puisque les opérations précédentes l’ont écrasé, et ouvrez à nouveau la première interface graphique de BrainBeats pour sélectionner les paramètres principaux. Cliquez sur Outils > BrainBeats > 1er niveau (niveau sujet). Sélectionnez Heartbeat-evoked potentials (HEP) comme analyse à exécuter, PPG comme type de données cardiaques, et cliquez sur le bouton pour afficher la liste des canaux permettant de sélectionner le canal PPG. Conservez les options Visualiser les sorties (recommandé) et Enregistrer les sorties sélectionnées, puis cliquez sur OK.
- La deuxième fenêtre de l’interface graphique s’affiche d’une manière similaire à l’étape 2.2 . La seule différence est que nous pouvons maintenant affiner le paramètre pour traiter le signal PPG au lieu du signal ECG. Cliquez sur OK pour exécuter avec les paramètres par défaut.
- Un message d’avertissement s’affiche, demandant la confirmation de la suppression du canal ECG supplémentaire qui a été détecté. Encore une fois, c’est normal. Cliquez sur Oui. Par défaut, la boîte à outils prétraite le signal PPG, détecte les ondes d’impulsion pour obtenir les intervalles RR, identifie les artefacts RR, le cas échéant, et les interpole (Figure 10). Étapes 2.7. à 2.12. sont effectués, et les mêmes tracés et sorties sont générés, mais sur la base des ondes R détectées à partir du signal PPG (voir Figure 11 et Figure 12).
REMARQUE : Les ondes R sont détectées en utilisant la pente du signal dans une fenêtre spécifiée. Les impulsions potentielles sont ensuite signalées lorsque la pente dépasse un seuil dynamique, qui est ajusté en fonction de l’historique de détection et des caractéristiques du signal. L’algorithme effectue ensuite une recherche dans une période de fermeture de l’œil pour déterminer la pente maximale, puis le début de l’onde d’impulsion est déterminé par seuillage. Les pics de l’onde R sont identifiés comme les vallées proches du début, et leurs emplacements sont enregistrés. L’algorithme parcourt l’ensemble du signal, ajustant en permanence les seuils de détection et identifiant les pics d’onde R, qui sont ensuite utilisés pour calculer les intervalles RR. - Pour les utilisateurs avancés, effectuez toutes les étapes ci-dessus avec les paramètres par défaut avec les lignes de commande suivantes (voir le script du tutoriel dans le référentiel BrainBeats pour plus d’options) :
eeglab ; fermer; % Lancer EEGLAB sans l’interface graphique
main_path = fileparts(which('eegplugin_
BrainBeats.m')) ; CD(main_path) ;
EEG = pop_loadset('filename','dataset.set',
'filepath',fullfile(main_path, 'sample_data')) ; %Load l’exemple de jeu de données
EEG = brainbeats_process(EEG,'analyse',
'hep', 'heart_signal', 'PPG', 'heart_channels,
{'PPG'},'clean_eeg',true) ; % Lancez BrainBeats 1er niveau pour traiter le fichier pour l’analyse HEP avec les paramètres par défaut
3. Extraction des caractéristiques EEG et HRV
- Chargez à nouveau le même ensemble de données (voir étape 1.5 ; Cliquez sur Fichier > Charger le jeu de données existant > Sélectionner le fichier dataset.set) car il a été écrasé par les opérations précédentes et ouvrez à nouveau l’interface graphique principale pour sélectionner les paramètres principaux (étape 2.1 ; Cliquez sur Outils > BrainBeats > 1er niveau). Sélectionnez Extraire les caractéristiques EEG et HRV pour le type d’analyse, ECG pour le type de signal cardiaque, puis sélectionnez ECG dans la liste des étiquettes d’électrodes. Cliquez sur OK.
- La deuxième fenêtre de l’interface graphique s’affiche comme à l’étape 2.2, mais avec des paramètres différents pour le prétraitement de l’EEG et l’extraction des caractéristiques HRV et EEG. Définissez ces paramètres comme décrit ci-dessous. Cliquez sur OK pour lancer avec les paramètres par défaut.
- Dans la section VRC, cliquez sur le bouton freq. options pour sélectionner la méthode de calcul de la puissance de la VFC (réglée par défaut sur le périodogramme de Lomb-Scargle normalisé), le chevauchement de la fenêtre (réglé par défaut sur 25%), et pour effectuer une normalisation de deuxième niveau (non réglée par défaut ; voir la note ci-dessous pour plus de détails).
- Dans la section des fonctionnalités EEG, cliquez sur le bouton freq. options pour affiner certains paramètres, tels que la plage de fréquence globale sur laquelle calculer la densité spectrale de puissance (PSD ; par défaut = 1-40 Hz), les unités (décibels, μV2/Hz, ou normalisées par la puissance totale), le type de fenêtre (par défaut = hamming), le chevauchement des fenêtres (par défaut = 50%), la longueur de la fenêtre (par défaut = 2 s), et les types de limites de fréquence pour chaque bande.
REMARQUE : La puissance de la VFC est calculée par défaut à l’aide du périodogramme normalisé de Lomb-Scargle, qui ne nécessite pas de rééchantillonnage (ce qui permet de mieux préserver l’information d’origine) et traite mieux les données échantillonnées de manière non uniforme, les données manquantes et le bruit (typique des intervalles NN)68. La version normalisée échelonne la puissance en fonction de la variance du signal, ce qui permet d’obtenir des résultats moins sensibles aux différents niveaux de bruit, plus axés sur l’intensité relative des composants périodiques et plus comparables entre différents enregistrements ou sujets. Parmi les autres méthodes disponibles, citons le parodogramme de Lomb-Scargle non normalisé, la méthode de Welch et la transformée de Fourier rapide (FFT). Un rééchantillonnage est automatiquement effectué pour les méthodes Welch et FFT afin de créer les séries chronologiques nécessaires échantillonnées régulièrement. Une normalisation de deuxième niveau peut être appliquée en divisant la puissance de chaque bande de fréquence par la puissance totale, ce qui permet d’obtenir une mesure plus intuitive de la contribution relative de chaque composante de fréquence à la puissance globale. Il est désactivé par défaut car il n’a de sens que lorsque les quatre bandes sont disponibles, nécessitant au moins 24 h de signal. Ces algorithmes sont adaptés de la boîte à outils de traitement du signal cardiovasculairePhysionet 46.
- Le même message d’avertissement s’affiche, demandant la confirmation de la suppression du canal PPG supplémentaire qui a été détecté. Encore une fois, c’est normal. Cliquez sur Oui. BrainBeats commencera à prétraiter les données ECG et à extraire les intervalles NN à l’identique, comme à l’étape 2.4. Ensuite, il extrait les caractéristiques de variabilité de la fréquence cardiaque (VFC) à partir des intervalles NN dans les domaines temporel (SDNN, RMSSD, pNN50), fréquentiel (ULF, VLF, LF, HF, rapport LF :HF, puissance totale) et non linéaire (Poincaré, moyenne du signal redressé de phase, entropie floue et dimension fractale).
REMARQUE : BrainBeats vérifie automatiquement la longueur du fichier pour s’assurer que les exigences minimales sont respectées (par exemple, l’alimentation ULF-HRV nécessite 24 h de données), envoie des messages d’avertissement si ce n’est pas le cas et n’exporte pas ces fonctionnalités pour éviter les estimations peu fiables. BrainBeats suit les lignes directrices et les recommandations pour estimer les mesures de VFC27,45. - BrainBeats prétraite les données EEG comme à l’étape 2.7 . (voir Figure 3). Les artefacts volumineux sont détectés automatiquement dans les données continues à l’aide de l’algorithme de reconstruction du sous-espace d’artefacts (ASR)50,52 (critère SD par défaut défini sur 30 et utilisant 80 % de la RAM disponible pour augmenter la vitesse). Ces gros artefacts sont supprimés des données EEG (voir Figure 13). Pour ajuster ces paramètres dans l’interface graphique, sélectionnez les champs Seuil de rejet des segments défectueux avec ASR et RAM disponible à utiliser pour l’ASR.
REMARQUE : Il n’est pas nécessaire de synchroniser dans le temps les séries chronologiques EEG et cardiovasculaires pour le mode des caractéristiques, car les caractéristiques sont estimées sur chaque signal séparément. Ainsi, les artefacts EEG peuvent être supprimés directement des données EEG (en rouge, figure 13), contrairement au mode HEP, où les époques contenant des artefacts ont été rejetées pour les deux séries chronologiques, car la synchronisation temporelle avec la précision ms est essentielle pour cette méthode. - Effectuez l’ICA en utilisant les mêmes algorithmes et paramètres que pour HEP (voir étape 2.11.), sauf que cette fois-ci, les composants cardiaques sont retirés s’ils sont détectés avec un niveau de confiance de 99% (ils ont été conservés pour HEP/HEO car nous ne voulons pas supprimer les signaux cardiaques pertinents).
- La case Domaine fréquentiel est cochée à l’étape 3.2., de sorte que BrainBeats extrait les caractéristiques suivantes du domaine fréquentiel : la densité spectrale de puissance (PSD) moyenne pour les bandes de fréquences delta (1-3 Hz), thêta (3-7 Hz), alpha (8-13 Hz), bêta (13-30 Hz) et gamma (30+ Hz), la fréquence alpha individuelle (IAF) et l’asymétrie alpha sur toutes les paires d’électrodes disponibles (symétriques). Vérifiez ces fonctionnalités.
REMARQUE : La conversion PSD en décibels (dB) facilite la comparaison des résultats entre les enregistrements et les sujets. Les limites de fréquence peuvent être définies aux limites conventionnelles (par exemple, 8-13 Hz prédéfinies pour la bande alpha) ou aux limites individualisées, qui sont détectées à partir de la distribution de la densité spectrale de puissance pour tenir compte des différences interindividuelles69 (par exemple, 7,3-12,6 Hz pour la bande alpha). L’algorithme a été conçu pour la bande alpha et ne fonctionne pas aussi bien pour les autres bandes, en particulier lorsque les pics ne sont pas présents dans la distribution spectrale de puissance. La fréquence alpha individuelle (IAF) est estimée à l’aide du centre de gravité alpha pour mieux traiter les pics divisés ou les pics ambigus69. L’asymétrie alpha est calculée selon les lignes directrices (fenêtre de Hamming de 2 s avec un chevauchement de 50 %, logarithme de la puissance alpha du canal gauche moins logarithme de la puissance alpha du canal droit)47. Par conséquent, les valeurs positives indiquent une puissance alpha plus grande à gauche qu’à droite et vice versa. L’asymétrie alpha peut être normalisée en divisant la puissance alpha de chaque électrode par la puissance alpha additionnée aux bornes de toutes les électrodes47. Les paires symétriques sont obtenues à l’aide des distances thêta, ce qui nécessite que les données EEG contiennent les coordonnées des électrodes. - La case Domaine non linéaire est cochée à l’étape 3.2., BrainBeats extrait l’entropie floue et la dimension fractale pour chaque canal EEG.
REMARQUE : On pense que les caractéristiques du domaine non linéaire capturent la dynamique complexe et non linéaire du cerveau qui sont manquées par les mesures spectrales et sont particulièrement prometteuses pour l’étude des interactions entre divers systèmes corporels 70,71,72. L’entropie floue est plus fiable et plus robuste que ses alternatives (entropie d’échantillon et entropie approchée) mais nécessite des temps de calcul plus longs (en particulier avec de longues séries chronologiques EEG avec des taux d’échantillonnage élevés). Pour résoudre ce problème, lorsque les signaux EEG durent plus de 2 minutes avec une fréquence d’échantillonnage supérieure à 100 Hz, ils sont automatiquement sous-échantillonnés (ou décimés lorsque le facteur n’est pas un entier) à 90 Hz (c’est-à-dire correspondant à une fréquence de Nyquist de 45 Hz, pour correspondre au filtre passe-bas par défaut et éviter autant que possible les artefacts de bruit de ligne). De plus, le calcul parallèle est activé par défaut lors de l’estimation des caractéristiques EEG, ce qui réduit le temps de calcul, en particulier lorsque de nombreux canaux EEG sont disponibles. - Sélectionnez Visualiser les sorties dans la première interface graphique (voir l’étape 3.1) pour générer un graphique affichant la densité spectrale de puissance (PSD) pour les données HRV et EEG (Figure 14) ainsi que les topographies du cuir chevelu affichant certaines caractéristiques EEG (Figure 15).
REMARQUE : Vous pouvez également trouver certaines sorties de prétraitement dans EEG.brainbeats.preprocessing et tous les paramètres utilisés dans EEG.brainbeats.parameters. Nous encourageons les utilisateurs à rendre compte de ces résultats dans des publications scientifiques afin d’accroître la reproductibilité des résultats. - Gardez la case Enregistrer les sorties cochée dans la première fenêtre de l’interface graphique pour enregistrer toutes les fonctionnalités exportées dans le fichier EEGLAB .set dans EEG.brainbeats.features et les enregistrer dans un fichier .mat dans le même dossier où le jeu de données a été chargé.
- BrainBeats prend en charge la fonction d’historique d’EEGLAB. À la fin de toutes les opérations, tapez eegh dans la fenêtre de commande de MATLAB pour afficher la ligne de commande qui vous permettra de répéter toutes les étapes ci-dessus via une seule ligne de commande, avec les paramètres qui ont été sélectionnés manuellement dans l’interface graphique, permettant une automatisation et une réplication faciles des opérations.
- Pour les utilisateurs avancés, effectuez toutes les étapes ci-dessus à l’aide de la commande suivante :
eeglab ; fermer; % Lancer EEGLAB sans l’interface graphique
main_path = fileparts(which('eegplugin_BrainBeats.m')) ; CD(main_path) ;
EEG = pop_loadset('filename','dataset.set',
'filepath',fullfile(main_path, 'sample_data')) ; % de chargement de l’exemple de jeu de données
EEG = brainbeats_process(EEG,' analyse',
'caractéristiques','heart_signal','ECG', 'heart_channels',{'ECG'},'clean_eeg',true) ; - Les étapes précédentes ont extrait les caractéristiques de la VFC du signal ECG. Utilisez les étapes suivantes pour extraire les caractéristiques de la VFC du signal PPG (les caractéristiques EEG sont les mêmes).
- Chargez à nouveau le même ensemble de données (étape 1.5.) puisqu’il a été écrasé par les opérations et ouvrez à nouveau l’interface graphique principale (étape 2.5.). Sélectionnez Extraire les caractéristiques EEG et HRV pour l’analyse, puis sélectionnez PPG pour le type de signal cardiaque et PPG pour le nom du canal. Cliquez sur OK.
- La 2ème fenêtre de l’interface graphique affiche désormais les paramètres de prétraitement de PPG et d’extraction des fonctionnalités HRV et EEG. Cliquez sur OK pour exécuter avec les paramètres par défaut. Les paramètres sont décrits à l’étape 2.17.
- Un message d’avertissement s’affichera, demandant la confirmation de la suppression du canal ECG détecté. Il faut s’y attendre, car la boîte à outils n’est pas conçue pour analyser simultanément l’ECG et le PPG (ou d’autres canaux auxiliaires) et le conserver dans l’ensemble de données entraînera de graves erreurs (par exemple, suppression d’artefacts, mauvaise décomposition de l’ICA, etc.). Cliquez sur Oui.
- BrainBeats prétraite le signal PPG et estime les intervalles NN comme à l’étape 2.5. Vérifiez les caractéristiques HRV extraites des intervalles NN, comme à l’étape 3.2. sauf que les intervalles NN ont maintenant été obtenus à partir du signal PPG. Les signaux EEG sont prétraités comme à l’étape 3.2. BrainBeats trace la PSD (Figure 16). La seule différence ici est la PSD estimée à partir des intervalles NN obtenus à partir du PPG par opposition à l’ECG.
- Pour les utilisateurs avancés, effectuez toutes les étapes ci-dessus à l’aide de la commande suivante :
eeglab ; fermer; % Lancer EEGLAB sans l’interface graphique
main_path = fileparts(which('eegplugin_BrainBeats.m')) ; CD(main_path) ;
EEG = pop_loadset('filename','dataset.set','filepath',
fullfile(main_path, 'sample_data')) ; %Load l’exemple de jeu de données
EEG = brainbeats_process(EEG,'analyse',
'fonctionnalités','heart_signal', 'PPG’heart_channels',{'PPG'},'clean_eeg',true) ;
4. Extrayez les artefacts cardiaques des signaux EEG.
- Chargez l’exemple de jeu de données (voir étape 1.5.).
- Ouvrez la fenêtre principale de l’interface graphique en cliquant sur Outils > BrainBeats > 1er niveau (niveau du sujet) et sélectionnez Extraire les artefacts cardiaques des signaux EEG pour le type d’analyse, ECG pour le type de signal cardiaque et sélectionnez ECG dans la liste des étiquettes d’électrode. Cliquez sur OK.
- La 2ème fenêtre de l’interface graphique affiche les paramètres de prétraitement. Réglez le bruit de la ligne électrique sur 50 Hz (Europe), modifiez le niveau de confiance si nécessaire, cochez la case Mode Boost (bêta) et cliquez sur OK pour exécuter avec les paramètres par défaut puisque les signaux EEG de l’ensemble de données d’échantillon ne sont pas prétraités.
REMARQUE : le niveau de confiance pour détecter les composants cardiaques est défini sur 80 % par défaut, ce qui peut être trop faible ou trop élevé pour certains ensembles de données. L’augmentation de cette valeur augmentera les chances de détecter les composants cardiaques, mais diminuera la fiabilité de cette détection. Le mode Boost (bêta) est facultatif et vise à améliorer les performances de classification en étalant le signal cardiaque dans les signaux EEG. - Un message d’avertissement s’affiche, demandant la confirmation de la suppression du canal PPG supplémentaire qui a été détecté. Il faut s’y attendre, car la boîte à outils n’est pas conçue pour analyser simultanément l’ECG et le PPG (ou d’autres canaux auxiliaires) et le conserver dans l’ensemble de données entraînera de graves erreurs (par exemple, suppression d’artefacts, mauvaise décomposition de l’ICA, etc.). Cliquez sur Oui.
- Le signal ECG est filtré par bande passante pour éliminer les dérives de fréquence lentes inférieures à 1 Hz et les bruits à haute fréquence supérieurs à 20 Hz (avec un filtre FIR à phase zéro non causal). Prétraitez les signaux EEG comme à l’étape 3.4.
- Effectuez une analyse en composantes indépendantes (ICA) à l’aide de l’algorithme préconditionné ICA pour les données réelles (PICARD). Pour modifier cette option, choisissez l’algorithme Infomax standard ou l’algorithme Infomax modifié pour la réplication à partir de l’interface graphique à l’étape 4.3 . Ensuite, classifiez automatiquement les composants indépendants avec ICLabel. Si un composant est classé comme composant cardiaque avec un niveau de confiance de 80 %, il est, par défaut, détecté et extrait automatiquement des données EEG.
- Conservez la case Visualiser les sorties dans la première interface graphique principale (étape 4.2.) pour visualiser la topographie du cuir chevelu du composant retiré (Figure 17 à gauche) et la série chronologique EEG finale (en bleu, Figure 17 à droite) après l’extraction du composant cœur (en rouge, Figure 17 à droite).
REMARQUE : le canal ECG est conservé pour la visualisation afin de confirmer l’extraction des composants liés à l’ECG, mais il est supprimé après cette étape car il ne contient plus d’informations pertinentes. - Pour les utilisateurs avancés, effectuez ces étapes à l’aide des lignes de commande suivantes :
eeglab ; fermer; % Lancer EEGLAB sans l’interface graphique
main_path = fileparts(which('eegplugin_BrainBeats.m')) ; CD(main_path) ;
EEG = pop_loadset('filename','dataset.set','filepath',
fullfile(main_path, 'sample_data')) ;
EEG = brainbeats_process(EEG,'analyse','rm_heart',
'heart_signal', 'ECG', ...
'heart_channels',{'ECG'},'clean_eeg',true,'vis_cleaning',false,...
'conf_thresh',.8,'boost',true) ;
Résultats
Tout d’abord, le plug-in BrainBeats a été utilisé pour prétraiter les données EEG et ECG, identifier et supprimer les artefacts, et analyser les potentiels évoqués par les battements cardiaques (HEP) et les oscillations (HEO). BrainBeats a réussi à détecter les intervalles RR à partir du signal ECG et de certains artefacts RR (Figure 2). BrainBeats a également signalé dans la fenêtre de commande que 11/305 (3,61 %) des pulsations étaient signalées comme des artefacts et int...
Discussion
Étapes critiques du protocole
Les étapes critiques sont décrites aux étapes 1.1 à 1.4. Des avertissements et des messages d’erreur sont mis en place à divers endroits de la boîte à outils pour aider les utilisateurs à comprendre pourquoi ils peuvent rencontrer des problèmes (par exemple, l’emplacement des électrodes n’est pas chargé dans les données EEG, la longueur du fichier est trop courte pour calculer une mesure fiable de la VFC à ultra-basse fréquence, la qualité du signal ...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
L’Institut des Sciences Noétiques a soutenu ces recherches. Nous remercions les développeurs des algorithmes open-source originaux qui ont été adaptés pour développer certains des algorithmes de BrainBeats.
matériels
| Name | Company | Catalog Number | Comments |
| EEGLAB | Swartz Center for Computational Neuroscience (SCCN) | Free/Open-source | |
| MATLAB | The Mathworks, Inc. | Requires a license | |
| Windows PC | Lenovo, Inc. |
Références
- von Bertalanffy, L. . General system theory Foundations, development, applications. , (1968).
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Bean, B. P. Nitrendipine block of cardiac calcium channels: high-affinity binding to the inactivated state. Proc Natl Acad Sci U SA. 81 (20), 6388-6392 (1984).
- Fuchs, T. . Ecology of the brain: The phenomenology and biology of the embodied mind. , (2017).
- Napadow, V., et al. Brain correlates of autonomic modulation: Combining heart rate variability with fMRI. NeuroImage. 42 (1), 169-177 (2008).
- Chang, C., Cunningham, J. P., Glover, G. H. Influence of heart rate on the BOLD signal: The cardiac response function. NeuroImage. 44 (3), 857-869 (2009).
- Gianaros, P. J., Sheu, L. K. A review of neuroimaging studies of stressor-evoked blood pressure reactivity: Emerging evidence for a brain-body pathway to coronary heart disease risk. NeuroImage. 47 (3), 922-936 (2009).
- Burch, G. E., DePasquale, N. P. . A history of electrocardiography. No 1. , (1990).
- Allen, J. Photoplethysmography and its application in clinical physiological measurement. Physiol Meas. 28 (3), 1 (2007).
- Cohen, M. X. Where does EEG come from and what does it mean. Trends Neurosci. 40 (4), 208-218 (2017).
- Cannard, C., Brandmeyer, T., Wahbeh, H., Delorme, A. Self-health monitoring and wearable neurotechnologies. Handb Clin Neurol. 168, 207-232 (2020).
- Gramann, K., Ferris, D. P., Gwin, J., Makeig, S. Imaging natural cognition in action. Int. J. Psychophysiol. 91 (1), 22-29 (2014).
- Jungnickel, E., Gehrke, L., Klug, M., Gramann, K. Chapter 10 - MoBI-Mobile brain/body imaging. Neuroergonomics. , 59-63 (2019).
- Al, E., et al. Heart-brain interactions shape somatosensory perception and evoked potentials. Proc Natl Acad Sci. 117 (19), 10575-10584 (2020).
- Banellis, L., Cruse, D. Skipping a beat: Heartbeat-evoked potentials reflect predictions during interoceptive-exteroceptive integration. Cereb Cortex Commun. 1 (1), (2020).
- Baranauskas, M., Grabauskaitė, A., Griškova-Bulanova, I., Lataitytė-Šimkevičienė, B., Stanikūnas, R. Heartbeat evoked potentials (HEP) capture brain activity affecting subsequent heartbeat. Biomed Signal Process. Cont. 68, 102731 (2021).
- Candia-Rivera, D., et al. Neural responses to heartbeats detect residual signs of consciousness during resting state in postcomatose patients. J Neurosci. 41 (24), 5251-5262 (2021).
- Jiang, H., et al. Brain-heart interactions underlying traditional Tibetan buddhist meditation. Cereb cortex. 30 (2), 439-450 (2020).
- Kumral, D., et al. Attenuation of the heartbeat-evoked potential in patients with atrial fibrillation. JACC Clin Electrophysiol. 8 (10), 1219-1230 (2022).
- Thakor, N. V., Tong, S. Advances in quantitative electroencephalogram analysis methods. Annu Rev Biomed Eng. 6, 453-495 (2004).
- Thayer, J. F., Åhs, F., Fredrikson, M., Sollers, J. J., Wager, T. D. A meta-analysis of heart rate variability and neuroimaging studies: Implications for heart rate variability as a marker of stress and health. Neurosci Biobehav Rev. 36 (2), 747-756 (2012).
- Mather, M., Thayer, J. F. How heart rate variability affects emotion regulation brain networks. Curr Opin Beha. Sci. 19, 98-104 (2018).
- Kemp, A. H., Quintana, D. S. The relationship between mental and physical health: Insights from the study of heart rate variability. Int J Psychophysiol. 89 (3), 288-296 (2013).
- Daneshi Kohan, M., Motie Nasrabadi, A., Shamsollahi, M. B., Sharifi, A. EEG/PPG effective connectivity fusion for analyzing deception in interview. Signal Image Video Process. 14 (5), 907-914 (2020).
- Übeyli, E. D., Cvetkovic, D., Cosic, I. Analysis of human PPG, ECG and EEG signals by eigenvector methods. Digit Signal Process. 20 (3), 956-963 (2010).
- Zambrana-Vinaroz, D., Vicente-Samper, J. M., Manrique-Cordoba, J., Sabater-Navarro, J. M. Wearable epileptic seizure prediction system based on machine learning techniques using ECG, PPG and EEG signals. Sensors. 22 (23), 9372 (2022).
- Shaffer, F., Ginsberg, J. P. An overview of heart rate variability metrics and norms. Front Public Health. 5, 258 (2017).
- Coan, J. A., Allen, J. J. B. The state and trait nature of frontal EEG asymmetry in emotion. The asymmetrical brain. , 565-615 (2003).
- Hagemann, D., Hewig, J., Seifert, J., Naumann, E., Bartussek, D. The latent state-trait structure of resting EEG asymmetry: replication and extension. Psychophysiology. 42 (6), 740-752 (2005).
- Widge, A. S., et al. Electroencephalographic biomarkers for treatment response prediction in major depressive illness: A meta-analysis. Am J Psychiatry. 176 (1), 44-56 (2019).
- Olbrich, S., Arns, M. EEG biomarkers in major depressive disorder: Discriminative power and prediction of treatment response. Int Rev Psychiatry. 25 (5), 604-618 (2013).
- Kumar, Y., Dewal, M. L., Anand, R. S. Epileptic seizures detection in EEG using DWT-based ApEn and artificial neural network. Signal Image Video Process. 8, 1323-1334 (2014).
- Acharya, U. R., et al. Automated diagnosis of epileptic EEG using entropies. Biomed Signal Process Control. 7 (4), 401-408 (2012).
- de Aguiar Neto, F. S., Rosa, J. L. G. Depression biomarkers using non-invasive EEG: A review. Neurosci Biobehav Rev. 105, 83-93 (2019).
- Cannard, C., Wahbeh, H., Delorme, A. Electroencephalography correlates of well-being using a low-cost wearable system. Front Hum Neurosci. 15, 736 (2021).
- Tarvainen, M. P., Niskanen, J. P., Lipponen, J. A., Ranta-aho, P. O., Karjalainen, P. A. Kubios HRV - Heart rate variability analysis software. Comput. Methods Programs Biomed. 113 (1), 210-220 (2014).
- Demski, A. J., Soria, M. L. ECG-kit: a Matlab toolbox for cardiovascular signal processing. Journal of Open Research Software. 4 (1), e8 (2016).
- Klug, M., et al. The BeMoBIL pipeline for automated analyses of multimodal mobile brain and body imaging data. BioRxiv. , (2022).
- Perakakis, P. . HEPLAB. , (2023).
- Grosselin, F., Navarro-Sune, X., Raux, M., Similowski, T., Chavez, M. CARE-rCortex: A Matlab toolbox for the analysis of CArdio-REspiratory-related activity in the Cortex. J Neurosci Meth. 308, 309-316 (2018).
- Luck, S. J., Gaspelin, N. How to get statistically significant effects in any ERP experiment (and why you shouldn't). Psychophysiology. 54 (1), 146-157 (2017).
- Alday, P. M. How much baseline correction do we need in ERP research? Extended GLM model can replace baseline correction while lifting its limits. Psychophysiology. 56 (12), e13451 (2019).
- Delorme, A. EEG is better left alone. Sci Rep. 13 (1), 2372 (2023).
- Widmann, A., Schröger, E., Maess, B. Digital filter design for electrophysiological data - a practical approach. J Neurosci Methods. 250, 34-46 (2015).
- Pham, T., Lau, Z. J., Chen, S. H. A., Makowski, D. Heart Rate Variability in Psychology: A Review of HRV Indices and an Analysis Tutorial. Sensors. 21 (12), 3998 (2021).
- Vest, A. N., et al. An open source benchmarked toolbox for cardiovascular waveform and interval analysis. Physiol Meas. 39 (10), 105004 (2018).
- Smith, E. E., Reznik, S. J., Stewart, J. L., Allen, J. J. B. Assessing and conceptualizing frontal EEG asymmetry: An updated primer on recording, processing, analyzing, and interpreting frontal alpha asymmetry. Int J Psychophysiol Off J Int Organ Psychophysiol. 111, 98-114 (2017).
- Dong, L., et al. MATLAB toolboxes for reference electrode standardization technique (REST) of scalp EEG. Front Neurosci. 11, 601 (2017).
- Candia-Rivera, D., Catrambone, V., Valenza, G. The role of electroencephalography electrical reference in the assessment of functional brain-heart interplay: From methodology to user guidelines. J Neurosci Methods. 360, 109269 (2021).
- Mullen, T. R., et al. Real-time Neuroimaging and cognitive monitoring using wearable dry EEG. IEEE Trans Biomed Eng Spec Issue Wearable Technol. 62 (11), 2553-2567 (2015).
- Chang, C. Y., Hsu, S. H., Pion-Tonachini, L., Jung, T. P. Evaluation of artifact subspace reconstruction for automatic EEG artifact removal. 40th Ann Int Conf IEEE Eng Med Biol Soc. , 1242-1245 (2018).
- Miyakoshi, M. Artifact subspace reconstruction: a candidate for a dream solution for EEG studies, sleep or awake. Sleep. 46 (12), 241 (2023).
- Kim, H., et al. ICA's bug: How ghost ICs emerge from effective rank deficiency caused by EEG electrode interpolation and incorrect re-referencing. Front Signal Process. 3, 1064138 (2023).
- Frank, G., Makeig, S., Delorme, A. A Framework to evaluate independent component analysis applied to EEG signal: testing on the Picard algorithm. ArXiv. , (2022).
- Ablin, P., Cardoso, J. F., Gramfort, A. Faster independent component analysis by preconditioning with Hessian approximations. IEEE Trans Signal Process. 66 (15), 4040-4049 (2018).
- Pion-Tonachini, L., Kreutz-Delgado, K., Makeig, S. ICLabel: An automated electroencephalographic independent component classifier, dataset, and website. NeuroImage. 198, 181-197 (2019).
- Bigdely-Shamlo, N., Mullen, T., Kothe, C., Su, K. M., Robbins, K. A. The PREP pipeline: standardized preprocessing for large-scale EEG analysis. Front Neuroinformatics. 9, 12 (2015).
- Maris, E., Oostenveld, R. Nonparametric statistical testing of EEG- and MEG-data. J Neurosci Methods. 164 (1), 177-190 (2007).
- Pernet, C. R., Latinus, M., Nichols, T. E., Rousselet, G. A. Cluster-based computational methods for mass univariate analyses of event-related brain potentials/fields: A simulation study. J Neurosci Methods. 250, 85-93 (2015).
- Pernet, C. R., Chauveau, N., Gaspar, C., Rousselet, G. A. LIMO EEG: A toolbox for hierarchical LInear MOdeling of ElectroEncephaloGraphic data. Comput Intell Neurosci. 2011, 1-11 (2011).
- Pernet, C., et al. Electroencephalography robust statistical linear modelling using a single weight per trial. Aperture Neuro. 2022 (7), 51 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I., Busch, N. A. Pupillometry and electroencephalography in the digit span task. Sci. Data. 9 (1), 325 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I. EEG, pupillometry, ECG and photoplethysmography, and behavioral data in the digit span task and rest. OpenNeuro. , (2024).
- Clifford, G. . Signal processing methods for heart rate variability. , (2002).
- Pan, J., Tompkins, W. J. A real-time QRS detection algorithm. IEEE Trans. Biomed. Eng. 3, 230-236 (1985).
- Maess, B., Schröger, E., Widmann, A. Highpass filters and baseline correction in M/EEG analysis. Commentary on: "How inappropriate highpass filters can produce artefacts and incorrect conclusions in ERP studies of language and cognition. J. Neurosci. Methods. 266, 164-165 (2016).
- Park, H. D., Blanke, O. Heartbeat-evoked cortical responses: Underlying mechanisms, functional roles, and methodological considerations. NeuroImage. 197, 502-511 (2019).
- Lomb, N. R. Least-squares frequency analysis of unequally spaced data. Astrophys Space Sci. 39, 447-462 (1976).
- Corcoran, A. W., Alday, P. M., Schlesewsky, M., Bornkessel-Schlesewsky, I. Toward a reliable, automated method of individual alpha frequency (IAF) quantification. Psychophysiology. 55 (7), e13064 (2018).
- Chen, W., Zhuang, J., Yu, W., Wang, Z. Measuring complexity using FuzzyEn, ApEn, and SampEn. Med Eng Phys. 31 (1), 61-68 (2009).
- Cannard, C., Delorme, A. . An open-source EEGLAB plugin for computing entropy-based measures on MEEG signals. , (2022).
- Lau, Z. J., Pham, T., Chen, S. H. A., Makowski, D. Brain entropy, fractal dimensions and predictability: A review of complexity measures for EEG in healthy and neuropsychiatric populations. Eur J Neurosci. 56 (7), 5047-5069 (2022).
- Costa, M., Goldberger, A. L., Peng, C. -. K. Multiscale entropy analysis of biological signals. Phys Rev E Stat Nonlin Soft Matter Phys. 71 (2), 021906 (2005).
- Humeau-Heurtier, A. multiscale entropy approaches and their applications. Entropy. 22 (6), 644 (2020).
- Armañac-Julián, P., et al. Reliability of pulse photoplethysmography sensors: Coverage using different setups and body locations. Front Electron. 3, 906324 (2022).
- Catrambone, V., Greco, A., Vanello, N., Scilingo, E. P., Valenza, G. Time-resolved directional brain-heart interplay measurement through synthetic data generation models. Ann Biomed Eng. 47, 1479-1489 (2019).
- Georgieva-Tsaneva, G., Gospodinova, E., Gospodinov, M., Cheshmedzhiev, K. Portable sensor system for registration, processing and mathematical analysis of PPG signals. Appl Sci. 10 (3), 1051 (2020).
- Kim, J. H., Park, S. E., Jeung, G. W., Kim, K. S. Detection of R-peaks in ECG signal by adaptive linear neuron (ADALINE) artificial neural network. MATEC Web Conf. 54, 10001 (2016).
- Lei, R., Ling, B. W. K., Feng, P., Chen, J. Estimation of heart rate and respiratory rate from PPG signal using complementary ensemble empirical mode decomposition with both independent component analysis and non-negative matrix factorization. Sensors. 20 (11), 3238 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon