Écosystème modèle à base d’agarose pour la culture de méthanotrophes dans un contre-gradient méthane-oxygène
Dans cet article
Résumé
Un protocole est décrit pour la préparation d’un écosystème modèle simple qui recrée le gradient de compteur méthane-oxygène trouvé dans l’habitat naturel des bactéries oxydantes de méthane aérobies, permettant l’étude de leur physiologie dans un contexte spatialement résolu. Les modifications apportées aux essais biochimiques courants pour l’utilisation avec l’écosystème modèle à base d’agarose sont également décrites.
Résumé
Les bactéries aérobies oxydantes du méthane, connues sous le nom de méthanotrophes, jouent un rôle important dans le cycle biogéochimique. Les méthanotrophes occupent une niche environnementale spécifique au sein des gradients de compteur méthane-oxygène trouvés dans les sols et les sédiments, ce qui influence leur comportement au niveau individuel et communautaire. Cependant, les méthodes conventionnelles d’étude de la physiologie de ces micro-organismes qui atténuent les gaz à effet de serre utilisent souvent des cultures planctoniques homogènes, qui ne représentent pas avec précision les gradients spatiaux et chimiques trouvés dans l’environnement. Cela entrave la compréhension des scientifiques sur le comportement de ces bactéries in situ. Ici, un écosystème modèle simple et peu coûteux appelé la seringue de gradient est décrit, qui utilise de l’agarose semi-solide pour recréer les gradients abrupts de compteur méthane-oxygène caractéristiques des habitats naturels des méthanotrophes. La seringue à gradient permet de cultiver des souches méthanotrophes et d’enrichir des consortiums mixtes oxydant le méthane à partir d’échantillons environnementaux, révélant des phénotypes uniquement visibles dans ce contexte spatialement résolu. Ce protocole fait également état de divers essais biochimiques qui ont été modifiés pour être compatibles avec la matrice d’agarose semi-solide, ce qui peut être utile aux chercheurs qui cultivent des microorganismes dans d’autres systèmes à base d’agarose.
Introduction
Les micro-organismes vivant à une interface anoxique-oxique jouent souvent des rôles écologiques importants1. Un exemple est celui des bactéries aérobies oxydant le méthane (méthanotrophes), qui existent dans les contre-gradients de méthane et d’oxygène dans les sols et les sédiments2. Ces micro-organismes possèdent des caractéristiques métaboliques et physiologiques uniques qui leur permettent d’exploiter les gradients de gaz présents dans leur environnement et font l’objet de recherches en cours depuis des décennies 3,4,5. À l’heure actuelle, la plupart des recherches publiées sur les méthanotrophes et les communautés oxydantes de méthane sont basées sur des travaux avec des cultures planctoniques homogènes qui ne parviennent souvent pas à saisir les gradients spatiaux et chimiques inhérents à leurs habitats microbiens naturels. Cette limitation entrave notre compréhension de la physiologie microbienne et notre capacité à relier l’information génomique aux traits phénotypiques.
Ce protocole fait état d’un écosystème modèle simple, basé sur le laboratoire, qui crée des conditions reproductibles pour l’étude à la fois de méthanotrophes spécifiques, tels que la souche LW13 de Methylomonas sp., et de communautés oxydantes de méthane directement à partir d’échantillons de sol environnementaux. Il est important de noter que la culture dans la seringue à gradient permet d’obtenir des phénotypes spécifiques au contre-gradient qui ne sont pas présents dans les cultures planctoniques homogènes6, mettant en évidence la capacité du système à dévoiler de nouveaux aspects de la physiologie méthanotrophe. Inspirée des écosystèmes modèles 7,8,9 précédemment publiés, la seringue à gradient est une méthode simplifiée qui peut être utilisée pour collecter des informations chimiques et moléculaires à partir de micro-organismes cultivés à l’aide de cette approche.
Les procédures rapportées pour les analyses génétiques, chimiques et moléculaires ont été modifiées pour fonctionner de manière fiable sur des cultures microbiennes cultivées dans une matrice d’agarose semi-solide. Ces procédures peuvent également être utiles pour analyser les bactéries cultivées dans d’autres systèmes à base d’agarose semi-solide, tels que ceux utilisés pour les essais de nage en gélose molle bactérienne. L’adaptation de ces analyses à des contextes spatialement résolus pourrait ouvrir de nouvelles voies pour l’étude de la vie microbienne dans des environnements plus pertinents sur le plan écologique.
Protocole
Les détails des réactifs et de l’équipement utilisés dans l’étude sont énumérés dans la table des matériaux.
1. Préparation et extrusion de seringues à gradient
REMARQUE : La préparation de la seringue à gradient doit être effectuée à l’aide d’une technique stérile.
- Utiliser plusieurs colonies de Methylomonas sp. LW13 fraîchement cultivées sur une plaque pour inoculer 6 mL de milieu à sels minéraux nitrés (NMS) dans un tube de verre de 18 mm x 150 mm. Scellez le tube à l’aide d’un bouchon de sérum et d’un joint à sertir en aluminium, et ajoutez du méthane à l’aide d’une seringue à une atmosphère finale de 50 % (v/v) de méthane dans l’air. Agitez cette culture liquide planctonique à 200 tr/min à température ambiante jusqu’à ce qu’elle soit trouble (environ un jour).
- Passage des cultures liquides 1:10 dans des milieux frais. Continuer la croissance des cultures liquides de méthanotrophes jusqu’à la croissance en phase logarithmique (DO600 de ~0,5) et ajuster à une DO600 = 1,0.
- Préparez les seringues en retirant le piston qui les accompagne et en les conservant dans un récipient stérile. Fixez un embout filtrant stérile en PTFE à la seringue et placez-le dans un support de tube à essai standard avec l’embout vers le bas.
- Pour chaque seringue de 10 ml, bien mélanger 1 mL de cellules de l’étape 1.2 avec 5 mL de NMS et 4 mL d’agarose fondue (0,5 % m/v, refroidi à 55 °C) dans un tube conique stérile. Ces volumes peuvent être augmentés pour remplir plusieurs seringues en parallèle.
- Verser lentement ou à l’aide d’une pipette sérologique pour ajouter le mélange à chaque seringue, jusqu’au repère de 8 mL. Laisser l’agarose se solidifier à l’intérieur des seringues (~15 min), puis boucher avec un bouchon en caoutchouc butyle stérile de 20 mm. Fixez le bouchon à la seringue à l’aide de ruban adhésif de laboratoire et étiquetez-le avec le contenu de la seringue.
- Pour ajouter du méthane dans l’espace libre de la seringue, remplissez une grande seringue (60 ml) avec 100 % de CH4 et fixez un embout filtrant en PTFE (0,2 μm, 25 mm) relié à une aiguille stérile (23 G). Percez le bouchon en caoutchouc avec la grande seringue et percez une deuxième aiguille stérile à travers le bouchon pour créer une sortie de gaz.
- Appuyez sur le piston de la grande seringue pour permettre à 20 ml de CH4 à 100 % de s’écouler dans l’espace de tête, en prenant soin de retirer l’aiguille de sortie lorsqu’il reste 1 à 2 ml de CH4 dans la grande seringue pour éviter le reflux d’oxygène par l’aiguille de sortie.
- Incuber les seringues à 18 °C, en répétant les étapes 1.6 et 1.7 tous les jours pour reconstituer le méthane.
- Pour extruder de l’agarose, remplacez l’embout du filtre en PTFE par une aiguille stérile de 23 G et remplacez le bouchon en caoutchouc par le piston de seringue fourni. Appuyez lentement sur le piston pour distribuer des incréments de 1 ml dans des tubes de microcentrifugation stériles séparés de 1,5 ml.
2. Détermination des concentrations de gaz à contre-gradient
- Mesure du gradient d’oxygène dissous
- À l’aide d’une lame de rasoir, trancher la largeur d’une seringue remplie d’agarose (préparée en suivant les étapes 1.1 à 1.8) près du filtre en PTFE. Fixez la seringue ouverte à un pousse-seringue orienté vers une microélectrode de type Clark, l’extrémité ouverte faisant face à l’extrémité de l’électrode.
- Ajustez les réglages du pousse-seringue pour déplacer la seringue vers la microélectrode à un débit de 1 mL/min (0,6 cm/min) ; commencer à enregistrer les mesures d’oxygène dissous sur le logiciel Unisense Logger dès que le pousse-seringue commence à bouger.
- Mesure du gradient de méthane
- Immédiatement avant l’extrusion, remplacez l’embout du filtre de la seringue par un robinet d’arrêt unidirectionnel relié à une aiguille de 23 G, et remplacez rapidement le bouchon en caoutchouc par un piston de seringue. Ajouter huit aliquotes d’agarose de 1 mL pour séparer les fioles étanches au gaz de 12 mL sous vide et laisser les échantillons s’équilibrer à température ambiante pendant 1 h.
- Équilibrez les flacons d’échantillons en fonction de la pression atmosphérique en les ouvrant et en les refermant immédiatement ou en perçant et en retirant rapidement une aiguille. Injecter 500 μL d’espace libre dans un chromatographe en phase gazeuse avec détection par ionisation de flamme (GC-FID) à l’aide d’une seringue étanche au gaz. Créez une courbe d’étalonnage dérivée des étalons CH4 pour convertir l’aire de pic (pA*min) en μmol/L.
3. Comptage des cellules dans la seringue à gradient
- Cytométrie en flux
- Extruder 1 mL de segments d’agarose à partir de seringues à gradient inoculées avec le LW13 de type sauvage ou mutant, comme indiqué à l’étape 1.9. Préparez et extrudez également de l’agarose à partir d’une seringue stérile et acellulaire comme contrôle négatif.
- Ajouter 0,75 mL de NaCl à 0,85 % (m/v) dans l’eau à tous les échantillons d’agarose extrudé et homogénéiser par vortex. De plus, diluez les échantillons à l’échelle 1:10 en transférant 100 μL dans un nouveau tube de microcentrifugation et en ajoutant 900 μL de la solution saline.
- Ajouter 3 μL d’un mélange 1:1 de colorants SYTO9 et d’iodure de propidium, puis incuber dans l’obscurité à température ambiante pendant 15 min. Pour déterminer les cellules par mL d’agarose, sonicer la suspension de billes de comptage des microsphères dans un bain-marie pendant 5 min. Ensuite, ajoutez 10 μL de suspension à chaque échantillon avant l’analyse par cytométrie en flux.
- Analysez les échantillons à l’aide d’un cytomètre en flux10,11 avec les paramètres suivants : déclenchement sur fluorescence verte, débit de 10 μL/s et taux d’analyse des particules inférieur à 1 000 particules/s.
- Comparez SPC vs. Tracé par points FITC entre les échantillons de contrôle acellulaires et les échantillons d’agarose inoculés pour dessiner des portes de tension d'« événement bactérien » qui excluent les particules d’agarose de fond. De plus, dessinez des portes de tension pour les billes de comptage des microsphères, qui doivent être cohérentes entre les échantillons.
- Pour déterminer la concentration de cellules dans chaque segment d’agarose à l’intérieur de la seringue à gradient, utilisez l’équation suivante, en notant que le facteur de dilution pour le protocole ci-dessus est de 17,7275 et que 10-6 mL est le volume d’une microbille de microsphère.
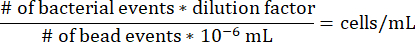
- Comptage des unités formant des colonies dans la seringue à gradient
- Extruder 1 mL de segments d’agarose dans des tubes de microcentrifugation séparés et stériles de 2 mL, ajouter 800 μL de NMS et agiter pendant 10 s pour faciliter le pipetage.
- Préparez une plaque stérile à 96 puits en ajoutant 180 μL de SGEN dans chaque puits. Ajouter 20 μL d’échantillons d’agarose diluée dans chaque puits de la première colonne et pipeter pour mélanger.
- À l’aide d’une pipette multicanaux, diluez dix fois les échantillons en série en transférant 20 μL de la première rangée de puits dans la deuxième rangée de puits et en pipetant 10 fois pour mélanger. Continuez ce processus jusqu’à la dernière rangée de l’assiette.
- Étiquetez les plaques à grille carrée contenant la gélose NMS ou le milieu de votre choix. À l’aide d’une pipette multicanaux, prélever 5 μL d’une colonne de la plaque à 96 puits sur la plaque de gélose. Selon la taille de la plaque de gélose, plusieurs colonnes peuvent être repérées sur la même plaque.
- Incuber des plaques à moins de 40 % de méthane dans l’air et les faire croître à 18 °C. Comptez les colonies bactériennes après 2-3 jours et déterminez les unités formant colonies par millilitre (UFC/ml).
4. Tests de détection de biomolécules
- Dosage des polysaccharides
- Extruder 1 mL de segments d’agarose dans des tubes de microcentrifugation séparés de 2 mL et mélanger avec 1 mL d’une solution de Na2CO3 à 1 % (m/v) dans de l’eau. Chauffer les échantillons à 80 °C pendant 30 min avec vortex toutes les 5 à 10 min, suivi d’une centrifugation à 4 000 x g à 4 °C pendant 20 min.
- Recueillir le surnageant combiné à trois volumes d’éthanol à 100 % et incuber à 20 °C pendant au moins 2 h (ou toute la nuit).
- Recueillir les polysaccharides précipités à l’éthanol par centrifugation à 16 100 x g à 4 °C pendant 30 min. Retirez le surnageant et faites sécher la pastille à l’air. Remettre la pastille en suspension dans 100 μL d’eau déminéralisée.
- Mesurer la teneur relative en polysaccharides de chaque segment d’agarose à l’aide d’un dosage colorimétrique à l’acide phénol-sulfurique12,13. Combinez 50 μL de l’extrait remis en suspension avec 150 μL d’acide sulfurique concentré et 30 μL de phénol à 5 % (v/v) dans de l’eau dans une plaque transparente à 96 puits.
- Mesurez l’absorbance à 490 nm à l’aide d’un lecteur de microplaques et calculez la teneur relative en polysaccharides de chaque segment d’agarose en pourcentage de l’absorbance du segment d’agarose le plus proche du filtre PTFE.
- Dosage des protéines
- Extruder l’agarose dans des tubes de microcentrifugation séparés de 1,5 mL et transférer 100 μL de chaque aliquote dans des tubes à essai en verre.
- Déterminer la concentration totale en protéines à l’aide du protocole en éprouvette d’un kit de dosage des protéines BCA. Préparez des étalons d’albumine BSA avec de l’agarose extrudé d’une seringue stérile comme diluant.
- Dosage de l’ADN extracellulaire
- Extruder l’agarose dans des tubes de microcentrifugation séparés de 1,5 mL et transférer 20 μL de chacun dans des tubes de microcentrifugation de 0,2 mL.
- Mesurez les concentrations d’ADN à l’aide du kit de dosage haute sensibilité 1x dsDNA disponible dans le commerce, conformément au protocole du fabricant.
5. Extraction de l’ARN
- Préparez le tampon d’extraction en combinant les éléments suivants dans 800 mL d’eau exempte de RNase : 2,0 g de CTAB, 2,0 g de polyvinylpyrrolidone (PVP 40), 81,8 g de NaCl, 100 mM de Tris-HCl (pH 8,0) et 20 mM d’EDTA. Porter le volume à 1 L et autoclave ; Conserver à 4 °C.
- Aliquote le tampon d’extraction préparé et ajoute 1 % (v/v) de concentration finale de bêta-mercaptoéthanol juste avant l’utilisation. Réchauffez le tampon à 65 °C à l’aide d’un bain-marie ou d’un bloc chauffant.
- Divisez chaque seringue à gradient en sections de 1 mL en extrudant de l’agarose dans des tubes de microcentrifugation séparés sans RNase de 2 mL en suivant la procédure décrite à l’étape 1.9.
- Centrifuger les échantillons à 21 000 x g, 4 °C, pendant 15 minutes et jeter le surnageant en gardant les échantillons sur de la glace.
- Ajouter 600 μL de tampon d’extraction préchauffé à chaque granule de 1 mL d’agarose extrudé. Ajouter environ 200 μL de billes de zircone ou de silice et homogénéiser les échantillons pendant 3 minutes à 30 Hz/s à l’aide d’un batteur à billes, en faisant une pause à mi-chemin pour placer les échantillons sur de la glace pendant 2 minutes.
- Centrifuger les échantillons à 15 000 x g, 4 °C, pendant 2 min pour réduire la formation de mousse. Extraire des échantillons en ajoutant 600 μL de chloroforme : alcool isoamylique (24:1) dans des tubes et en faisant tourbillonner pendant 10 s.
- Centrifuger les échantillons à 15 000 x g, 4 °C, pendant 8 min. Transférez avec précaution la phase aqueuse supérieure dans un nouveau tube de microcentrifugation sans RNase et ajoutez 600 μL de chloroforme : alcool isoamylique (24:1) à la phase supérieure transférée et vortex pendant 10 s.
- Centrifuger les échantillons à 15 000 x g, 4 °C, pendant 8 min et transférer la nouvelle phase aqueuse supérieure dans un nouveau tube de microcentrifugation sans RNase. Ajouter un volume égal d’isopropanol à la phase supérieure transférée et incuber les échantillons pendant plusieurs heures à -20 °C. En option : les échantillons peuvent être laissés à -20 °C pendant la nuit.
- Prélever les précipités contenant de l’ARN en centrifugeant les échantillons à 16 100 x g, 4 °C pendant 30 min.
- Jeter le surnageant et laver la pastille avec 300 μL d’éthanol froid à 75 % (v/v) fabriqué avec de l’eau sans RNase et centrifuger à 16 100 x g, 4 °C, pendant 5 min.
- Lavez à nouveau les granulés en suivant l’étape 5.10.
- Après avoir retiré le surnageant d’éthanol, laissez les granulés sécher à l’air libre pendant 15 min. Dissoudre les granulés dans 100 μL d’eau sans RNase.
- Traitez les échantillons avec de la DNase I à 37 °C pendant 30 min en suivant le protocole du fabricant.
- Inactiver la DNase I en ajoutant 300 μL de phénol acide : chloroforme : IAA (125:24:1, pH 4,5). Agiter pendant 10 s et incuber à température ambiante pendant 5 min.
- Centrifuger à 16 100 x g, 4 °C, pendant 5 min et conserver la phase aqueuse supérieure en la transférant dans un nouveau tube de microcentrifugation sans RNase.
- Facultatif : Regrouper l’ARN du même segment dans toutes les seringues répliquées en combinant les phases supérieures dans un tube conique.
- Ajouter 1 volume d’isopropanol égal au volume de la phase supérieure contenant l’ARN et ajouter 7,5 M de LiCl à une concentration finale de 0,8 M, en inversant plusieurs fois pour mélanger.
- Incuber les échantillons pendant plusieurs heures à -20 °C. En option : les échantillons peuvent être laissés à -20 °C pendant la nuit.
- Centrifuger les échantillons à 16 100 x g, 4 °C, pendant 15 min et jeter soigneusement le surnageant. Lavez deux fois la pastille contenant de l’ARN en ajoutant de l’éthanol froid à 70 % (v/v) dans de l’eau exempte de RNase, en centrifugeant à 16 100 x g, 4 °C, pendant 5 min, et en éliminant le surnageant.
- Laissez les granulés sécher à l’air libre à température ambiante pendant 10 min et mettez-les en suspension dans 50 μL d’eau sans RNase. Conservez les échantillons d’ARN à -80 °C.
- Facultatif, en fonction de la qualité de l’ARN : les échantillons peuvent être repurifiés à l’aide d’un kit de purification d’ARN pour éliminer les petits ARN (<200 nt).
- Pour confirmer que les échantillons d’ARN ne contiennent pas d’ADN résiduel, utilisez 1 μL d’ARN purifié comme matrice pour l’amplification par PCR à l’aide d’amorces universelles du gène de l’ARNr bactérien 16S 27F/1492R. Inclure deux réactions PCR supplémentaires contenant 1 μL d’ADN génomique (dilué 1:100) ou d’eau sans nucléase pour servir de témoins positifs et négatifs, respectivement.
- Exécutez les produits sur un gel d’agarose TBE à 1 % en utilisant l’électrophorèsesur gel 14.
REMARQUE : Les échantillons d’ARN sans contamination par l’ADN doivent entraîner l’absence d’une bande dans la voie d’échantillon. Si les échantillons d’ARN montrent une contamination par l’ADN, retraitez les échantillons à partir de l’étape 5.13. L’ARN est prêt pour l’analyse en aval et peut éventuellement être analysé pour quantifier son intégrité en mesurant l’indice d’intégrité de l’ARN (RIN).
Résultats Représentatifs
Ici, l’écosystème modèle de seringue à gradient a été utilisé pour cultiver une seule souche (la souche méthanotrophe Methylomonas sp. LW13) (Figure 1A)6, mais il peut également être utilisé pour enrichir une communauté microbienne oxydant le méthane par inoculation directe dans le sol (Figure 1B). La présence d’un gradient de compteur méthane-oxygène a été validée en mesurant la concentration de méthane et d’oxygène dans des seringues acellulaires et inoculées (figure 1C). Pour les seringues à gradient inoculées LW13, un contre-gradient s’est formé dans la journée suivant le rinçage de la seringue, qui s’est accentué au cours des trois jours d’incubation. Au cours de la même période, une bande horizontale s’est formée à la même profondeur à laquelle les deux substrats gazeux ont atteint leurs concentrations les plus faibles (figure 1A). Le gradient de gaz abrupt et l’appauvrissement en méthane et en oxygène au-delà de la profondeur de la bande horizontale ont montré que LW13 métabolisait le méthane de manière aérobie et produisait un phénotype non observé en culture planctonique homogène. Ce phénotype est également produit par d’autres bactéries méthanotrophes isolées à partir du même échantillon environnemental que LW136. La variation dépendante de la souche dans le moment et la profondeur du développement de la bande horizontale entre différentes souches méthanotrophes suggère que la bande horizontale était affectée par le comportement spécifique de chaque microbe lorsqu’il est cultivé dans un contexte spatialement résolu6.
Le nombre de cellules dans l’ensemble du bouchon d’agarose a été mesuré à l’aide de la cytométrie en flux et du nombre de colonies (UFC/mL) (figure 2A). Cette méthode a été utilisée pour comparer la distribution cellulaire et la survie de LW13 de type sauvage à une souche mutante de LW13 contenant une délétion du gène de la fucose 4-O-acétyltransférase (OAT), dont il a été précédemment démontré qu’elle influençait le développement de la bande horizontale6. Le mutant ΔOAT de LW13 a eu une croissance globale plus faible dans la seringue à gradient sur 6 jours par rapport au type sauvage, un effet qui n’a pas été observé dans les cultures planctoniques homogènes des mêmes souches6. La souche mutante ne formait pas la même bande horizontale distincte que le type sauvage LW13 lorsqu’elle a été cultivée dans la seringue à gradient (figure 2B). Le nombre de cellules et l’apparence de la bande horizontale ont été restaurés à des niveaux similaires à ceux du type sauvage lors de la complémentation génique dans la souche mutante. Ces résultats démontrent que la seringue à gradient peut être utilisée pour lier des gènes à des phénotypes spécifiques uniquement présents dans le gradient compteur méthane-oxygène.
Diverses techniques génétiques, chimiques et moléculaires ont été adaptées pour être utilisées avec des bactéries cultivées dans une matrice d’agarose semi-solide. L’écosystème du modèle de seringue à gradient peut être facilement utilisé pour les tests de quantification biomoléculaire standard avec l’inclusion de l’agarose non inoculée comme contrôle négatif. La concentration de trois biomolécules différentes que l’on trouve couramment dans les substances polymères extracellulaires et les biofilms a été mesurée : les polysaccharides, les protéines et l’ADN extracellulaire15 (Figure 3). Dans les segments de seringue inoculés LW13, la bande horizontale présentait significativement plus de polysaccharides que les autres segments, sans augmentation significative des protéines ou de l’ADN extracellulaire.
Le séquençage de l’ARN a été utilisé pour mesurer les différences transcriptionnelles dans LW13 se développant à différentes profondeurs de la seringue. L’extraction robuste de l’ARN a été réalisée à l’aide d’un tampon d’extraction à base de CTAB, suivi d’étapes conventionnelles d’extraction de phénol : chloroforme et de précipitation. Les résultats de l’analyse RNA-seq ont ensuite été utilisés pour identifier les gènes impliqués dans la production de la bande polysaccharidique horizontale. Ces résultats indiquent que l’agarose semi-solide indispensable à la création d’un écosystème modèle spatialement résolu n’empêche pas de poursuivre les analyses biochimiques qui sont généralement réservées aux cultures planctoniques et sur plaques.
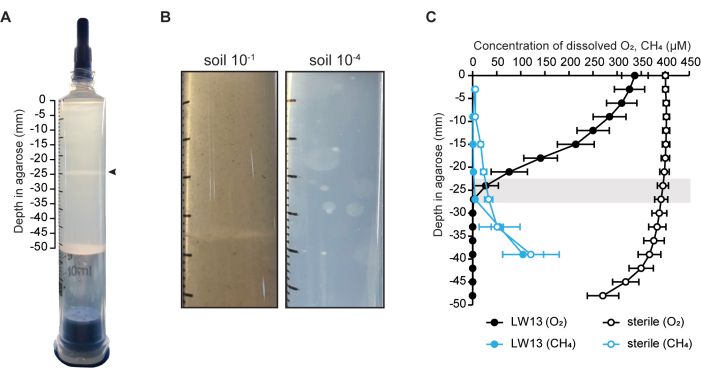
Figure 1 : L’écosystème du modèle de seringue à gradient. (A) La seringue à gradient inoculée avec le méthanotrophe Methylomonas sp. LW13. Une bande horizontale distincte (pointe de flèche) se forme dans les deux jours suivant le rinçage de la seringue avec 100 % de méthane. (B) Gros plan de seringues à gradient inoculées avec de la terre diluée 10-1 et 10-4 et incubées pendant deux semaines. Les seringues à gradient contenant plus de sol dilué ont donné lieu à des colonies sphériques dans l’ensemble de l’agarose, tandis que les inoculums de sol plus concentrés ont donné lieu à une bande distincte. (C) Caractérisation du gradient compteur méthane-oxygène dans des seringues à gradient stérile et inoculées LW13 après trois jours d’incubation. La barre grise indique la plage de profondeurs à laquelle la bande polysaccharidique a été localisée ; les données montrent la moyenne ± ET de trois expériences indépendantes avec trois répétitions techniques chacune. Les panneaux (A) et (C) ont été modifiés à partir de Beals et al.6. Veuillez cliquer ici pour voir une version agrandie de cette figure.
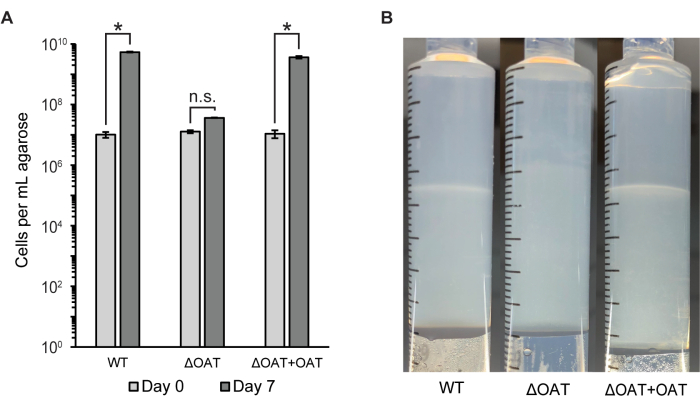
Figure 2 : Quantification du type sauvage et du mutant LW13 après incubation dans la seringue à gradient. (A) Le nombre total de cellules LW13 par mL d’agarose extrudée récupérée à partir de seringues à gradient au jour 0 et au jour 7, mesuré par cytométrie en flux. ΔOAT contient une délétion du gène fucose 4-O-acétyltransférase, qui s’est avéré être fortement exprimé dans les cellules situées à la profondeur de la bande polysaccharidique. ΔOAT+OAT contient le gène OAT inséré à un endroit distal du génome ΔOAT. *, significativement différent (test t hétéroscédastique bilatéral, α = 0,05) ; N.S., pas significativement différent. Les données montrent la moyenne ± ET de trois expériences indépendantes avec deux répétitions techniques chacune. (B) Développement d’une bande horizontale dans des seringues à gradient inoculées avec LW13 de type sauvage, ΔOAT ou ΔOAT+OAT après sept jours d’incubation. Cette figure a été modifiée à partir de Beals et al.6. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure 3 : Quantification des biomolécules à des profondeurs croissantes de la seringue à gradient. Teneur relative en polysaccharides (%), concentration en protéines (μg/mL) et concentration en ADN (ng/μL) dans huit sections de seringues à gradient inoculées avec LW13 incubées pendant sept jours (cercles remplis ; les cercles ouverts montrent les valeurs des seringues à gradient stériles). Les données montrent la moyenne ± ET de trois expériences indépendantes avec trois répétitions techniques chacune. En ce qui concerne la teneur relative en polysaccharides, * indique une différence significative par rapport au témoin stérile à profondeur équivalente (test t hétéroscédastique bilatéral, α = 0,05). Pour les concentrations de protéines et d’ADN, * indique une différence significative par rapport à la section contenant la bande horizontale (ANOVA à un facteur avec analyse post hoc de Tukey-Kramer) ; N.S., non significatif. La figure a été modifiée à partir de Beals et al.6. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Méthodes de culture méthanotrophe
Les méthanotrophes sont étudiés depuis des décennies pour comprendre leur physiologie, leur comportement individuel et communautaire dans l’environnement naturel, et leur potentiel d’atténuation du méthane dans les applications industrielles. Tout au long de ces études, une grande partie des recherches menées ont été menées à l’aide de cultures planctoniques homogènes où le contexte spatial est perdu. L’écosystème modèle de seringue à gradient a été développé pour reproduire en laboratoire le contre-gradient méthane-oxygène caractéristique des habitats méthanotrophes naturels, permettant aux chercheurs d’étudier les méthanotrophes poussant dans un environnement qui ressemble davantage à l’endroit où ces organismes ont évolué.
Au cours des 30 dernières années, les chercheurs ont recréé le gradient compteur méthane-oxygène en laboratoire à l’aide de diverses méthodes, souvent dans le but principal d’isoler et de classer les méthanotrophes des consortiums mixtes oxydant le méthane. Ces méthodes peuvent être divisées en deux approches, toutes deux impliquant l’utilisation de chambres opposées de méthane et d’oxygène : suspendre un sol relativement non perturbé sur une membrane 16,17,18, ou inoculer de petites quantités de sol ou de culture bactérienne pure dans un milieu minimal dans de l’agarose 7,8,19. La méthode de la seringue à gradient décrite ici combine l’approche basée sur la seringue de Dedysh et ses collègues9 avec la culture de méthanotrophes provenant de travaux antérieurs d’Amaral et Knowles8, et de Schink et ses collègues7. Cette dernière méthode a jeté les bases de la culture de méthanotrophes dans un contre-gradient méthane-oxygène et a utilisé un flux continu de méthane et d’oxygène de chaque côté du bouchon d’agarose. Bien que cela fournisse un environnement plus constant, cette approche ajoute de la complexité à la configuration expérimentale et nécessite des sources de gaz dédiées.
En revanche, la seringue à gradient décrite ici repose sur un rinçage quotidien de la seringue pour fournir du méthane frais, un processus qui prend moins d’une minute par seringue, tout en fournissant un accès continu à l’oxygène atmosphérique grâce à un embout filtrant stérile en PTFE. Cette méthode plus simple pourrait permettre une adoption plus large de cet écosystème modèle pour l’étude des méthanotrophes dans un contexte spatialement résolu. Le protocole décrit détaille également les analyses chimiques et moléculaires qui peuvent être effectuées directement sur les bactéries incubées dans l’agarose semi-solide. Par conséquent, il n’est pas nécessaire d’exciser et de cultiver les bactéries en dehors de la matrice d’agarose avant l’analyse, ce qui permet de préserver les conditions de gradient de gaz au moment de l’échantillonnage.
Remarques sur le protocole
Étant donné que les bactéries sont cultivées à l’intérieur d’une seringue volumétrique en polypropylène, les chercheurs peuvent utiliser le piston de seringue qui l’accompagne pour segmenter avec précision et reproductibilité le bouchon d’agarose tout en maintenant l’intégrité spatiale de la matrice d’agarose qui reste dans le corps de la seringue. Sans la conception étanche à l’air inhérente à la seringue, les bouchons d’agarose devraient être retirés du corps de la seringue et tranchés, ce qui introduirait une incertitude quant au volume des segments d’agarose et libérerait des quantités non quantifiables de méthane et d’oxygène dissous dans l’atmosphère. L’extrusion d’agarose à l’aide d’une aiguille stérile simplifie la préparation des échantillons et permet d’homogénéiser les segments extrudés sans cisailler les cellules bactériennes. Cette méthode permet aux chercheurs de diviser chaque seringue à gradient inoculée en au moins huit segments d’agarose et d’effectuer des expériences parallèles sur des méthanotrophes se développant dans une gamme de concentrations d’oxygène et de méthane.
En optimisant l’extraction de l’ARN à partir d’agarose à haute teneur en polysaccharides, il a été constaté que des réactifs courants comme le thiocyanate de guanidium et le TRIzol conduisaient à la gélification de l’agarose, qui obstruait les colonnes de purification et résistait à la granulation par centrifugation. Les faibles rendements et la faible qualité de l’ARN étaient également préoccupants, car les grosses molécules de polysaccharides peuvent piéger les acides nucléiques, tandis que les petits polysaccharides peuvent coprécipiter avec l’ARN20. Au lieu de cela, un tampon d’extraction contenant le tensioactif cationique CTAB a été utilisé, qui solubilise les membranes lipidiques20 ; et NaCl, qui empêche la formation de complexes CTAB-acide nucléique et permet aux acides nucléiques de précipiter mais maintient les polysaccharides en solution21. Les RNases ont été dénaturées par l’inclusion de β-mercaptoéthanol dans le tampon CTAB. Pour l’expérience RNA-seq, une étape facultative de purification sur colonne a été incluse pour exclure les petits ARN (<200 nucléotides) avant la préparation de la banque.
Limites et considérations
Alors que la NMS et l’agarose fournissent une matrice moyenne minimale pour la culture des bactéries méthanotrophes, la seringue à gradient décrite ici ne recrée que les gradients de gaz des habitats méthanotrophes, mais pas d’autres gradients présents dans ces environnements tels que les métaux traces22, la salinité23 ou d’autres nutriments24. Il est possible que ces gradients puissent être ajoutés à un système similaire à l’avenir. De plus, le volume de la seringue (8 mL d’agarose) limite la biomasse totale par seringue, ce qui nécessite la mise en commun de plusieurs seringues pour certaines analyses (comme décrit à l’étape 5.16). Bien que la seringue à main segmente commodément l’agarose en aliquotes de 1 ml, sa taille limite également l’espace libre à environ 4 ml, limitant ainsi la quantité de méthane en vrac qui peut être stockée pour les microbes cultivés. Étant donné que les taux d’oxydation du méthane sont proportionnels au taux de croissance du méthanotrophe aérobie25, il est recommandé de réapprovisionner quotidiennement le méthane dans l’espace de tête. Bien que cela puisse encore entraîner des périodes de limitation du méthane, ces périodes sont reproductibles en laboratoire et imitent probablement les situations trouvées dans les environnements naturels.
Lors de l’utilisation de la seringue à gradient, la présence de polysaccharides d’agarose nécessite quelques ajustements aux dosages utilisés pour analyser les méthanotrophes cultivés dans ce système. Par exemple, les protocoles nécessitant le transfert de petits volumes d’agarose extrudée nécessitent plusieurs étapes de dilution avec une homogénéisation complète entre chaque dilution pour un pipetage précis. De plus, dans des cas tels que le dosage des polysaccharides où les polysaccharides inhérents à la matrice d’agarose réagissent avec le réactif acide sulfurique-phénol, l’inclusion d’un contrôle négatif d’agarose stérile et acellulaire est essentielle. Les premières tentatives d’atténuer ces problèmes en incluant l’enzyme hydrolysante de l’agarose, la β-agarase, ont échoué et ont introduit une variable inconnue dans les expériences biologiques. L’utilisation de plusieurs réplicats techniques, la dilution complète, l’homogénéisation et l’inclusion de témoins peuvent être utilisés pour atténuer la plupart des défis inhérents à la matrice d’agarose.
Applications
En plus des études sur une seule souche, la seringue à gradient peut soutenir la co-culture de plusieurs souches, et le sol peut être utilisé comme inoculum à la place de la culture bactérienne pure. La conception simple de l’écosystème du modèle de seringue à gradient se prête à la culture d’autres types de micro-organismes qui existent à l’interface entre les environnements anoxiques et oxiques en utilisant un substrat gazeux différent, tel que H2 ou CO, à la place du méthane. En résumé, l’utilisation d’un écosystème modèle simple et résolu spatialement permet aux chercheurs d’étudier la physiologie unique et les adaptations métaboliques des micro-organismes anoxiques-oxiques et peut être utilisée pour relier les gènes aux phénotypes des organismes.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par un financement de démarrage du département de chimie de l’Université de l’Utah et le NSF CAREER Award #2339190. Nous remercions les membres du Puri Lab pour les discussions utiles. Nous remercions Rachel Hurrell (Université de l’Utah) pour ses conseils initiaux concernant l’expérience de cytométrie en flux.
matériels
| Name | Company | Catalog Number | Comments |
| 1% Gas mix analytical standard | Supelco | 22561 | 1% each component in nitrogen: carbon monoxide, carbon dioxide, hydrogen, methane and oxygen |
| 100% Methane | Airgas | ME CP300 | chemically pure grade |
| 15 ppm Gas mix analytical standard | Supelco | 23470-U | 15 ppm each component in nitrogen: methane, ethane, ethylene, acetylene, propane, propylene, propyne, and n-butane |
| 1x Nitrate mineral salts | see CAS numbers below | Dissolve the following in Mili-Q water and autoclave: 0.2 g/L MgSO4·7H2O, 0.2 g/L CaCl2·6H2O, 1 g/L KNO3, and 30 μM LaCl3. Before use, add trace elements to a 1X final concentration and phosphate buffer (pH 6.8) to a final concentration of 5.8 mM. | |
| 23 G needle | BD Biosciences | 305194 | sterile, Luer-Lok |
| 500x Trace elements | see CAS numbers below | Dissolve the following in Milli-Q water: 1.0 g/L Na2-EDTA, 2.0 g/L FeSO4·7H2O, 0.8 g/L ZnSO4·7H2O, 0.03 g/L MnCl2·4H2O, 0.03 g/L H3BO3, 0.2 g/L CoCl2·6H2O, 0.6 g/L CuCl2·2H2O, 0.02 g/L NiCl2·6H2O, and 0.05 g/L Na2MoO·2H2O. | |
| 96 Well plate | CELLTREAT | 229596 | sterile |
| Acid phenol:chloroform:IAA (125:24:1) | Invitrogen | AM9720 | pH 4.5 |
| Agarose | Fisher Scientific | BP160 | molecular biology grade, CAS 9012-36-6 |
| Aluminum crimp seals | VWR | 30618-460 | 20 mm |
| Bead beater | Qiagen | 9003240 | TissueLyser III |
| Butyl rubber stopper | Chemglass Life Science | 50-143-854 | 20 mm, blue |
| Chloroform:isoamyl alcohol (24:1) | Millipore Sigma | 25666 | BioUltra, for molecular biology |
| Clark-type O2 microelectrode | Unisense | OX-500 | |
| DEPC-treated water | Thermo Scientific | R0601 | |
| DNase I (Ambion) | Invitrogen | AM2222 | |
| Flow cytometer | Beckman Coulter | CytoFLEX | |
| Gas chromatograph (flame ionization detection) | Agilent | 6890N | |
| Gastight analytical syringe | Hamilton | 81220 | 1750 TLL |
| Gastight analytical syringe needle | Hamilton | 7729-07 | 22 G, metal hub needle, 2 in, point style 5 |
| Gas-tight vials | Labco | 938W | Exetainer vial: 12 mL, round bottom |
| Glass culture tubes | Bellco Glass | 2048-00150 | 18 x 150 mm |
| LiCl precipitation solution (7.5 M) | Invitrogen | AM9480 | |
| One-way stopcock | VWR | MFLX30600-00 | inlet port: female luer, outlet port: male luer lock |
| Petri dish, square | Fisher Scientific | FB0875711A | 100 x 100 mm |
| Phosphate buffer, 0.2 M (pH 6.8) | see CAS numbers below | Dissolve the following in Milli-Q water and autoclave: 12.24 g/L KH2PO4, 26.29 g/L Na2HPO4 · 7H2O | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| PTFE syringe filter tip | Thermo Scientific | 03-050-469 | hydrophobic, pore size: 0.2 µm, diameter: 4 mm |
| Qubit 1x dsDNA High Sensitivity Assay Kit | Invitrogen | Q33230 | |
| Qubit 4 Fluorometer | Invitrogen | Q33238 | |
| RNA Clean & Concentrator-5 | Zymo Research | R1013 | |
| Serum stopper | Fisher Scientific | 03-340-302 | 20 mm |
| Syringe | BD Biosciences | 302995 | Luer-Lock, 10 mL, single use, sterile |
| Syringe pump | New Era Pump Systems Inc. | 1000-US | NE-1000 one channel programmable |
| SYTO9, propidium iodide, microspheres | Invitrogen | L34856 | LIVE/DEAD BacLight Bacterial Viability Kit |
| Zirconia/silica beads | BioSpec Products | 11079101z | 0.1 mm diameter |
| Chemical reagents | CAS number | ||
| CaCl2·6H2O | 7774-34-7 | ||
| CoCl2·6H2O | 7791-13-1 | ||
| Concentrated sulfuric acid | 7664-93-9 | ||
| CTAB, cetrimonium bromide | 57-09-0 | ||
| CuCl2·2H2O | 10125-13-0 | ||
| Ethanol | 64-17-5 | ||
| FeSO4·7H2O | 7782-63-0 | ||
| H3BO3 | 10043-35-3 | ||
| Isopropanol | 69-63-0 | ||
| KH2PO4 | 7778-77-0 | ||
| KNO3 | 7757-79-1 | ||
| LaCl3 | 10099-58-8 | ||
| MgSO4·7H2O | 10034-99-8 | ||
| MnCl2·4H2O | 13446-34-9 | ||
| Na2CO3, sodium carbonate | 497-19-8 | ||
| Na2-EDTA | 139-33-3 | ||
| Na2HPO4 · 7H2O | 7782-85-6 | ||
| Na2MoO·2H2O | 10102-40-6 | ||
| NaCl, sodium chloride | 7647-14-5 | ||
| NiCl2·6H2O | 7791-20-0 | ||
| Phenol (90% solution in water) | 108-95-2 | ||
| PVP40, polyvinylpyrrolidone | 9003-39-8 | ||
| Tris-HCl | 1185-53-1 | ||
| ZnSO4·7H2O | 7446-20-0 | ||
| β-Mercaptoethanol | 60-24-2 |
Références
- Brune, A., Frenzel, P., Cypionka, H. Life at the oxic-anoxic interface: microbial activities and adaptations. FEMS Microbiol Rev. 24 (5), 691-710 (2000).
- Auman, A. J., Stolyar, S., Costello, A. M., Lidstrom, M. E. Molecular characterization of methanotrophic isolates from freshwater lake sediment. Appl Environ Microbiol. 66 (12), 5259-5266 (2000).
- Whittenbury, R., Phillips, K. C., Wilkinson, J. F. Enrichment, isolation and some properties of methane-utilizing bacteria. J Gen Microbiol. 61 (2), 205-218 (1970).
- Koo, C., Rosenzweig, A. Biochemistry of aerobic biological methane oxidation. Chem Soc Rev. 50 (5), 3424-3436 (2021).
- Strong, P. J., Xie, S., Clarke, W. P. Methane as a resource: Can the methanotrophs add value. Environ Sci Technol. 49 (7), 4001-4018 (2015).
- Beals, D. G., Puri, A. W. Linking methanotroph phenotypes to genotypes using a simple spatially resolved model ecosystem. ISME J. 18 (1), wrae060 (2024).
- Bussmann, I., Rahalkar, M., Schink, B. Cultivation of methanotrophic bacteria in opposing gradients of methane and oxygen. FEMS Microbiol. Ecol. 56 (3), 331-344 (2006).
- Amaral, J. A., Knowles, R. Growth of methanotrophs in methane and oxygen counter gradients. FEMS Microbiol Lett. 126 (3), 215-220 (1995).
- Danilova, O. V., et al. A new cell morphotype among methane oxidizers: a spiral-shaped obligately microaerophilic methanotroph from northern low-oxygen environments. ISME J. 10 (11), 2734-2743 (2016).
- Ou, F., McGoverin, C., Swift, S., Vanholsbeeck, F. Absolute bacterial cell enumeration using flow cytometry. J Appl Microbiol. 123 (2), 464-477 (2017).
- Krause, S. M. B., et al. Lanthanide-dependent cross-feeding of methane-derived carbon is linked by microbial community interactions. Proc Natl Acad Sci USA. 114 (2), 358-363 (2017).
- Masuko, T., et al. Carbohydrate analysis by a phenol-sulfuric acid method in microplate format. Anal Biochem. 339 (1), 69-72 (2005).
- Felz, S., Al-Zuhairy, S., Aarstad, O. A., van Loosdrecht, M. C. M., Lin, Y. M. Extraction of structural extracellular polymeric substances from aerobic granular sludge. J Vis Exp. (115), e54534 (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), e3923 (2012).
- Costa, O. Y. A., Raaijmakers, J. M., Kuramae, E. E. Microbial extracellular polymeric substances: Ecological function and impact on soil aggregation. Front Microbiol. 9, 1636 (2018).
- Sinke, A. J. C., Cottaar, F. H. M., Buis, K., Keizer, P. Methane oxidation by methanotrophs and its effects on the phosphate flux over the sediment-water interface in a eutrophic lake. Microb Ecol. 24 (3), 259-269 (1992).
- Murase, J., Frenzel, P. A methane-driven microbial food web in a wetland rice soil. Environ Microbiol. 9 (12), 3025-3034 (2007).
- Reim, A., Lüke, C., Krause, S., Pratscher, J., Frenzel, P. One millimetre makes the difference: High-resolution analysis of methane-oxidizing bacteria and their specific activity at the oxic-anoxic interface in a flooded paddy soil. ISME J. 6 (11), 2128-2139 (2012).
- Rahalkar, M., Bussmann, I., Schink, B. Methylosoma difficile gen. nov., sp. nov., a novel methanotroph enriched by gradient cultivation from littoral sediment of Lake Constance. Int J Syst Evol Microbiol. 57 (5), 1073-1080 (2007).
- Wang, L., Stegemann, J. P. Extraction of high-quality RNA from polysaccharide matrices using cetlytrimethylammonium bromide. Biomaterials. 31 (7), 1612 (2010).
- Liyanage, N. M. N., Chandrasekara, B. C. H. W. M., Bandaranayake, P. C. G. A CTAB protocol for obtaining high-quality total RNA from cinnamon (Cinnamomum zeylanicum Blume). 3 Biotech. 11 (4), 201 (2021).
- Semrau, J. D., DiSpirito, A. A., Gu, W., Yoon, S. Metals and methanotrophy. Appl Environ Microbiol. 84 (6), e02289-e02317 (2018).
- Zhang, S., et al. Salinity significantly affects methane oxidation and methanotrophic community in Inner Mongolia lake sediments. Front Microbiol. 13, 1067017 (2023).
- Fox, A. L., Trefry, J. H. Nutrient fluxes from recent deposits of fine-grained, organic-rich sediments in a Florida estuary. Front Mar Sci. , (2023).
- He, L., et al. A methanotrophic bacterium to enable methane removal for climate mitigation. Proc Natl Acad Sci. 120 (35), e2310046120 (2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon