Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un flux de travail simple et rapide pour la quantification des structures externes de la drosophile adulte
Dans cet article
Résumé
Ici, nous présentons un flux de travail rapide et peu coûteux pour l’imagerie haute résolution des yeux de la drosophile adulte afin de quantifier les motifs et les défauts de croissance. Nous décrivons notre protocole de préparation d’échantillons par montage ponctuel, acquisition d’images haute résolution et analyse d’images.
Résumé
L’œil composé de la drosophile est un tissu précisément modelé qui a révélé les mécanismes moléculaires et les processus biologiques qui déterminent la morphogenèse. Il s’agit d’une structure simple d’yeux unitaires répétitifs, appelée ommatidies, qui est utilisée pour caractériser les interactions génétiques et les fonctions des gènes. Les mutations qui affectent l’architecture de l’œil peuvent être facilement détectées et analysées ; Par conséquent, ce système est fréquemment utilisé dans les institutions défavorisées. L’analyse phénotypique supplémentaire comprend souvent un microscope électronique à balayage (MEB) pour générer des images à fort grossissement adaptées à l’analyse quantitative. Cependant, les MEB sont coûteux et nécessitent des réactifs coûteux ; la préparation des échantillons s’étend sur plusieurs jours ; Et, souvent, ils ont besoin de personnel à temps plein pour la préparation des échantillons et l’entretien des instruments. Cela limite leur utilité dans les institutions sous-financées ou pendant l’austérité budgétaire. En entomologie, l’utilisation de la technologie d’imagerie numérique à haute résolution est une pratique courante pour l’identification et la caractérisation des espèces. Cet article décrit une méthode qui combine des stratégies et permet l’imagerie numérique à haute résolution des structures de la drosophile adulte et l’analyse quantitative à l’aide du logiciel ouvert ImageJ. Le flux de travail est une alternative rapide et conviviale pour les étudiants qui remédie aux limites des installations de recherche sous-financées et sous-financées grâce à une approche rentable et rapide de l’analyse phénotypique quantitative.
Introduction
Drosophila melanogaster est un puissant organisme modèle génétique qui est utilisé depuis des décennies pour élucider les voies de signalisation moléculaire et les comportements cellulaires. De nombreuses voies de signalisation conservées au cours de l’évolution, essentielles au développement multicellulaire, ont été identifiées pour la première fois et leur mécanisme d’action défini chez la drosophile. Environ 65 à 75 % de tous les gènes associés aux maladies humaines ont des orthologues chez la drosophile 1,2. L’œil de la drosophile adulte est un modèle important qui a permis des criblages génétiques impartiaux qui ont facilité la découverte d’importants gènes conservés impliqués dans des maladies humaines, notamment le cancer 3,4, la neurodégénérescence5 et les troubles métaboliques6.
L’œil de la drosophile est composé de ~800 yeux unitaires, appelés ommatidies, qui sont disposés avec précision dans un motif hexagonal sur la surface de l’œil adulte7. Chaque ommatidium est composé de huit neurones photorécepteurs qui occupent un emplacement distinct dans un trapèze asymétrique. Ceux-ci sont soutenus par quatre cellules coniques non neurales et deux cellules pigmentaires primaires qui sécrètent le cristallin et le pseudo-cône pour concentrer la lumière sur les rhabdomères sensibles à la lumière des neurones photorécepteurs. Les ommatidies voisines sont séparées par une seule rangée de cellules de réseau interommatidiennes, composée de cellules pigmentaires secondaires, de cellules pigmentaires tertiaires et de complexes de poils mécanosensoriels 8,9,10.
Les perturbations du développement oculaire sont visibles chez les yeux adultes sous la forme d’une augmentation ou d’une diminution de la taille de l’œil, d’une abondance ou d’une structure anormale des lentilles ou des poils, ou d’un « œil rugueux » où le motif hexagonal normalement invariant est perturbé de sorte qu’une rangée d’ommatidies ne peut plus être suivie à la surface de l’œil. Ces phénotypes peuvent être notés au niveau des tissus macroscopiques à l’aide de microscopes à dissection. L’analyse détaillée des phénotypes comprend traditionnellement la microscopie électronique à balayage suivie d’une analyse quantitative d’images11. Cependant, la microscopie électronique à balayage nécessite des instruments coûteux, des réactifs coûteux, une préparation d’échantillons qui s’étend sur des jours et, souvent, du personnel à temps plein.
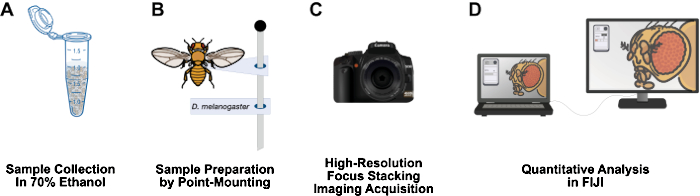
Figure 1 : Flux de travail pour l’imagerie des structures de la drosophile adulte. (A) Recueillir et fixer la drosophile adulte dans de l’éthanol à 70 %. (B) Préparer les échantillons pour l’imagerie par montage ponctuel et fixation sur des broches. (C) Acquérir des images haute résolution grâce à l’empilement et à l’intégration de la mise au point. (D) Quantifier les images à l’aide des FIDJI. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Cet article présente un flux de travail relativement peu coûteux, qui a un temps de préparation d’échantillon court, qui peut facilement être installé sur une paillasse de laboratoire de 3 pieds, qui ne nécessite pas de matières dangereuses et qui pourrait être un ajout à longue durée de vie aux laboratoires de recherche sur la drosophile (Figure 1). Le montage ponctuel est une technique entomologique utilisée pour sécher à l’air libre et conserver les petits insectes à corps mou, tels que la drosophile12. Cette méthode repose sur la combinaison d’objectifs de microscope avec des appareils photo reflex numériques haute résolution pour des grossissements efficaces de 10x à 1 000x. La profondeur de champ limitée inhérente à la macrophotographie est surmontée par l’empilement de mise au point : assembler une série d’images avec le plan focal se déplaçant à travers l’échantillon d’intérêt13. Cette méthode produit des images à haute résolution adaptées à la quantification des phénotypes et pourrait facilement être adaptée à d’autres structures d’intérêt, telles que l’aile, la patte, le thorax et l’abdomen. Le flux de travail d’analyse d’images utilise le programme gratuit d’analyse d’images FIJI (NIH ImageJ). Cette méthodologie rend la préparation d’échantillons, l’imagerie haute résolution et l’analyse accessibles aux étudiants de premier cycle et aux scientifiques des établissements défavorisés.
Protocole
1. Collecte et fixation de la drosophile adulte
- Mettez en place des croisements de drosophiles ou sélectionnez des souches et placez-les dans des flacons contenant de la nourriture pour mouches. Incuber les flacons à la température désirée (généralement 25 °C) jusqu’à ce que les mouches se soient développées et que les adultes se rapprochent (~10-14 jours à 25 °C).
- Anesthésez les mouches avec du CO2 et placez-les sur un tampon de CO2 .
- Triez les mouches à l’aide d’une plume et sélectionnez les individus ayant le phénotype souhaité (par exemple, des ailes droites). Fabriquez un trieur de mouches à plumes en taillant une plume d’oie pour qu’elle s’insère dans l’extrémité effilée d’une pipette sérologique de 1 ml.
- Préparez un tube de microcentrifugation de 1,7 mL avec 1 mL d’éthanol à 70 %. Placez les mouches sélectionnées dans le tube de microcentrifugation et mettez-les sur de la glace. Conservez les tubes de microcentrifugation à 4 °C pendant la nuit (Figure 2A).
REMARQUE : Ne conservez pas les mouches dans de l’éthanol pendant plus de 24 heures. Le stockage à long terme des mouches dans de l’éthanol à 70 % entraînera une perte de pigment pour les yeux et le corps.
2. Préparation de l’échantillon par montage ponctuel
REMARQUE : Les drosophiles sont des insectes à corps mou qui deviennent cassants et s’effondrent lorsqu’ils sont séchés à l’air ; Par conséquent, ce protocole exige que les échantillons soient imagés le jour même de leur montage. Travaillez en petits groupes de ~5 mouches à la fois pour éviter la perte d’échantillon. Augmentez le nombre d’échantillons dans un ensemble en fonction de l’efficacité. Les échantillons qui nécessitent plus de temps avant l’imagerie peuvent être déshydratés par une série de concentrations croissantes d’hexaméthyldisilazane (HMDS)14.
- Découpez de petites pointes triangulaires (7,1 mm x 2,7 mm) dans du papier cartonné d’archives de 65 lb à l’aide d’une perforatrice spécialisée. Préparez les pointes en pliant la pointe (la plus étroite 25 %) à un angle de 90° à l’aide d’une pince à pointe fine Dumont #5 (figure 2B).
- À l’aide d’une pince à pointe fine Dumont #5, retirez les mouches des tubes de la microcentrifugeuse (étape 1.4). Épongez doucement les mouches avec un tissu de laboratoire non pelucheux pour éliminer l’excès d’éthanol. Positionnez chaque mouche sur son côté gauche sur une fiche sous un microscope à dissection.
REMARQUE : Retirez les mouches du tube en les tenant à une structure anatomique qui n’est pas la zone d’intérêt - lors de l’imagerie de la tête, nous tenons des échantillons par l’aile ou la patte. Ne tenez pas les échantillons par l’abdomen, car cette structure est utilisée pour coller la mouche sur la pointe de la carte. - Préparez la colle à peau en ajustant sa consistance à la viscosité souhaitée. Mélangez 1 à 2 gouttes de colle de peau avec 1 à 2 gouttes d’eau déminéralisée (DI) et mélangez avec une pipette de transfert sur une fiche. Prenez une pointe de carte préparée à l’extrémité large à l’aide d’une pince et placez une petite quantité de colle diluée sur la pointe pliée de la pointe en la tamponnant dans le mélange colle-eau (Figure 2C).
REMARQUE : La colle doit être étalable mais pas coulante. - Appliquez l’extrémité pliée de la pointe sur la face antérieure de l’abdomen droit, autour des segments abdominaux 2 et 3 (figure 2C). Avant que la colle ne sèche, effectuez de légers ajustements sur la mouche de sorte que l’axe antéro-postérieur de la mouche soit perpendiculaire à la pointe courbée de la pointe.
- Insérez une goupille de montage n° 3 dans l’extrémité large de la pointe de la carte (Figure 2D) et fixez-la à un bloc d’épingle à insectes (Figure 2E). Étiquetez chaque épingle ou rangée d’épingles avec le génotype correspondant.

Figure 2 : Préparation de l’échantillon. (A) Les drosophiles adultes sont triées en fonction de marqueurs phénotypiques et rassemblées dans des tubes de microcentrifugation étiquetés contenant 70 % d’éthanol sur de la glace. Les mouches sont stockées à 4° C pendant la nuit. (B) Les pointes de la carte en papier sont préparées en pliant l’extrémité étroite à 90° du reste de la carte à l’aide d’une paire de pinces #5. (C) Les mouches sont récupérées des tubes et brièvement laissées sécher à l’air. La colle de peau est appliquée sur la petite extrémité pliée de la pointe de la carte préparée et collée sur la mouche adulte au niveau des segments abdominaux 2-3. (D) Les spécimens sont montés, avec une étiquette d’identification, sur une épingle à insectes en acier inoxydable #3. (E) Les échantillons épinglés sont stockés sur une carte d’échantillonnage jusqu’à ce qu’ils soient prêts pour l’acquisition de l’image. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Acquisition d’images par empilement de mise au point haute résolution
- Acquérez des photographies haute résolution d’yeux de mouches à l’aide d’un système d’imagerie d’empilement de mise au point assemblé et personnalisé.
- Prenez des photos avec un boîtier d’appareil photo reflex numérique doté d’un téléobjectif 70-200 mm connecté à un objectif Apo Microscope 20x via un adaptateur d’objectif 77 mm.
- Assurez-vous que l’échantillon est éclairé par un flash à travers un diffuseur (Figure 3).
- Contrôlez le positionnement Z à l’aide d’un contrôleur Stackshot et d’un rail macro.
- Connectez l’appareil photo, le flash et la platine motorisée à un trépied en aluminium anodisé robuste.
- Positionnez chaque échantillon monté sur un cardan de platine universel, la tête orientée de manière à ce que l’œil soit tourné vers l’objectif. Ajustez la position de la tête en déplaçant doucement la tête avec la pince.
ATTENTION : Des ajustements importants et rapides peuvent entraîner une décapitation accidentelle. - Avec l’appareil photo connecté à un ordinateur portable, ajustez les paramètres d’acquisition dans le logiciel. Photographiez des spécimens avec un grossissement de 20x avec les paramètres suivants : puissance du flash 1/32, vitesse d’obturation 1/200, ouverture F2.8 et ISO 400. Assurez-vous que toutes les fonctions de mise au point automatique et de stabilisation d’image sont désactivées.
REMARQUE : ces paramètres équilibrent l’éclairage optimal du flash, la vitesse d’obturation et la profondeur de champ. Ils devraient être ajustés pour d’autres grossissements et/ou combinaisons de lentilles. - Définissez l’emplacement d’enregistrement de la pile d’images résultante (10 à 50 images) dans le dossier de fichiers souhaité. Assurez-vous d’une capacité de stockage suffisante pour les images (~8,5 Mo par image).
- Ajustez les paramètres de la pile de mise au point sur l’unité de commande Stackshot en mode Distance automatique .
- Réglez la taille du pas sur 5 μm et calculez le nombre de pas en réglant les positions de début et de fin de la pile de mise au point.
- Visualisez l’échantillon en mode LiveView et avec l’appareil photo en mode de prise de vue automatique pour identifier les positions de démarrage et d’arrêt.
- Déplacez le rail de manière à ce que la partie la plus proche de l’échantillon soit nette (position de départ définie), puis déplacez-vous jusqu’à l’endroit où l’élément d’intérêt le plus éloigné est net (position finale définie).
- Remettez l’appareil photo en mode de prise de vue manuel et démarrez l’acquisition d’images à partir de l’unité de commande Stackshot.
REMARQUE : Le temps d’acquisition de l’image dépend de la taille de l’échantillon. Plus la profondeur de champ nécessaire pour les grands échantillons est grande, plus il y a de coupes incluses dans la pile d’images, ce qui prolongera le temps d’acquisition global. - Ouvrez les fichiers dans le logiciel d’empilement de focus référencé. Générez une image empilée en cliquant sur Pile | Aligner et empiler tout (PMax).
- Enregistrez l’image finale sur le disque dur de l’ordinateur en tant que fichier .tif en cliquant sur Fichier | Enregistrer l’image de sortie.
REMARQUE : En fonction de la résolution du fichier d’image empilé et du nombre d’échantillons imagés, des disques durs externes de grande taille (1 To) peuvent être nécessaires pour la sauvegarde des images. Dans ce protocole, les images empilées font environ 100 Mo chacune avant d’être compressées.

Figure 3 : Acquisition d’images. (A) Appareil d’imagerie avec des pièces étiquetées comme suit : a) Corps d’appareil photo reflex numérique ; b) téléobjectif ; c) Objectif et adaptateur Apo Microscope 20x ; d) Flash ; e) Diffuseurs à lentille et à dôme ; f) Contrôleur Stackshot, Macro Rail et Platine rotative ; g) Cardan de scène universel ; h) Trépied. (B) Appareil d’imagerie avec diffuseur de lumière en place. (C) Gros plan de l’échantillon monté en position pour l’imagerie. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Flux de travail d’analyse FIJI pour calculer le contour des yeux adultes
- Pour l’analyse d’images, procurez-vous le logiciel FIJI15 sur le site Web de ImageJ.net.
- Choisissez des images pour l’analyse où l’œil est centré et aligné avec un éclairage adéquat et un flou périphérique minimal, indiquant un alignement correct avec la caméra.
- Calibrez l’échelle de l’image.
- Téléchargez l’image de la barre d’échelle pour un grossissement de 20x qui correspond à 500 μm. Alternativement, au moment de l’acquisition de l’image, photographiez une règle en utilisant les mêmes paramètres. Ouvrez l’image dans FIJI Software.
- Mesurez la longueur de la barre d’échelle (Figure 4A). Utilisez l’outil Ligne droite pour tracer exactement la ligne. Cliquez sur Analyser | Mesurer. Cette distance de pixel est équivalente à 500 μm (Figure 4B).
- Calculez les pixels par micron. Utilisez cette option pour convertir les mesures de pixels en mesures micrométriques.
- Ouvrez le fichier d’image empilée dans FIJI (Figure 4C).
- Sélectionnez Loupe dans la barre d’outils pour agrandir la zone de mise au point. Essayez de remplir l’écran avec l’œil et la cuticule de la tête qui l’entoure immédiatement (Figure 4D).
- Sélectionnez Sélectionner un outil à main levée dans la barre d’outils. Délimitez la région rétinienne le plus près possible, en suivant les contours de la rangée la plus externe d’ommatidies (Figure 4E). Pour supprimer une partie de la sélection, maintenez le bouton d’option enfoncé et sélectionnez les pixels à supprimer. Pour ajouter à la sélection, maintenez les boutons Option et Maj enfoncés et sélectionnez les pixels à ajouter.
- Pour calculer la surface, sélectionnez Analyser | Mesurez à partir du menu supérieur (Figure 4F). Une nouvelle fenêtre apparaîtra avec les paramètres area, mean, minimum et maximum . Copiez et collez ces données dans une feuille de calcul pour la documentation et la conversion des mesures en pixels en mesures micrométriques.
- Effectuer des analyses statistiques appropriées.
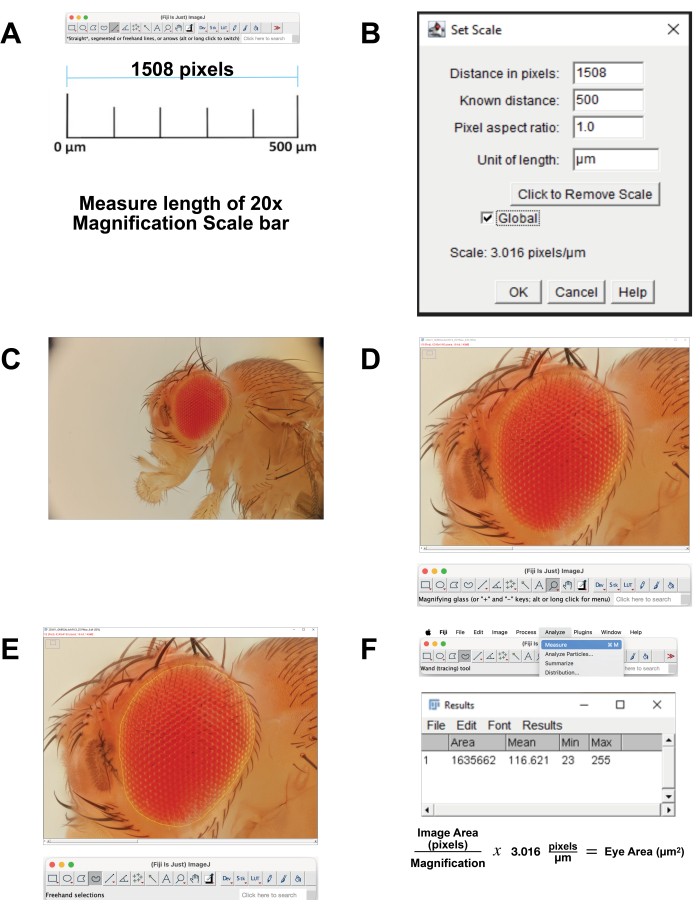
Figure 4 : Analyse de l’image en FIDJI. (A) Mettre à l’échelle l’image originale. Téléchargez l’image d’étalonnage et mesurez la longueur de la barre de 500 μm. (B) Ajustez la mise à l’échelle à l’aide de la fonction Set Scale. (C) Ouvrez l’image empilée. (D) Agrandissez l’image de sorte que l’œil soit centré et presque plein écran. (E) Utilisez l’outil Sélection à main levée pour tracer le contour de l’œil à la frontière entre la rangée la plus externe d’ommatidies et la cuticule environnante. (F) Mesurer la surface dans la région sélectionnée est calculé en cliquant sur Analyser | Mesurer | Aire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
L’œil de la drosophile est un excellent système modèle pour l’étude de la structure des tissus, du contrôle de la croissance et de la mort cellulaire. Nous avons récemment publié une étude sur la façon dont le pH intracellulaire (pHi) influence la croissance des tissus. Tout d’abord, nous avons établi un système génétique où la surexpression de l’échangeur sodium-proton DNhe2 (l’orthologue de NHE1 chez les mammifères) dans l’œil en développ...
Discussion
Nous décrivons ici une méthode de préparation d’échantillons, d’imagerie à haute résolution et d’analyse des structures de la drosophile adulte. L’œil de la drosophile est un système modèle génétiquement traitable qui a fourni des informations essentielles sur les mécanismes moléculaires sous-jacents à des maladies, notamment le cancer19, la neurodégénérescence20 et les maladies métaboliques
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Les auteurs tiennent à remercier les membres du laboratoire de pH de Grillo-Hill pour leurs discussions et leur soutien. Nous remercions Tim Andriese, Randy Kirschner, Kitty (Ngoc-Huong) Nguyen, Marco Parent, Jonny Shaloub et Librado Veliz pour leur excellent soutien technique. Ce travail a été soutenu par les bourses NIH SC3GM132049 et 1R16GM153640 (BKGH), une bourse de recherche professeur-étudiant en biotechnologie de la CSU (LM et BKGH), et des fonds de démarrage du College of Science et du Département des sciences biologiques de l’Université d’État de San José (FJL). Une mention spéciale va à Bernd Becker pour sa débrouillardise et son aide tout au long de ce processus. Nous remercions la communauté BioIcons (https://bioicons.com/) de nous avoir fourni des icônes de haute qualité pour nos figurines et en particulier à Servière pour l’icône de la pipette, et DBCLS pour les icônes de drosophile, de forceps et de microscope électronique de bureau utilisées dans Figure 1 et Figure 2, qui sont sous licence CC-BY 4.0 Unported. Nous remercions également la communauté SciDraw (https://scidraw.io/) de fournir des icônes de haute qualité pour nos figurines, en particulier Diogo Losch De Oliveira (doi.org/10.5281/zenodo.3925953), qui sont sous licence Creative Commons 4.0 (CC-BY).
matériels
| Name | Company | Catalog Number | Comments |
| 1 mL serological pipette | ThermoFisher Scientific | 170353N | |
| 1.7 mL microcentrifuge tubes | Genesee Scientific | 24-282LR | |
| 20x Apo Microscope Objective | Mitutoyo Corp. | 378-804-3 | |
| Archival 65 lb cardstock | Neenah, Inc. | 91901 | |
| Canon EF 70-200 mm USM II telephoto lens | Canon | 3044C002 | |
| Canon EOS 6D Mark II DSLR Camera Body | Canon | 1897C002 | |
| Diffuser Dome | Macroscopic Solutions | PA-DIF-GIM-SM | |
| Diffuser for Mitutoyo M Plan APO Objectives | Macroscopic Solutions | mitutoyo-diffusers | |
| Drosophila vials and plugs | Genesee Scientific | 32-117BF | |
| Dumont #5 fine-tip forceps | Fisher Scientific | NC9889584 | |
| Goose feathers | Amazon | B01CMMJI6U | |
| Heavy-Duty Anodized Aluminum Tripod | Really Right Stuff, LLC | TFA-32G | |
| Kimwipes | Fisher Scientific | 06-666A | lint-free lab tissue |
| Levenhuk M1000 Plus Digital Camera | Levenhuk | 70358 | |
| No. 3 mounting pin | Indigo Instruments | 33414-3 | |
| Nutri-Fly Bloomington Drosophila media | Genesee Scientific | 66-113 | fly food |
| Point-Punch | M.C. Mieth Manufacturing, Inc. | 448Detail | |
| Screwknob Clamp | Really Right Stuff, LLC | SK-Clamp | For attaching the macro rail to the tripod |
| Stackshot Controller and Macro Rail | Cognisys Inc. | ST3X_100_BUNDLE | |
| Step-down Ring Adapter | RAF Camera | 763461174207 | Lens adapter to connect the microscope objective to the camera lens |
| Titebond Glue | Franklin International | 5013 | |
| Yongnuo YN-24-EX Macro Twin Lite Flash | Shenzhen Yongnuo Photographic Equipment Co. | YN-24EX | |
| Software | |||
| Canon EOS Utility (v. 3.16.1). | Canon | acquisition software | |
| FIJI | National Institutes of Health | Fiji is released as open source under the GNU General Public License. FIJI Version 2.14.0/1.54f | |
| GraphPad Prism | GraphPad Software, Boston, Massachusetts USA | Prism Version 10.3.1 | |
| Zerene Stacker (v.1.04) | Zerene Systems, LLC | Focus Stacking Software |
Références
- Rubin, G. M., et al. Comparative genomics of the eukaryotes. Science. 287 (5461), 2204-2215 (2000).
- Ugur, B., Chen, K., Bellen, H. J. Drosophila tools and assays for the study of human diseases. Dis Model Mech. 9 (3), 235-244 (2016).
- Hodgson, J. A., Parvy, J. -. P., Yu, Y., Vidal, M., Cordero, J. B. Drosophila larval models of invasive tumorigenesis for in vivo studies on tumour/peripheral host tssue interactions during cancer cachexia. Int J Mol Sci. 22 (15), 8317 (2021).
- Lam Wong, K. K., Verheyen, E. M. Metabolic reprogramming in cancer: mechanistic insights from Drosophila. Dis Model Mech. 14 (7), dmm048934 (2021).
- Bonini, N. M. A perspective on Drosophila genetics and its insight into human neurodegenerative disease. Front Mol Biosci. 9, e1060796 (2022).
- Drummond-Barbosa, D., Tennessen, J. M. Reclaiming Warburg: using developmental biology to gain insight into human metabolic diseases. Development. 147 (11), dev189340 (2020).
- Ready, D. F., Hanson, T. E., Benzer, S. Development of the Drosophila retina, a neurocrystalline lattice. Dev Biol. 53 (2), 217-240 (1976).
- Johnson, R. I. Hexagonal patterning of the Drosophila eye. Dev Biol. 478, 173-182 (2021).
- Weasner, B. P., Kumar, J. P. The early history of the eye-antennal disc of Drosophila melanogaster. Genetics. 221 (1), iyac041 (2022).
- Pichaud, F., Casares, F. Shaping an optical dome: The size and shape of the insect compound eye. Semin Cell Dev Biol. 130, 37-44 (2022).
- Oster, I. I., Crang, R. E. Scanning electron microscopy of Drosophila mutant and wild type eyes. Trans Am Microsc Soc. 91 (4), 600-602 (1972).
- Gibb, T. J., Oseto, C. . Insect collection and identification: Techniques for the field and laboratory. , (2019).
- Mertens, J., Roie, M. V., Merckx, J., Dekoninck, W. The use of low cost compact cameras with focus stacking functionality in entomological digitization projects. ZooKeys. 712, 141-154 (2017).
- Brown, B. V. A further chemical alternative to critical-point-drying for preparing small (or large) flies. Fly Times. 11, 10 (1993).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Method. 9 (7), 676-682 (2012).
- Grillo-Hill, B. K., Choi, C., Jimenez-Vidal, M., Barber, D. L. Increased H+ efflux is sufficient to induce dysplasia and necessary for viability with oncogene expression. eLife. 4, e03270 (2015).
- White, K. A., Grillo-Hill, B. K., Barber, D. L. Cancer cell behaviors mediated by dysregulated pH dynamics at a glance. J Cell Sci. 130 (4), 663-669 (2017).
- Peralta, J., et al. Drosophila Nhe2 overexpression induces autophagic cell death. Mol Biol Cell. 35 (7), br13 (2024).
- Munnik, C., Xaba, M. P., Malindisa, S. T., Russell, B. L., Sooklal, S. A. Drosophila melanogaster: A platform for anticancer drug discovery and personalized therapies. Front Genet. 13, 949241 (2022).
- Nitta, Y., Sugie, A. Studies of neurodegenerative diseases using Drosophila and the development of novel approaches for their analysis. Fly. 16 (1), 275-298 (2022).
- Pletcher, R. C., et al. A genetic screen using the Drosophila melanogaster TRiP RNAi collection to identify metabolic enzymes required for eye development. G3: Genes|Genomes|Genetics. 9 (7), 2061-2070 (2019).
- Bangi, E., et al. A personalized platform identifies trametinib plus zoledronate for a patient with KRAS-mutant metastatic colorectal cancer. Sci Adv. 5 (5), eaav6528 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon