Coltura a lungo termine di fette organotipiche del midollo spinale di topo come piattaforma per la convalida del trapianto di cellule nella lesione del midollo spinale
In questo articolo
Riepilogo
In questo articolo, forniamo un metodo riproducibile per generare e mantenere a lungo termine fette organotipiche del midollo spinale trapiantate con cellule staminali neurali come modello ex vivo per testare terapie di sostituzione cellulare.
Abstract
Mancano ancora cure risolutive per le lesioni del midollo spinale (SCI), a causa della complessa fisiopatologia. Uno degli approcci rigenerativi più promettenti si basa sul trapianto di cellule staminali per sostituire il tessuto perso e promuovere il recupero funzionale. Questo approccio dovrebbe essere ulteriormente esplorato meglio in vitro ed ex vivo per verificarne la sicurezza e l'efficacia prima di procedere con sperimentazioni sugli animali più costose e dispendiose in termini di tempo. In questo lavoro, mostriamo la creazione di una piattaforma a lungo termine basata su fette organotipiche del midollo spinale (SC) di topo trapiantate con cellule staminali neurali umane per testare terapie di sostituzione cellulare per le SCI.
Le colture organotipiche SC standard vengono mantenute per circa 2 o 3 settimane in vitro. Qui descriviamo un protocollo ottimizzato per la manutenzione a lungo termine (≥30 giorni) fino a 90 giorni. Il terreno utilizzato per la coltura a lungo termine di fette di SC è stato ottimizzato anche per il trapianto di cellule staminali neurali nel modello organotipico. Le cellule staminali neuroepiteliali umane derivate da SC (H-SC-NES) portatrici di un reporter di proteina fluorescente verde (GFP) sono state trapiantate in fette di SC di topo. Trenta giorni dopo il trapianto, le cellule mostrano ancora l'espressione di GFP e un basso tasso di apoptosi, suggerendo che l'ambiente ottimizzato ha sostenuto la loro sopravvivenza e integrazione all'interno del tessuto. Questo protocollo rappresenta un solido riferimento per testare in modo efficiente le terapie di sostituzione cellulare nel tessuto SC. Questa piattaforma consentirà ai ricercatori di eseguire un pre-screening ex vivo di diverse terapie di trapianto cellulare, aiutandoli a scegliere la strategia più appropriata prima di procedere con gli esperimenti in vivo .
Introduzione
La lesione traumatica del midollo spinale (SCI) ha conseguenze fisiche, psicologiche ed economiche devastanti per i pazienti e gli operatori sanitari1. Molti tentativi sono stati fatti per promuovere la rigenerazione assonale nella lesione midollare con diversi approcci 2,3,4 e alcuni effetti benefici sono stati dimostrati dalla formazione di relè neuronali tra neuroni prossimali e distali nel sito di lesione attraverso terapie di sostituzione cellulare. L'interesse per le terapie cellulari è ancora in crescita5 poiché le cellule trapiantate possono svolgere molti ruoli, tra cui fornire supporto trofico, modulazione immunitaria, rigenerazione dei circuiti neurali persi attraverso l'induzione della plasticità, sostituzione cellulare e rimielinizzazione degli assoni6.
Recentemente, lo sforzo principale nel campo si è concentrato sulle cellule staminali/progenitrici neurali umane (NSC/NPCs)7. Diversi studi suggeriscono che le NSC/NPC modulano la risposta degli astrociti8, promuovono la secrezione di fattori prorigenerativi 9,10 e sostituiscono le cellule neuronali mancanti nella LMC11,12. Tuttavia, gli studi che supportano la differenziazione delle cellule trapiantate in neuroni funzionali sono ancora scarsi. Inoltre, la sopravvivenza e la differenziazione delle cellule trapiantate nel midollo spinale danneggiato (SC) sono basse13, probabilmente perché le cellule trapiantate impiegano diverse settimane, anche mesi, per differenziarsi in vivo. Inoltre, gli studi attuali non hanno completamente chiarito molti aspetti biochimici, molecolari, cellulari e funzionali delle terapie di sostituzione cellulare. In questo contesto, sono necessari modelli semplici, veloci ed economici per studiare i meccanismi dell'attecchimento cellulare, la capacità delle cellule trapiantate di proliferare, differenziarsi in tipi specifici o sottopopolazioni di cellule e formare sinapsi con i neuroni residenti.
L'integrazione degli studi istologici nella registrazione elettrofisiologica e nella profilazione del trascrittoma e del proteoma è necessaria per una piena comprensione della cascata molecolare che si verifica dopo il trapianto di cellule. Questo sicuramente accelererà la progettazione e la convalida di nuove terapie di sostituzione cellulare in modelli preclinici e studi clinici. Infatti, fino ad oggi, l'uso di roditori, animali di grossa taglia e primati non umani è stato utile per chiarire molti processi cellulari dopo il trapianto14. Tuttavia, a causa dell'alto costo, dell'alto impatto etico, nonché della complessità dell'organismo, il loro utilizzo spesso non è semplice o non è adeguato a svelare i processi biochimici e molecolari. Inoltre, possono presentare molti svantaggi correlati a differenze biologiche, sia interspecie (metabolismo) che variabilità intraspecie (sesso, età). Questi fattori, insieme a fattori esterni come situazioni di stress, potrebbero alterare l'esito di un esperimento e la loro prevedibilità in termini di traduzione terapeutica nell'uomo 15,16,17.
Pertanto, molti gruppi impiegano colture cellulari 2D in vitro e fette organotipiche ex vivo (colture ex vivo) oltre ai modelli animali. La coltura cellulare 2D è il sistema più comunemente utilizzato per lo studio di processi biologici specifici a livello di singola cellula e/o popolazione cellulare. Tuttavia, le colture cellulari monostrato non riflettono la complessità che si trova in un intero organismo. La mancanza di strutture tissutali e di ambiente fisiologico non consente ai sistemi di coltura 2D di emulare completamente gli aspetti strutturali, morfologici e funzionali chiave del tessuto studiato 18,19,20.Le colture organotipiche possono superare alcuni di questi problemi. I modelli organotipici si basano sull'espianto di un frammento di un tessuto o di un organo e sul suo mantenimento ex vivo per un periodo limitato21,22. In particolare, vengono generate fette di tessuto espiantato con uno spessore preciso che permette ai nutrienti di raggiungere facilmente quasi tutte le cellule delle fette. Possono essere generati da varie regioni del sistema nervoso centrale, come l'ippocampo, l'ipotalamo, il cervelletto, il talamo, la corteccia cerebrale, la substantia nigra e lo striato e il midollo spinale23. Le colture organotipiche conservano l'architettura tissutale, la distribuzione spaziale delle cellule, la diversità cellulare e l'ambiente (cioè la composizione della matrice extracellulare) dell'organo di origine. Inoltre, conservano l'attività neurale originale, le connessioni tra le cellule e, in particolare, i circuiti a breve distanza dopo l'espianto.
Questi aspetti forniscono alcuni vantaggi per le colture ex vivo rispetto sia alle colture monostrato che ai modelli animali. Mantengono le caratteristiche tissutali chiave riscontrate in vivo ma con la riduzione dei costi e la possibilità di eseguire diversi tipi di esperimenti molecolari, cellulari e funzionali con un'accurata regolazione dei parametri ambientali della coltura 24,25,26,27,28,29. Le fette organotipiche possono anche essere sfruttate per sviluppare modelli per diversi disturbi neurologici assomigliando alle caratteristiche istopatologiche chiave di condizioni specifiche30. Inoltre, la conservazione dell'ambiente tissutale multicellulare originale li rende piattaforme appropriate per lo screening di farmaci e per testare molecole e materiali neuroprotettivi e neuro-rigenerativi.
In questo lavoro, proponiamo l'uso di colture organotipiche di SC come modello per ottimizzare i trapianti di NSC. Questo non è banale poiché sono necessarie condizioni di coltura ottimali per garantire la sopravvivenza sia dell'ospite (tessuto SC) che del trapianto (NSC) per settimane. Diversi gruppi di ricerca hanno innestato in colture organotipiche, derivate dal cervello e derivate da SC, vari tipi di cellule. La maggior parte dei lavori ha mostrato il trapianto di cellule staminali mesenchimali31,32,33, cellule di rivestimento olfattivo34 o NSC 35,36,37,38,39,40 e ha valutato le interazioni delle cellule trapiantate con le cellule ospiti, la sopravvivenza dell'intero sistema e se le cellule trapiantate si differenziano in neuroni o cellule simili ai neuroni all'interno dell'ambiente tissutale ex vivo 32,33,41. Alcuni di loro hanno valutato il potenziale rigenerativo delle cellule dopo il trapianto, osservando la loro crescita assonale all'interno del tessuto 37,40,41, la capacità mielinizzante dei precursori trapiantati di oligodendrociti 42, la migrazione delle cellule trapiantate nel tessuto ospite43 e se le cellule trapiantate rilasciavano fattori che spingevano verso un ambiente pro-rigenerativo31. Una limitazione degli studi attuali è che non esplorano l'attecchimento per un periodo a lungo termine.
Considerando che le NSC sembrano richiedere diverse settimane per differenziarsi in vivo44,45, questo studio si concentra su come generare e mantenere fette di SC di topo a lungo termine (≥30 giorni) per un massimo di 90 giorni. Si è scoperto che le fette conservano la loro struttura anatomica originale e mantengono un tasso di apoptosi basso e stabile nel tempo e un'elevata vitalità cellulare. Abbiamo osservato l'espressione diffusa dei marcatori neuronali RNA binding fox-1 homolog 3 (RBFOX3) e neurofilament light chain (NFL), con quest'ultimo che mostra una tendenza crescente di germinazione assonale attorno alle fette nel tempo, attestando la loro condizione di salute. Inoltre, abbiamo trapiantato con successo nelle SC-slice cellule staminali neuroepiteliali umane derivate da FGP (H-SC-NES) nelle prime fasi del differenziamento neuronale. L'innesto di NSC è stato mantenuto per 30 giorni dopo il trapianto e le cellule hanno mostrato espressione di GFP per tutto il periodo in coltura. Anche il tasso di apoptosi delle cellule al giorno post-trapianto (DPT) 30 è risultato essere in linea con il valore del tasso di apoptosi osservato a DPT 7 nelle stesse cellule40. Le cellule sembravano innestarsi nell'ambiente tissutale e sopravvivevano fino a diverse settimane.
In sintesi, i nostri dati dimostrano che è possibile mantenere in coltura fette organotipiche di SC per 3 mesi senza compromettere la loro citoarchitettura originale e l'ambiente tissutale. Soprattutto, possono essere sfruttati per testare terapie cellulari prima di procedere con un esperimento in vivo , riducendo così i costi e i tempi sperimentali. Qui, illustriamo in dettaglio tutti i passaggi per generare fette organotipiche di SC di topo e come mantenerle per periodi di lungo periodo (≥30 giorni). Inoltre, spieghiamo in modo approfondito come eseguire il trapianto di NPC nelle fette e come mantenerle per l'analisi a valle.
Protocollo
Le procedure sugli animali sono state eseguite nel rigoroso rispetto dei protocolli approvati dal Ministero della Salute e dal Comitato Etico locale dell'Università di Pisa, in conformità alla Direttiva 2010/63/UE (licenza di progetto n. 39E1C. N.5Q7 uscito il 30/10/2021). I topi C57BL/6J sono stati tenuti in un ambiente regolato (23 ± 1 °C, 50 ± 5% di umidità) con un ciclo luce-buio di 12 ore con cibo e acqua ad libitum.
Tutto il lavoro relativo alle cellule h-SC-NES è stato eseguito secondo le linee guida NIH per l'acquisizione e la distribuzione di tessuti umani per scopi di ricerca biomedica e con l'approvazione dei Comitati di Indagine Umana e dei Comitati Etici Istituzionali di ciascun istituto da cui sono stati ottenuti i campioni. L'approvazione finale è stata ottenuta dal Comitato per la Bioetica dell'Università di Pisa (Revisione n. 29/2020). Gli esemplari umani anonimizzati sono stati forniti dalla sovvenzione congiunta MRC/Wellcome Trust (099175/Z/12/Z), Human Developmental Biology Resource (www.hdbr.org). È stato ottenuto il consenso informato appropriato e tutte le informazioni non identificative disponibili sono state registrate per ciascun campione. Il tessuto è stato manipolato in conformità con le linee guida etiche e le normative per l'uso a scopo di ricerca del tessuto cerebrale umano stabilite dal NIH (http://bioethics.od.nih.gov/humantissue. html) e dalla Dichiarazione WMA di Helsinki (http://www.wma.net/en/30publications/10policies/b3/index.html).
1. Preparazione di soluzioni e attrezzature per l'isolamento e la coltura del midollo spinale (SC)
- Soluzione di rivestimento per inserti a membrana
- Preparare la soluzione di rivestimento (Tabella 1): una soluzione acquosa con 0,1 mg di mL-1 di collagene, 0,01 mg di mL-1 di poli-L-lisina e 0,01 mg di mL-1 di laminina.
- Posizionare ciascun inserto a membrana in una piastra da 35 mm o in una piastra a 6 pozzetti.
- Aggiungere alla parte superiore della membrana 1 mL di soluzione di rivestimento: incubare la soluzione per 4 ore a temperatura ambiente (RT); quindi, rimuoverlo e lasciare asciugare l'inserto della membrana per una notte (ON). Conservare gli inserti della membrana a 4 °C fino al loro utilizzo.
NOTA: Tutti i passaggi devono essere eseguiti in condizioni sterili. Il rivestimento della membrana deve essere conservato per un massimo di 1 settimana a 4 °C prima dell'uso per evitare la degradazione delle proteine e un'adesione non ottimale della fetta alla membrana.
- Preparazione dei terreni: terreno organotipico, terreno di dissezione, terreno di innesto
- Preparare il terreno organotipico (OM, Tabella 1).
- Preparare il mezzo di dissezione come descritto nella Tabella 1.
- Preparare il mezzo di innesto (GM, ottimizzato da Onorati et al.46, Tabella 1).
NOTA: Le soluzioni devono essere preparate in condizioni sterili e appena prima del loro utilizzo (1 giorno prima o lo stesso giorno dell'esperimento).
- Preparazione del materiale per la chirurgia
- Nella cabina di biosicurezza, tenere a portata di mano lo stereomicroscopio di dissezione e gli strumenti chirurgici: due paia di microforbici, due paia di pinzette diritte e due paia di pinzette curve.
- Nella cabina di biosicurezza, preparare lo strumento tritatutto dotandolo di una lama per tagliare l'SC a fette. Ruotare una vite del tritatutto per sollevare il braccio metallico dove deve essere posizionata la lama. Posizionare la lama nel sito designato, abbassare il braccio metallico con la lama fino a quando non tocca il piatto di taglio e fissarlo serrando la vite di sicurezza con una chiave esagonale fino a quando la lama non è saldamente fissata. Ruotare la vite micrometrica allo spessore della fetta desiderato (generalmente 350 μm). Controllare se la lama è posizionata esattamente perpendicolarmente al piatto di taglio.
NOTA: Per eseguire correttamente il taglio è necessario un posizionamento perpendicolare preciso della lama rispetto al piatto di taglio. - Preparare nella cabina di biosicurezza: due pipette di plastica Pasteur (necessarie per spostare la SC isolata e le fette), almeno quattro piastre da 35 mm e due da 60 mm e una scatola di ghiaccio fresco.
- Sterilizzare tutti gli strumenti con etanolo al 70% e UV (un ciclo di 20 minuti) appena prima del loro utilizzo per preservare la sterilità della coltura.
2. Isolamento della SC di topo e generazione di slice
- Isolamento del mouse SC
- Sacrificare i cuccioli di topo postnatale del giorno 3 (P3) secondo la licenza del progetto.
- Attraverso una laparotomia della linea mediana con le microforbici, isolare la regione lombare della colonna vertebrale dal resto del corpo del topo e metterla in un mezzo di dissezione freddo in un piatto da 35 mm.
- Utilizzando uno stereomicroscopio da dissezione e le microforbici, tagliare la spina dorsale lungo l'asse sagittale e utilizzare una pinzetta diritta per rimuovere delicatamente la SC dalla cavità della spina dorsale.
- Staccare con cura le meningi dalla regione lombare isolata del SC utilizzando una pinzetta dritta.
- Trasferire e incubare la regione lombare SC isolata in terreno di dissezione freddo e fresco per 10-15 minuti prima di procedere con la fase successiva.
- Generazione di sezioni
- Utilizzando una pipetta Pasteur in plastica, prelevare la regione lombare SC isolata dal mezzo di dissezione e posizionarla sul piatto di taglio dello strumento trituratore, perpendicolarmente alla lama.
NOTA: Il corretto posizionamento dell'SC rispetto alla lama (perpendicolare) è essenziale per generare correttamente le fette SC. - Rimuovere il mezzo di dissezione residuo sul ponte attorno alla SC con l'aiuto della pipetta Pasteur e della carta assorbente sterile. Procedere con il sezionamento automatizzato SC.
- Una volta generate le fette, mettere del terreno di dissezione fresco con una pipetta Pasteur sul piatto di taglio con le fette. Quindi, raccogliere le fette in una piastra da 35 mm con terreno di dissezione fresco e incubarle per 15 minuti.
- Durante l'incubazione della fetta, lavare la superficie degli inserti della membrana 3 volte con OM utilizzando una pipetta di plastica Pasteur. Quindi, lasciare 1 mL di OM sul fondo di ciascun inserto della membrana.
- Controllare le fette sotto lo stereomicroscopio di dissezione. Seminare il numero desiderato di fette sugli inserti a membrana condizionati trasferendole con una pipetta di plastica Pasteur.
- Spostare le fette nell'orientamento preferito e nella posizione desiderata sugli inserti della membrana con l'aiuto delle pinzette diritte. Rimuovere l'eccesso di terreno con una pipetta Pasteur per permettere alle fette di aderire meglio alla superficie della membrana.
NOTA: Lo spostamento e l'orientamento delle fette con le pinzette diritte devono essere eseguiti delicatamente per evitare danni ai tessuti o all'inserto della membrana. - Dopo 30 minuti di incubazione a 37 °C, trasferire l'inserto in una nuova capsula di Petri.
NOTA: Toccare l'anello di plastica ma non le membrane durante il cambio del fluido. - Aggiungere 1 mL di OM fresco integrato con fattore neurotrofico derivato da linee cellulari gliali (GDNF) 100 ug mL-1 nella parte inferiore dell'inserto della membrana.
- Incubare le fette a 37 °C. Riferirsi al primo giorno di coltura come giorno ex vivo (DEV) 0.
- Utilizzando una pipetta Pasteur in plastica, prelevare la regione lombare SC isolata dal mezzo di dissezione e posizionarla sul piatto di taglio dello strumento trituratore, perpendicolarmente alla lama.
3. Coltura a lungo termine di fette organotipiche
- Mantenere le fette in coltura a 37 °C fino ai punti temporali desiderati.
- Sostituire il mezzo con OM nuovo a DEV 1 come descritto nei passaggi 2.2.7-2.2.8.
- Al DEV 3, passare il terreno al GM per creare l'ambiente appropriato per il trapianto di cellule staminali il giorno successivo. Sostituire il fluido con GM fresco ogni 48 ore (es. DEV 5, DEV 7...).
- Aggiungere GDNF fresco (concentrazione finale 100 μg mL-1) al terreno ogni giorno fino a DEV 7. Dopo DEV 7, aggiungerlo solo quando il mezzo viene cambiato (passaggio 3.3).
4. Coltura cellulare h-SC-NES
NOTA: le cellule h-SC-NES vengono mantenute in coltura in presenza di fattori di crescita (terreno NES, fase 4.1.1). Prima del trapianto, le cellule vengono piastrate in condizione di predifferenziazione per 7 giorni rimuovendo i fattori di crescita dal terreno (terreno di predifferenziazione: terreno NES senza fattore di crescita dei fibroblasti 2 (FGF-2) e fattore di crescita epidermico (EGF), passaggio 4.1.2). Quindi, le cellule vengono piastrate in condizioni di differenziazione (Terreno di differenziazione, passaggio 4.1.3) per 2 giorni prima del trapianto. La differenziazione è supportata dall'aggiunta di integratori neurotrofici (fattore neurotrofico derivato dal cervello, BDNF) al mezzo di differenziazione. Il mantenimento, la scissione, la pre-differenziazione e la differenziazione delle celle h-SC-NES12,46 sono descritte in dettaglio di seguito.
- Preparazione dei terreni: NES, predifferenziazione e terreni di differenziazione
- Preparare il terreno di mantenimento delle celle h-SC-NES (terreno NES, Tabella 1).
- Preparare il terreno di predifferenziazione cellulare h-SC-NES (Terreno di pre-differenziazione, Tabella 1).
- Preparare il terreno di differenziazione cellulare h-SC-NES (Terreno di differenziazione, Tabella 1). Aggiungere BDNF fresco quando si cambia il terreno o quando le cellule vengono piastrate per la prima volta nella condizione di differenziazione.
NOTA: Tutti i terreni devono essere preparati in condizioni sterili e devono essere filtrati con filtri da 0,22 μm.
- Soluzione di rivestimento per celle h-SC-NES
NOTA: le cellule h-SC-NES sono mantenute in supporti di coltura rivestiti di POLFN (POLFN = Poli-L-Ornitina, Laminina, Fibronectina).- Preparare la soluzione di rivestimento in una provetta: una soluzione di poli-L-ornitina con laminina (5 μg mL-1) e fibronectina (1 μg mL-1).
- Trasferire la soluzione di rivestimento preparata nel supporto di coltura cellulare, avendo cura di aggiungerne una quantità sufficiente a coprire l'intera superficie del supporto di coltura cellulare. Incubare il rivestimento per 1 ora a 37 °C o per una notte a 4 °C.
- Rimuovere la soluzione di rivestimento dai supporti di coltura cellulare.
NOTA: La soluzione di POLFN può essere riciclata altre due volte, ma la laminina e la fibronectina devono essere aggiunte ogni volta fresche. - Lavare il rivestimento 3 volte con acqua sterile per colture cellulari. Conservare i rivestimenti a 4 °C o utilizzarli.
NOTA: I rivestimenti devono essere utilizzati entro 1 settimana. Successivamente, i rivestimenti sono considerati scaduti a causa dei processi di degradazione delle proteine aggiunte.
- Manutenzione delle celle h-SC-NES
- Mantenere le cellule h-SC-NES in coltura in terreno NES. Controlla le cellule ogni giorno al microscopio per monitorare quando raggiungono la confluenza.
- Cambiare mezzo terreno ogni 2 giorni: togliere metà del terreno condizionato e aggiungere quello fresco (considerare un tasso di evaporazione del 20%).
- Se le celle raggiungono la confluenza, procedere con la divisione, come descritto al punto 4.4.
- Passaggio cellulare h-SC-NES
NOTA: Le celle sono suddivise come segue12:- Rimuovere il terreno condizionato e lavare una volta le celle con soluzione salina tamponata con fosfato di Dulbco (DPBS) senza Ca2+/Mg2+.
- Rimuovere il DPBS e aggiungere la soluzione di tripsina/EDTA alle cellule per eseguire il distacco enzimatico. Incubare le cellule a 37 °C per 30 s - 1 min.
- Dopo l'incubazione, controllare le cellule al microscopio: se non sono staccate, picchiettare leggermente il supporto di coltura cellulare per eseguire il distacco meccanico e incubarle a 37 °C per altri 30 s.
- Dopo l'incubazione, inattivare la tripsina/EDTA aggiungendo 4 volumi di DPBS/soluzione di siero fetale bovino (FBS) (10% vol/vol) al supporto della coltura cellulare con cellule e tripsina/EDTA. Pipettare delicatamente la soluzione sulla superficie di supporto della coltura cellulare su e giù per aiutare tutte le cellule a staccarsi. Raccogliere la sospensione cellulare in una provetta.
- Centrifugare la sospensione cellulare a 200 × g per 3 minuti. Scartare il surnatante e risospendere il pellet in terreno NES fresco.
- Contare le cellule e piastarle su ogni nuovo supporto di coltura rivestito con POLFN a una densità di 0,5-1 × 105 cellule/cm2.
- Aggiungere Y-27632 (10 μM) e posizionare le celle a 37 °C. Controllali ogni giorno fino alla confluenza e poi dividili di nuovo per la manutenzione/espansione o la banca cellulare.
- predifferenziamento cellulare h-SC-NES
- Dividi le celle come descritto nel passaggio 4.4.
- Piastra le cellule sui supporti di coltura cellulare rivestiti di POLFN a una densità di ̴0,5-1 × 105 cellule/ cm2 in terreno di predifferenziazione. Aggiungere Y-27632 (10 μM) dopo la divisione. Chiama il primo giorno in giornata di predifferenziazione in vitro (DIV) 0.
- Cambiare metà del terreno ogni 2-3 giorni (vedere il passaggio 4.3.2).
- Mantenere le cellule in condizione di predifferenziazione fino al DIV 7 e poi procedere con il passaggio 4.6.
- Differenziazione cellulare h-SC-NES
- Al DIV 7 di predifferenziazione, dividere le cellule come descritto nel passaggio 4.4.
- Piastra le cellule su supporti di coltura cellulare rivestiti di POLFN a una densità di ̴1-1,5 × 105cellule/cm2in terreno di differenziazione. Aggiungere Y-27632 (10 μM) e BDNF (30 ng mL-1) dopo la divisione.
- Dopo 2 giorni di differenziazione (DIV 10), dividere le cellule per il trapianto in fette.
5. Trasduzione cellulare h-SC-NES con vettori lentivirali portatori di GFP
NOTA: La trasduzione cellulare viene eseguita durante la fase di mantenimento delle cellule h-SC-NES. Quando le cellule sono trasdotte correttamente, queste possono essere espanse e vengono applicati i protocolli di predifferenziazione e differenziazione precedentemente descritti (passaggi 4.5 e 4.6).
- Preparazione del terreno di trasduzione cellulare
NOTA: Il terreno di trasduzione cellulare viene preparato mescolando un volume specifico di terreno NES e un volume preciso di preparazione di stock di vettori lentivirali (LVS) in base a diversi parametri: il MOI desiderato (molteplicità di infezione = rapporto tra il numero di particelle virali e il numero di cellule ospiti in un dato mezzo di infezione); il numero di celle piastre; la concentrazione iniziale del preparato LVS (=LVS PFU, unità formante placche); la superficie del recipiente di coltura utilizzato.- Calcolare il volume corretto di preparato LVS da aggiungere al terreno NES, in base al MOI scelto utilizzando le equazioni (1) e (2).
(n cellule su piastra/cm2) × cm2 di supporto per coltura cellulare × MOI = LVS PFU per il MOI scelto (1)
LVS PFU : Tot LVS Vol iniziale (μL) = LVS PFU per MOI : LVS Vol da aggiungere al terreno (μL)(2)
NOTA: L'LVS PFU (PFU iniziale di LVS) e il volume totale iniziale di LVS sono forniti dal produttore. L'LVS PFU per il MOI scelto è calcolato come descritto nell'equazione (1). Pertanto, possiamo ottenere il volume di preparazione LVS che deve essere aggiunto al volume totale di terreno NES (in base al supporto della coltura cellulare) per il MOI scelto, come descritto nell'equazione (2).
Esempio: abbiamo utilizzato MOI 3, in base a precedenti esperienze di laboratorio (il MOI potrebbe variare a seconda della linea cellulare utilizzata e della preparazione virale). Se il MOI desiderato è 3, il numero di cellule da placcare è 0,5 × 105/cm2 e il supporto di coltura è un MW24 da 1 pozzetto (2 cm2), supponendo che l'LVS PFU/TU iniziale (unità di formazione della placca/unità di trasduzione) sia 25 × 106 PFU in 1 mL (1.000 μL = volume LVS iniziale), i calcoli sono i seguenti:
Celle piastrate in 1 pozzetto-MW24 (2 cm2) = 0,5 × 105 celle × 2 cm2 = 1 × 105 celle
1 × 105 celle × 3 (MOI) = 3 × 105 PFU = LVS PFU per MOI 3
25 × 106 PFU:1.000 μL = 3 × 105 PFU:x μL
x μL = 12 μL = volume LVS da aggiungere al terreno
Pertanto, per trasdurre le cellule con il mezzo di trasduzione cellulare con MOI 3 in 1 pozzetto-MW24, aggiungere 12 μL di preparazione LVS iniziale al terreno NES (238 μL) preparato per 1 pozzetto di MW24. Il volume totale finale è di 250 μl.
NOTA: Il terreno viene solitamente preparato fresco il giorno della trasduzione in condizioni sterili.
- Calcolare il volume corretto di preparato LVS da aggiungere al terreno NES, in base al MOI scelto utilizzando le equazioni (1) e (2).
- Protocollo di trasduzione h-SC-NES
- Piastre h-SC-NES a basso passaggio su piastra a 24 pozzetti multipli rivestita in POLFN (o in qualsiasi altro supporto di coltura) a una densità di 0,5 × 105/cm2 in terreno NES.
- Il giorno successivo, raccogliere il terreno NES condizionato dai pozzetti in cui sono state piastrate le cellule. A seconda del supporto di coltura scelto, aggiungere alle cellule il volume più basso di terreno di trasduzione cellulare fresco necessario per coprire uniformemente la superficie di semina (ad esempio, 250 μL/pozzetto di una piastra a 24 multipozzetti).
- Quindi, incubare le cellule h-SC-NES per 6 ore a 37 °C. Successivamente, aggiungere alle cellule il terreno condizionato precedentemente raccolto (200 μL/pozzetto di una piastra a 24 multipozzetti) e incubare le cellule ON a 37 °C.
- Il giorno successivo, lavare una volta le cellule h-SC-NES con DPBS ed eseguire un cambio totale del terreno (terreno NES).
- I giorni successivi, controllare le cellule al microscopio a fluorescenza per osservare l'espressione della GFP.
- Espandi le cellule h-SC-NES per la conservazione delle cellule e il trapianto.
6. Trapianto di cellule in fette di SC e co-coltura
- Preparazione di microaghi di vetro
- Utilizzare un estrattore per ottenere aghi sottili dai capillari di vetro borosilicato. Impostare l'estrattore come segue: HEAT 990, PULL 350.
NOTA: Da un capillare è possibile ricavare due aghi sottili.
- Utilizzare un estrattore per ottenere aghi sottili dai capillari di vetro borosilicato. Impostare l'estrattore come segue: HEAT 990, PULL 350.
- Preparazione cellulare per il trapianto
- Dividi le celle come descritto nel passaggio 4.4.
NOTA: Se le cellule non esprimono un reporter fluorescente, etichettarle con un colorante per il tracciamento delle cellule per monitorarle utilizzando un microscopio a fluorescenza dopo il trapianto e durante la coltura a lungo termine. Seguire il protocollo del produttore scelto per la fase di etichettatura. - Contare le cellule dopo la divisione e centrifugare a 200 × g per 3 minuti. Sospendere il pellet ottenuto con terreno fresco + Y-27632 (10 μM) per avere la concentrazione di celle desiderata (di solito un intervallo compreso tra 30.000-50.000 celle μL-1).
- Trasferire la sospensione cellulare in una provetta da 500 μl o 1,5 ml e posizionarla sul ghiaccio. Le cellule sono pronte per il trapianto.
- Dividi le celle come descritto nel passaggio 4.4.
- Trapianto di cellule in fette organotipiche
NOTA: Eseguire trapianti di cellule h-SC-NES nelle fette organotipiche SC di topo utilizzando un microiniettore d'aria e microaghi di vetro.- Caricare un microago di vetro con 4 μL di sospensione cellulare utilizzando una micropipetta e puntali per microcaricatori.
NOTA: Evitare la formazione di bolle d'aria nell'ago poiché potrebbe ostacolare il processo di microiniezione. Se si formano delle bolle, rimuoverle con la micropipetta. - Posizionare l'ago nel supporto assegnato del microiniettore e rompere la punta dell'ago utilizzando le pinzette dritte.
NOTA: Rompere l'ago di vetro più vicino alla punta per evitare la formazione di grossi fori. - Prima del trapianto nelle fette, impostare i parametri della microiniezione. Impostare la pressione a 10 psi.
NOTA: Il valore della pressione può essere modificato in base al microiniettore e alle osservazioni dell'operatore: la pressione deve essere sufficiente per microiniettare la sospensione cellulare, evitando danni ai tessuti. - Su un vetrino calibrato, mettere una goccia di olio minerale con una pipetta Pasteur e microiniettare la sospensione cellulare nella goccia. Il diametro della sfera di sospensione cellulare ottenuta nella goccia d'olio è correlato a uno specifico volume di microiniezione. Modificare i parametri di microiniezione secondo necessità per raggiungere un diametro della sfera di sospensione cellulare di 0,2 mm per l'iniezione di 4nL.
- Dopo aver impostato il volume corretto, microiniettare rapidamente la sospensione cellulare nelle fette. Controllare sotto lo stereomicroscopio a fluorescenza la presenza delle cellule nelle fette per verificare che la microiniezione/trapianto sia andata a buon fine.
NOTA: La sospensione cellulare a volte potrebbe ostruire l'ago: in questo caso, provare a rimuovere l'ostruzione della sospensione cellulare modificando i parametri di iniezione o caricare un nuovo ago con sospensione cellulare fresca. - Dopo il trapianto, posizionare le fette a 37 °C e 5% di CO2fino al punto temporale desiderato ed eseguire il cambio del terreno a giorni alterni come descritto nei passaggi 2.2.7-2.2.8.
- Caricare un microago di vetro con 4 μL di sospensione cellulare utilizzando una micropipetta e puntali per microcaricatori.
7. Colorazione in immunofluorescenza
- Giorno 1
- Rimuovere il terreno dal fondo della membrana dell'inserto e lavare le fette 3 volte con DPBS preriscaldato.
- Fissare le fette con formaldeide (FA) al 4% preriscaldata: rimuovere il DPBS e aggiungere 1,5 mL di FA al 4% sul fondo dell'inserto a membrana con le fette. Dopo 15 minuti di incubazione in RT, aggiungere 1 mL in più di FA al 4% sulla superficie superiore dell'inserto di membrana e incubare per 15 minuti in RT. Tempo di fissazione totale: 30 minuti in RT
- Togliere il 4% di FA e lavare le fette per 3 x 10 min con DPBS.
- Tagliare la membrana dell'inserto circonferenzialmente con un coltello chirurgico, separare la membrana con le fette dalla componente plastica dell'inserto e procedere con le fasi di immunofluorescenza.
NOTA: Dopo questo passaggio, la membrana con le fette galleggia in DPBS nel piatto. - Permeabilizzare con 1 mL/membrana di una soluzione con Triton allo 0,7% in DPBS per 10 minuti a RT.
- Rimuovere la soluzione di permeabilizzazione e incubare i campioni per 4 ore a 4 °C con 1 mL/membrana di soluzione bloccante composta da 0,5% Triton e 10% FBS in DPBS.
- Rimuovere la soluzione bloccante e aggiungere alle fette gli anticorpi primari alla loro diluizione di lavoro, ad esempio l'anticorpo anti-neurofilamento di topo (NFL), 1:500; coniglio anti-NFL, 1:500; anticorpo anti-RBFOX3 (NeuN) di coniglio, 1:400; caspasi-3 antiattiva di coniglio (aCASP3), 1:400; Nuclei anti-umani di topo, (Hu-Nu), 1:400; coniglio anti-Hu-Nu, 1:400; topo anti-GFP, 1:400 (come riportato Tabella 1) in 1 mL di soluzione anticorpale composta da 0,5% Triton, 1% FBS in DPBS. Incubare ON a 4 °C.
- Giorno 2
- Lavare le membrane per 3 x 10 minuti con 1-2 ml di DPBS.
- Incubare la membrana con anticorpi secondari (ad esempio, anticorpo secondario IgG (H+L) anti-topo di capra, Alexa Fluor 488, 1:500; Anticorpo secondario IgG (H+L) di capra anti-coniglio, Alexa Fluor 568, 1:500; Anticorpo secondario IgG (H+L) di capra anti-topo, Alexa Fluor 647, 1:500; Anticorpo secondario IgG di coniglio di capra (H+L), Alexa Fluor 647, 1:500 come riportato Tabella 1) e Hoechst/DAPI per nuclei diluiti in 1 mL/membrana di soluzione anticorpale (Triton 0,5% + FBS 1% in DPBS) per 3 ore a RT.
NOTA: Proteggere accuratamente i campioni dalla luce per evitare lo sbiancamento secondario degli anticorpi durante l'incubazione e nei passaggi successivi. - Rimuovere la soluzione anticorpale e lavare per 3 x 10 minuti con DPBS (1-2 mL).
- Sostituire il DPBS con un DPBS nuovo e conservarlo a 4 °C in condizioni di protezione dalla luce.
- Al termine del protocollo di immunofluorescenza, montare le membrane su vetrini. Versare una goccia di 200 μl di soluzione di montaggio su un vetrino. Con l'aiuto di una pinzetta dritta, trasferire la membrana galleggiante dal piatto da 35 mm su un vetrino coprioggetti e poi trasferire la membrana sul vetrino con la soluzione di montaggio.
- Mettere una goccia di 100 μL di soluzione di montaggio su un nuovo vetrino coprioggetti e coprire la membrana con esso, fissandola sul vetrino. Lasciarlo asciugare per una notte sotto la cappa chimica in condizioni di protezione dalla luce.
- Conservare i campioni a 4 °C al buio o eseguire l'analisi di imaging.
8. Saggio vivo/morto
- Preparare la soluzione di lavoro aliquotando 700 μl per piatto di terreno fresco e aggiungere alla corretta diluizione di lavoro il Sytox (ad esempio, Componente B, 1:2.000) e la Calceina AM (ad esempio, Componente A, 1:2.000).
NOTA: Poiché i reagenti sono sensibili alla luce, proteggere la soluzione di lavoro dalla luce. - Valutare il volume medio sul fondo della membrana e aggiungere il Sytox e la Calceina AM alla stessa diluizione di lavoro descritta al punto 8.1.
- Aggiungere 2 gocce da 30 μl ciascuna sulla parte superiore di ciascuna fetta della soluzione di lavoro preparata al punto 8.1.
NOTA: Proteggere il piatto dalla luce posizionandolo al buio. - Incubare le fette per 30 minuti a RT.
- Dopo l'incubazione, ritagliare la membrana circonferenzialmente dall'inserto con un coltello chirurgico: dopodiché, la membrana con le fette galleggia nella soluzione di lavoro.
- Posizionare la membrana senza la soluzione di montaggio capovolta su un vetrino coprioggetti con l'aiuto di una pinzetta dritta e aggiungere 100 μL di DPBS sopra le membrane per mantenerle idratate.
- Acquisisci immagini dal vivo utilizzando il microscopio confocale il più velocemente possibile.
NOTA: Aggiungere 2 gocce da 40 μl ciascuna di DPBS sulla parte superiore della membrana ogni 30 minuti durante l'acquisizione dell'immagine per evitare che la membrana si secchi.
9. Imaging
- Imaging confocale di campioni fissati
- Per l'analisi qualitativa, acquisire immagini utilizzando un microscopio confocale con i seguenti parametri di acquisizione: impostare l'opzione Immagine grande (scegliere: 4 x 4), utilizzare l'obiettivo 10x, nessuna pila e una risoluzione di 3.634 x 3.634 pixel.
- Per l'analisi quantitativa (aCASP3, Calceina e Sytox per le fette e aCASP3 per le cellule), acquisire le immagini utilizzando il microscopio confocale con i seguenti parametri di acquisizione: obiettivo 20x, risoluzione di 1.024 x 1.024 pixel con un passo Z di 3 μm.
- Imaging dal vivo di fette trapiantate utilizzando lo stereomicroscopio
NOTA: Acquisisci immagini utilizzando lo stereomicroscopio in modalità campo chiaro ed epifluorescenza.- Utilizzando l'impostazione del campo chiaro , acquisire immagini delle fette (1 obiettivo x con zoom 3x utilizzato qui).
NOTA: Modificare la luce a seconda del microscopio utilizzato e utilizzare le fibre ottiche se necessario. - Utilizzando l'impostazione della fluorescenza , acquisire immagini delle cellule trapiantate con lo stesso obiettivo e zoom utilizzati per le fette (vedere il passaggio 9.2.1). Utilizzare i seguenti parametri per l'acquisizione: guadagno 1, esposizione 200-500 ms, offset -10.
- Utilizzando l'impostazione del campo chiaro , acquisire immagini delle fette (1 obiettivo x con zoom 3x utilizzato qui).
- Imaging dal vivo dopo il saggio vivo/morto con il microscopio confocale
- Acquisizione di immagini utilizzando un microscopio confocale con i seguenti parametri di acquisizione: obiettivo 20x, risoluzione 1.024 x 1.024 pixel con passo Z di 3 μm.
10. Analisi delle immagini da parte di ImageJ
- Analisi dell'area NFL, RBFOX3 e DAPI
- Aprire il software ImageJ (https://imagej.net/software/imagej/).
- Aprire l'immagine del file facendo clic su File | apri | seleziona file | apri.
- Nella finestra pop-up, seleziona Visualizzazione stack | Hyperstack e modalità colore | Impostazione predefinita (con scalabilità automatica).
- Nella barra degli strumenti, seleziona Immagine | Colore | Canali divisi.
- Scegli i canali desiderati da analizzare: canale verde per NFL (marcatore assonale), canale rosso per RBFOX 3 (marcatore neuronale) e canale blu per DAPI (colorazione nucleare).
- Per l'analisi NFL, procedi con i seguenti passaggi: nella barra degli strumenti seleziona Immagine | Regola | Soglia | Seleziona i parametri (sfondo scuro, algoritmo, ad es. predefinito) e sposta il cursore sulla barra dei valori (sotto/sopra) per coprire e circoscrivere tutta l'area dei neuriti (i neuriti sono evidenziati in bianco su uno sfondo scuro) | Imposta | Applicare.
- Seleziona dalla barra degli strumenti lo strumento di tracciamento Bacchetta e usalo per definire automaticamente l'area bianca coperta da NFL. Premi Analizza | Misura | Valore dell'area in μm2.
- Per l'analisi DAPI e RBFOX3, procedere con i seguenti passaggi: nella barra degli strumenti, selezionare Immagine | Regola | Soglia | Selezionare i parametri (sfondo bianco, algoritmo, ad es. default) e spostare il cursore sulla barra dei valori (sotto/sopra) per coprire e circoscrivere tutta l'area RBFOX3 o DAPI | Imposta | Applicare.
- Nella barra degli strumenti, seleziona Elabora | FFT | Filtro passa-banda. Utilizzare la barra del valore di soglia per regolare l'area bianca coperta da RBFOX3 o DAPI, corrispondente al loro segnale di fluorescenza.
- Dalla barra degli strumenti, selezionare lo strumento di tracciamento Bacchetta e utilizzarlo per definire automaticamente l'area coperta da RBFOX3 o DAPI. Premi Analizza | Misura | Valore dell'area in μm2.
- Analisi dell'apoptosi con ImageJ
- Aprire il software ImageJ (https://imagej.net/software/imagej/).
- Apri l'immagine del file Z-stack facendo clic su File | apri | seleziona file | apri.
- Nella finestra pop-up, seleziona Visualizzazione stack | Hyperstack e modalità colore | Impostazione predefinita (con scalabilità automatica).
- Nella barra degli strumenti, seleziona Immagine | Colore | Canali divisi.
- Scegli i canali desiderati: canale rosso per aCASP3 (marcatore di apoptosi da analizzare) e blu o ciano per DAPI o Hu-Nu per i nuclei. Quindi, sovrapponi i canali selezionando nella barra degli strumenti Immagine | Colore | Unisci i canali | Crea composito.
- Trascina la barra Z nella parte inferiore dell'immagine per sfogliare la pila Z dell'immagine e identificare le pile nella regione centrale delle sezioni con positività aCASP3.
- Nella barra degli strumenti, seleziona Plugin | Analizza | Contatore di celle.
- Nella finestra pop-up aperta, selezionare Inizializza per preparare l'immagine per il conteggio; quindi, selezionare un tipo di contatore (ad esempio, Tipo 1) e rinominarlo come oggetto da contare (ad esempio, celle aCASP3+ ). Rinominare altri tipi di contatori come descritto sopra per contare altri oggetti (ad esempio, celle DAPI+ o Hu-Nu+ per il numero totale di celle).
- Nella finestra pop-up, selezionare il tipo di contatore corrispondente all'oggetto da contare (ad esempio, celle aCASP3+ ), quindi selezionare lo strumento Punto nella barra degli strumenti e iniziare a contare manualmente il numero di celle apoptotiche, positive per aCASP3, facendo clic su ciascuna di esse positive nell'immagine aperta.
- Seleziona un altro tipo di contatore nella finestra del contatore di celle e inizia a contare il numero totale di celle (celle DAPI+ , per le sezioni; Cellule Hu-Nu+ , per le cellule trapiantate).
- Analisi del saggio Live/Dead di ImageJ
- Aprire il software ImageJ (https://imagej.net/software/imagej/).
- Apri l'immagine del file Z-stack facendo clic su File | apri | seleziona file | apri.
- Nella finestra pop-up, seleziona Visualizzazione stack | Hyperstack e modalità colore | Impostazione predefinita (con scalabilità automatica).
- Nelle barre degli strumenti, seleziona Immagine | Colore | Canali divisi.
- Scegli i canali desiderati: canale verde per Calceina (marcatore di vitalità da analizzare) e canale ciano per Sytox (marcatore morto). Quindi, sovrapponi i canali selezionando nella barra degli strumenti Immagine | Colore | Unisci i canali | Seleziona Crea composizione.
- Trascina la barra Z nella parte inferiore dell'immagine per sfogliare la pila Z dell'immagine e identificare le pile nella regione centrale delle sezioni con positività Calcein e Sitox.
- Nella barra degli strumenti, seleziona Plugin | Analizza | Contatore di celle.
- Nella finestra pop-up aperta, selezionare Inizializza per preparare l'immagine per il conteggio; quindi selezionare un tipo di contatore (ad esempio, Tipo 1) e rinominarlo come oggetto da contare (ad esempio, celle Calcein+ ). Rinominare altri tipi di contatori come descritto sopra se è necessario contare un altro oggetto (ad esempio, celle Sytox+ ).
- Nella finestra pop-up, selezionare il tipo di contatore corrispondente all'oggetto da contare; quindi seleziona lo strumento Punto nella barra degli strumenti e inizia a contare manualmente il numero di celle di Calcein+ , facendo clic su ciascuna di esse positive nell'immagine aperta.
- Selezionare un altro tipo di contatore nella finestra del contatore di celle e contare le celle Sytox+ come descritto per Calcein.
11. Grafici e analisi statistiche
- Esegui tutte le analisi statistiche e traccia i grafici utilizzando il software che preferisci.
Risultati Rappresentativi
I metodi descritti consentono la costituzione di fette organotipiche SC da topi allo stadio P3 e il loro mantenimento in coltura per un tempo prolungato in condizioni di salute. Inoltre, mostriamo un protocollo per il trapianto di cellule nelle fette e per la loro co-coltura per un massimo di 30 giorni (Figura 1). In primo luogo, mostriamo l'ottimizzazione delle condizioni di coltura e un protocollo adatto alla coltura prolungata delle fette di SC con cellule trapiantate (Figura 2A). Le fette vengono generate e mantenute da DEV 0 fino a DEV 2 nell'OM, che è stato originariamente proposto come mezzo ottimale per la manutenzione delle sezioni SC47. Tuttavia, a causa della presenza di proteine sieriche, questo mezzo potrebbe essere subottimale per sostenere la differenziazione neuronale e la maturazione delle cellule precursori neurali trapiantate. Infatti, al DEV 3, abbiamo testato il passaggio dall'OM al GM, una formulazione contenente Neurobasal plus B27, che supporta la sopravvivenza neurale, e senza siero, che inibisce il corretto differenziamento neuronale, promuovendo invece un destino gliale48,49.
La Figura 2B mostra i risultati ottenuti commutando il mezzo a DEV 3 da OM a GM, rispetto alle fette SC che non hanno ricevuto l'interruttore (le fette di controllo sono state coltivate in OM). Abbiamo utilizzato la distribuzione del segnale NFL all'interno delle fette come marcatore per l'integrità neuronale (Figura 2B, C). Le fette a DEV 7 erano sane in entrambe le condizioni di coltura, mostrando la distribuzione diffusa del neurofilamento (NFL, in verde) al loro interno. Al DEV 10, le fette coltivate in GM sembravano essere più sane rispetto alle fette di controllo coltivate in OM, come documentato dalla distribuzione delle colorazioni NFL. Abbiamo anche stimato l'area NFL+ (% Area NFL+ /Area DAPI+ ) delle fette mostrate nelle immagini rappresentative della Figura 2B. L'area NFL+ stimata è rappresentata negli istogrammi della Figura 2C, confermando che il segnale NFL è distribuito diffusamente nelle fette a DEV 7 in entrambe le condizioni. Tuttavia, a DEV 10, l'area stimata coperta dalla colorazione NFL diminuisce per la condizione di coltura OM.
Questi dati suggeriscono che il passaggio al GM a DEV 3 è ben tollerato per la coltura prolungata di fette di SC (DEV 10). Come passo successivo, abbiamo testato GM in momenti più prolungati: DEV 30, DEV 60 e DEV 90. Come mostrato nella Figura 3A, B, le fette sono state mantenute sane in coltura fino a DEV 90. La colorazione NFL è stata trovata ampiamente presente nelle fette in ogni momento, con una germinazione diffusa attorno alle fette di neuriti che partono dalla regione centrale. Infatti, abbiamo stimato l'area NFL+ delle fette mostrate nella Figura 3A ed è aumentata nel tempo, come mostrato negli istogrammi della Figura 3B. Abbiamo anche osservato positività al marcatore neuronale RBFOX3, fornendo un'altra linea di evidenza del differenziamento neuronale delle fette. Ad ogni punto temporale, abbiamo anche controllato il tasso di apoptosi valutando in diverse fette il numero di cellule positive ad aCASP3 (Figura 4A, B). L'analisi è stata eseguita come descritto nella sezione 10.2 del protocollo. Il tasso di apoptosi (% di cellule aCASP3+ /numero totale di cellule DAPI+ ) è risultato molto basso in ogni punto temporale (0,85 ± 0,52%, 0,71 ± 0,27%, 0,66 ± 0,45% per DEV 30, 60 e 90, rispettivamente) senza differenze significative tra i tre punti temporali considerati (valore p > 0,05, Figura 4B). Questi dati suggeriscono che il tasso di apoptosia associato ad aCASP3 rimane stabile nel tempo e, insieme all'ampia distribuzione di NFL nelle fette (Figura 4A), confermano la sopravvivenza delle fette in ogni punto temporale.
A supporto dei dati precedenti, abbiamo anche eseguito un saggio vivo/morto per valutare la vitalità delle fette nei tre diversi punti temporali. Abbiamo usato la Calceina (colorazione verde) per marcare le cellule vitali e metabolicamente attive e Sytox (colorazione ciano) per valutare la morte cellulare. Come mostrato negli istogrammi della Figura 4C, la percentuale di cellule metabolicamente attive aumenta leggermente da DEV 30 a DEV 90 (93,17 ± 5,21%, 96,43 ± 3,02%, 96,33 ± 3,10% rispettivamente per DEV 30, 60 e 90), stabilizzandosi tra gli ultimi due punti temporali (DEV 30 vs DEV 60 p-value = 0,018; DEV 30 vs DEV 90 p-value = 0,027; DEV 60 vs DEV 90 p-value = 0,99). Abbiamo riscontrato bassi livelli di morte cellulare che sono diminuiti nel tempo (6,83 ± 5,21%, 3,57 ± 3,02%, 3,66 ± 3,10% per DEV 30, 60 e 90, rispettivamente) e una differenza significativa è stata riscontrata tra DEV 30 e i punti temporali successivi, DEV 60 e DEV 90 (DEV 30 vs DEV 60 p-value = 0,018; DEV 30 vs DEV 90 p-value = 0,027; DEV 60 vs DEV 90 p-value = 0,99). Questi dati, in associazione con il tasso di apoptosi, confermano la sopravvivenza della fetta nel tempo e supportano l'efficacia del protocollo di coltura a lungo termine eseguito.
Una volta stabilita la fattibilità della coltura prolungata delle fette di SC, abbiamo sfidato il sistema mediante trapianto di cellule h-SC-NES nelle prime fasi del differenziamento neuronale. Abbiamo testato le cellule h-SC-NES perché hanno mostrato risultati promettenti per il trattamento della lesione midollare12. La procedura di trapianto di cellule h-SC-NES nelle fette di SC di topo è descritta nella sezione 6 del protocollo. Le fette SC e le cellule h-SC-NES trapiantate sono state mantenute fino al DPT 30. Le cellule sono state innestate al DIV 10 di differenziazione (stadio di precursore neurale) in fette organotipiche DEV 4, come mostrato nello schema del protocollo della Figura 5A. Le cellule trapiantate sono state monitorate per l'espressione di GFP in coltura per un massimo di 30 giorni. La Figura 5B mostra immagini dal vivo rappresentative, a diversi DPT, di una fetta SC con cellule GFP+ trapiantate. L'espressione stabile della GFP nel tempo (Figura 5B e Figura 6A) suggerisce che le cellule sono sopravvissute nel tessuto SC nelle condizioni di coltura precedentemente ottimizzate. Abbiamo anche controllato il tasso di apoptosi delle cellule trapiantate come descritto nella sezione 10.2 del protocollo. Il tasso di apoptosi (% di cellule aCASP3+ /numero totale di cellule Hu-Nu+ ) è risultato molto basso (0,44 ± 0,34%) dopo 30 DPT (Figura 6B). Inoltre, il tasso di apoptosi a DPT 30 è risultato essere in linea con quello trovato per lo stesso tipo di cellule a DPT 7, come precedentemente riportatoa 40, documentando che le colture si stabilizzano nel tempo.
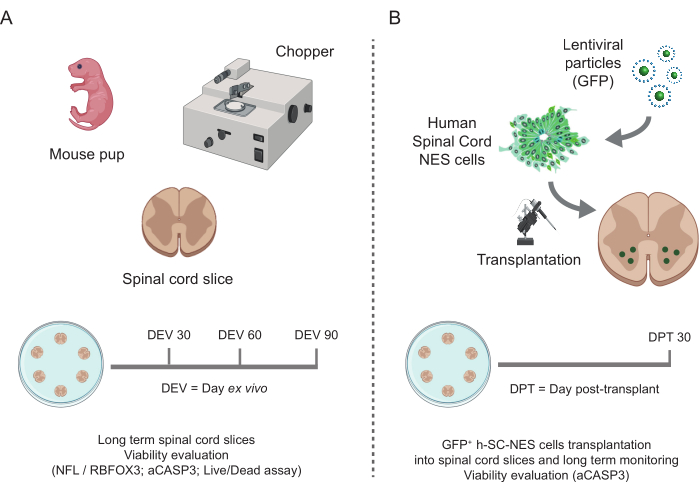
Figura 1: Flusso di lavoro del protocollo. Schema rappresentativo che mostra il flusso di lavoro generale del protocollo eseguito. (A) A sinistra, uno schema che riassume la generazione di fette di SC di topo da SC isolate di cuccioli di topo a P3 e la coltura a lungo termine di fette organotipiche di SC. (B) A destra, uno schema che riassume il trapianto di cellule h-SC-NES che esprimono GFP in fette di topo SC-organotipiche. Le cellule innestate vengono mantenute per 30 giorni dopo il trapianto. Abbreviazioni: h-SC-NES = staminali neuroepiteliali derivati dal midollo spinale umano; GFP = proteina fluorescente verde; DEV = giorno ex vivo; DPT = giorno post-trapianto; NFL = catena leggera a neurofilamenti; RBFOX3= omologo 3 di fox-1 legante l'RNA; aCASP3 = Caspasi-3 attiva; SC= midollo spinale. Clicca qui per visualizzare una versione più grande di questa figura.
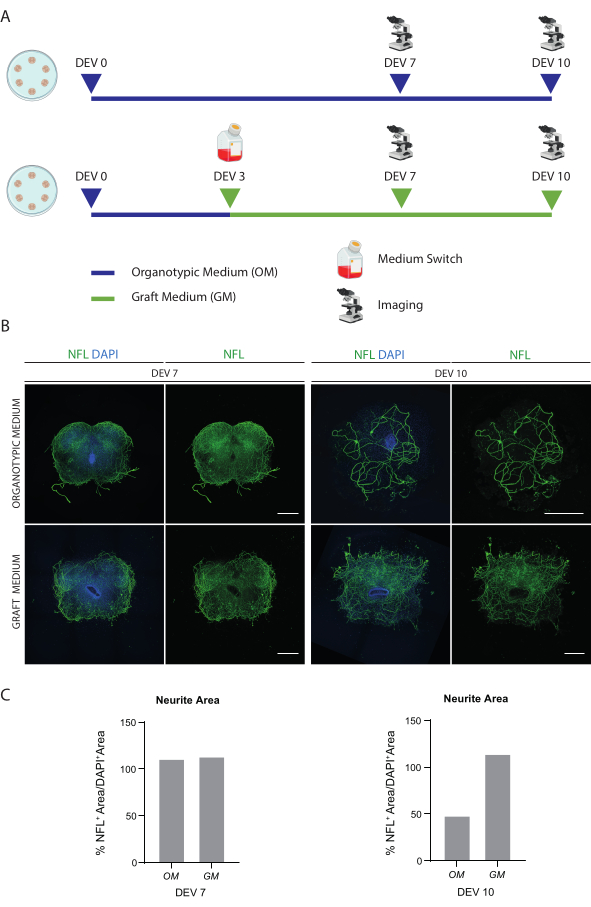
Figura 2: Ottimizzazione delle condizioni di coltura a lungo termine. (A) Schema rappresentativo del protocollo per testare OM e GM. L'OM viene mantenuto fino a DEV 7-10 per il gruppo di controllo. Il mezzo viene commutato al GM a DEV 3 per le fette trattate; quindi, vengono fissati a DEV 7-10 per il confronto con i controlli. (B) Immagini rappresentative che confrontano fette organotipiche di SC di topo a DEV 7 e 10 coltivate in diverse condizioni. Le fette sono colorate per il neurofilamento marcatore del citoscheletro (NFL, verde). L'ampia distribuzione della colorazione NFL in fette coltivate con GM suggerisce una sopravvivenza globale e una differenziazione. I nuclei sono controcolorati con DAPI. Barra della scala = 500 μm. (C) Istogrammi rappresentativi della stima dell'area coperta da NFL nelle fette mostrate in Figura 1B. Al DEV 10, la superficie della NFL diminuisce nella condizione di coltura OM. Abbreviazioni: DEV = giorno ex vivo; DAPI = 4',6-diamidino-2-fenilindo; NFL = catena leggera a neurofilamenti.; OM = mezzo organotipico; GM = mezzo di innesto; SC = midollo spinale. Clicca qui per visualizzare una versione più grande di questa figura.
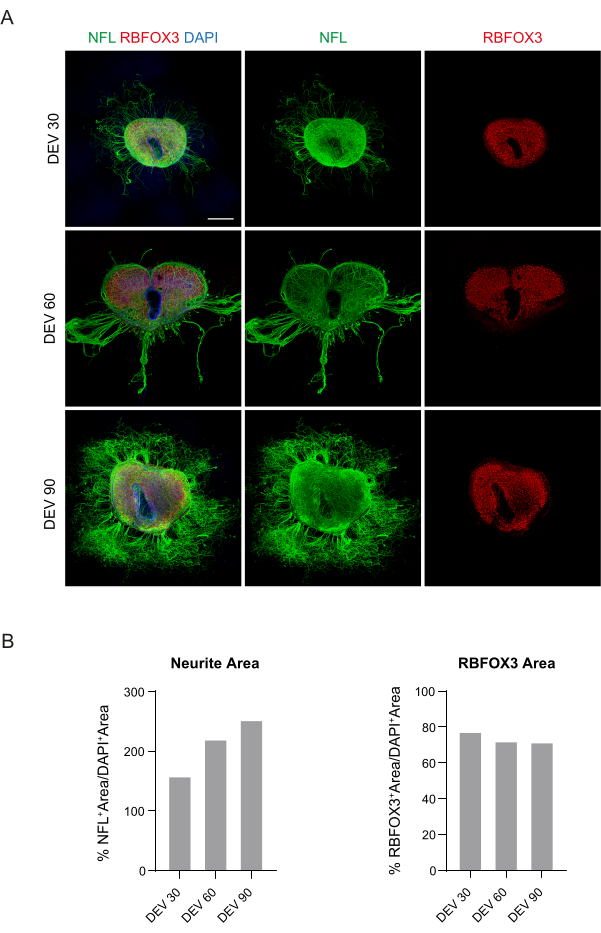
Figura 3: Fette organotipiche SC di topo coltivate a lungo termine. (A) Le fette vengono mantenute in coltura fino a DEV 90. Il test di immunofluorescenza rivela un'ampia distribuzione del neurofilamento marcatore citoscheletrico (NFL, verde) e del marcatore neuronale nucleare RBFOX3 (rosso), attestando la loro condizione di salute e identità neuronale dopo una coltura a lungo termine. Da notare, gli assoni NFL+ spuntano diffusamente attorno alle fette nel tempo. I nuclei sono controcolorati con DAPI. Barra della scala = 500 μm. (B) Istogrammi rappresentativi della stima dell'area e del tempo NFL+ e dell'area RBFOX3+ delle fette mostrate nel pannello A. L'area dei neuriti NFL+ aumenta nel tempo. Abbreviazioni: DEV = giorno ex vivo; DAPI = 4',6-diamidino-2-fenilindo; NFL = catena leggera a neurofilamenti; SC = midollo spinale; RBFOX3= Omologo 3 di fox-1 legante l'RNA. Clicca qui per visualizzare una versione più grande di questa figura.
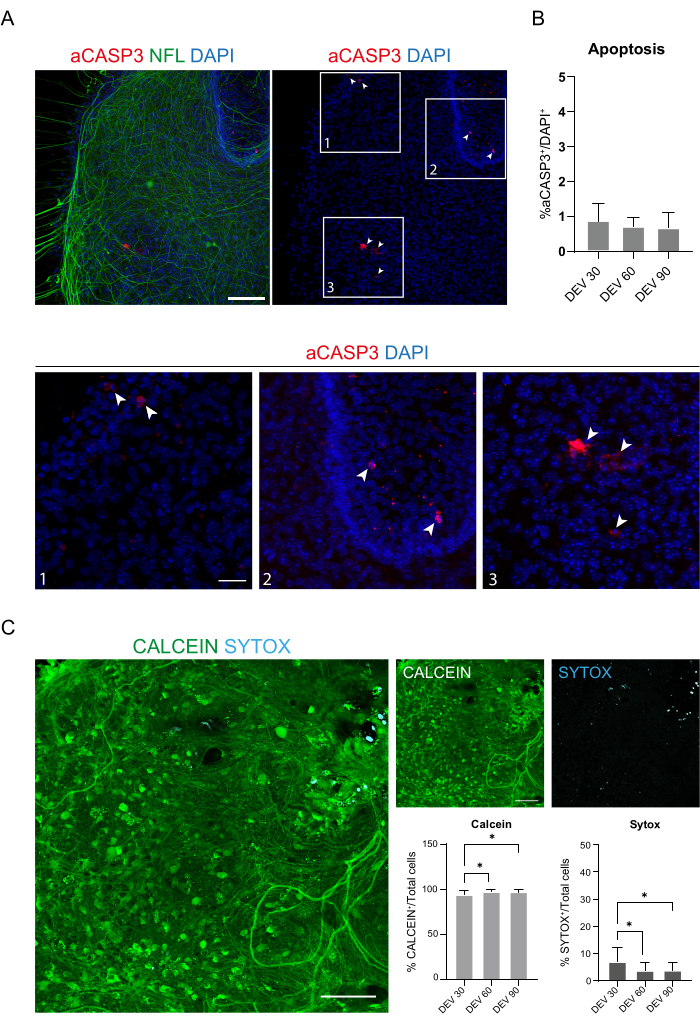
Figura 4: Valutazione della vitalità cellulare nelle fette SC nel tempo. (A) Immagini rappresentative di fette organotipiche a DEV 60 colorate per aCASP3 (rosso) e NFL (verde). Barra della scala = 100 μm. NFL mostra un modello diffuso. Cellule rare sono positive per il marcatore apoptotico aCASP3 (Inserti: 1-2-3). (B) Analisi del tasso di apoptosi in fette in diversi punti temporali. Media ± SD, N (si replica) = 6 fette, n (celle totali) > 1.000 per ogni fetta, test di Kruskal-Wallis, confronto multiplo, p-value > 0,05. Il tasso di apoptosi è stabile nel tempo. Nei riquadri 1-2-3 del pannello A, è possibile osservare i dettagli delle cellule positive per aCASP3 (colorazione rossa, frecce bianche). Piccoli punti rossi etichettano i detriti cellulari e i nuclei picnotici. Barra di scala = 50 μm. (C) Immagini rappresentative del saggio vivo/morto eseguito su fette SC a DEV 90: le cellule metabolicamente attive sono etichettate in verde con Calceina, mentre le cellule morte e danneggiate sono etichettate in azzurro chiaro (ciano) con Sytox. I due istogrammi mostrano la % di celle positive per Calceina (a sinistra) e Sytox (a destra) sul numero totale di celle. Per entrambe le ± medie SD, N (si replica) = 6 fette, n (celle totali) > 1.000 per ogni fetta, test di Kruskal-Wallis, confronto multiplo, DEV 30 vs DEV 60 p-value = 0,018; DEV 30 vs DEV 90 p-value = 0,027; DEV 60 vs DEV 90 p-value > 0,99. Abbreviazioni: DEV = giorno ex vivo; DAPI = 4',6-diamidino-2-fenilindo; NFL = catena leggera a neurofilamenti; SC = midollo spinale; aCASP3 = caspasi-3 attiva. Clicca qui per visualizzare una versione più grande di questa figura.
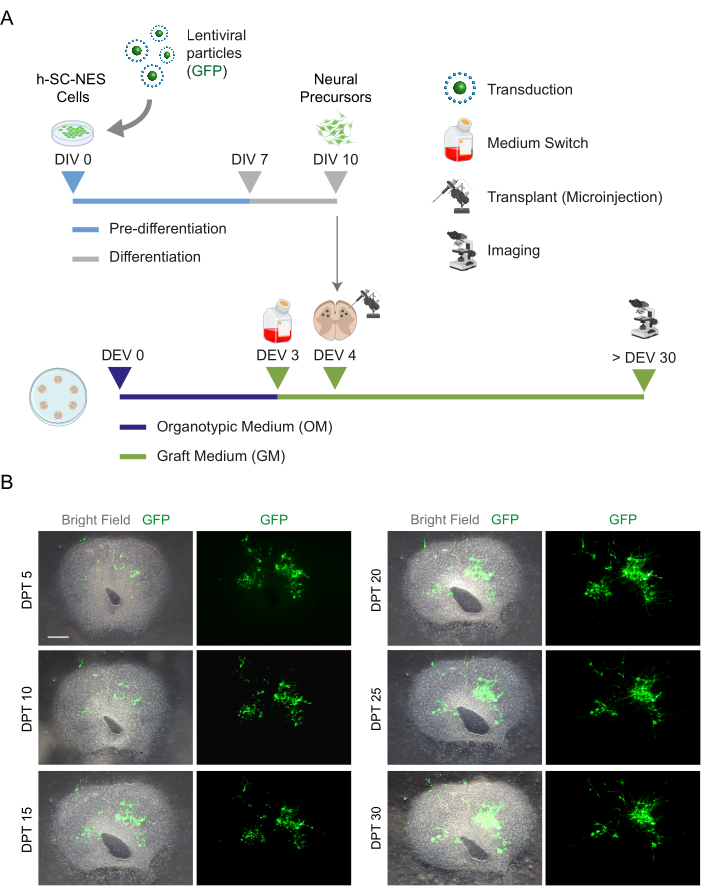
Figura 5: Trapianto di cellule h-SC-NES in fette organotipiche di topo. (A) Schema rappresentativo del protocollo di trapianto. Le cellule vengono trapiantate come precursori neurali al DIV 10 di differenziazione in fette organotipiche DEV 4. (B) Immagini rappresentative di fette organotipiche di topo trapiantate con cellule h-SC-NES che esprimono GFP nel tempo fino a DPT 30. Le cellule vengono trasdotte con un vettore lentivirale che trasporta il gene GFP . L'espressione di GFP nel tempo conferma la loro vitalità e adattamento all'ambiente della fetta. Barra della scala = 500 μm. Abbreviazioni: DIV = primo giorno in pre-differenziazione; h-SC-NES = staminali neuroepiteliali derivati dal midollo spinale umano; GFP = proteina fluorescente verde; DEV = giorno ex vivo; OM = mezzo organotipico; GM = mezzo di innesto; DPT = giorni dopo il trapianto. Clicca qui per visualizzare una versione più grande di questa figura.
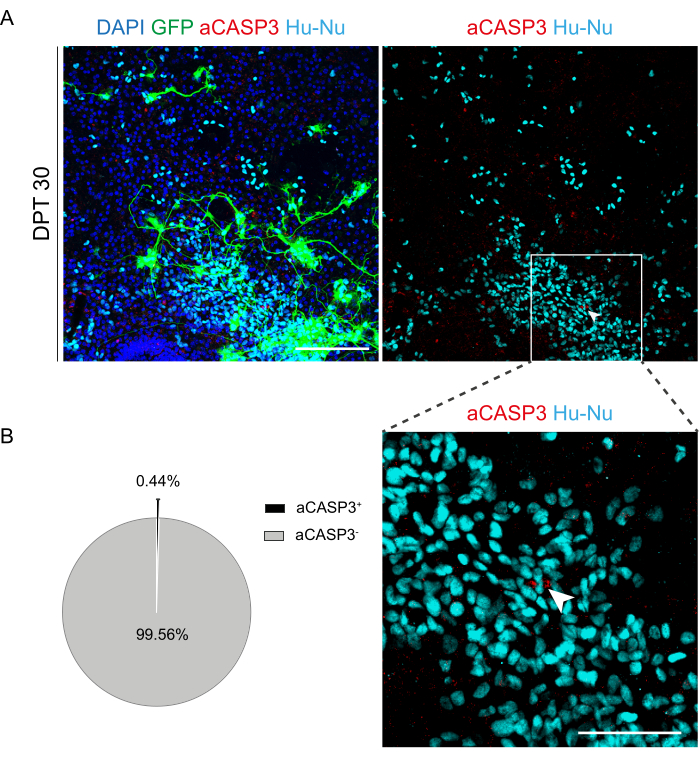
Figura 6: Valutazione del tasso di apoptosi delle cellule h-SC-NES trapiantate dopo 30 giorni dal trapianto. (A) Immagine rappresentativa di una fetta organotipica di topo trapiantata con cellule h-SC-NES che esprimono GFP. Le cellule vengono trasdotte con un vettore lentivirale che trasporta il gene GFP per monitorarle nelle fette dopo il trapianto. L'espressione di GFP nel tempo conferma la loro vitalità e adattamento all'ambiente della fetta. Il punto temporale visualizzato è DPT 30; le cellule vengono colorate per i nuclei umani (ciano) e aCASP3 (rosso). Barra della scala = 150 μm. (B) A sinistra, grafico a torta rappresentativo dell'analisi dell'apoptosi di cellule trapiantate in fette a DPT 30 (N (si replica) = 5 fette, n (cellule) = 5.000) e a destra, un inserto di cellule Hu-Nu+ e un dettaglio di una cellula positiva ad aCASP3 (freccia bianca). Barra di scala = 75 μm. Piccoli punti rossi etichettano i detriti cellulari e i nuclei picnotici. Abbreviazioni: h-SC-NES = staminali neuroepiteliali derivati dal midollo spinale umano; GFP = proteina fluorescente verde; DPT = giorno post-trapianto; DAPI = 4',6-diamidino-2-fenilindo; NFL = catena leggera a neurofilamenti; aCASP3 = caspasi-3 attiva; Hu-Nu = nuclei umani. Clicca qui per visualizzare una versione più grande di questa figura.
Tabella 1: Composizione delle soluzioni utilizzate in questo protocollo. Clicca qui per scaricare questa tabella.
Discussione
Non esiste ancora un trattamento efficace per i pazienti con lesione midollare. Sono stati testati diversi approcci e uno dei più promettenti si basa su una strategia rigenerativa di sostituzione cellulare. Attualmente, i progressi nel campo della medicina rigenerativa richiedono nuove piattaforme per testare l'efficacia e la sicurezza dei trapianti cellulari, da soli o in combinazione con altri approcci. La loro convalida preclinica è essenziale per proseguire gli studi clinici. Le colture organotipiche SC sono una piattaforma utile per studiare diversi aspetti della neurodegenerazione, della rigenerazione neurale e del neurosviluppo e per studiare l'efficacia di nuovi approcci terapeutici23. In particolare, le caratteristiche specifiche delle colture organotipiche, come il mantenimento dell'istoarchitettura originale e della composizione cellulare e del microambiente, sono vantaggiose per svelare le dinamiche del trapianto, come l'attecchimento, l'integrazione, il differenziamento e la maturazione cellulare.
Coerentemente con i protocolli pubblicati, le fette organotipiche SC possono essere mantenute in coltura per circa 2-3 settimane in condizioni di salute, il che ne limita l'uso per le indagini a lungo termine e lo screening funzionale necessari per i programmi di test della terapia cellulare. L'esplorazione di processi importanti come la differenziazione e la maturazione verso il corretto destino delle cellule trapiantate all'interno del tessuto SC richiede un monitoraggio a lungo termine. Questi processi cellulari sono fondamentali durante i trapianti comuni in modelli animali. La disponibilità di un sistema ex vivo che imiti molte caratteristiche presenti in vivo sarebbe utile nella fase di screening preclinico.
Per questo motivo, in questo lavoro, proponiamo un metodo di coltura organotipica SC ottimale a lungo termine (≥30 giorni) che consente di mantenere fette di SC vitali fino a 90 giorni, triplicando il loro consueto periodo di coltura. Inoltre, mostriamo un attecchimento stabile delle cellule h-SC-NES all'interno delle fette SC e il mantenimento della coltura del trapianto fino a 30 giorni. Abbiamo monitorato l'attecchimento cellulare nel tempo osservando l'espressione di GFP per verificare la sopravvivenza cellulare fino a DPT 30. Dopo 30 DPT, abbiamo valutato il tasso di apoptosi cellulare. In letteratura, è stata riportata la valutazione dell'apoptosi delle cellule h-SC-NES trapiantate in sezioni SC a 7 DPT40. Qui, abbiamo esteso l'analisi dell'apoptosi cellulare a DPT 30 per confrontare il tasso di apoptosi rispetto al punto temporale precedente (DPT 7). Abbiamo scoperto che i nostri dati sono in linea con la letteratura, suggerendo che le cellule h-SC-NES trapiantate sopravvivono anche in un momento successivo se vengono mantenute nelle condizioni di coltura ottimizzate nel nostro lavoro. Questa piattaforma ex vivo a lungo termine migliorata, da sola e nella configurazione del trapianto, aiuterà i ricercatori nello screening preclinico per i trapianti di cellule staminali per la lesione midollare. Ciò consentirà loro di identificare la migliore cellula candidata per ulteriori studi in vivo che promuovono il successo dei trapianti. Inoltre, dopo lo screening iniziale, le fette organotipiche di SC potrebbero anche essere utilizzate in parallelo agli studi in vivo per confermare e corroborare le dinamiche e i comportamenti cellulari a lungo termine osservati in modelli animali o per supportare studi meccanicistici.
Il nostro protocollo descrive in dettaglio come generare questo modello organotipico a lungo termine, ma dovrebbero essere discussi anche alcuni passaggi critici. Per quanto riguarda la generazione delle colture organotipiche SC, ci sono alcune sfide durante l'intervento chirurgico e le prime fasi della coltura. Una procedura chirurgica ben eseguita è essenziale per generare fette che mantengano l'istoarchitettura originale. Se la SC si rovina durante l'isolamento, le fette possono perdere la loro tipica struttura anatomica e il danno tissutale può indurre un eccessivo insulto pro-infiammatorio che porta a condizioni malsane e morte cellulare. La fase più impegnativa durante l'intervento chirurgico è l'estrazione della SC dalla spina dorsale e la rimozione delle meningi dalla SC isolata. Il successo di questi passaggi dipende dall'esperienza dell'operatore; Pertanto, si consiglia un periodo di formazione prima di iniziare con gli esperimenti.
Anche la sezione coronale della SC attraverso un elicottero è una fase impegnativa. L'SC isolato deve essere posizionato sul piatto di taglio esattamente perpendicolarmente alla lama. L'operatore deve inoltre posizionare la lama perpendicolarmente al piatto di taglio. Queste precauzioni sono necessarie per garantire la generazione di fette riproducibili tra lo stesso e diversi esperimenti. Un altro problema importante è che il tempo per l'intervento chirurgico è limitato: l'intera procedura di generazione della fetta deve richiedere ~30 minuti. Se l'operatore dedica più tempo alla chirurgia e al taglio, il tessuto SC ne risentirà e questo può compromettere il successo della coltura e le fasi successive dell'esperimento.
Una volta che le fette sono state posizionate sulla membrana di coltura, è importante nutrirle correttamente. Il GDNF è necessario per sostenere il recupero e la sopravvivenza dei tessuti. Il taglio con un tritatutto è traumatico per il tessuto e, per questo motivo, le fette vengono posizionate subito dopo il taglio in un mezzo di dissezione ghiacciato per ripulire l'eccesso di molecole pro-infiammatorie e che promuovono la morte. Quindi, le fette vengono posizionate sulle membrane di coltura (inserti di coltura cellulare) con terreno fresco modificato con GDNF per favorire un recupero più rapido e l'adesione della fetta alla membrana. GDNF dovrebbe essere aggiunto al terreno ogni giorno per la prima settimana in coltura a causa della sua breve emivita50,51. Abbiamo osservato che le fette necessitano della presenza continua di GDNF durante i primi giorni di coltura per promuovere il recupero e la vitalità dei tessuti. In ogni caso, poiché la presenza di GDNF è importante per l'intero periodo di coltivazione, è fortemente sconsigliato interrompere la somministrazione di GDNF in momenti successivi.
Durante la prima settimana di coltura, è anche importante controllare le fette macroscopicamente a occhio e al microscopio. Il tessuto traslucido e la trasparenza dei bordi sono segni di una corretta adesione delle fette alla membrana e del tessuto vitale. Il tessuto necrotico apparirà estremamente bianco alla prima vista macroscopica e le aree necrotiche appariranno grigio scuro al microscopio. Dopo alcune settimane di coltura, la morfologia del tessuto può cambiare: i movimenti cellulari e l'adesione del tessuto alla membrana possono influenzare questo processo. Abbiamo osservato, ad esempio, la perdita del lume centrale in alcune fette piene di cellule e la perdita della morfologia del corno dorsale e ventrale. Questo accade principalmente con fette più piccole, mentre la maggior parte di esse manterrà una struttura anatomica vicina a quella originale. Le fette vengono solitamente generate dalla regione lombare o toracica perché in questo modo possono avere le dimensioni adeguate per mantenere nel tempo la loro istoarchitettura originaria: se sono troppo piccole, perdono la loro architettura mentre, se troppo grandi, la regione centrale può andare incontro a necrosi. Pertanto, abbiamo utilizzato la regione lombare dei cuccioli di topo per generare fette di dimensioni adeguate per una coltura ottimale a lungo termine ma, in linea di principio, possono essere considerati altri segmenti. Inoltre, abbiamo scelto di utilizzare la regione lombare, perché le regioni ventrale e dorsale sono più distinguibili l'una dall'altra. Inoltre, questa regione presenta aree tissutali con una percentuale più elevata di motoneuroni e materia grigia, che sono siti di interesse per le terapie di sostituzione cellulare nella lesione midollare. Per quanto riguarda il trapianto di cellule nelle fette, il problema principale è legato alla rottura della punta del microago di vetro. Se il foro per il passaggio delle cellule è troppo grande, può causare danni al tessuto SC durante la microiniezione. Se è troppo piccolo, l'impilamento delle cellule può ostruire l'ago, ostacolando il processo di trapianto. La procedura di trapianto deve essere completata entro 1 ora per ridurre al minimo la sofferenza e la morte delle cellule.
Il protocollo proposto fornisce uno strumento ottimale e versatile per diversi tipi di indagini. Qui, applichiamo la nostra piattaforma a lungo termine per convalidare il trapianto di cellule h-SC-NES nelle prime fasi di differenziazione all'interno del tessuto SC di topo per 30 giorni. La principale novità dell'approccio proposto è l'ottimizzazione del protocollo di co-coltura. I componenti della GM sostengono la sopravvivenza neuronale a lungo termine delle fette di SC e delle cellule h-SC-NES trapiantate. Infatti, gli OGM, essendo un terreno privo di siero, sostengono la differenziazione delle cellule trapiantate verso il destino neuronale rispetto al terreno precedentemente utilizzato per la coltura organotipica di fette47.
Per quanto riguarda i modelli proposti per la lesione midollare, gli esperimenti vengono solitamente eseguiti su topi adulti. Finora, le differenze più importanti tra SC neonatale e adulto sono legate al più alto potenziale rigenerativo riscontrato nel neonato rispetto ai topi adulti52. Tuttavia, tali differenze non hanno alcun impatto sul tipo di protocollo che stiamo proponendo, poiché qui ci concentriamo sulla risposta delle cellule trapiantate all'ambiente del tessuto ospite piuttosto che sulle capacità di rigenerazione dei neuroni residenti. Un'altra differenza tra topi neonatali e adulti dopo una lesione midollare è legata alla formazione della cicatrice gliale che si verifica negli adulti. Questo aspetto non viene preso in considerazione nel modello proposto, che non considera i complessi processi fisiopatologici conseguenti alle lesioni primarie e secondarie.
Per quanto riguarda le applicazioni, la piattaforma potrebbe essere utilizzata anche per studiare l'integrazione tra le cellule trapiantate con i circuiti residenti presenti nel modello organotipico SC. Gli strumenti di ingegneria genetica erano già utilizzati nel SNC per valutare la connettività sinaptica e potrebbero essere sfruttati a questo proposito 53,54,55. In particolare, l'integrazione potrebbe essere studiata e validata valutando la formazione di sinapsi tra le cellule trapiantate e il tessuto SC ex vivo. Queste colture organotipiche a lungo termine potrebbero anche essere sfruttate per testare agenti neuroprotettivi e neurorigenerativi o nuove molecole/materiali o per studiare malattie neurodegenerative che coinvolgono la SC. Per studiare specifiche malattie neurodegenerative, il protocollo deve essere adattato per la coltura di fette di SC generate da modelli rilevanti, come topi transgenici portatori di specifiche mutazioni associate alla patologia, allo stadio rilevante per la patologia (ad esempio, neonatale, giovanile, adulto). In conclusione, il nostro protocollo e le colture organotipiche in generale, essendo espianti di uno specifico organo, presentano caratteristiche che colmano il divario tra le colture cellulari 2D e i modelli in vivo, confermandosi uno strumento prezioso sia per la ricerca di base che per i test preclinici.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Lo studio è stato sostenuto dalla Fondazione Wings for Life (WFL-IT- 20/21), dal Piano Nazionale di Ripresa e Resilienza (PNRR) Next-Generation EU-Mission 4 component 2, dall'investimento n. 1.4-CUP N. B83C22003930001 (Tuscany Health Ecosystem-THE, Spoke 8), e da Marina Romoli Onlus. Questo manoscritto riflette solo i punti di vista e le opinioni degli autori, né l'Unione Europea né la Commissione Europea possono essere considerati responsabili per essi. Dati e metadati sono disponibili su Zenodo 10.5281/zenodo.10433147. Le immagini sono state generate con il https://www.biorender.com/ Biorender.
Materiali
| Name | Company | Catalog Number | Comments |
| anti-cleaved Caspase-3, (Asp175) (5A1E) (Rabbit) | Cell Signaling Technology | 9661S | 1:400 |
| anti-GFP (Mouse) - monoclonal | Sigma/Merck | G6539 | 1:400 |
| anti-Human Nuclei (Mouse) - monoclonal, clone 235-1 | Sigma/Merck | MAB1281 | 1:400 |
| anti-Human Nuclei (Rabbit) | NeoBiotechnologies | RBM5-346-P1 | 1:400 |
| anti-NeuN (RBFOX3) (Rabbit) - polyclonal | Sigma/Merck | ABN78 | 1:400 |
| anti-NFL (Mouse) | Sigma/Merck | MAB1615 | 1:400 |
| anti-NFL H-Phospho (Rabbit) -polyclonal | Biologend | 840801 | 1:500 |
| Aqua Polymount | Poly-sciences | 18606-20 | |
| B-27 | Gibco | 17504-044 | |
| BDNF | Gibco | PHC7074 | |
| Blades | Leica | 118364227 | |
| Cell culture graded water | Sigma/Merck | W3500-500ML | |
| Collagen from rat tail | Sigma/Merck | C7661 | |
| Confocal microscope - A1 Confocal Microscope (Eclipse Ti) | Nikon | ||
| D(+)-Glucose | Sigma/Merck | G7021 | |
| Dissecting Forceps | World Precision Instruments | 15915 | |
| DMEM/F12 | Gibco | 31330 | |
| DPBS | Sigma/Merck | D8537 | |
| EGF | Sigma/Merck | gf144 | |
| FBS | Gibco | 10270-106 | |
| FGF-2 | Stemgent | 03-0002 | |
| GDNF | Sigma/Merck | SRP3200 | |
| Glass capillaries, 3.5" | Drummond Scientific Company | 3-000-203-G/X | |
| Glutamax | Gibco | 35050-038 | |
| Goat-anti Mouse IgG Alexa Fluor 488 | Thermo Fisher Scientific | A11029 | |
| Goat-anti Mouse IgG Alexa Fluor 647 | Thermo Fisher Scientific | A21236 | 1:500 |
| Goat-anti Rabbit IgG Alexa Fluor 568 | Thermo Fisher Scientific | A11011 | 1:500 |
| Goat-anti Rabbit IgG Alexa Fluor 647 | Thermo Fisher Scientific | A21244 | 1:500 |
| Graph Pad-Prism | Dotmatics | Software for Statistical Analysis | |
| HBSS | Gibco | 14025-050 | 1:500 |
| HEPES | Gibco | 15630-056 | |
| Hoechst 33342 | Thermo Fisher Scientific | H3570 | |
| Horse Serum | Gibco | 16050-122 | |
| Insulin | Sigma/Merck | I9278 | |
| Laminin | Sigma/Merck | L2020 | |
| Lentiviral prep | Addgene | 17446-LV | |
| L-Glutamine | Thermo Fisher Scientific | 25030024 | |
| LIVE/DEAD Viability/Cytotoxicity assay kit | Thermo Fisher Scientific | L32250 | |
| McIlwain Tissue Chopper | World Precision Instruments | ||
| MEM | Gibco | 11090-081 | |
| Microloader tips | Eppendorf | 5242956003 | to load cells in the needle for transplantation |
| Microscope slides | VWR | 631-0909 | |
| Millicell cell culture membrane | Sigma/Merck | PICM0RG50 | |
| Miscroscope cover glasses | VWR | ECN 631-1572 | |
| N-2 | Gibco | 17502-048 | |
| Neurobasal | Gibco | 21103-049 | |
| Penicillin/Streptomycin | Thermo Fisher Scientific | 15140122 | |
| Petri dish (35mm) | VWR | 734-2317 | |
| PFA | Sigma/Merck | P6148-500G | |
| Plastic pasteur pipette | Sarstedt | 86.1171.010 | |
| Pneumatic PicoPump | World Precision Instruments | PV830 | Microinjector for transplantation |
| Poly-L-lysine | Sigma/Merck | P4707 | |
| Scalpel blade No 10 Sterile Stainless Steel | VWR International | SWAN3001 | |
| Scalpel handle #3 | World Precision Instruments | 500236 | |
| Spring Scissors | World Precision Instruments | 501235 | |
| Stereomicroscope for imaging and acquisition | Nikon | SMZ18 | |
| Stereomicroscope for surgery | VWR | ||
| Triton X-100 | Merck | T8787 | |
| Tweezers-Dumont #5-inox | World Precision Instruments | 501985 | |
| Vannas Scissors, 8.5 cm | World Precision Instruments | 500086 | |
| Vertical micropipette puller | Shutter Instrument | P-30 | |
| Y-27632 | R&D Systems | 1254/50 |
Riferimenti
- Ding, W., et al. Spinal cord injury: The global incidence, prevalence, and disability from the Global Burden of Disease Study 2019. Spine. 47 (21), 1532-1540 (2022).
- Yang, B., et al. Strategies and prospects of effective neural circuits reconstruction after spinal cord injury. Cell Death Dis. 11 (6), 439 (2020).
- Liu, K., et al. PTEN deletion enhances the regenerative ability of adult corticospinal neurons. Nat Neurosci. 13 (9), 1075-1081 (2010).
- Anderson, M. A., et al. Required growth facilitators propel axon regeneration across complete spinal cord injury. Nature. 561 (7723), 396-400 (2018).
- de Freria, C. M., Van Niekerk, E., Blesch, A., Lu, P. Neural stem cells: promoting axonal regeneration and spinal cord connectivity. Cells. 10 (12), 3296 (2021).
- Badner, A., Siddiqui, A. M., Fehlings, M. G. Spinal cord injuries: how could cell therapy help. Expert Opin Biol Ther. 17 (5), 529-541 (2017).
- Assinck, P., Duncan, G. J., Hilton, B. J., Plemel, J. R., Tetzlaff, W. Cell transplantation therapy for spinal cord injury. Nat Neurosci. 20 (5), 637-647 (2017).
- Ishii, K., et al. Neutralization of ciliary neurotrophic factor reduces astrocyte production from transplanted neural stem cells and promotes regeneration of corticospinal tract fibers in spinal cord injury. J Neurosci Res. 84 (8), 1669-1681 (2006).
- Zhang, Y. W., Denham, J., Thies, R. S. Oligodendrocyte progenitor cells derived from human embryonic stem cells express neurotrophic factors. Stem Cells Dev. 15 (6), 943-952 (2006).
- Faulkner, J., Keirstead, H. S. Human embryonic stem cell-derived oligodendrocyte progenitors for the treatment of spinal cord injury. Transpl Immunol. 15 (2), 131-142 (2005).
- Kadoya, K., et al. Spinal cord reconstitution with homologous neural grafts enables robust corticospinal regeneration. Nat Med. 22 (5), 479-487 (2016).
- Dell' Anno, M. T., et al. Human neuroepithelial stem cell regional specificity enables spinal cord repair through a relay circuit. Nat Commun. 9 (1), 3419 (2018).
- Wu, S., FitzGerald, K. T., Giordano, J. On the viability and potential value of stem cells for repair and treatment of central neurotrauma: overview and speculations. Front Neurol. 9, 602 (2018).
- Nardone, R., et al. Rodent, large animal and non-human primate models of spinal cord injury. Zoology. 123, 101-114 (2017).
- Hartung, T. Thoughts on limitations of animal models. Parkinsonism Relat Disord. 14, (2008).
- Shanks, N., Greek, R., Greek, J. Are animal models predictive for humans. Philosophy, Ethics, and Humanities in Medicine. 4 (1), 2 (2009).
- Dawson, T. M., Golde, T. E., Lagier-Tourenne, C. Animal models of neurodegenerative diseases. Nat Neurosci. 21 (10), 1370-1379 (2018).
- Hayden, P. J., Harbell, J. W. Special review series on 3D organotypic culture models: Introduction and historical perspective. In Vitro Cell Dev Biol Anim. 57 (2), 95 (2021).
- Jensen, C., Teng, Y. Is it time to start transitioning from 2D to 3D cell culture. Front Mol Biosci. 7, 33 (2020).
- Mirbagheri, M., et al. Advanced cell culture platforms: a growing quest for emulating natural tissues. Materials Horizons. 6 (1), 45-71 (2019).
- Gähwiler, B. H. Organotypic monolayer cultures of nervous tissue. J Neurosci Methods. 4 (4), 329-342 (1981).
- Stoppini, L., Buchs, P. -. A., Muller, D. A simple method for organotypic cultures of nervous tissue. J Neurosci Methods. 37 (2), 173-182 (1991).
- Pandamooz, S., Nabiuni, M., Miyan, J., Ahmadiani, A., Dargahi, L. Organotypic spinal cord culture: a proper platform for the functional screening. Mol Neurobiol. 53 (7), 4659-4674 (2016).
- Fuller, L., Dailey, M. E. Preparation of rodent hippocampal slice cultures. CSH Protoc. 2007, (2007).
- Gertz, C. C., Lui, J. H., LaMonica, B. E., Wang, X., Kriegstein, A. R. Diverse behaviors of outer radial glia in developing ferret and human cortex. J Neurosci. 34 (7), 2559-2570 (2014).
- Ballerini, L., Galante, M. Network bursting by organotypic spinal slice cultures in the presence of bicuculline and/or strychnine is developmentally regulated. Eur J Neurosci. 10 (9), 2871-2879 (1998).
- Avossa, D., Rosato-Siri, M. D., Mazzarol, F., Ballerini, L. Spinal circuits formation: a study of developmentally regulated markers in organotypic cultures of embryonic mouse spinal cord. Neuroscience. 122 (2), 391-405 (2003).
- Lossi, L., Merighi, A. The use of ex vivo rodent platforms in neuroscience translational research with attention to the 3RS philosophy. Front Vet Sci. 5, 164 (2018).
- Nogueira, G. O., Garcez, P. P., Bardy, C., Cunningham, M. O., Sebollela, A. Modeling the human brain with ex vivo slices and in vitro organoids for translational neuroscience. Front Neurosci. 16, 838594 (2022).
- Qi, X. R., et al. Human brain slice culture: a useful tool to study brain disorders and potential therapeutic compounds. Neurosci Bull. 35 (2), 244 (2019).
- Park, H. W., et al. Human mesenchymal stem cell-derived Schwann cell-like cells exhibit neurotrophic effects, via distinct growth factor production, in a model of spinal cord injury. Glia. 58 (9), 1118-1132 (2010).
- Charrière, K., Risold, P. Y., Fellmann, D. In vitro interactions between bone marrow stromal cells and hippocampal slice cultures. C R Biol. 333 (8), 582-590 (2010).
- Jeong, D. K., Taghavi, C. E., Song, K. J., Lee, K. B., Kang, H. W. Organotypic human spinal cord slice culture as an alternative to direct transplantation of human bone marrow precursor cells for treating spinal cord injury. World Neurosurg. 75 (3-4), 533-539 (2011).
- Riggio, C., et al. Generation of magnetized olfactory ensheathing cells for regenerative studies in the central and peripheral nervous tissue. Int J Mol Sci. 14 (6), 10852-10868 (2013).
- Kamei, N., et al. Neural progenitor cells promote corticospinal axon growth in organotypic co-cultures. Neuroreport. 15 (17), 2579-2583 (2004).
- Kamei, N., et al. NGF released from transplanted neural progenitor cells promote corticospinal axon growth in organotypic cocultures. Spine. 32 (12), 1272-1278 (2007).
- Hamasaki, T., et al. Magnetically labeled neural progenitor cells, which are localized by magnetic force, promote axon growth in organotypic cocultures. Spine. 32 (21), 2300-2305 (2007).
- Kim, H. M., Lee, H. J., Lee, M. Y., Kim, S. U., Kim, B. G. Organotypic spinal cord slice culture to study neural stem/progenitor cell microenvironment in the injured spinal cord. Exp Neurobiol. 19 (2), 106-113 (2010).
- Liu, X., Chu, T. H., Su, H., Guo, A., Wu, W. Neural progenitor cell apoptosis and differentiation were affected by activated microglia in spinal cord slice culture. Neurol Sci. 35 (3), 415-419 (2014).
- De Vincentiis, S., et al. Low forces push the maturation of neural precursors into neurons. Small. 19 (30), 2205871 (2023).
- Abouelfetouh, A., Kondoh, T., Ehara, K., Kohmura, E. Morphological differentiation of bone marrow stromal cells into neuron-like cells after co-culture with hippocampal slice. Brain Res. 1029 (1), 114-119 (2004).
- Sypecka, J., Koniusz, S., Kawalec, M., Sarnowska, A. The organotypic longitudinal spinal cord slice culture for stem cell study. Stem Cells Int. 2015, 471216 (2015).
- Tanvig, M., et al. A brain slice culture model for studies of endogenous and exogenous precursor cell migration in the rostral migratory stream. Brain Res. 1295, 1-12 (2009).
- Tennstaedt, A., et al. Human neural stem cell intracerebral grafts show spontaneous early neuronal differentiation after several weeks. Biomaterials. 44, 143-154 (2015).
- Vogel, S., et al. The in vivo timeline of differentiation of engrafted human neural progenitor cells. Stem Cell Res. 37, 101429 (2019).
- Onorati, M., et al. Zika virus disrupts phospho-TBK1 localization and mitosis in human neuroepithelial stem cells and radial glia. Cell Rep. 16 (10), 2576-2592 (2016).
- Vyas, A., et al. An in vitro model of adult mammalian nerve repair. Exp Neurol. 223 (1), 112-118 (2010).
- Brewer, G. J., Torricelli, J. R., Evege, E. K., Price, P. J. Optimized survival of hippocampal neurons in B27-supplemented neurobasal, a new serum-free medium combination. J Neurosci Res. 35 (5), 567-576 (1993).
- De Vries, G. H., Boullerne, A. I. Glial cell lines: an overview. Neurochem Res. 35 (12), 1978-2000 (2010).
- Ziv-Polat, O., et al. The role of neurotrophic factors conjugated to iron oxide nanoparticles in peripheral nerve regeneration: in vitro studies. Biomed Res Int. 2014, 267808 (2014).
- Mesa-Infante, V., Afonso-Oramas, D., Salas-Hernández, J., Rodríguez-Núñez, J., Barroso-Chinea, P. Long-term exposure to GDNF induces dephosphorylation of Ret, AKT, and ERK1/2, and is ineffective at protecting midbrain dopaminergic neurons in cellular models of Parkinson's disease. Mol Cell Neurosci. 118, 103684 (2022).
- Montero, A. M., Huang, A. H. The regenerative capacity of neonatal tissues. Development. 149 (12), (2022).
- Feng, L., Kwon, O., Lee, B., Oh, W. C., Kim, J. Using mammalian GFP reconstitution across synaptic partners (mGRASP) to map synaptic connectivity in the mouse brain. Nat Protoc. 9 (10), 2425-2437 (2014).
- Il Choi, D., Kaang, B. -. K. Interrogating structural plasticity among synaptic engrams. Curr Opin Neurobiol. 75, 102552 (2022).
- Choi, J. -. H., et al. Interregional synaptic maps among engram cells underlie memory formation. Science. 360 (6387), 430-435 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon