このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
スペクトル的に解決された二光子顕微鏡を用いたGタンパク質共役受容体相互作用の in vivo定量で
要約
スペクトル的に解決された二光子顕微鏡イメージングシステムを採用することにより、フェルスター共鳴エネルギー移動(FRET)効率のピクセルレベルのマップは、ホモオリゴマー複合体を形成するために仮定膜受容体を発現する細胞に対して得られる。効率マップをFRETから、我々が検討されてオリゴマー複合体に関する化学量論的情報を推定することができます。
要約
情報が両方の利点を産業用途だけでなく、基本的な基本的な生物学的知識を高めるに蓄積されたので、生きた細胞内のタンパク質相互作用の研究は、研究の重要な領域です。電子励起状態におけるドナー分子と近隣のアクセプター分子との間のフェルスター(蛍光)共鳴エネルギー移動は、(FRET)頻繁に生細胞におけるタンパク質間相互作用の研究のために利用されている。興味のあるタンパク質は、蛍光プローブの2種類のタグ付けと生物細胞で発現されています。蛍光プローブは、一般的にレーザー光を使用して、励起され、蛍光プローブから発せられる蛍光発光のスペクトル特性を収集し、分析される。タンパク質相互作用の程度に関する情報は、分光放射データに埋め込まれています。一般的に、細胞を正確に細胞内で関心のある各地域のタンパク質相互作用の程度を定量化するために十分なスペクトル情報を蓄積するために、回数をスキャンする必要があります。しかし、これらの領域の分子組成は、細胞全体の平均には明らかに、FRET効率の定量的な値の空間的な決定を制限する、取得プロセスの過程で変更される可能性があります。スペクトル的に解決された二光子顕微鏡を用いて、我々は、関心のサンプルの唯一の完全な励起のスキャン後のスペクトル解決のイメージの完全なセットを取得することができます。このピクセルレベルのスペクトルデータから、細胞全体に効率のFRETのマップが計算されます。細胞全体に効率のFRETの実験で得られた分布するオリゴマー複合体におけるFRETの単純な理論を適用することにより、単一のスペクトル解決のスキャンは、研究の下でオリゴマー複合体に関する化学量論と構造情報を明らかにする。ここでは、蛍光プローブの2種類のタグが付けられた膜の受容体(滅菌2α-因子受容体)を発現する生物細胞( 酵母 Saccharomyces cerevisiae)の準備の手順を説明します。さらに、我々はスペクトル解決二光子顕微鏡イメージングシステムを用いて蛍光データの収集に関与する重要な要素を示しています。このプロトコルの使用はそれに接続されている蛍光マーカーと生細胞内で発現可能なタンパク質の任意のタイプを研究するために拡張することができる。
プロトコル
1。プラスミドの設計
興味のあるタンパク質は、次に説明するよう、二つの異なる蛍光標識の一つに融合されています。蛍光標識は、タンパク質1のホモオリゴマー形成に関する細胞内のタンパク質の位置だけでなく、定量的な情報に関する情報を提供していません。蛍光共鳴エネルギー移動(FRET)2-4の技術は5-10タンパク質相互作用に関する情報を蓄積するために使用されます。二つの分子場合のエネルギーの伝達は、双極子-双極子相互作用11を介して光学的に励起分子(通常はドナーと呼ばれる、D)から興奮していない分子(通常はアクセプターと呼ばれる、)に発生する可能性があるという原理を利用したFRETの互いに近接して来る。次のように融合タンパク質をコードするプラスミドを設計では、2つの主要機能は、留意する必要がある。
- 蛍光タグの互換性のあるペアを選択すると、研究をFRETで最も重要なのです。ドナータグの発光スペクトルとアクセプタのタグの励起スペクトル、およびレーザーパルスのスペクトルの中心波長と励起スペクトルの間に大きな分離との間の波長でかなりの重複:理想的には、蛍光タグの組み合わせは、2つの基準を満たしている必要があります。アクセプターの(すなわち、大きなストークスシフト)。ドナー14および黄色蛍光タンパク質(YFP)12またはそれの変異受容体として金星15と呼ばれるようにGFP 2:緑色蛍光タンパク質12、13の2つの変種が特によく、これらの条件を満たすのに適しています。セルリアン16は、やや控えめな結果とも、ドナーとしても使用することができる。
- 定量的に分子複合体の唯一のドナーがその競合が複数のドナーとつのアクセプタを含む複合体で、同じ受容体にエネルギーを転送するためのドナーの間で発生しないこと、(平均で)時間で励起されている場合、画像はそれが最善であるFRETの。これは、理論とデータ分析のプロセス17の単純化につながる。そのため、信号レベルは通常低いです。これを補うために、目的のタンパク質の発現レベルが比較的高いはずです。高い発現レベルが細胞内蛋白質の遺伝子を運ぶためにプラスミドの高コピーを使用して実現されています。
2。酵母の変換
それは、目的のタンパク質の構造を発現する細胞集団の割合を最大化するために有利である。この目的のために、我々は二つの異なるプラスミドを、選択マーカーのトリプトファンと一つのマーカウラシルを含むもので、トリプトファンまたはウラシルを生成することができない酵母細胞( 出芽酵母 )、の栄養要求株を変換する。プラスミドはまた、2つの蛍光プローブの一つでタグ付けされた目的のタンパク質を発現するようにセルの命令を運ぶ遺伝子が含まれています。形質転換細胞はまた、トリプトファンおよびウラシルを欠く固体培地のプレート上で増殖されています。細胞の少なくとも3つのタイプを変換する必要があります:目的のタンパク質を発現する細胞は、蛍光プローブ、アクセプター蛍光プローブでタグ付けだけタンパク質を発現するドナー蛍光プローブと細胞でタグ付けされた唯一のタンパク質を発現する細胞の両方のタイプでタグ付け。
- 固形培地のプレートを準備(酵母窒素ベース、W / Oアミノ酸、1.6 g / Lの酵母合成ドロップアウト中W / Oウラシル及びトリプトファン、1.7 g / Lの2%[W / V]グルコース、2%[W / V]細胞形質転換の前に少なくとも一日に形質転換された細胞用増殖培地として使用する寒天)。彼らはあらゆる汚染物質の自由残っている場合は固体培地プレートは、調製後2ヶ月のためにまで使用することができます。
- 各プラスミドのペアが変換されるためには、三角フラスコにYPD(10g / Lのバクト酵母エキス、20g / lのバクトペプトン、2%[W / V]グルコース)10mlを加え。形質転換される酵母細胞の栄養要求株のコロニーをYPDをIncoluate。
- サンプルの軌道旋回のアクションを提供する研究室シェーカー、で酵母培養液を含む三角フラスコを置き、30℃一晩インキュベート℃、翌朝は、定期的に少量を削除することによって、細胞培養の成長の監視を開始文化とその光学密度(OD)をテスト。細胞培養の成長は、変革の時に0.5〜1.0のODすなわち、中期対数期にある必要があります。
- 形質転換するプラスミドそれぞれペアの滅菌マイクロ遠心チューブにプラスミドの両方のタイプの預金5μL。
- 各プラスミドのペアが変換されるためには、空のマイクロ遠心チューブにサケ精子DNA(5 mg / ml)を5μLを加える。水没5分間沸騰したお湯でサケ精子DNAを含むマイクロチューブの下半分を。ポッピングOPからマイクロチューブのキャップを防ぐためにエン、のみ浸す水の微量遠心管の下半分。沸騰したお湯から削除した後、少なくとも2分間、氷中にサケ精子DNAを含むマイクロチューブを置きます。
- プラスミドを含有する各マイクロ遠心チューブにサケ精子DNAの5μLを追加。
- それは半ば対数相(すなわちODが0.5と1.0の間に読んで)に達したことを確認するために成長している酵母培養液のODを測定します。
- 2分間1000xgで酵母懸濁液を遠心してください。
- 上清を捨て、ボルテックスミキサーを用いて滅菌脱イオン水に酵母ペレットを再懸濁します。
- 2分間1000xgで再び懸濁液を遠心してください。
- 上清を捨て、TEで0.1M LiOAc(pH8.0の1.02グラムLiAc、pH7.4の1Mトリスを10mL、及び10 mLの0.1 M EDTA)の溶液中でペレットを再懸濁します。 TEの0.1M LiOAcの体積は、YPDでオリジナルの酵母の培養液量の1 / 100と同じでなければなりません。
- プラスミドを含有する微量遠心管のそれぞれに、細胞を100μLを配布する。各チューブは、プラスミドと細胞を混合するために、穏やかにフリックします。
- マイクロチューブの各々に[W / V]ポリエチレングリコール(PEG 4000)ソリューション44%400μLを追加。
- 優しく細胞とプラスミドとのPEG 4000を混在させる。細胞を壊すことを避けるために、この時点ではボルテックスミキサーを使用しないでください。穏やかに混合するために、マイクロキャップと人差し指と親指の間のマイクロ遠心チューブの下部を固定し、徐々にチューブを逆さにしてから、徐々に直立の位置に戻し管をもたらす。その円筒形の軸の周りにチューブを90 °回転させて、ゆっくりと再び反転。このプロセスの細胞はマイクロチューブの壁の表面全体の面積を下にスライドするように回数を繰り返します。
- 45分間30℃インキュベーターにマイクロチューブを置きます。
- インキュベーション45分後、インキュベーターから細胞を除去し、14に記載されて穏やかに混合手順を繰り返します。
- 42℃の水浴中にマイクロチューブをセットし、15分間反応させます。もう一度、のみ浸す水の微量遠心管の下半分を。
- 42℃のインキュベーション後、マイクロチューブに滅菌脱イオン水1 mLを加える。
- 5秒のための卓上型微量遠心でチューブを遠心する。 micropipetterを使用してチューブから上清を取り除く。
- マイクロ遠心チューブの各々に滅菌脱イオン水100μLを加えると逆のプロセスを使用して細胞を滅菌脱イオン水を混ぜる。
- そのマイクロ遠心チューブに加え、特定のプラスミドを選択する増殖培地のプレートに、単一のマイクロ遠心チューブの内容を追加。 4月6日滅菌ガラスビーズ(直径1mm)を追加します。細胞は寒天の増殖培地の表面に広がっている順序で形質転換された酵母細胞やガラスビーズの液滴を含むプレートを振る。形質転換細胞の各セットに対してこの手順を繰り返します。
- 5日 - 3、30℃インキュベーターでプレートを置きます。 3〜5日後、1〜3 mm径の複数の酵母のコロニーは、増殖培地の表面上に見えるようになります。この時、細胞はスペクトル的に解決された二光子顕微鏡で画像化する準備が整いました。
3。スペクトル的に解決された二光子顕微鏡のキャリブレーション
これらの研究で使用されているスペクトル解決二光子顕微鏡は、他の場所で18日、19日詳細に記載されている。 〜750から830 nmの範囲の波長のフェムト秒パルスを生成するサファイアレーザー:簡単に言うと、高出力固体連続波レーザは、モードロックチタンをポンプするために使用されます。レーザービームは、試料の回折限界のスポットへの高NA顕微鏡対物によって焦点を当てています。励起では、2光子励起の確率は低いが原因でビームの近焦点領域で発生します。直交コンピュータ制御のスキャニングミラーのペアは、2つの異なる方向(プロトコルを通して、x方向と y方向と称す)における試料の平面内にビームの焦点を変換するために使用されます。光はEM - CCDアレイのピクセルに入射を下回る前に、サンプルの照射ボクセルからバック伝播蛍光発光は、発光のパスに置かれた透過型グレーティングによるスペクトル成分に分散されている。従って、ビームの焦点領域内の任意の蛍光分子/分子の完全なスペクトルプロファイルは、CCDアレイの一次元(y方向)に沿って得られる。 twoスキャンミラーの一つの動きを使用して、レーザーの焦点の位置は、y方向に垂直な線に沿ってサンプル内でスキャンすることができます。このシングルラインスキャン中にCCDカメラを開いているのシャッターを保持することにより、蛍光分子の解像度のスペクトルプロファイルスキャンの全体のラインに沿ってidingは、サンプル全体のレーザービームの単一掃引で収集されます。セル内の別のy位置にこのような性質の複数のラインスキャンを蓄積すると、サンプル内に存在する蛍光錯体の多数のスペクトルプロファイルを与える。ラインスキャンから得られた画像は、異なる波長18、19でのサンプルの複数のXY蛍光強度空間マップを与えるために再構築されます。正確に再構築し、XY蛍光強度空間マップに対応する実際の波長を計算するために、ラインスキャンプロトコルは、十分に特徴付けられた蛍光発光スペクトルを用いてサンプルを使用して校正する必要があります。
- スペクトル的に解決された二光子顕微鏡は、場所の数に関心のサンプル全体に励起光の焦点を走査することにより撮像された細胞におけるオリゴマー複合体に関するスペクトル情報を蓄積。
- 直交スキャンミラーのペアは、2つの異なる方向にサンプル間のレーザビームの焦点を変換するために使用されます。スキャンミラーの動きはコンピュータ制御とCCDカメラからの蛍光強度のデータの抽出と同期です。
- ラインスキャンから、光の特定の波長に対応する蛍光強度の空間マップを再構築することができます。正確にXY蛍光強度空間マップを再構築し、これらのマップのそれぞれに対応する実際の波長を計算するために、ラインスキャンプロトコルは、フルオレセイン溶液を用いて校正する必要があります。
- キャリブレーション手順を開始するには、中央ドナータグの最大励起波長、GFP 2を 2倍である800nm の波長で励起スペクトル。中央励起スペクトルをするために、群速度分散を変更するには、レーザー共振器内にあるプリズムを翻訳。プリズムは、リニアモーター駆動ステージを制御するコンピュータにマウントされています。
- コンピュータプログラムを使用すると、カメラはダークノイズを減らすために、最低達成可能な温度に、CCDチップの温度を下げるためにCCDチップにコマンドを送信する、とのインタフェースです。
- 顕微鏡スライド上にフルオレセイン溶液のピペット10μL。サンプルの薄層を均一にカバーし、顕微鏡のスライドの間の領域に分散されるように、カバースリップでカバー。
- カバースリップの表面上に液浸オイルの小滴を配置
- 今XYZの並進ステージに顕微鏡スライドを固定し、手動で光軸方向にステージ移動を制御するリニアアクチュエータを調整することによって、光軸方向にスライド/ステージを翻訳。顕微鏡対物は、イマージョンオイルの滴に接触するまでスライド/ステージを翻訳。
- 放出された光は、CCDアレイを打つようにデータ収集のビデオモードに切り替え、カメラを制御するコンピュータプログラムを使用することをリアルタイムでコンピュータの画面上に表示されます。ゆっくりと、レーザービームの焦点に試料をもたらすために光軸方向に平行移動ステージを制御するマイクロメータを調整します。
- フルオレセインのサンプルにフォーカスがある場合に、排出は、CCDアレイ上にシャープな線として表示されます。 CCDアレイからカメラを制御するコンピュータプログラムを使用してコンピュータへのピクセル輝度の読み出しをダウンロードしてください。画像Jの強度値のダウンロード行列を開き、蛍光領域を介して線を描画することにより、CCDアレイ上の位置の関数としてピクセル強度を測定。画像Jのコマンドは、フルオレセインのサンプルの発光スペクトルを示すプロットを作成する→プロットプロファイルを分析に使用します。
- CCD上のスペクトルの次元に沿って正確に1つのピクセルによって蛍光スペクトルのピークの動きのサンプルの結果内で隣接する位置とy位置など、y方向のレーザーフォーカスの移動を制御するミラーの増分パラメータを調整する配列。 、サンプル内の異なる2つのy位置のためのフルオレセインのスペクトル強度の値の画像をダウンロードする画像Jと強度の画像を開く、および画像Jのカーソルを使用して蛍光スペクトルの各ピークのピクセル位置を見つけることによってこれを監視します。
- CCDオープンのシャッターを残して、x方向に試料を横切るレーザーフォーカスをスキャン。スキャンの線に沿って各ボクセルから発せられた光は、CCDアレイに入射して落ちるはず。
- このラインスキャンで得られたCCDアレイからのデータを格納して、CCDアレイのピクセルを消去します。
- このセクションのステップ11で決定した金額によってy方向にレーザーの焦点の位置を移動します。
- もう一度CCD ARのシャッターを開いたまま、サンプル全体でx方向にレーザービームをスキャン線とデータを格納する。
- 物理的な領域まで、このラインスキャンの手順を繰り返します〜単一生体細胞の大きさより50%大きく、レーザー光によって照らされています。
- 行の特定のラインスキャン画像の数と波長との関係は、異なる波長で複数のXY蛍光強度空間マップを得るために画像を再構成するために使用する必要があります。特定の波長(λj)のXY蛍光発光画像を取得するには、この波長に対応する最初の行のスキャンから得られた画像上に行番号を見つける。その後、後続のラインスキャン画像の隣接する行には、その特定の波長の蛍光発光画像の次の行に対応する。その波長におけるサンプルの xy蛍光強度空間マップを得るために、この波長に対応するすべての画像の行を積み重ねる。他のすべての入手の波長に対して、この手順を繰り返します。
- それぞれのXY蛍光強度の空間マップに関連付けられている波長を計算するには、背景が画像のインデックス(j)の関数として各再構成画像の正規化された蛍光強度を訂正決定。カメラの画素のスペクトル次元の位置と放出された光子の波長との関係はほぼ線形であるため再構成されたXY蛍光強度の空間マップの画像と対応する波長との関係も線形と次のように計算されます。
 mの値は次のように表されている場所:
mの値は次のように表されている場所: 
上記の定式化では、シンボルλjは j 番目 の再構成画像の波長を表しています。 λmaxとλの値が½フルオレセインのサンプルの発光スペクトルから抽出され、それぞれ、蛍光サンプルの蛍光強度が最大値の最大値と半分の時の波長に対応しています。画像のインデックスjの 最大値とjは ½それぞれ、最大蛍光強度と最大の半分を有する再構成画像に対応しています。
4。興味の生体試料に関するデータの収集
- あなたのサンプルのデータを収集するには、まず、形質転換酵母のコロニーをインキュベーターからプレートを取り外します。形質転換細胞全幅半値全幅(FWHM)の少なくとも3つのタイプがあるはずです。
- 蛍光プローブの両方のタイプでタグ付けされた目的のタンパク質を発現する細胞
- ドナー蛍光プローブでタグ付けだけタンパク質を発現する細胞、および
- アクセプター蛍光プローブでタグ付けだけタンパク質を発現する細胞。
- マイクロ遠心チューブに100mMのKClを100μLを加える。マイクロピペットの先端を使用して、ドナーとアクセプター蛍光プローブの両方でタグ付けされたタンパク質を発現する細胞のプレートの3-5酵母のコロニーをこすり落とす、およびこれらの細胞を100mMのKClを接種する。
- 細胞懸濁液の10μLを削除し、新鮮な顕微鏡スライド上に分配する、カバーグラスで液滴をカバーし、カバースリップの表面に油の液滴を配置。
- 顕微鏡対物に到達するレーザー光を遮断するレーザービームの経路にシャッターを手動で閉じます。
- 顕微鏡対物は、イマージョンオイルの滴に接触するまで、XYZ移動ステージに顕微鏡スライドを固定し、光軸方向にスライド/ステージを翻訳。
- サンプルの広視野画像が深刻なため、排出経路の透過型グレーティングの存在がぼやけされます。したがって、透過型グレーティングを先行する光路に小さな半値幅でバンドパスフィルタを配置。広視野光照明をオンにし、データ収集のカメラのビデオモードにアクセスしてください。細胞が焦点に持って来られるまで、カメラの画面上のセルの画像を見ながら徐々に光軸方向の並進ステージを翻訳
- レーザービームの焦点の位置に単一のセルを実現するために x または y方向にステージを翻訳。
- 広視野の照明源をオフにしてください。排出パスからバンドパスフィルタを削除します。
- 同時にCCDアレイで受信した信号を見ながら、短い時間(<1秒)のためのレーザービームをブロック解除します。蛍光信号はレーザービームがセルに入射する時にCCDアレイで検出されている場合、この細胞の完全な蛍光データ収集のスキャンを実行します。それは、カリフォルニア州の決定と同じスキャンパラメータ、線とyの増分の具体数を、使用することが重要です。秤動の手順は、以前に示した。
- ドナーのタグとアクセプターの両方のタグに接続されているタンパク質を発現する細胞の数が多いため、セルの位置と蛍光データ収集のプロセスを繰り返します。
- 両方の受容体でタグ付けされたタンパク質を発現する細胞の蛍光画像を蓄積した後に、アクセプター蛍光プローブでタグ付けだけタンパク質を発現するドナー蛍光プローブと細胞でタグ付けされた唯一のタンパク質を発現する細胞の両方のためにプロセス全体を繰り返します。
- セクション3で説明した手順を使用して、データの各セットのスペクトル解決XY蛍光強度空間マップを得るためにすべてのスキャンを再構築する。
5。画像の解析
XY蛍光強度空間マップ、蛍光生体細胞の単一のスキャンからの結果、のそれぞれのピクセルは、その特定のピクセルに対応する励起のボクセル内に存在する分子複合体の完全なスペクトルプロファイルが含まれています。興味の蛋白質が互いに相互作用している場合は、このスペクトルプロファイルは、ドナーとアクセプター蛍光プローブの両方からの信号の混合物が含まれます。興味のあるタンパク質間の相互作用の性質を決定するために、セル内のこれらのタンパク質複合体の多数からのスペクトルプロファイルは、サンプリングされ、見かけ上は、励起状態の割合として定義された効率を(E app)を、FRETをする必要があります。ドナーの各ピクセルの、FRETを経由してアクセプター蛍光プローブに転送するようになる蛍光プローブを計算する必要があります。
- 唯一のドナー蛍光プローブでタグ付けされたタンパク質を発現する細胞は、セクション4で説明した再構成画像の各々のバックグラウンド補正蛍光強度を決定し、最大値に正規化する。これらの再構成画像の各々が別々の波長(λj)に対応しているため、正規化蛍光強度値のこのコレクションは、I D(λj)はドナープローブの測定された発光スペクトルに対応しています。
- アクセプタープローブの測定された発光スペクトルを得るための唯一のアクセプター蛍光タグでタグ付けされたタンパク質を発現する細胞の場合は1、I(λj)を繰り返します。アクセプタープローブが、それはほとんど直接レーザー光源を用いて励起されていないように選択されているので、アクセプタのみサンプルから発せられる信号は、セル固有の自家蛍光信号と同じ桁数におそらく低くしてとなります。したがって、このように算出された測定されたアクセプタの蛍光スペクトルは、先に進む前に、自家蛍光の寄与を除去するために修正する必要があるかも知れません。
- 手順1と手順2で取得した正規化スペクトルを用いて、スペクトルの関係I(λj)は= kのDAを使用してアクセプター蛍光プローブでタグ付けされたドナー蛍光プローブとタンパク質でタグ付けされた両方のタンパク質を発現する細胞の蛍光発光スペクトルの各ピクセルを分解するI I(λj)は蛍光スペクトルを測定したそれぞれの特定のピクセルを表す。D(λj)は+ K AD I A(λJ) K DAと k ADの値は、受容体の存在下でそれぞれドナー、5の存在下でアクセプターからドナーからの蛍光発光に比例しています。
- 式を用いて、各ピクセルでの細胞内のタンパク質の相互作用をマッピングする明白なFRETの効率を計算し、
 。ここで、Q、D、Qはドナー蛍光プローブとアクセプター蛍光プローブの量子収率はそれぞれであり、そしてW D及びwは 、それぞれ19、20、正規化ドナーとアクセプターの発光スペクトルの積分です。
。ここで、Q、D、Qはドナー蛍光プローブとアクセプター蛍光プローブの量子収率はそれぞれであり、そしてW D及びwは 、それぞれ19、20、正規化ドナーとアクセプターの発光スペクトルの積分です。 - 最後に、特定のE アプリの値の発生回数はE アプリケーションの特定の値に対してプロットされた各ピクセルのためのE アプリの計算値を使用してヒストグラムを作成します。
6。ヒストグラム解析
E アプリのヒストグラムから量的情報を抽出するためには、それぞれのヒストグラムは、理論的には次の式に従って、ガウス関数の和でフィットされています。 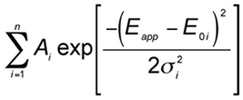 、
、
iは振幅です、 私の標準偏差、およびE 0I最も可能性は 、i 番目のガウス関数のFRET効率σは。異なるオリゴマーのサイズsと設定はE 0I及びそれらの1の間のさまざまな相関関係の異なる数字(n)は、17につながる。例えば、菱形四量体のために、五つの峰を表1に記載されている式で与えられる、予測されています。
fiveガウスピークの中央値のそれぞれとペアの間に表1の関係は菱形状の四量体のために予測効率のFRET。 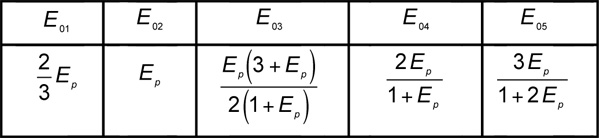
E pに対応するものを- -他の4つがE pの値から計算される間、それは1つだけE 0I値は、データのフィッティングの過程で調整する必要があることを意味します。
- オリゴマーのモデルを仮定し、E アプリのヒストグラム1をフィッティングに使用されるガウス分布の数を識別します。 E P、iを調整することでヒストグラムをフィット、 およびi、σ。ヒストグラムの相対的な配置はモデルによって固定されていることに注意してください。フィットのカイ二乗を最小化したり、他のいくつかの機能的適合度。
- 別の可能性オリゴマーのモデルとリピートステップ番号1を前提としています。
- 最高使用されるパラメータの数のデータをフィットモデルを選択してください。これは統計的検定を(そのような自由度の数あたりのカイ二乗など)を使用して必要になる場合があります。
代表的な結果
図1に示す滅菌2α-因子(Ste2p)21、22を発現する酵母細胞で実行スペクトル解決二光子顕微鏡の測定値から計算結果K DA、K AD、およびE アプリの空間マップです。
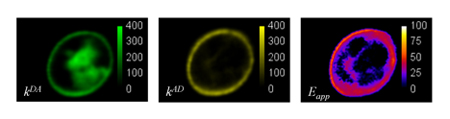
図1。無菌2α-因子受容体を発現する酵母細胞。ドナー(K DA)とアクセプター(K AD)信号の2次元の地図をスペクトル的に解決さ二光子を用いて撮影した画像の各ピクセル位置に適用されるスペクトルのデコンボリューションの手順から得られた無菌2α-因子受容体(Ste2p)を発現する酵母細胞の顕微鏡。蛍光強度は、その値にして規模をインセットとして表示されるに従って、偽色が割り当てられます。見かけフレット効率の二次元マップはk、DAと k ADの画像を用いて計算した。
ヒストグラムプロットは、図1に示すように、細胞のE アプリのマップから抽出された値を用いて調製した。図2に描か図1で描かれたセルの測定されたE アプリ値 (円)に対応するフィットヒストグラムのプロットです。赤色の実線は、個々のガウス関数(緑実線)の合計を使用して測定データに最も近似して表します。
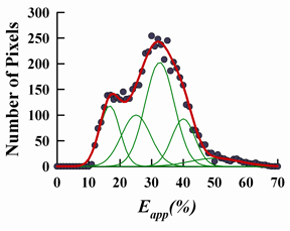
図2。滅菌2α-因子受容体を発現する酵母細胞のためのE アプリの値のヒストグラムプロットは、図 1で描かれたセルのE アプリの測定値(円)に対応するヒストグラムプロットが表示されます。個々のガウス関数(緑色の線)は、(赤色の実線)の測定値をシミュレートするために加算されます。ガウス関数のパラメータは:E 01 = 16.7、1 = 118.9、1 = 3.2、σ、E 02 = 25.1、2 = 100.0、σ2 = 4.6、E 03 = 32.6; 3 = 202.6、σ3 = 4.6; E 04 = 40.1、4 = 92.6、4 = 3.7、σ、E 05 = 50.1、5 = 16.0で、5 = 7.9、σ。
ガウス間の関係は表1に示す図2のヒストグラムで示されたデータに合わせて必要なことを意味します。ガウス関数の間の数と相関関係が菱形状の四量体のオリゴマー複合体の予想されるE アプリの値の間の数と相関関係に対応しています。したがって、それはSte2pオリゴマー複合体が in vivo 19 の菱形状の四量体の形を仮定していること、スペクトル解決二光子の測定から明らかである。
ディスカッション
本発表では、我々はin vivoでのタンパク質のオリゴマー複合体についてのサイズと構造情報を決定する方法を示します。提示されたデータは、酵母細胞で発現し、特定の膜受容体(すなわちSte2p)から得ていたが、方法は、それがセルの任意のタイプで発現するタンパク質の任意の種類、その蛋白質であることのみを規定する適用可能なすべての点で網羅です適切な蛍光マーカーでタグ?...
開示事項
謝辞
この作品は、UW -ミルウォーキーの研究成長戦略大綱、生物医学と医療技術のためのウィスコンシン州研究所、そしてブラッドリー財団によってサポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| Peptone | Fisher Scientific | BP1420 | |
| Yeast Extract | Fisher Scientific | BP1422 | |
| Polyethlyene Glycol 4000 | Hampton Research | HR2-605 | |
| Yeast Nitrogen Base w/o (NH4)2SO4 and amino acids | Fisher Scientific | DF0335-15-9 | |
| Yeast Synthetic Drop-out Medium Supplements | Sigma-Aldrich | Y2001 | |
| D-Glucose | Fisher Scientific | D16-1 | |
| Agar | Fisher Scientific | S70210 | |
| Ammonium Sulfate | Fisher Scientific | A702-500 | |
| Potassium Chloride | Acros Organics | 424090010 | |
| Leucine | Fisher Scientific | BP385 | |
| Histidine | Fisher Scientific | BP382 | |
| Plan Achromat Infinity Corrected 100x Oil Immersion Objective NA=1.43 | Nikon Instruments | ||
| Spectrally resolved two photon microscope |
参考文献
- Raicu, V., Diaspro, A. . Nanoscopy and Multidimensional Optical Fluorescence Microscopy. , (2010).
- Lakowicz, J. R. . Principles of Fluorescence Spectroscopy. , (2006).
- Raicu, V., Popescu, A. . Integrated Molecular and Cellular Biophysics. , (2008).
- Selvin, P. R. The renaissance of fluorescence resonance energy transfer. Nat Struct Biol. 7, 730-734 (2000).
- Raicu, V., Jansma, D. B., Miller, R. J., Friesen, J. D. Protein interaction quantified in vivo by spectrally resolved fluorescence resonance energy transfer. Biochem J. 385, 265-277 (2005).
- Maurel, D. Cell-surface protein-protein interaction analysis with time-resolved FRET and snap-tag technologies: application to GPCR oligomerization. Nat Methods. 5, 561-567 (2008).
- Shyu, Y. J., Suarez, C. D., Hu, C. D. Visualization of AP-1 NF-kappaB ternary complexes in living cells by using a BiFC-based FRET. Proc Natl Acad Sci USA. 105, 151-156 (2008).
- Demarco, I. A., Periasamy, A., Booker, C. F., Day, R. N. Monitoring dynamic protein interactions with photoquenching FRET. Nat Methods. 3, 519-524 (2006).
- Wallrabe, H., Periasamy, A. Imaging protein molecules using FRET and FLIM microscopy. Curr Opin Biotechnol. 16, 19-27 (2005).
- Wlodarczyk, J. Analysis of FRET signals in the presence of free donors and acceptors. Biophys J. 94, 986-1000 (2008).
- Förster, T. Experimentelle und theoretische Untersuchung des zwischenmolekularen bergangs von Elektronenanregungsenergie. Z. Naturforsch. A: Astrophys Phys Phys Chem. 4, 321-321 (1949).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2, 905-909 (2005).
- Giepmans, B. N., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312, 217-2124 (2006).
- Zimmermann, T., Rietdorf, J., Girod, A., Georget, V., Pepperkok, R. Spectral imaging and linear un-mixing enables improved FRET efficiency with a novel GFP2-YFP FRET pair. FEBS Lett. 531, 245-249 (2002).
- Nagai, T. A variant of yellow fluorescent protein with fast and efficient maturation for cell-biological applications. Nat Biotechnol. 20, 87-90 (2002).
- Rizzo, M. A., Springer, G. H., Granada, B., Piston, D. W. An improved cyan fluorescent protein variant useful for FRET. Nat Biotechnol. 22, 445-449 (2004).
- Raicu, V. Efficiency of Resonance Energy Transfer in Homo-Oloigomeric Complexes of Proteins. J Biol Phys. 33, 109-127 (2007).
- Raicu, V., Fung, R., Melnichuk, M., Chaturvedi, A. . , (2007).
- Raicu, V. Determination of supramolecular structure and spatial distribution of protein complexes in living cells. Nat Photonics. 3, 107-113 (2009).
- Rath, S., Sullivan, A. P., Stoneman, M. R., Raicu, V. Microspectroscopic method for determination of size and distribution of protein complexes in vivo. Proc of SPIE. 7378, 737829-737829 (2009).
- Kurjan, J. Pheromone response in yeast. Annu Rev Biochem. 61, 1097-1129 (1992).
- Overton, M. C., Chinault, S. L., Blumer, K. J. Oligomerization of G-protein-coupled receptors: lessons from the yeast Saccharomyces cerevisiae. Eukaryot Cell. 4, 1963-1970 (2005).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved