このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
膵島質量、サイズ分布とアーキテクチャのコンピュータ支援大規模可視化と定量化
要約
免疫組織化学染色した膵臓標本の大規模な調達と分析の新たなコンピュータ支援の方法が説明されています:セクション全体の(1)仮想スライスのキャプチャを、大規模データの(2)質量分析、2D仮想スライスの(3)復興(4)3Dの膵島のマッピング、および(5)数理解析。
要約
膵島は、β-細胞(インスリン)、α細胞(グルカゴン)、および外分泌組織の中に埋め込まれ、1を構成しているデルタ-細胞(ソマトスタチン)のようないくつかのホルモンを分泌する内分泌細胞から構成されるユニークな微小器官である - 全体膵臓の2%。体と膵臓の重量との間には密接な関係があります。全β-細胞塊はまた、体内でインスリンの需要を補うために比例して増加する。何がこの比例拡大は島のサイズ分布であるエスケープします。人間のような大型動物は、この微小器官を機能させるために一定のサイズ制限があることを示唆し、マウスと同様の膵島のサイズ分布を共有します。比例的に大きな島を生成するために大規模な動物の膵臓の無力は、膵島の数の増加と、自分たちの全体的な膵島のサイズ分布に大きな島の割合の増加によって補償される。また、膵島は、異なる種間の細胞構成とアーキテクチャにと、様々な病態生理学的条件の下で同じ種の中で印象的な可塑性を示す。本研究では、我々はそのような動的な生物学的プロセスや複雑な構造の研究では大規模で異種データ収集の分析を可能にする分析プロセスの自動化を容易にするために生物学的画像データの分析のための新しいアプローチを説明します。このような研究は、原因について、公正なサンプリングの技術的な問題に妨げと大規模データを生成する正確小島生物学の生物学的プロセスの複雑さを捉えるために設定されています。ここではサンプルの入手が限られて(またはサンプルのコレクションを最小限にする)と標準的な実験的な設定の中で公平な"代表的な"データを収集するため、および膵島の複雑な三次元構造を正確に分析する方法を示しています。コンピュータ支援自動化は、大規模データセットの収集と分析を可能にし、またデータの公平な解釈を保証します。さらに、膵島のサイズ分布と空間座標(すなわちX、Y、Z -位置)の正確な定量ができないだけ膵島の構造や組成の正確な可視化につながるだけでなく、私たちは変更の条件に、開発と適応の間にパターンを識別することができます数学的モデリングを介して。本研究で開発した手法は、同様に他の多くのシステムと生物の研究に適用可能である。
プロトコル
1。免疫組織化学的染色像の仮想スライスを作成する
- StereoInvestigatorを開きます。顕微鏡のホルダーにサンプルを保持するクリーンなスライドを配置し、"取得"し、"ライブ映像"(取得→ライブ画像)をクリックして、ステレオインベでそれを視覚化する。各チャネルの曝露レベルを決定する;私たちの具体的な例では、チャンネル2はCy7 for DAPI、GFPのチャネル3、RFPのチャンネル4、Cy5とのチャネル5とチャネル6に使用されます。露出のレベルを決定するために、蛍光の強度を表示する"ビデオのヒストグラム"ウィンドウを使用します。適切な蛍光強度に到達しているときにビデオのヒストグラムの右端にある強度の尾オフ。曝露レベルは、"カメラ設定"ウィンドウで変更することができます。ビデオのヒストグラムは、顕微鏡のフォーカスホイールで集中することができます。
- 仮想スライスを作成する前に、基準点を迎える離れてサンプルから点、で画面をクリックして、サンプルの輪郭の周囲にクリックして、サンプルを輪郭。輪郭が完了したら、右クリックし、開始点と終了点を接続するために"閉じる等高線"を選択します。
- 輪郭がクローズされると、サンプル用の仮想スライス(取得→仮想スライスを取得)をキャプチャします。オープン時の仮想スライスウィンドウのオプション、"取得のハイスピード"だけでなく、隣にあるボックスをオフに焦点を自動選択する"マニュアル。"ファイルを保存します。
- 仮想スライス定焦点にする、すなわち、サイトが仮想スライスのプレビューで、選択することにより、キャリブレーション(右クリック→サイトのリストをフォーカスするために追加)し、手動でいくつかのランダムなセクションを集中させる必要があります。いくつかのサイトがフォーカスされているときは、仮想スライス開始(右クリック→定焦点にすると仮想スライスを開始します)。最初のサンプルの仮想スライスが完了したら、次のチャンネルに切り替え、それに応じて露出を調整します。各チャネルごとにステップ4と5を繰り返します。
2。コンピュータ支援2次元解析
膵島の定量化
- 画像は、最初の分析のためにロードされたイメージを準備してから、そのような細胞組成(すなわちβ-、α-、およびデルタ - 細胞集団、および島の各エリアとして膵島の機能を定量化する"IHCVS、"名前のImageJのマクロを使用して処理されます合計値の領域)。スタックは、個々のチャネルが免疫組織化学的染色に基づいて別々の画像を表示ImageJの、で、その別々のチャンネルに分割されます。各画像は、自動的に閾値画像の8ビットの白黒画像、またはマスクが、生成された後、閾値処理される。別々のチャンネルの画像は、複合マスクに算術的に加算され、そしてImageJのビルトインパーティクル分析は、合成画像上で実行されます。
- 単一のβ細胞より小さい粒子排除しながらROIは次に識別されます(<170μmの2;原ら、2003;エリアは〜15μmの直径を用いて計算)。結果としてROIを分析し、ImageJを用いて定量され、定量化は、境界(領域を取り巻く距離)、真円度(数値1.0は完全な円を描いている丸みの度合い)、フェレーの直径(エリア内で最も長い距離)とセンターを含めることができます。分析された各領域の座標は、そのように膵島の分布を数学的にこれらのパラメータをさまざまに組み合わせて分析し、3次元グラフにプロットすることができる、データはそのうちの数学的モデリングに使用されます。単一の仮想スライス画像は30秒で分析されます。複数の画像は、ImageJのマクロ言語でサポートされているループの構文に画像処理と解析のスクリプトを埋め込むことにより、分析することができます。ループは、指定されたディレクトリ内のすべてのファイルを開き、画像を分析し、Excelファイルなどの新しい場所に結果を出力します。
- 集計結果は、画像にラベルが付いた対応する番号と共に、そのような面積、周囲、真円度、フェレーの直径、及び各島のための膵島の中心としてデータを格納し、Excelスプレッドシートに保存されます。
計算解析およびヒストグラムのセットアップ
- 一度起動すると自動的に実行されるMathematicaのスクリプトは、エントリの数十万人にのぼる、収集されたスプレッドシートのデータを処理するために使用されます。実行される重要なプロセスの一つは、サイズ分布を調べるために、また、データに外れ値を決定するために使用されるヒストグラムを構築する周波数解析、です。スクリプトの下、総膵島面積を測定し、他のパラメータと比較して、特に時間のポイントや膵臓の年齢に、それに応じてグラフで表示されます。
3。二次元免疫組織化学的染色仮想スライス画像の三次元再構成
仮想スライスの三次元再構成
- そのディレクトリ(すなわち、フォルダ)にスクリプトを("im_jp2_2_tiff")にコピーしますJP2画像が含まれています。コンソールに"./im_jp2_2_tiff"と入力してEnterキーを押すことによって、Linuxのシェルでスクリプトを実行します。このスクリプトを実行すると、自動的に仮想スライスから三次元スタックの構築と定量化に使用されるフォーマットであるTIFFファイル、にディレクトリでJP2画像のすべてを変換します。 (注:それが正常に実行できるように、スクリプトにテキストファイルを変換するスクリプトが実行されない場合に"chmod + x im_jp2_2_tiff"のタイプ、)
- 撮影した画像のすべてのJP2からTIFFファイルに変換されているときに、彼らはImageJの解析に使用する準備が整いました。
- ImageJのと、最初のサンプル(この場合は5枚の画像:DAPI、GFP、RFP、Cy5で、Cy7)のためにすべての画像を開いて、1つのイメージ(画像→色→マージチャンネル)として、それらをマージ。試料の各々に対して、このプロセスを繰り返します。サンプルのすべてが単一の画像として結合されているときは、"多角形選択"ツールで不要な領域を選択して(編集→塗りつぶし)でそれらを埋めることで画像をきれいに。 (必要に応じて、小さいサイズに画像サイズを変更します)。その後、RGBカラー画像(イメージ→タイプ→RGBカラー)にそれぞれの画像を変換する。最後に、必要に応じて、それらはさらに、"スタックレッグ"プラグインを使用して整列することができる後に、画像の外スタック(スタックのイメージ→スタック→画像)を作成します。最終的に、チャンネルをマージして、画像を組み合わせることで全体の膵臓のランゲルハンス島のすべての3Dモンタージュを作成します。画像を表示するには、"プラグイン"し、をクリックし、"3Dビューア。"
4。膵島のマッピング
画像のスタックの収集
- 顕微鏡下で全体のマウント膵島を位置づける。
- Slidebookソフトウェアを開きます。 "Ctrl + Shiftキー+ E"を押して"コントロールをキャプチャし、フォーカスを"アクセス"フォーカスのコントロール"ウィンドウでは、膵島の大きさに応じて、20倍または40倍、に目標を設定します。水浸対物レンズを使用している場合は水を適用します。
- 島を見つけるために顕微鏡の接眼レンズを使用するためには、"ビン""フォーカスのコントロール"ウィンドウで、2xおよびUser1にフィルタセットを設定することを設定します。 "排出量の選択は、"100%の目とニュートラルデンシティに1に設定する必要があります。 "GFP EY"をクリックし、"開くフルーア。"
- Slidebookのサンプルを視覚化するために、フォーカスに戻すウィンドウを制御し、"ライブ"とクリックして"フィルタセット"切り替える"GFP DSを。"中央にカメラ1ウィンドウ内の膵島を可能な限りジョイスティックを使用してください。どこかのセルを簡単に識別できる膵島の中心部に近い深さに焦点を当てる。
- で"フォーカスコントロール]ウィンドウ、""Zのタブ"を選択します。基準点としての現在の深さを設定するには、ウィンドウの左上隅に設定]をクリックします。機能が明確に識別できるれた膵島の一番上の深さにスクロールし、クリックして、スコープのコントロールを使用して、"トップを設定してください。"膵島の一番下までスクロールし、クリックして"設定ボトムを。"基準点に戻るには"Go"をクリックしてください。
- [キャプチャ]ウィンドウで、設定された"ビンのファクターは""2倍"とする"フィルタセット"を設定する"ライブを。" "3D"(とのみ3D)に、"キャプチャの種類"を設定します。ウィンドウの右側に、"トップとボトムの位置を使用"をクリックします。 "キャプチャした後、ポイントを参照に戻る"をクリックします。に、"ステップサイズ"を設定して"3。"
- 下にある"フィルタセットボックス、"DAPI DSU、GFP DSU、RFP DSU、およびCy5 DSUを選択。それぞれについて、"露出を調整し、"選択した露出が適していることを確認するために"Test"をクリック下の"ベスト検索"をクリックします。キャプチャを開始する"スタート"をクリックします。
- キャプチャが完了すると、必要に応じてレベルを調整し、白にRFPのための表示色を変更。スライドを保存し→エクスポート→TIFFシリーズの表示に進みます。最後にダッシュでスライドの名前を入力して保存します。個々のTIFFファイルの数十が作成されているので、別のフォルダに各膵島の画像のスタックを維持すると便利かもしれません。
マッピング画像のスタック
- ステレオ調査官を開きます。画像スタックを(ファイル→画像のスタック→画像のスタックを開く)開いて、イメージを視覚化する。 3.00μmまでの"イメージスケーリングのメニュー、"セット"の画像間の距離"。 Slidebookで2Xビニングを補正するために、"上書きXとYのスケーリング"とスケーリング"XとYのソースに指定されたユーザーを"選択し、XとYの両方のために0.65を入力してください
- センターへの関心の膵島の途中でクリックして画像"マクロウィンドウ。"適切なマーカーを使用して少なくとも1つのセルをマーク。 β-細胞が緑色に表示される、α細胞が赤色表示され、デルタ細胞は白色表示されます。使用マーカー2(中空円)α細胞をマークするβ細胞、マーカー5(中空の三角形)をマークすると、マーカー6(デルタ細胞に中空の三角形)。細胞は、サンプルをDAPIで染色されている場合は青色に表示される彼らの核の中心部にマークされている必要があります。
- 少なくとも一つのセルがマークされたら、上部メニューのアイコンを使用して、"直交View"を表示。 "Z -フィルター"と確認して"対称を。"範囲のhould 15.00に設定する。 0.0 Zのレベルから、マウスのホイールを使用して膵島をスクロールし、適切なマーカーで各セルをマーク。各セルは、その深さの中央に一度だけマークする必要があります。 Z -の各レベルをマーキング仕上げ時には、"Z -フィルター"チェックボックスをオフにすることができます。これはZ -レベルのすべてのマーカーのすべてが表示されます。
- すべてのセルにマークが設定されているときは、"DAT"ファイルとしてファイルを保存し、→エクスポートトレースをファイルに進みます。 "TXT"ファイルとトレースを保存します。新しい島をマップしようとする前にワークスペースを消去するには、"新規データファイルを"クリック。
5。代表的な結果:
免疫組織化学染色した膵臓のサンプルの仮想スライスの準備よりは両方一緒に膵島(図1A)として、個別に別々のチャネルで、全体の膵臓の内分泌細胞(α、β、およびδ-細胞)のすべてを検査するものを可能にします(図1B)。コンピュータのプログラムやスクリプトの助けを借りて、大規模データの質量分析は、これらの仮想スライスを行うことができます。特に、複合マスク(図1C)の粒子解析では、膵島面積、周囲(地域を取り巻く距離)、循環(1.0は完全な円を表す真円度、)などのパラメータが含まれている統計表として出力し、 (図1D)検出されたそれぞれの島のためのフェレーの直径(エリア内で最も長い距離)。合計膵島数とサイズ分布のヒストグラムと同様にα-、β-、およびデルタ - セル領域の詳細な比較の生産におけるこれらのイメージの結果の大規模解析。さらに、各仮想スライスは約5μmの深さで撮影され、そして個々の2D仮想スライスのすべては、さらに全体の膵臓サンプルの3D再構成を作成するために積まれている。膵島マッピングは、3Dでの島を捉えるだけでなく、別のインスタンスを示していますが、さらに詳細なコンピューター支援解析。膵島のマッピングは、異なる島のキャプチャ(図2A)およびα-、β-、および3D(図2C、D)の膵島を可視化するために様々なZ -平面(図2B)におけるδ-細胞のマーキングの後続で構成されています。マップされた膵島の自動化された数学的解析は、細胞間の距離(図2E)と細胞間の距離の分布(図2F)の累積確率を含めて、それらの細胞構成とアーキテクチャを表示します。
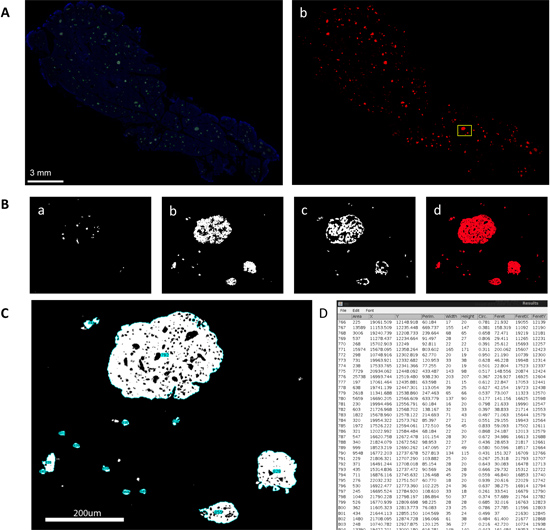
図1仮想スライスを使用して膵島の分布の大規模なキャプチャと分析。ヒト膵臓部のA.バーチャルスライスビュー。 A.インスリンの免疫組織化学的染色(緑)、グルカゴン(赤)、ソマトスタチン(白)とDAPI(青)。 B.自動二値化後の8ビットマスクを変換する。箱入りの面積は、各チャネルのBB回数で拡大されます。 A.デルタ細胞、Bのβ-細胞、C. α-細胞、およびD.合成画像をマージ。 C.パーティクル分析は、複合マスク時に実行。小細胞クラスターを含む各膵島の構造が(青色のハイライト)番号が付けられていることに注意してください。 C.に示すようにタグに対応するIDを持つ個々の構造物について測定された様々なパラメータのD.統計表、

図2。免疫組織化学的仮想スライスの分析。エリア、真円度とフェレーの直径などのパラメータによって、各島の大きさと形状分布を示す図1のAの3次元の散布図。携帯膵島構成とサイズを示す図1のBの3次元散布図。図1から全体の人間のセクション分析のC.膵島のサイズ分布は、対数正規確率密度分布に装着。携帯組成比のD.数理解析(β-細胞における緑、青に赤とデルタ細胞におけるα-細胞)図1の各膵島有効径ビン用。 E. A.ランダムサンプリング免疫組織化学的分析の膵島のサイズ分布(左)。仮想スライスの分析の膵島のサイズ分布(右)。 B.ランダムサンプリング免疫組織化学的分析(赤)と仮想スライス(青)の対数正規プロットの比較。
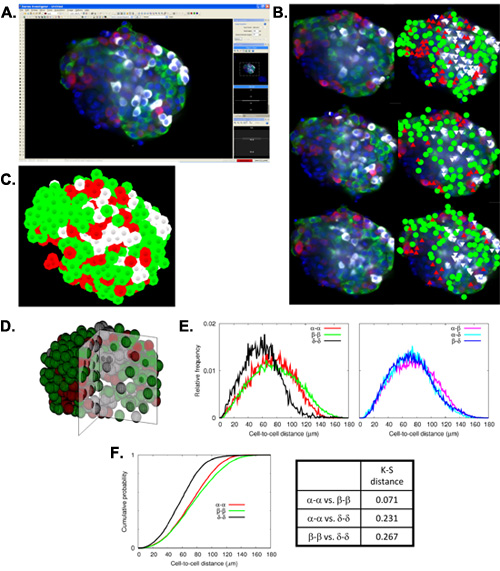
図3。膵島のマッピングと細胞構成と建築の数学的分析。 :ステレオ-調査官にアップロードされたヒト膵島の画像の3次元再構成されたスタック(赤の緑、α-細胞におけるβ-細胞、白のデルタ細胞、および青色の核)から単一の焦点面を示すスクリーンキャプチャ。 B:蛍光画像(左)と10μmの間隔で示すように、3つの異なった焦点面内の対応するマップされたデータ(右)。 C:3D再構築された膵島のマッピングデータの代表ビュー。 D:小島マッピングによって得られた座標に基づいて四半期スライス膵島の三次元再構成。 E:細胞構成とアーキテクチャの数理解析。 LEFT。単一の細胞集団の2つのセルの間に細胞間の距離の相対度数。右側。二つの異なる細胞集団の間で細胞間の距離の相対度数。 F:コルモゴロフスミルノフ(KS)テスト。残しました。 α-へ-α、β-に-β、およびデルタからデルタ細胞に対する細胞間距離分布の累積確率。右側。対応する三つの累積確率のためのKSの距離。
ディスカッション
コンピュータ支援大規模可視化と定量化は、膵島の研究で4つのキーポイントを買う余裕がここに提示さ:(1)膵標本の大規模解析は、全体的な膵島のサイズ分布と膵島のアーキテクチャの包括的なビューを提供します。 (2)3次元再構成と細胞構成と建築の数学的解析は、さらに膵島内の内分泌細胞の空間配置の検討を促進する。 (3)異なる種の間で、様々な病態生理学的条件下で、同じ...
開示事項
謝辞
研究は、シカゴの糖尿病研究研修センター(動物モデルコア)、およびKovlerファミリー財団からの贈り物の大学に米国公衆衛生サービスグラントDK - 081527、DK - 072473とDK - 20595によってサポートされています。
資料
| Name | Company | Catalog Number | Comments | |
| Fluorescent microscope | Microscope | Olympus Corporation | IX-81 | |
| Stereo Investigator | Program | MBF Bioscience | ||
| MIP-GFP mice | Mice | Jackson Laboratory | ||
| Mathematica | Program | Wolfram | ||
| Image J | Program | National Institutes of Health | ||
| Slidebook | Program | Olympus |
参考文献
- Steiner, D. J., Kim, A., Miller, K., Hara, M. Pancreatic islet plasticity - Interspecies comparison of islet architecture and composition. ISLETS. 2, 135-145 (2010).
- Kim, A., Miller, K., Jo, J., Wojcik, P. l., Kilimnik, G., Hara, M. Islet architecture - a comparative study. ISLETS. 1, 129-136 (2009).
- Kilimnik, G., Kim, A., Jo, J., Miller, K., Hara, M. Quantification of pancreatic islet distribution in situ in mice. Am J Physiol Endocrinol Metab. 297, E1331-E1338 (2009).
- Hara, M., Dizon, R. F., Glick, B. S., Lee, C. S., Kaestner, K. H., Piston, D. W., Bindokas, V. P. Imaging pancreatic beta-cells in the intact pancreas. Am J Physiol Endocrinol Metab. 290, E1041-E1047 (2006).
- Hara, M., Wang, X., Kawamura, T., Bindokas, V. P., Dizon, R. F., Alcoser, S. Y., Magnuson, M. A., Bell, G. I. Transgenic mice with green fluorescent protein-labeled pancreatic beta -cells. Am J Physiol Endocrinol Metab. 284, E177-E183 (2003).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved