このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
初期段階のマウス胚を用いて組織特異的遺伝子のためのクロマチン免疫沈降アッセイ
要約
我々はマウス胚組織における組織特異的遺伝子発現の開始中または後に組織特異的遺伝子に要因の相互作用を識別するために、クロマチン免疫沈降(ChIP)法を示しています。それは正常な胚発生時に発生するとして、このプロトコルは、組織特異的遺伝子の活性化の研究に広く適用可能であるべきである。
要約
クロマチン免疫沈降(ChIP)はタンパク質を識別するための強力なツールです:1-3生きた細胞のコンテキストで発生するクロマチンの相互作用。この手法は、広く一次組織で、組織培養細胞の中で利用し、より少ない程度にされています。特に開発の早い時間にげっ歯胚組織、チップのアプリケーションは、組織の限られた量と胚の細胞と組織型の不均一性によって複雑になる。ここでは、解離胎生8.5(E8.5)の胚を用いてChIPを行うための方法を提示する。クロマチンの相互作用:単一のE8.5胚からの断片化クロマチンは、コントロールおよび特定の蛋白質の調査のための調査員に十分な材料を可能にするfiveアリコート、するに分けることができます。
組織特異的遺伝子発現プログラムの仕様中にクロマチンの相互作用:我々は、タンパク質を文書化し始めるためにこのテクニックを利用してきた。すべての相互作用が発生するかどうかといった区別なく、クロマチンの相互作用、のサブセット、または単セルのタイプ(S):結果は、タンパク質の検出であるため、胚の細胞型の不均一性は、必ずしもこの手法の適用を制限する。しかし、組織特異的遺伝子発現の開始時または、以下の組織特異的遺伝子の検査は、2つの理由で実現可能です。最初に、組織特異的因子の免疫沈降は、必ずしも要因が発現する細胞の種類からクロマチンを分離します。遺伝子の活性化に関連付けられている第二に、コアクチベーターとヒストンの免疫沈降を含む翻訳後修飾は、遺伝子と遺伝子がされているか、アクティブ化された細胞のタイプの遺伝子調節配列で発見されるべきである。テクニックは、ほとんどの組織特異的遺伝子の活性化のイベントの調査に適用可能であるべきである。
後述の例では、我々は、骨格筋特異的遺伝子のプロモーターにおける結合因子を調べるためにE8.5及びE9.5のマウス胚を利用した。体幹と四肢の骨格筋が形成する元となる前駆組織である体節は、、E8.5 - 9.5 4,5で存在している。 Myogeninは、骨格筋の分化6月9日に必要な調節因子である。データはmyogeninがE8.5とE9.5の胚においてそれ自身のプロモーターに関連付けられていることを示しています。 myogeninが唯一の開発6,10のこの段階で体節で表されているため、データはそれ自身のプロモーターとmyogeninの相互作用は既にE8.5胚における骨格筋の前駆細胞で発生したことを示している。
プロトコル
1。胚の単離
注 :マウスを含むすべての操作が適切な動物のケアと使用ポリシーとプロトコルに従い実施すること
- 雌マウスの交配のプラグインの存在交配後の朝にチェックして、別のケージに入れて、スタッドの男性から交尾の雌を分離。嵌合プラグが観測された日の正午は、開発の胎生0.5(E0.5)とみなされます。
- E8.5で、(または必要な段階、異なる場合)、承認されたプロトコルを使用して、マウスを犠牲にする。
- 70%エタノールで安楽死させた動物の腹を濡らし、腹腔を開く。胚の存在を示す着床部位が各子宮角(図1A)の長さに沿って配置"糸に通したビーズ"として見られている。はさみを使用すると、両方の子宮角を取り除く分離するために個別に各移植部位を切断(図1B)と室温(RT)を含む100mMペトリ皿の中に置いてください解剖の培地(DMEM、10%ウシ胎児血清、20mMのHEPES、pH7.4、中、60μg/ mlのペニシリン、2mMのストレプトマイシン)。
- 鉗子を使用して、子宮組織(図1C)から各胚および周囲の膜を除去し、新鮮な解剖の培地の中に置いてください。
- 鉗子を用いて解剖顕微鏡下で個々の胚を隔離する。開発のこの段階で、胚はコンタクトdeciduum組織、内臓卵黄嚢と羊膜頭頂卵黄嚢に囲まれています。羊膜は胎児と直接接触している透明な膜です。内臓卵黄嚢は、羊膜と頭頂卵黄嚢の間に位置しており、容易に胚を育てる著名な血管の存在によってE8.5 - E9.5の胚で区別される。その周囲の膜からそれぞれの胚を取り出して、余分な血液を除去するために解剖のメディアを含む新しいプレートに個別に転送する。発達段階は胚から胚へと同腹の間で異なることがあります。代表E8.5胚及び代表E9.5の胚は、図1Dに示されています。
- 解剖のメディア(上記のステップ4で説明)の200μlを含む1.5mlのエッペンドルフチューブにそれぞれの胚を移す。
2。隔離された胚の均質化
- 各エッペンドルフチューブに、200μlの量(DPBS 1X Dubbeccoのリン酸で希釈緩衝生理食塩水)にコラゲナーゼII型の20台を追加。
- ゆっくりと20分間100rpmで37℃のシェーカーにサンプルを振る。
- 細胞の塊を破壊するためにピペッティングしてよく懸濁します。
- 1.5mlのエッペンドルフチューブの上に配置滅菌セルストレーナー(40μmのメッシュの大きさ)の上に細胞懸濁液を適用します。
- すぐに分離を完了するために、セルストレーナーの上に室温1X DPBS 600μlを適用する。ストレーナーを捨てる。
- 4で試料を遠心° Cを5分で4000 × gで。
- 上清を捨てる。
- RT 1XのDPBS 1ml中にペレットを再懸濁する。
- 室温で1分間4000 × gで試料を遠心分離します。
- 上清を捨てる。
- RT郭清のメディアを200μlにペレットを再懸濁します。
- 胚細胞を数える。平均3〜5 × 10 6 cells/E8.5胚があります。
3。クロスリンクしたクロマチン
- 1%のホルムアルデヒドの最終濃度の200μlをサンプルに37%ホルムアルデヒドの5.6μlを添加する。
- 室温で10分間サンプルをインキュベートする。
- 4℃で3分間4000 × gで試料を遠心分離します。
- 上清を捨てる。
- 80μlの/ mlのプロテアーゼ阻害剤カクテル(PIC)を含む冷1X DPBSで細胞を再懸濁します。
- 4で試料を遠心℃で3分間4000 × gで。
- 上清を捨てる。このステップでは、サンプルは-80℃で凍結することができます冷凍、架橋試料は数ヶ月間安定です。
4。超音波処理
- RT SDS溶解緩衝液(1μlのPIC/800液総量で新鮮な付加PICと50mMのトリス- HCl(pH 8.1)、10mMのEDTA及び1%SDS)100μlに細胞ペレットを再懸濁します。再懸ときは、再懸濁を促進するためのRT SDS溶解バッファーの追加の100 ulを加える。ピペッティングし、反転によってよく混ぜる。
- 氷上で10分間細胞懸濁液をインキュベートする。
- 200〜DiagenodeのBioruptorを使用して500塩基対にせん断DNAに溶解した細胞を超音波洗浄™UCD200(5分× 8回:高(320ワット)電源オン30秒on/30秒の振幅の設定はオフ)。サンプルは、氷スラリーで囲む必要があります。サンプルは4 ° Cを超える温めたり、フリーズしたりしないでください。利用可能なsonicatorsの多くの種類があります。超音波処理の条件は、約500 bpの長さに架橋DNAが切断されるため、各ソニケーターのために最適化する必要があります。
- 4で超音波処理試料を遠心℃で10分間、14000 × gで。
- T新鮮なエッペンドルフチューブを氷上で冷却した上清をケース温度。
- 5アリコートの最大に超音波処理サンプルを分ける。我々は経験的にone E8.5胚は5アリコートに分割できることを決定した。小規模または大規模なサンプルサイズは、それに応じて拡大縮小することができます。このステップでは、サンプルは-80℃で保存することができます℃でさらに使用するまで。
- クロスリンクが逆になるステップ6まで-20℃での入力や店舗として使用するための準備one分量。 1μl/800で新鮮な付加PICを含むチップ希釈バッファー(16.7 mMのトリス- HCl(pH 8.1)、1.2 mMのEDTA、1.1%トリトンX - 100、167 mMのNaClおよび0.01%SDS)で10倍、他のアリコートを希釈するμlのBuffer。
5。事前にクリアし、免疫沈降
- サーモン、精子のDNA /タンパク質4時またはGアガロース- 50%のスラリー° C、回転と、1時間の75μlの事前クリア希釈上清。またはプロテインGビーズをチップに使用する抗体によって決定されるべきどちらタンパク質の使用。プロテインGビーズがヤギの抗体またはマウスIgG 1抗体のために推奨されているのに対し、例えば、タンパク質ビーズを、ウサギの抗体をお勧めします。詳細については、ビーズの製造業者の推奨事項を参照してください。
- 1分間2500 × gで4℃で遠心する。
- 新しい、事前に冷却エッペンドルフチューブに上清を移す。
- 事前にクリアアリコートに抗体(4μgの/サンプル)を追加し、4℃で一晩インキュベート° Cを回転した。
6。クロマチンタンパク - ビーズ複合体を洗う
- 4時に各バイアルとインキュベートにサケ精子DNA /プロテインAまたはGアガローススラリーの60μlを加え、℃で1時間の回転とC。上記の手順3で事前にクリアするのに使用されるのと同じビーズの種類を使用してください。
- 1分間1,000 × gで4℃で遠心する。
- 注意深く上清を除去して捨てる。氷の上にDNA -タンパク質 - ビーズ複合体をしてください。
- としての洗浄バッファー1、2、3、4で以下の指示ペレットを再懸濁し、洗浄する。 1〜3 ° Cとこれらのバッファで洗浄を4で実行する必要がある4に冷やしすべきバッファ℃を洗うバッファ4は、このバッファーで室温と洗浄にあるはず洗浄は室温で行ってください。各洗浄は適切な温度で回転で5分間です。 4℃(洗浄バッファー4回洗浄し、室温)1分間1000 × gでの各洗浄、遠心分離に続いて、その後、慎重に吸引したり、上清を取り除く。
バッファ1:低塩の洗浄緩衝液(20mMトリス- HCl(pH 8.1)、2mMのEDTA、1%トリトンX - 100、
150mMのNaCl及び0.1%SDS)、1ミリリットル× 2回洗浄。
バッファ2:高塩の洗浄緩衝液(20mMトリス- HCl(pH 8.1)、2mMのEDTA、1%トリトンX - 100、
500mMのNaCl及び0.1%SDS)、1ミリリットル× 2回洗浄。
バッファ3:塩化リチウム(LiCl)の洗浄緩衝液(10mMのトリス- HCl(pH 8.1)、1mMのEDTA、1%IGEPAL - CA630、
0.25 M塩化リチウム、1%デオキシコール酸(ナトリウム塩))、1ミリリットル× 1洗浄。
バッファ4:TE緩衝液(10mMトリス- HCl(pH 8.1)、1mMのEDTA)、1ミリリットル× 2回洗浄。
7。クロマチン - 抗体複合体の溶出
- 新しく調製した溶出バッファー(1%SDS、0.1 M 炭酸水素ナトリウム)を250μlで洗浄したクロマチンタンパク-ビーズ複合体を再懸濁します。その後、回転で15分間室温でインキュベートする5秒間激しくボルテックスサンプル、。
- 室温で1分間2500 × gで遠心し、新しいエッペンドルフチューブに溶出液を移す。
- ステップ1と2を繰り返し、同じエッペンドルフチューブに溶出液を組み合わせる。溶出液を室温で維持する必要があります。このステップの完了時に、溶出液をさらに使用するまで-20℃で保存することができます。
8。クロスリンクを逆にしてDNAを回収する
- 500μlの溶出液(入力制御のための40μL/ mL)について、20μlの5 M NaClを追加します。
- ℃で一晩に4時間、水浴で65℃溶出液を加熱する。
- 各サンプル(入力制御を含む)10μlのDNA断片のサイズをチェックするために0.1から1 kbpの範囲をまたがるサイズマーカーの隣の2%アガロースゲル上で実行することができます。 DNAがスメアではなく鋭いバンドとして表示されます。ゲルの実行中にサンプルの残りは65 ° Cで残すことができます。
- QIAquickゲル抽出キット(キアゲン)を用いて各サンプルからDNAを回収する。 QIAクイックスピンカラムと500μlのサンプルにイソプロパノール200μlのシリカメンブレンにDNAを吸着を最適化するために、600μlのQGのバッファを(キットに付属)を追加します。 QGのバッファは、サンプルのpHを簡単に測定が可能pH指示薬が含まれていることに注意してください。サンプルの色が紫に黄色から判明した場合(pH値> 7.5)を混合した後、試料と混合するための3 M酢酸ナトリウム10μlの液(pH 5.2)に追加します。これは、列のビーズへのDNA結合を最大化するために混合物のpHを減らすことができます。
- 列をロードする前に、任意のSDSの沈澱物が存在するかどうかを判断するために混合物を確認してください。 SDS沈殿が発生した場合、サンプルはで暖めすべき42℃の水浴沈殿が消えるまで。
- QIAクイックスピンカラム(キットに含まれる紫色の列)に混合物を700μlをロードし、1分間14000 × gで室温で遠心。先に進む前に、カラムからのフロースルーを捨て、残りのサンプルでこの手順を繰り返します。
- カラムを洗浄するために500μlのQGのバッファを追加。 2分間14000 × gで室温で遠心する。次に進む前に、カラムからのフロースルーを捨てる。
- メーカーの指示に従ってエタノール追加されているPE洗浄緩衝液(キットに付属)の700μlを持つカラムを洗浄します。 1分間14000 × gで室温で遠心する。次に進む前に、カラムからのフロースルーを捨てる。
- 完全にカラムからPEのバッファの残りを除去するために遠心分離のステップを繰り返します。フロースルーとコレクションチューブを捨てる。
- 60μlのEBのバッファー(キットに付属の溶出バッファー)を加えることで新鮮なエッペンドルフチューブにカラムからDNAを溶出する。
- 1分間14000 × gで室温で遠心する。溶出したDNAを検出するまで-20℃で保存することができます。回収された物質260の測定値は通常、一定分量あたり〜6月7日UGの合計回復のために、90〜120 ngの/ ULの濃度を示している。しかし、これらのサンプルの280分の260比はRNAがサンプル中に存在することを示唆し、通常は> 1.8です。こうして回収されたDNAの正確な量が少なくなることがあります。
9。回収したDNAの解析
- SDSはPCR Taqポリメラーゼの活性を妨害することができます。 SDSはPCRを実行する前に完全に削除する必要があります。 1分間14000 × gで4℃、残存SDSと遠心° Cを沈殿させるために氷の上にDNAをインキュベートする。新しいエッペンドルフtube.Thisのステップに上清を移すことを強くお勧めします。
- DNAの溶出液は、従来のPCR法によって、または、解析を行うそれぞれの配列に特異的なプライマーを用いた定量的リアルタイムPCR(Q - PCR)のいずれかによって検出することができます。全DNAの溶出液から5〜10μlのは、1つのPCRまたはQ - PCR反応に用いることができる。入力制御を用いたPCRは、他の試料の量と比較してDNAの5〜10倍希釈で行うことができます。 PCRおよびQ - PCRの条件は、しかし、出発原料の限られた量は、追加のPCRサイクルを必要とする、変化します。従来のPCR検出のために、我々は40サイクルから始め、必要に応じて調整することをお勧めします。
10。代表的な結果
我々は、E8.5とE9.5の胚(図2)の両方からChIPを行うためにこのプロトコルを使用している。結果はmyogeninがE8.5とE9.5の胚におけるmyogeninプロモーター上に存在していることを示している。沈降精製DNAは、従来のPCR(図2A)および定量的リアルタイムPCR(図2B)によって分析した。これとは対照的に、myogeninが発現していない卵黄嚢、のmyogeninプロモーターへの結合myogeninの兆候はなかった。インターフェロン-γ(IFNγ)myogenin結合部位に一致するシーケンスが含まれているプロモーターは、負のシーケンス制御として使用された。予想通り、myogeninは、テストされた組織サンプルの任意のIFNγプロモーターに結合されていませんでした。骨格筋の前駆体である、、E8.5および9.5胚では、myogeninは、特に体節(6,10図3)で表されます。したがって、結果はmyogeninがE8.5とE9.5での体節にmyogeninプロモーターに結合していることを示している。
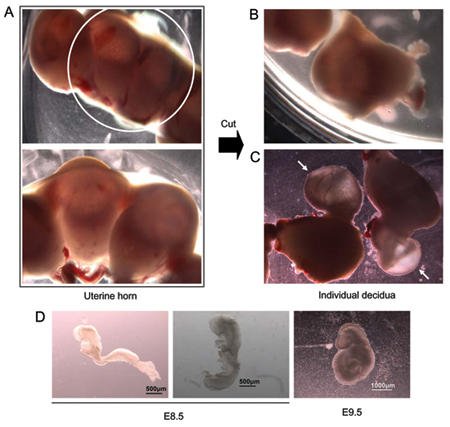
図1。E8.5胚の解剖。 ()絶縁子宮角(上部パネル、下部のパネルに示すように高い倍率)。 (B)子宮角は、個々の胚を含む個々の着床部位を分離するためにはさみで切る。 (C)胚は、解剖時の子宮組織から突出。矢印は、まだ余分な胚組織で覆われた胚をマーク。同じごみから(D)二E8.5胚。左の胚は回転のプロセスを開始していません。右側の胚は回転を受けています。右端 - 代表E9.5の胚。
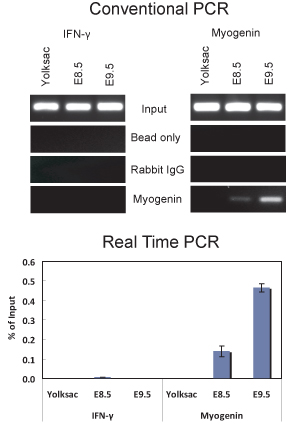
図2。ChIPアッセイがE8.5とE9.5の胚におけるmyogeninプロモーターへの結合myogeninを示す。 myogenin抗体や非特異的IgGは転写の開始部位に-79から69の相対的にmyogeninプロモーターの部分を増幅するプライマーを使用して実行されたを使用してChIPの実験から精製したDNA5μlの中(上)従来のPCR解析その-12転写開始部位の〜1075塩基対上流に位置するmyogenin結合部位に一致するシーケンスが含まれているIFNγプロモーターの部分を増幅するプライマーに位置するmyogenin結合部位が含まれています。使用されるIFNγのプライマー配列は、5' - GCT GAC TCA AGA CCC CGA GGC - 3'および5' - TGA GGA TGG GGC AGG AGG CC - 3'であった。 ()で使用したのと同じサンプルの(下)定量的リアルタイムPCR解析。市販標準偏差 - データが入力+ /の%としてプロットされます。
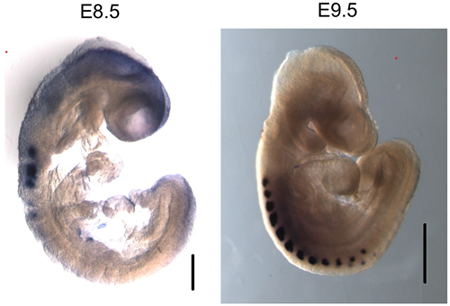
図3。Myogeninが特にE8.5及びE9.5の胚の体節に発現している。 myogeninのin situハイブリダイゼーション全体のマウントは、体節内の特定のmRNAの発現を示しています。 200μm程度 - E8.5画像のサイズのバー。 E9.5画像のサイズのバー - 500μmの。
ディスカッション
説明したChIPのプロトコールでは、我々は、筋原性レギュレータmyogeninが単一のE8.5及びE9.5の胚に存在する骨格筋の前駆体の組織におけるmyogeninプロモーターに関連付けられていることを示している。先行研究では、広範囲にin vitroでのゲルシフト実験の初期にはin vitroで翻訳や細菌生産myogeninと11月20日標的遺伝子の調節配列の該当する部分をコードする放射性標識DNA...
開示事項
謝辞
この作品は2009年のアメリカの回復と再投資法を通じて授与資金を含むANI、、とNIH R01 GM87130でたたくのNIH R01 GM56244によってサポートされていました
資料
| Name | Company | Catalog Number | Comments |
| ChIP Assay Kit | Upstate, Millipore | 17-295 | |
| Collagenase Type II | Invitrogen | 17101015 | Dilution by 1 x PBS |
| Dulbecco’s modified eagle medium (DMEM) | GIBCO, by Life Technologies | 12100-061 | High glucose content |
| Dulbecco’s phosphate buffered saline 1X (DPBS) | GIBCO, by Life Technologies | 14190-144 | Calcium chloride free, Magnesium chloride free |
| Fetal bovine serum (FBS) | Mediatech, Inc. | 35-010-CV | |
| Gel extraction kit | QIAquick | 28704 | 50 reaction kit |
| Penicillin/streptomycin stock solution | GIBCO, by Life Technologies | 5000 μg/ml concentration | |
| Protease Inhibitor Cocktail | Sigma-Aldrich | P8340 | |
| Salmon sperm DNA /Protein A agarose | EMD Millipore | 16-157 | |
| myogenin antibody | Santa Cruz Biotechnology, Inc. | sc-576 | |
| Normal rabbit IgG | EMD Millipore | 12-370 | |
| Platinum PCR Supermix | Invitrogen | 11306-016 | |
| GoTaq Q-PCR master mix | Promega Corp. | A6001 |
参考文献
- Minard, M. E., Jain, A. K., Barton, M. C. Analysis of epigenetic alterations to chromatin during development. Genesis. 47, 559-572 (2009).
- Kuo, M. H., Allis, C. D. In vivo cross-linking and immunoprecipitation for studying dynamic Protein:DNA associations in a chromatin environment. Methods. 19, 425-433 (1999).
- Johnson, K. D., Bresnick, E. H. Dissecting long-range transcriptional mechanisms by chromatin immunoprecipitation. Methods. 26, 27-36 (2002).
- Yusuf, F., Brand-Saberi, B. The eventful somite: patterning, fate determination and cell division in the somite. Anat Embryol (Berl). 211, 21-30 (2006).
- Buckingham, M., Bajard, L., Chang, T., Daubas, P., Hadchouel, J., Meilhac, S., Montarras, D., Rocancourt, D., Relaix, F. The formation of skeletal muscle: from somite to limb. J Anat. 202, 59-68 (2003).
- Wright, W. E., Sassoon, D. A., Lin, V. K. Myogenin, a factor regulating myogenesis, has a domain homologous to MyoD. Cell. 56, 607-617 (1989).
- Edmondson, D. G., Olson, E. N. A gene with homology to the myc similarity region of MyoD1 is expressed during myogenesis and is sufficient to activate the muscle differentiation program. Genes Dev. 3, 628-640 (1989).
- Nabeshima, Y., Hanaoka, K., Hayasaka, M., Esumi, E., Li, S., Nonaka, I. Myogenin gene disruption results in perinatal lethality because of severe muscle defect. Nature. 364, 532-535 (1993).
- Hasty, P., Bradley, A., Morris, J. H., Edmondson, D. G., Venuti, J. M., Olson, E. N., Klein, W. H. Muscle deficiency and neonatal death in mice with a targeted mutation in the myogenin gene. Nature. 364, 501-506 (1993).
- Sassoon, D., Lyons, G., Wright, W. E., Lin, V., Lassar, A., Weintraub, H., Buckingham, M. Expression of two myogenic regulatory factors myogenin and MyoD1 during mouse embryogenesis. Nature. 341, 303-307 (1989).
- Brennan, T. J., Olson, E. N. Myogenin resides in the nucleus and acquires high affinity for a conserved enhancer element on heterodimerization. Genes Dev. 4, 582-595 (1990).
- Rosenthal, N., Berglund, E. B., Wentworth, B. M., Donoghue, M., Winter, B., Bober, E., Braun, T., Arnold, H. H. A highly conserved enhancer downstream of the human MLC1/3 locus is a target for multiple myogenic determination factors. Nucleic Acids Res. 18, 6239-6246 (1990).
- Braun, T., Gearing, K., Wright, W. E., Arnold, H. H. Baculovirus-expressed myogenic determination factors require E12 complex formation for binding to the myosin-light-chain enhancer. Eur J Biochem. 198, 187-193 (1991).
- Chakraborty, T., Brennan, T., Olson, E. Differential trans-activation of a muscle-specific enhancer by myogenic helix-loop-helix proteins is separable from DNA binding. J Biol Chem. 266, 2878-2882 (1991).
- French, B. A., Chow, K. L., Olson, E. N., Schwartz, R. J. Heterodimers of myogenic helix-loop-helix regulatory factors and E12 bind a complex element governing myogenic induction of the avian cardiac alpha-actin promoter. Mol Cell Biol. 11, 2439-2450 (1991).
- Brennan, T. J., Chakraborty, T., Olson, E. N. Mutagenesis of the myogenin basic region identifies an ancient protein motif critical for activation of myogenesis. Proc Natl Acad Sci U S A. 88, 5675-5679 (1991).
- Lassar, A. B., Davis, R. L., Wright, W. E., Kadesch, T., Murre, C., Voronova, A., Baltimore, D., Weintraub, H. Functional activity of myogenic HLH proteins requires hetero-oligomerization with E12/E47-like proteins in vivo. Cell. 66, 305-315 (1991).
- Chakraborty, T., Brennan, T. J., Li, L., Edmondson, D., Olson, E. N. Inefficient homooligomerization contributes to the dependence of myogenin on E2A products for efficient DNA binding. Mol Cell Biol. 11, 3633-3641 (1991).
- Cserjesi, P., Olson, E. N. Myogenin induces the myocyte-specific enhancer binding factor MEF-2 independently of other muscle-specific gene products. Mol Cell Biol. 11, 4854-4862 (1991).
- Braun, T., Arnold, H. H. The four human muscle regulatory helix-loop-helix proteins Myf3-Myf6 exhibit similar hetero-dimerization and DNA binding properties. Nucleic Acids Res. 19, 5645-5651 (1991).
- Serna, d. e. l. a., L, I., Ohkawa, Y., Berkes, C. A., Bergstrom, D. A., Dacwag, C. S., Tapscott, S. J., Imbalzano, A. N. MyoD targets chromatin remodeling complexes to the myogenin locus prior to forming a stable DNA-bound complex. Mol Cell Biol. 25, 3997-4009 (2005).
- Blais, A., Tsikitis, M., Acosta-Alvear, D., Sharan, R., Kluger, Y., Dynlacht, B. D. An initial blueprint for myogenic differentiation. Genes Dev. 19, 553-569 (2005).
- Cao, Y., Kumar, R. M., Penn, B. H., Berkes, C. A., Kooperberg, C., Boyer, L. A., Young, R. A., Tapscott, S. J. Global and gene-specific analyses show distinct roles for Myod and Myog at a common set of promoters. EMBO J. 25, 502-511 (2006).
- Ohkawa, Y., Yoshimura, S., Higashi, C., Marfella, C. G., Dacwag, C. S., Tachibana, T., Imbalzano, A. N. Myogenin and the SWI/SNF ATPase Brg1 maintain myogenic gene expression at different stages of skeletal myogenesis. J Biol Chem. 282, 6564-6570 (2007).
- Davie, J. K., Cho, J. H., Meadows, E., Flynn, J. M., Knapp, J. R., Klein, W. H. Target gene selectivity of the myogenic basic helix-loop-helix transcription factor myogenin in embryonic muscle. Dev Biol. 311, 650-664 (2007).
- Metivier, R., Penot, G., Hubner, M. R., Reid, G., Brand, H., Kos, M., Gannon, F. Estrogen receptor-alpha directs ordered, cyclical, and combinatorial recruitment of cofactors on a natural target promoter. Cell. 115, 751-763 (2003).
- Ausubel, F. M., Brent, R., Kingston, R. E., Moore, D. D., Seidman, J. G., Smith, J. A., Struhl, K. . Current Protocols in Molecular Biology. , (2010).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved