Method Article
ミッションターゲットIDのライブラリを使用してmiRNAのターゲットのゲノムワイドスクリーン
要約
ターゲットIDのライブラリは、miRNAのターゲットを識別するために使用されるクローニングされたcDNAのプラスミドベースのゲノムワイドなコレクションです。ここでは、その使用してアプリケーションを示しています。
要約
ターゲットIDのライブラリは、マイクロRNA(miRNA)のターゲットの検出と識別を支援するために設計されています。ターゲットIDのライブラリは、デュアル選択融合タンパク質を、チミジンキナーゼ-ゼオシン(TKzeo)から3'UTRの下流にクローニングしたプラスミドベースのゲノムワイドなcDNAライブラリーです。選択範囲の最初のラウンドは、安定な形質転換のために興味のあるmiRNAの導入に続いて、最後には、miRNAのターゲットを含むcDNAの選択です。選択されたcDNAは、シークエンシング(ターゲットIDライブラリワークフローと詳細については、図1-3を参照)によって識別されます。
人間のトランスクリプトームの広範なカバレッジを確保するために、ターゲットIDライブラリのcDNAは、複数のヒト組織および細胞株から調製したtotal RNAのプールを使用して、オリゴdTプライミングを経由して生成されました。 1.2キロバイトの平均サイズは、0.5〜4 kbまでのcDNAの範囲を、その結果と(プラスミドマップの図4を参照)p3TKzeoデュアル選択プラスミドにクローニングした。のLiで表される遺伝子標的braryはSigma-AldrichのWebページに記載されています。イルミナシーケンス( 表3)、の結果から、ライブラリがUCSC RefGeneで21518ユニークな遺伝子の16922(79%)、10または読み出し以上(66%)と14,000の遺伝子が含まれていることを示しています。
プロトコル
1。安定細胞株のターゲットIDのライブラリと選択したトランスフェクション
1。ゼオシンキル曲線
ゼオシンを安定的にトランスフェクトした細胞を選択するために使用されています。しかし、過剰なゼオシンは、ほとんどの種類の細胞に望ましくない表現型の応答を引き起こします。したがって、キル曲線分析は、最小致死量を確立するために実行する必要があります。
- プレート1.6×10のメディアの120μlを96ウェルプレートのウェルに4セル。
- 次の日は、50μg/ mlのから1 mg / mlに適切なウェルに至るまで増加する濃度でゼオシンを追加します。
- 2日ごとに生存率を調べます。
- 3日毎にゼオシンを含む培地を交換してください。希望の時間が完了した後、細胞死を引き起こすの選択試薬の最小濃度は、その細胞の種類や実験のために使用する必要があります。我々の結果は、500μg/ mlのゼオシンは、A549のためにHeLa細胞とMCF7細胞が最適であることを示している。
2。ヌクレオと選択を介してライブラリーのトランスフェクション
- どちら表現または興味のあるmiRNAの低レベルを表現していない細胞株を選択します。ターゲットIDライブラリの安定な発現が達成された後のmiRNAは、標的の選択については、セクションBに導入されます。
- 文化は/セルを展開します。我々は、ライブラリのトランスフェクション当たり2×10 7細胞で良好な結果を得ています。
- > 80%コンフルエントである細胞をトリプシン処理し、15 mlの滅菌スクリュー張りチューブに2×10 7個の細胞を転送します。
- ペレットは、5分間200 xgで細胞をトリプシン処理。
- 培地を除去し、HBSSまたは1X PBSで細胞ペレットを洗浄する。
- 5分間200×gで遠心分離し、洗浄液を吸引除去する。
- 細胞ペレットのステップを洗って繰り返します。
- 37完全培地2 mlのプレウォーム6ウェルプレート℃に
- 各トランスフェクションのために0.5 mlチューブごとにターゲットID·ライブラリー(10μlを超えない)の2μgを追加します。
- 2×10 6細胞あたりAmaxaのヌクレオ溶液100μl(細胞特異的)の上から15 mlのチューブに再懸濁します。たとえば、10 Nucleofectionsするための試薬1 mlを加える。
- 一度反応は、プラスミド2μgのに細胞100μlを追加します。ピペットで混ぜる。
- のNucleofectorキュベットに混合物を転送します。
- のNucleofector楽器にキュベットを挿入し、細胞株(細胞生存率よりも好ま高効率)に適した最適化されたプログラムを実行します。
- 予め温めておいた培地をピペットを埋める。同じピペット、6ウェルプレートへの転送で細胞を取る。各ヌクレオに適しごとに1つずつ繰り返します。
- 一晩のインキュベーションの成長室に戻ります。
- 翌日、培地を交換し、細胞が3-5日で回復することができます。
- としてキル曲線解析から決定ゼオシンの適切なレベルを含む完全培地で培地を交換します。
- モーゼオシン選択のためのnitor細胞(細胞が死ぬ)。
- 2〜3日ごとにゼオシンで培地を交換してください。
- かつて6ウェルプレート、通路、プール内のコンフルエントと大きなフラスコ内の細胞を展開します。
- 細胞の増殖のための時間の長さは、ユーザと依存性の細胞ラインであるが、それは非常に将来のスクリーニングのためにクライオ株式を生成するためにゼオシン耐性細胞を拡大することをお勧めします(〜2-3週間、細胞株に依存)。
2。 miRNAの発現コンストラクトでトランスフェクション·ライブラリー細胞は、安定細胞株のために選択し、miRNAのターゲットを選択します。
注:ゼオシン選択はもはや必要ない、または望まれている。 miRNAの発現およびガンシクロビル(ターゲット)の選択時にゼオシンする細胞の曝露は、miRNAのターゲットが失われる場合があります。
3。ピューロマイシン、G418、およびガンシクロビルキル曲線
- 細胞が安定的にターゲットIDライブラリを発現すると野生型のためのピューロマイシンまたはG418とガンシクロビルのKill曲線を実行します。細胞。
- プレート1.6×10 120μlの新鮮な培地で96ウェルプレートのウェルに4セル。次の日puromycin/G418の0.1から10μg/ mlの、または選択したウェルにガンシクロビル2から32μMから追加します。
- 2日ごとに生存率を調べます。 3日ごとに選択試薬を含む培地を交換してください。希望の時間が完了した後の細胞死を引き起こす*選択試薬の最小濃度は、その細胞の種類や実験のために使用する必要があります。我々の結果は、0.25から1μg/ mlのピューロマイシンは、A549用に最適であることをHeLa細胞を示し、MCF7細胞、ガンシクロビルMCF7細胞と8から16μMの0.3μg/ mlのG418は、A549、HeLa細胞、およびMCF7細胞に最適です。
*注:ガンシクロビルを選択すると、低速または無細胞増殖は、完全な細胞死することなく観察することができる。彼らは活発に分裂されていないかどうかを確認するために数日間の治療上の細胞を観察します。このような場合は、その後培地中のフェノールレッドが赤のままです。それはまた気を実行する必要があるかもしれません感受性の変化が観察された場合にはターゲットIDのライブラリのLL曲線解析は、細胞をトランスフェクトした。しかし、これは細胞の種類の特定に基づいて決定する必要があります。
4。ヌクレオとターゲットの選択を介してmiRNAのトランスフェクション
- 2×10 7細胞を達成するためにターゲットIDのライブラリセルを展開し、細胞が> 80%コンフルエントになったときにトリプシン/成長する。
- 15ミリリットル滅菌スクリューへの転送2×10 7細胞は、5分間200 xgで管とペレットを突破した。
- 培地を除去し、HBSSまたは1X PBSで細胞ペレットを洗浄する。
- 5分間200×gで遠心分離し、洗浄液を吸引除去する。
- 細胞ペレットのステップを洗って繰り返します。
- 37ウェルあたりの完全培地2ml℃でプレウォーム6ウェルプレート
- 各トランスフェクションのためのmiRNAの発現プラスミド(10μlを超えない)あたり0.5 mlチューブに2μgのを追加します。 Origene microRNA発現プラスミド(ネオマイシン - G418選択)またはpBABE-ピューロ(1764プラスミドの自己複製されたmiRNAの遺伝子; Addgene)が正常に使用されています。後者については、両側に約200 bpのmiRNAのヘアピンは、ヒトのDNAからPCRクローニングした。
- 2×10 6細胞あたりAmaxaのヌクレオ溶液100μl(細胞特異的)の上から15 mlのチューブに再懸濁します。例えば:10 Nucleofectionsについては、試薬1 mlを加える。
- 一度反応は、プラスミド2μgのに細胞100μlを追加します。ピペットで混ぜる。
- のNucleofectorキュベットに混合物を転送します。
- のNucleofector楽器にキュベットを挿入し、細胞株(細胞の生存率よりも好ま高効率)に適した最適化されたプログラムを実行します。
- 予め温めておいた培地をピペットを埋める。同じピペット、6ウェルプレートへの転送で細胞を取る。各ヌクレオに適しごとに1つずつ繰り返します。
- 一晩のインキュベーションの成長室に戻ります。
- 培地を交換し、細胞が3-5日で回復することができます。
- miRNAが構築物でトランスフェクションの前に行わキルカーブテストから決定されたピューロマイシンまたはG418の適切なレベルを含む完全培地で培地を交換します。
- ピューロマイシン/ G418選択(細胞が死ぬ)のセルを監視することができます。
- ピューロマイシン/ G418 2〜3日ごとに培地を交換してください。
- かつて6ウェルプレート、通路、プール内のコンフルエントと大きなフラスコ内の細胞を展開します。
- 細胞の増殖のための時間の長さはユーザーに依存しますが、それは非常に将来のスクリーニングのためにクライオ株式を生成するためにピューロマイシン/ G418耐性細胞を拡大することをお勧めします(〜2-3週間、細胞株に依存)。
- とは、miRNAが構築するトランスフェクションの前に行わキルカーブテストから決定されたピューロマイシン/ G418、ガンシクロビルの適切なレベル(GCV)で培地を交換してください。
- 選択のセル(セルdyin監視G、)を標的とTK-ZEOのノックダウンmiRNAの。
- GCVとピューロマイシン/ G418の存在下で細胞を展開します。
- GCV選択した細胞からゲノムDNAを準備します。
- PCRは、キットプライマー(PCR増幅を参照)の挿入を増幅する。
- 標準的なシーケンシングにTOPOTAベクター(Invitrogen)にクローンまたはディープシークエンシング用のPCR産物を提出してください。
3。 PCR-増幅、選択したライブラリに挿入し、そのシーケンス
注:この手順は、ガンシクロビルの選択を生き延びてきたPCR-増幅ライブラリターゲットに実行されます。
5。 GenElute哺乳類のゲノムDNA Miniprepキット(カタログ番号G1N10)または同等品を使用して染色体DNAを調製するためにガンシクロビル選択したセルを収集します。我々は、PCRでターゲット選択したセルからのDNAとの比較のために、ネガティブコントロールとして使用するターゲットIDのライブラリが含まれていない親細胞株からDNAを調製することをお勧めします。
- PCRアム増幅プライマー1および増幅プライマー2のゲノムDNAをplify。 PCR(製品番号P0982)のために - 成功した増幅は、JumpStart REDTaq ReadyMix反応ミックスを用いて得られたされています。別のポリメラーゼが使用されている場合、条件の最適化が必要になる場合があります。 PCRのサイクリング条件のサンプルのPCRセットアップおよび表2は表1を参照してください。
- 1%アガロースゲル上でPCR産物の2-5μlを解決します。予想される製品がいくつか目に見えるDNAがバンドの塗抹標本でなければなりません。ターゲットIDのライブラリなしで細胞からのゲノムDNAのPCR産物を制御するために比較します。
- クローニングおよび/またはPCR産物の深いシーケンスを続行します。増幅産物が全く観察されなかったり、DNAを制御するために同一である場合、PCR増幅の条件(すなわち、プライマー濃度、アニーリング温度、サイクル)を最適化することができます。
6。クローニングとシーケンシング
注:私達は非常にクローニングPCR産物をお勧めしますsは、その後キットに付属の増幅プライマーを用いてクローンの少なくとも96から標準的なシーケンシングを行う。この手順は、96ウェル一晩培養し、プラスミド精製システムを使用して正常に実行されています。ディープシーケンシングが望まれている場合であっても、クローニングとシーケンシング規格の予備的結果は、ディープシーケンシングの余分な費用が保証されているかどうかを判断するために品質チェックとして使用することができます。
- クローニングPCR増幅産物(上記)のTAクローニングキット製造業者のプロトコルに従ってください。
- 有能な細菌の細胞にクローンを変換し、抗生物質含有培地上で一晩を選択します。
- 適切な抗生物質を含む培養液で個々のコロニーを分離して成長します。
- プラスミドDNAを精製する。 6.5。増幅プライマーでシークエンシング反応を行います。
- (小説TRのヒトトランスクリプトーム(既知の転写産物)とヒトゲノムとの配列のBLASTアラインメントによる遺伝子標的を同定anscripts)*
*複製のトラブルシューティング:シーケンスからの挿入の数が多いプラスミド配列である場合は、次のようなクローニング/シーケンス条件を最適化:
- PCR増幅産物(インサート)の質と量
- 連結反応
- プラスミド調製(質と量)
- シークエンシング反応条件
4。代表的な結果
のmiR-373ターゲットのライブラリ画面
ミッションのターゲットIDライブラリの性能を評価するために、miR-373のターゲットは、MCF-7ライブラリを発現する細胞から選択された。それはほとんど、あるいは全く検出可能なのmiR-373(データは示さず)を発現するため、MCF-7が選ばれました。のmiR-373は、その生物学的興味のために選ばれた。のmiR-373発現は、非転移性細胞株[2]通常は、MCF-7腫瘍の浸潤や転移を促進します。さらに、のmiR-373のマウスからオーソログのmiR-290クラスターは、マウス胚性幹細胞に関与している細胞の維持には、[3]、およびmiR-373の家族は、miR-372は、誘導多能性幹細胞[4]に書き換え線維芽細胞を促進します。最後に、我々はジンクフィンガーは、プロモーターのmiR-373発現はMCF-7細胞におけるAAVS1サイトに構築するPGKを挿入するヌクレアーゼを使用し、RNAのマイクロアレイ解析(データは図示せず)によって潜在的なのmiR-373ターゲットのリストを生成します。
ターゲットIDのライブラリは、MCF-7細胞にトランスフェクトし、細胞の安定した人口を選択してゼオシン含有培地中で増幅した。得られた細胞(MCF-7ライブラリ)は安定のmiR-373を発現するコンストラクトをトランスフェクションおよびmiR-373のターゲットを発現する細胞を濃縮するためにガンシクロビル含有培地で選択した。我々は、ネガティブコントロールMCF-7ライブラリ細胞(のmiR-373を除く)デタッチと死んだ細胞に期待されるように丸めになっていないことを観察した。しかし、彼らは、foを観察したとしてフェノールレッド含有培地がオレンジ色に黄色の電源を入れていなかったため容易に検出された、成長を止めたrは細胞のmiR-373( 図5)を発現する 。細胞は、ガンシクロビル含有培地で展開され、標的配列は、生き残った細胞から調製したターゲットIDライブラリーcDNAインサートとDNAに隣接するプライマーを用いるPCRにより単離した。 PCR産物は、イルミナの塩基配列を決定した。
表4に示すように、我々は11076ユニークな遺伝子にマッピングされた17740719 cDNAの読み取りを、得られた。これらのユニークな遺伝子のうち、2898は40以上の読み取りで検出されたため、信頼性の高いヒットと考えられた。 cDNAインサートを増幅するために使用したPCRプライマーは40であり、300塩基離れて挿入部位から読み取るために13106469ベクトルが期待されていた。このように、それはほとんど得られた配列は、ゲノム材料のより伝統的な深いシーケンスとは異なり、ベクトルからであろうと予想されます。 14%は、ベクトルまたはcDNAのどちらかにマップされませんでしたが、NCBIの非冗長ヌクレオチドデータベース(すなわち、NRヒットを含む)に合わせなかったし、わずか1%はcDNAインサートがありませんでした。
最初の比較では、ターゲットIDのライブラリで指定された一意な遺伝子の10は以前に識別のmiR-373 TarBaseのターゲット( 表5)のリストにもあることがわかった。これらの10のmiR-373のターゲットは、以前に一時的に合成のmiR-373 [5]を模倣してトランスフェクションしたHeLa細胞からRNAをマイクロアレイにより同定した。さらに、我々はAAVS1サイト(データは図示せず)からのmiR-373を発現するMCF-7細胞ではダウンレギュレート、これらの同じ10個の遺伝子を検出しました。したがって、これらの10は、miR-373の可能性が高い有効なターゲットです。仕事は、潜在的なターゲットとRT-定量PCR、ウエスタンブロット、ルシフェラーゼレポーターアッセイによって実験的に選択されたヒット曲を検証する試みの一覧を特徴付けるために進行中です。
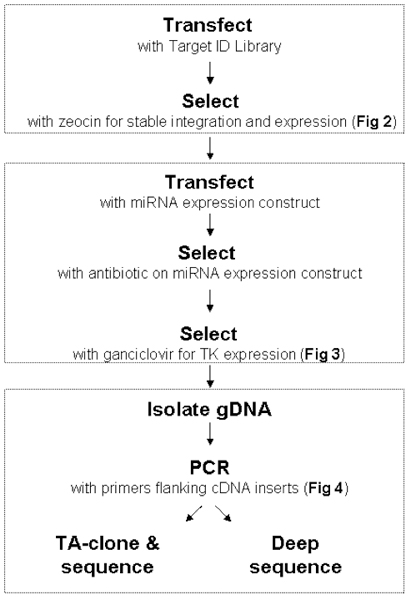
図1ターゲットIDライブラリのワークフロー。セクションAC:プロシージャ内のセクションを参照してください。各ステップは、に示し、詳細に記載されている 2と3を図。

図2。トランスフェクションおよびゼオシン選択。 ターゲットIDのライブラリは、プラスミド()、チミジンキナーゼゼオシン融合タンパク質(図4】TKzeo)の後、3'-UTRに挿入されたヒトcDNAとそれぞれのプールです。細胞は、ターゲットID·ライブラリー(B)をトランスフェクトし、3〜5日で回復するために許可されています。構文は、この回復期(C)の間にゲノムに組み込まれ、エンコードされたトランスクリプト(D)を表現することができます。回復後、細胞をゼオシン(E)にさらされています。安定的に統合されたターゲットIDの構成要素からTKzeo融合タンパク質を発現する細胞は、ゼオシン選択(F)を生き残る。トランスフェクト細胞(G)死ぬ。さらに、任意の細胞は、内在性のmiRNA又はOTHのターゲットである構築物を含むERの要因は、構造が死んでしまうのターゲットID(H)からTKzeoの発現を阻害する細胞内で発現。
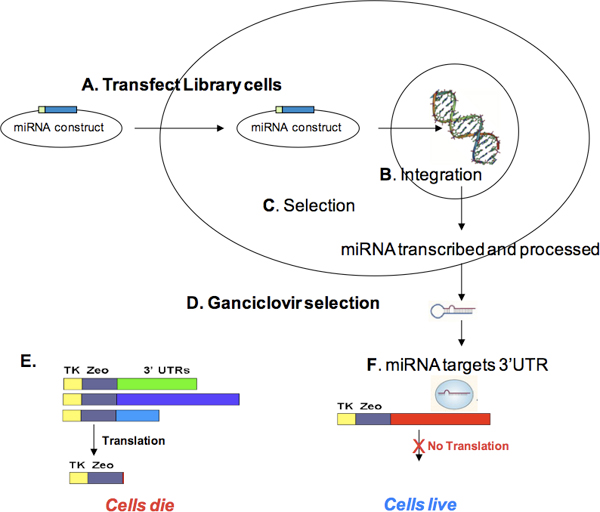
ターゲットID·ライブラリー(すなわち、ゼオシン、選択したセル)を含む図3。miRNAのトランスフェクションとガンシクロビル選択できます。セルは、選択可能なマイクロRNAの発現コンストラクト()でトランスフェクトされています。リカバリ中に、miRNA発現コンストラクトは、(B)を統合し、miRNAを構築する上でエンコードされた選択可能なマーカーを発現することができます。安定した統合(C)と細胞の増殖のために選択した後、細胞をガンシクロビル(D)で処理されています。ガンシクロビルの存在下でチミジンキナーゼ(TK)を産生する細胞(すなわち、TKzeoを発現する細胞がmiRNAの標的にされない構造)一方、(E)のmiRNAターゲットのでライブラリコンストラクトを含む細胞を死んでしまう ITESは、TKを生成しません、したがって、ガンシクロビル選択(F)を存続します。生き残った細胞は、ゲノムDNA孤立を成長させることができるし、cDNAを含むmiRNAの標的部位は、ターゲットIDの増幅プライマーを用いてPCR増幅。 PCR産物は、miRNAのターゲットを識別するために、人間のゲノムと塩基配列と整列させることができる。
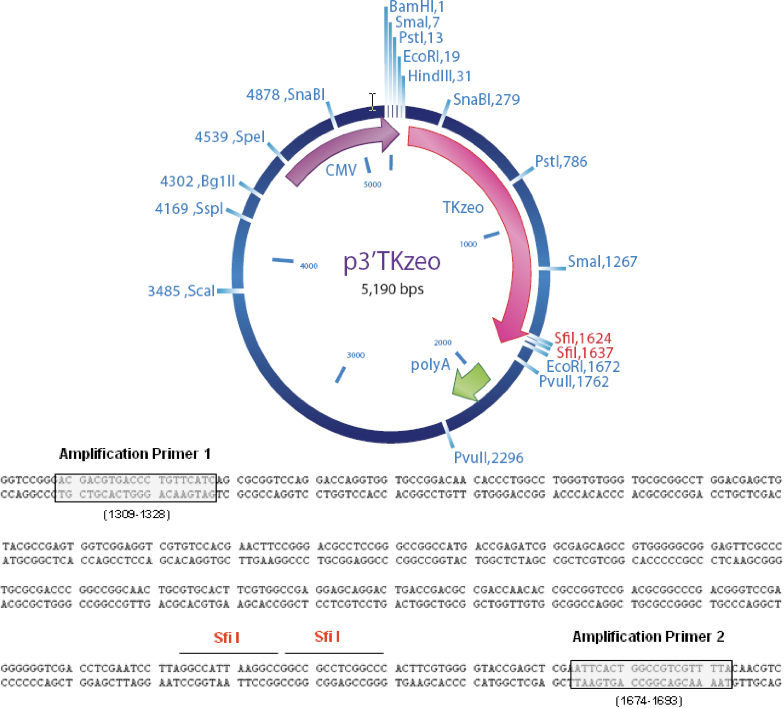
図4プラスミドマップおよび増幅プライマーの場所。 SFI私は、cDNAクローニングのサイトです。
プライマー配列
ミッションのターゲットID増幅プライマー1
5 ACGACGTGACCCTGTTCATC 3
ミッションのターゲットID増幅プライマー2
5 TAAAACGACGGCCAGTGAAT 3
ig5.jpgの "alt ="図5 "/>
図5細胞の増殖に対するガンシクロビルの影響。二十四ウェルプレートは、ターゲットIDのライブラリを含む二から十万MCF-7細胞またはMCF-7細胞を播種した。 24時間後、培地を0、8、または16μMガンシクロビルを含む培地と交換した。 15日後、プレートをブリリアントブルーR染色液(B6529)(6下井戸)のHBSSとステンドグラスで洗浄し、ウェルその後、(6上部のウェル)撮影しました。彼らはチミジンキナーゼ(TK)を発現しないため、ガンシクロビルは、ライブラリなしでMCF-7細胞には影響を与えないことに注意してください。一方、MCF-7ライブラリ細胞は急行TKを行うが、Brillinatブルーを取って生きている細胞で示すように、完全に、8または16μMでガンシクロビルによって殺されていません。しかし、ライブラリの細胞はその培地中のフェノールレッド色素の色によって証明されるように、ガンシクロビルで成長を止めないでください。逮捕された細胞は、その培地を酸性化しないし、色を変更する代わりに赤みを帯びたままであるか、アンジュ黄色。
| 試薬 | ボリューム/リアクション |
| ジャンプREDTaq ReadyMix反応ミックス | 10μlの |
| 増幅プライマー1(25μM) | 0.2μL |
| 増幅プライマー2(25μM) | 0.2μL |
| ゲノムDNA | 50から200 ngの |
| 水、分子グレード | 20μlのへ |
表1。
| 手順 | 温度。 | 時間 | サイクル |
| 初期変性 | 95°C | 5分。 | 1 |
| 変性 | 95°C | 30秒。 | パン= "3"> 40サイクル |
| *アニーリング | 62°C | 30秒。 | |
| 拡張 | 68°C | 2分。 | |
| 最後の拡張 | 68°C | 5分。 | 1 |
| ホールド | 4°C | ホールド |
表2。
*アニーリング温度は変わる場合がありますが、我々は、53と64の間の範囲のアニール温度で最高の増幅産物℃を観測した
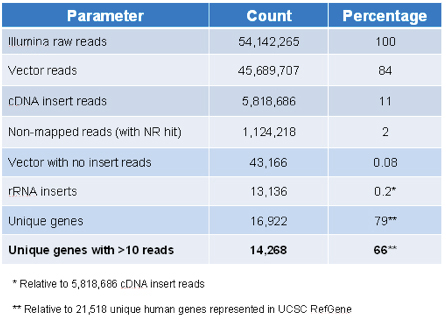
イルミナシーケンスによって表3。ターゲットIDのライブラリのコンテンツ。
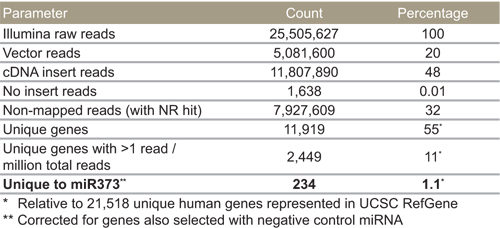
表4。イルミナシーケンスでのmiR-373選択したターゲット。
les/ftp_upload/3303/3303table5.jpgの "alt ="表5 "/>表5。以前にRNAマイクロアレイによって識別された10のターゲット。
ディスカッション
マイクロRNAは、mRNAの翻訳を阻害することにより転写後に遺伝子発現を調節すると、頻繁に、標的mRNAを([6]日)不安定に20から24ヌクレオチドのRNAである。単一のmiRNAは、開発と環境シグナルに対する細胞の応答を制御するために数百のmRNAを調節することができます。標的mRNAを識別し、検証することで、これらの経路におけるmiRNAの役割と機能を決定する上で不可欠である。動物のmiRNAとその標的部位に完全に相補的ではありませんが、しかし、ターゲットの同定は簡単ではありません。 "シード"領域は、miRNAの5 '末端から2〜7を基づいており、通常、そのターゲットに相補的である。しかし、そこに種子のルールには多くの例外があり、塩基対下流には、不完全種子の一致を補正することができます。コンピュータアルゴリズムの数は、シード·マッチングと下流の補償、ターゲット構造に基づいてmiRNAのターゲットを予測するために開発されているDポジション、シーケンスの保全、実験的に検証ターゲット([7]で検討)のために観察されている他のさまざまなパラメータを設定します。インシリコ予測に便利であると識別多くの有効なmiRNAのターゲットを実行しながら、ほとんどの遺伝子は実験的な検証テストに失敗すると予測し、多くの実際の目標は、予測されていません。コンピュータのアルゴリズムは以前に決定され、ターゲットの機能に基づいているためさらに、彼らは既に知られているものから逸脱してターゲットの検出を許可しません。
実験的なシステムの数が生きている細胞内で機能的なmiRNAのターゲットを識別または発見するために首尾よく使用されています。これらのグローバルなスクリーニング法は、マイクロアレイとRNA配列、RNAの共免疫沈降法(RIP)、および細胞培養におけるアミノ酸と安定同位体標識(SILAC)、プロテオミクスの方法([7]件)が含まれています。各メソッドには、長所と短所があります。標的miRNAはしばしばmRNAを不安定化し、その劣化、損失oにつながるので、それぞれのmiRNAを模倣または阻害剤を導入した後、mRNAレベルでのRゲインは、miRNAのターゲットを識別することができます。 mRNAのこの損失または利得を容易にマイクロアレイまたはディープシークエンシングにより検出されます。 mRNAの検出方法が簡素化され、タンパク質の検出法よりも高感度であるが、mRNAの検出が劣化しない任意のmiRNAのターゲットを欠場する。 SILACからのもの[8]またはリボソームプロファイリングでのRNA検出からmiRNAのターゲットの結果を比較するバーテルラボからの最近のレポート[9]は、哺乳動物のmiRNAが主にターゲットのmRNAレベルを減少させることによって行動することを示しています。ただし、個々のmiRNAのターゲットでの作業、他の多くのラボでは、蛋白質の変化がmRNAレベルの変化なしを検出した。検出可能なmRNAの損失と翻訳調節と思われるものの報告は、次のとおりです。のmiR-10bがPDCD4でHOXD10 [10]、p27kip1にmiR221/222 [11]、miR-21のp85β[13] [12]は、miR-126を、SIRT1上でのmiR-34 [14] PTEN [15]、のmiR - 中継台Arid4bの[16]は、miR-200C JAG1の[17]、およびmiR-299、297、567、上に、miR-21のVEGFA上のND 609 [18]。また、クランシーら[19]最近のlet - 7の標的部位を含む個々のmRNAアイソフォームが選択的に検出されたときにlet-7による翻訳調節にのみ検出されたことを報告した。すべてのmRNAアイソフォームが1つのmRNAとして検出されたときにそれがある - - として多くのマイクロアレイおよび塩基配列の解析で得られた後者は、少なくともいくつかのケースでは、並進規制が複合プロファイルに見落とされる可能性があることを示唆している。アルゴノート(通常は新生血管傷害)または他の関連タンパク質(RNA免疫沈降、またはRIP)を介してのmiRNA-mRNAの複合体の共免疫沈降に関係なく、調節機構のmiRNAターゲットを分離します。さらに、RIPは、相互作用を内在し、おそらく生物学的に関連が検出されます。しかし、それはすべてのmiRNA-mRNAの相互作用が機能的であり、RIPだけ一時的に関連付けたり、急速に劣化しているすべてのmRNAターゲットを逃すかどうかは知られていない。さらに、特異的なmiRNA-mRNAのパートナーはbioinformatiを推測する必要がありますすべてのmiRNA-mRNAのペアが一緒に共沈自動的ので。最後に、SILACは直接miRNAの調節、タンパク質自体の最終的な製品を識別しますが、小文字は区別されませんので、希少なタンパク質とタンパク質レベルの小さな倍の変化を蹴る。
現在のmiRNAのターゲット同定法の制限の光の中で、我々は別のメカニズムによって機能的なmiRNAのターゲットを識別するための追加のグローバルアッセイが必要になったと感じました。このニーズを満たすために、我々は、ヨープGakenとロンドン大学キングスカレッジのアジムMohamedaliによって発明された技術をライセンスされています。彼らの発明は、二重の選択融合タンパク質であり、具体的には、チミジンキナーゼゼオシン融合、その3'UTRにおける潜在的なmiRNAの標的配列のcDNAライブラリーで規制。 図2に示すように安定TKzeo-cDNA構築物でトランスフェクトした細胞と発現しているが、ゼオシンで選択することができます。興味のあるmiRNAを発現した後、そのmiRNAのの目標は、wiを選択することができます図3に示すように、ガンシクロビル番目。ガンシクロビルは、任意の細胞が関心のmiRNAのターゲットを欠いているTKzeo-cDNAを発現しているチミジンキナーゼを発現する細胞を殺す。標的cDNAは脇腹cDNAおよびPCR産物はターゲットを識別するために配列決定することができますガンシクロビルを使用してプライマーを生き残る細胞からのDNAのPCR増幅により単離することができる。
ミッションのターゲットIDライブラリ - 我々は機能的なヒトmiRNAターゲットのグローバルな識別と発見のための新しいツールにキングス·カレッジ·テクノロジを開発しました。ターゲットIDのライブラリを使用すると、ユーザーは、哺乳動物細胞培養、トランスフェクションと薬剤選択の一連の手順によってmiRNAのターゲットを特定することができます。ライブラリは、包括的であり、ヒト遺伝子の66から79パーセントが含まれています。初期の結果は、両方の前に発見されたほか、新たな目標は、ミッションのターゲットIDライブラリから単離することができることを示しています。
プロトコルは、いくつかの長さですが、哺乳動物細胞培養、トランスフェクション、薬剤選択、PCRおよびシークエンシング - - ターゲットIDのライブラリの使用は標準的な分子生物学研究室の技術を必要とするため、ほとんどの生物学者の手段の範囲内でなければなりません。我々は十分な注意が培養条件を最適化し、適切な実験デザインに支払われ、そして最も重要なのは、ターゲットIDのライブラリと成功を確実にするための選択の手順を実行する(kill-曲線)に最適な薬物濃度を決定するために、各セルの新しいタイプを事前にテストされることを示唆している。
すべてのmiRNAのターゲットの識別方法と同様に、それは非常にターゲットIDのライブラリーをスクリーニングすることによって識別されるターゲットは、マイクロアレイ、定量RT-PCR、レポーターアッセイ(すなわち、ルシフェラーゼ)、またはウェスタン分析などの二番目の方法、使用して検証することをお勧めします。
開示事項
作者は、提出された作品に関心を持っている営利団体と金銭的関係を宣言します。
謝辞
私たちは彼女のサポートと有益なトラブルシューティングの議論に参加するため、特に全体のシグマライフサイエンスミッションのターゲットIDライブラリの開発チームは、技術を発見し、ライセンス条件を交渉するためのケビンGutshallと、ヘザーHolemonに感謝します。我々はまた、自由にそれがクローニングされ得ることに大きなcDNAのバッチとその持続性を準備するために未発表の結果と提案、RxBiosciencesのQaziハミドを共有するためにロンドン大学キングスカレッジの博士ヨープGäkenに感謝します。我々は、cDNA配列とデータ解析を行うためのSAFCからナン·リンスコット·バハルに感謝します。
ミッションは、シグマアルドリッチバイオテクノロジーLPの登録商標です。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | コメント |
| JumpStartのREDTaq ReadyMix | Sigma-Aldrich社 | P0982 | |
| ガンシクロビル | Sigma-Aldrich社 | G2536 | |
| G418 | Sigma-Aldrich社 | G8168 | |
| ピューロマイシン | Sigma-Aldrich社 | P9620 | |
| TOPO TA | インビトロジェン | KNM4500-01 | |
| 哺乳類のゲノムDNA MiniprepキットをGenElute | Sigma-Aldrich社 | G1N10 | |
| 細胞特異的ヌクレオのkそれ | ロンザ | ||
| のNucleofector | ロンザ | AAD-1001 | |
| ゼオシン | インビトロジェン | R250-01 | |
| ターゲットidライブラリ | Sigma-Aldrich社 | MREH01 |
参考文献
- Huang, Q., Gumireddy, K., Schrier, M. The microRNAs miR-373 and miR-520c promote tumour invasion and metastasis. Nat. Cell Biol. 10 (2), 202-210 (2008).
- Lichner, Z., Pall, E., Kerekes, A. The miR-290-295 cluster promotes pluripotency maintenance by regulating cell cycle phase distribution in mouse embryonic stem cells. Differentiation. 81 (1), 11-24 (2011).
- Subramanyam, D., Lamouille, S., Judson, R. L. Multiple targets of miR-302 and miR-372 promote reprogramming of human fibroblasts to induced pluripotent stem cells. Nat. Biotechnol. 29 (5), 443-448 (2001).
- Lim, L. P., Lau, N. C., Garrett-Engele, P. Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs. Nature. 433 (7027), 769-773 (2005).
- Huntzinger, E., Izaurralde, E. Gene silencing by microRNAs: contributions of translational repression and mRNA decay. Nat. Rev. Genet. 12 (2), 99-110 (2011).
- Thomas, M. Desperately seeking microRNA targets. Nat. Struc. Mol. Biol. 17 (10), 1169-1174 (2010).
- Baek, D. The impact of microRNAs on protein output. Nature. 455 (7209), 71-71 (2008).
- Guo, H. Mammalian microRNAs predominantly act to decrease target mRNA levels. Nature. 466 (3708), 835-840 (2010).
- Ma, L. Tumour invasion and metastasis initiated my microRNA-10b in breast cancer. Nature. 449 (7163), 682-688 (2007).
- Visone, R., et al. MicroRNAs miR-221 and miR-222, both overexpressed in human thyroid papillary carcinomas, regulate p27Kip1 protein levels and cell cycle. Endocrine-Related Cancer. 14 (3), 791-798 (2007).
- Lu, Z., et al. MicroRNA-21 promotes cell transformation by targeting the programmed cell death 4 gene. Oncogene. 27 (31), 4373-4379 (2008).
- Guo, C., et al. The noncoding RNA, miR-126, suppresses the growth of neoplastic cells by targeting phosphatidylinositol 3-kinase signaling and is frequently lost in colon cancers. Genes Chrom. Cancer. 47 (11), 939-946 (2008).
- Yamakuchi, M., et al. miR-34a repression of SIRT1 regulates apoptosis. Proc. Natl. Acad. Sci. U.S.A. 105 (36), 13421-13426 (2008).
- Wickramasinghe, N. S., et al. Estradiol downregulates miR-21 expression and increases miR-21 target gene expression in MCF-7 breast cancer cells. Nucleic Acid Res. 37 (8), 2584-2595 .
- Ciaudo, C., et al. Highly dynamic and sex-specific expression of microRNAs during early ES cell differentiation. PLoS Genetics. 5, e1000620 (2009).
- Vallejo, D. M., et al. Targeting Notch signaling by the conserved miR-8/200 microRNA family in development and cancer cells. EMBO J. 30 (4), 756-769 (2011).
- Jafarifar, F., et al. Repression of VEGFA by CA-rich element-binding microRNAs is modulated by hnRNAP. L. EMBO J. 30 (7), 1324-1334 (2011).
- Clancy, J. L., et al. mRNA isoform diversity can obscure detection of miRNA-mediated control of translation. RNA. 17 (6), 1025-1033 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved