このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
単一分子蛍光顕微鏡による受容体ダイナミクスの高分解能時空間解析
要約
This protocol describes how to use total internal reflection fluorescence microscopy to visualize and track single receptors on the surface of living cells and thereby analyze receptor lateral mobility, size of receptor complexes as well as to visualize transient receptor-receptor interactions. This protocol can be extended to other membrane proteins.
要約
単一分子顕微鏡は、これらの側面( 例えば 、動力学、異なる状態や集団、一時的な相互作用の共存)は、一般的にアンサンブル測定で隠されている、などに関して、特に、シグナル伝達分子の挙動を解析するための強力なアプローチとして浮上している標準的な生化学的または顕微鏡の方法で得られたもの。従って、このような受容体 - 受容体相互作用などの動的イベントは、高時空間解像度で生細胞におけるリアルタイムで追跡することができる。このプロトコルは、小さくて明るい有機蛍光団と直接生きた細胞の表面上の単一の受容体を可視化する全反射蛍光(TIRF)顕微鏡を用いた標識に基づく方法について説明します。このアプローチは、いずれかが正確に、受容体を局在化受容体複合体のサイズを測定し、そのような一過性受容体 - 受容体相互作用などの動的イベントをキャプチャすることを可能にする。プロトコルは、PE方法の詳細な説明を提供しています試料調製、画像取得および画像解析を含む単一分子実験を、rform。一例として、2つのG-タンパク質共役受容体を分析するこの方法の適用、 すなわち 、β2 -アドレナリン及びγ-アミノ酪酸タイプB(GABA B)は受容体が報告されている。プロトコルは、他の膜タンパク質および異なる細胞モデル、トランスフェクション法および標識戦略に適合させることができる。
概要
受容体は、細胞表面感覚細胞外環境に位置しており、このような臭気物質、イオン、小神経伝達物質および大きいタンパク質ホルモンのような様々な刺激に応答する。細胞膜の流動性は、受容体及び他の膜タンパク質の移動を可能にする。これは、タンパク質複合体およびそのような機能単位にアセンブルし、細胞内部にシグナルを伝達する受容体によって使用されるような一過性のタンパク質 - タンパク質相互作用の発生の形成に必須である。例えば、細胞表面受容体1の最も大きなファミリーを構成するG-タンパク質共役受容体(GPCR)は、シグナル伝達の微調整調節に関与することが表示され、di-/oligomersを形成することが示唆されている重要な生理的および薬理学的結果2-5があるかもしれません。
単一分子顕微鏡で直接高spatiotempで可視化する大きな可能性を秘めている経口解像度の関連付けを含む生きている細胞の表面上に位置する個々の受容体の動的挙動は、二量体およびより高次の分子複合体6-10を形成する。これは通常、分子の数千から数百万の平均的挙動を報告し、標準的な生化学的および顕微鏡の方法に比べていくつかの利点を提供しています。
十分に明るく、光安定性、フルオロフォアとタンパク質標識は、単一分子顕微鏡検査のために不可欠です。このプロトコルは、共有結合し、細胞表面の受容体に小さな、明るい有機蛍光団を接続するために最近導入され、SNAPタグ11を利用しています。 SNAPは、不可逆的に蛍光団結合ベンジルグアニン(BG-フルオロフォア)誘導体で標識することができるヒトDNA修復酵素のO 6 -アルキルグアニン-DNAアルキルトランスフェラーゼに由来する20 kDのタンパク質タグである。 CLIP、SNAPに由来し、さらに設計タグは、代わりに蛍光団-Cで標識することができるonjugatedベンジルシトシン誘導体12。
本稿で報告されたプロトコルは、トランスフェクトし、ラベルの小さい有機蛍光団との11の受容体をSNAP-タグおよび生細胞10の表面上の単一の受容体または受容体複合体を視覚化するために全反射蛍光(TIRF)顕微鏡を使用する方法について説明します。細胞外SNAP-タグ化細胞表面タンパク質10の> 90%の標識効率の報告されたプロトコールをもたらす。受容体複合体のサイズおよび移動度を分析するために、ならびに一過性受容体 - 受容体相互作用を捕捉するために単一分子データを使用する方法に関するさらなる情報は、提供される。プロトコル全体の概略的なワークフローは、 図1に示されている。一例として、SNAP-タグ化Gタンパク質共役受容体(GPCR)を有するチャイニーズハムスター卵巣(CHO)細胞のトランスフェクションは、フルオロフォア-BG誘導体で標識し、続いてよくquantifへの応用として、yおよびモニタ受容体di-/oligomerizationが記載されている。このプロトコルは、他の細胞表面タンパク質と蛍光タグ( 例えば 、CLIP)、ならびに他のトランスフェクションおよび標識方法にまで拡張することができる。
プロトコル
1。サンプルの調製
- カバーガラス清掃
注:ヒュームフードの下で動作します。- 個々のカバースリップを分離し、カバースリップホルダーにガラス製カバースリップ(直径24mm)を配置するためにきれいなピンセットを使用してください。
- ビーカーにカバースリップでホルダーを置き、カバーグラスがカバーされるまで、クロロホルムを追加します。室温で1時間にわたり浴超音波処理器中で蒸発し、超音波処理を減らすためにアルミホイルでビーカーをカバーしています。ビーカーからカバースリップホルダーを取り、カバーグラスを乾燥させます。
- 代わりにクロロホルムを5M NaOH溶液でステップ1.1.2を繰り返します。
- 新しいビーカーにカバースリップホルダーを取り、蒸留水で3回洗浄する。 100%エタノールを充填したガラス細胞培養プレート中で洗浄し、カバースリップを置く。
- キャリブレーションサンプルの調製
- 適切な溶媒に蛍光染料を溶解させる。
- までの蛍光色素の1:10連続希釈液を調製1時から1 nMのフィルタに(0.22μm)の水を滅菌した。
- 100%エタノールに保存掃除カバースリップを取り、フィルター滅菌水で洗う。スポット別々に洗浄カバースリップ上の各蛍光色素希釈液20μl。カバーグラスを滅菌フードの下で乾燥させます。使用するまで、光とホコリからカバースリップを保護します。単一の蛍光分子(ステップ3を参照)の強度を推定するために、これらのサンプルを使用しています。
- トランスフェクション
- 10%ウシ胎児血清(FBS)を補充した1時01分ダルベッコ改変イーグル培地/栄養混合物F-12で培養CHO細胞(DMEM/F12)、100 U / mlペニシリンおよび37℃で100μg/ mlのストレプトマイシン、内5%のCO 2 注:自家蛍光を最小限にするために、実験を通じてフェノールレッドを含まない培地を使用してください。
- 、100%エタノール溶液から清浄カバースリップを取り、滅菌リン酸緩衝生理食塩水(PBS)でそれらを洗浄し、6ウェル細胞培養plの各ウェルに1カバースリップを置く食べた。
- トリプシン処理は、カウントし、3×10 5細胞/ウェルカバースリップを含有する6ウェル細胞培養プレート中の密度でシードCHO細胞。細胞が約達成するために、24時間インキュベーター(37℃、5%CO 2)で成長しましょう。トランスフェクションのための最適な細胞密度は80%コンフルエント。
- 各ウェルには、( 例えば 、2 -アドレナリン受容体βSNAP-タグ)所望のプラスミドDNAの2μgのを希釈し、500μlのOptiMEM培地を含む2別々のチューブ内の6μlのリポフェクトアミン2000。室温で5分間インキュベートする。
- 1チューブにステップ1.3.4からのソリューションを組み合わせて、トランスフェクション混合物を得るために混ぜる。室温で20分間トランスフェクション混合物をインキュベートする。
- インキュベーション(1.3.5)の間に、CHO細胞を取得し、予め温めた(37℃)PBSで二回洗浄する。 1ミリリットル/ 10%FBSを添加したフェノールレッドを含まないDMEM/F12培地のよくなく、抗生物質なしで、PBSを交換してください。
- 全体トランスフェクトを追加イオン混合物を穏やかに各ウェルに滴下して、ステップ1.3.5(1 ml)を加え、完全な混合を確実にするためにプレートを前後に揺する。
- CO 2 5%、37℃で2〜4時間インキュベートし、次のステップに直後に進むNOTE:これらのトランスフェクション条件は、受容体密度を達成するために最適化された<単一のに適している0.45個/μm2で、分子イメージング。異なるセル、構築物または試薬を使用する際に調整が必要になることがあります。
- タンパク質標識
- 1μMの最終濃度を得るために、10%FBSを補充した1ミリリットルDMEM/F12培地中のフルオロフォア-BGストック溶液1μlを希釈する。インキュベーターからトランスフェクトされた細胞を取り、あらかじめ温めておいた(37℃)PBSで2回洗浄します。 1μMの蛍光団のBG溶液1mlを含むPBSを交換し、37℃、5%CO 2インキュベーター内で20分間インキュベートする。
- インキュベーション後、細胞を3を洗浄10%FBSを補充したDMEM/F12培地中、37℃で5分間インキュベートした各時間に回ピンセットで(標識された細胞との)カバースリップを取り、イメージング室に配置します。
- 300μlの撮像緩衝液で2回洗浄する。新鮮なイメージングバッファー300μlのを追加し、画像化(その2)にすぐに進んでください。
2画像の取得
注:全反射蛍光を使用してください(TIRF)顕微鏡、油浸高開口数の対物レンズ( 例えば 、100X magnification/1.46開口数)を装備した、適切なレーザ(例えば405nm、488nmの、561 nmおよび645ナノメートルダイオードレーザ)、電子増倍電荷結合素子(EMCCD)カメラ、インキュベーター、単一蛍光分子を可視化する温度制御。
- すなわち 、所望の顕微鏡のパラメータを設定します。、レーザーライン、全反射角度が(このパラメータは、溶込みを制御エバネッセント場の深されたTiON)、露出時間、フレームレート、動画10あたりの画像数。温度ドリフトや結露を避けるために、常にオンヒーター/インキュベーターと温度制御を維持する。
- 顕微鏡の100倍の対物レンズにイマージョンオイルの滴を入れてください。顕微鏡の試料ホルダーに標識した細胞を画像化室を配置し、明視野照明を使用して焦点の細胞をもたらす。
- 全反射照明に切り替えます。しかし、同時に、所望の細胞を探索する光退色を最小にできるように、できるだけ低いレーザパワーを保つ。
- 所望のセルを選択し、細かいピントを調整します。シングルフルオロフォアの可視化を可能にするレベルにレーザーパワーを設定します。画像シーケンスを取得し、。TIFFなどの生イメージシーケンスファイルを保存します。
3。キャリブレーション(ガラス及び単量体/二量体受容体のコントロールにシングルフルオロフォア)
- 各校正SAMPLを組み立てるイメージングチャンバーに1.2で説明したように、電子用意しました。顕微鏡で各サンプルを配置し、単一のステップで漂白十分に分離回折限界スポットを含むサンプルを選択します。 注:これらのスポットは、蛍光色素の単一分子を表す。
- 工程2に記載されTIRF画像シーケンスを取得する重要 :同じ撮像パラメータは、較正のためのものも含め全ての実験に使用されなければならない。
- 4.1から4.2に詳述され検出および追跡分析を行う。 4.2.6で説明したように各粒子の強度を抽出します。これらのデータから、平均値(μ)および単一蛍光体の強度の標準偏差(σ)を計算します。
- オプション:単量体細胞表面受容体( 例えば CD86)をトランスフェクトした細胞上で同じ分析を行い、N-末端SNAP 10の1つまたは2つのいずれかのコピーで標識して蛍光団-BG誘導体で標識された。 Folloガラス上の単一のフルオロフォアのために上記の手順ワット。 Calebiro、D. らの説明に従って、標識効率を推定する。10
4。画像解析
- 画像シーケンスの準備
- 画像をトリミングする画像処理ソフトウェア( 例えば、ImageJは ) を使用します。
- 独立した個々のフレームを保存します。TIFF画像を新規フォルダに、各画像フレーム番号に示す。
- 細胞の輪郭に沿って関心領域(ROI)を描き、ImageJの内のメジャーツールまたは別のソフトウェアで同様のツールを使用して、細胞表面の面積を測定する。細胞表面面積でムービーの冒頭粒子の総数で割ることによって粒子密度を計算するためにこの値を使用します。
- 粒子検出および追跡
注:このようなU-トラック 13として使用する非商用のソフトウェアは、MATLABでの作業自動的に検出し、単一の受容体粒子を追跡するための環境を、。
注:U- トラックアルゴリズムは、複数の仮説の追跡手法に基づいています。このアプローチは、1フレーム内で指定された粒子は、次のフレームで所定の粒子に対応している表示され、消えたり/他の粒子から/で分割併合する個々の確率が割り当てられているコスト行列を、構築することにより、フレーム間の粒子をリンクします。世界的に、コストを最小化する解、 すなわち 、最も確率の高い1は、最終的に選択されています。また、これは一時的に消え、粒子、蛍光体の点滅によって引き起こされる典型的な現象を追跡することができます。 U-トラック (2.1.0)の最新バージョンは、これらの分析の実行を容易にするグラフィック·ユーザ·インタフェースがあります。- 映画の選択インターフェイスを開くには、MATLABコマンドプロンプトで、「movieSelectorGUI」。作成するために指示に従ってください以前に保存した別々の画像から始まる新しいムービーデータベース(4.1.2を参照)。
- 粒子検出および追跡するために必要な画素サイズ以下で、秒単位の時間間隔、開口数、カメラのビット深さ及びフルオロフォアの発光波長を提供する。映画のデータベースを保存します。
- 映画の選択インタフェースから、オブジェクトの種類として、「単粒子 」 を選択、分析を実行します。粒子検出および追跡に使用されるパラメータを定義することができる場所に新しいウィンドウが表示されます。デフォルトのパラメータで始まります。 。検出および/ またはトラッキングの品質はオプション ( 例えば 、いくつかの粒子が検出されたか、トラックが断片化されていない)満足のいくものであれば、後は、これらのパラメータを調整します。トラッキング設定の下で、保存するために「 マトリックス形式にエクスポート追跡結果 " をチェック「trackedFeaturedInfoと呼ばれる単一の行列(フィールド内のすべての粒子の座標と振幅」)。これらのパラメータの詳細については、U-トラックのマニュアルを参照してください。
- 検出アルゴリズムを実行します。このアルゴリズムは、自動的に各回折限界スポット局所強度極大の周りの顕微鏡の点広がり関数に等しい標準偏差が2次元ガウス関数をフィッティングすることによって( すなわち 、単一の受容体/受容体複合体)の背景の上の位置と強度を決定。その後、追跡アルゴリズムを実行します。 。マットファイルで分析結果を格納します。
- トラックを視覚化し、検出および追跡の品質をチェックするために、U-トラックパッケージに含まれる「movieViewer "ルーチン、または類似したカスタムのものを使用してください。
- 各フレームで追跡粒子の位置と振幅( すなわち 、強度)を表示します。matファイルを開きます。ステップ4.2.4で生成されたデータはtracksFinal ARのtracksCoordAmpCGフィールドに含まれています線、および/またはtrackedFeaturedInfo中。粒子の総数から4.1.3で測定された細胞表面面積でこの値を除算粒子密度を算出し検出した。
- オプション:受容体の粒子の動きを分析すること(4.2.6参照)を時間をかけて、粒子の座標を使用します。 Matlabのまたは類似のソフトウェアを使用して、平均二乗変位(MSD)と拡散係数(D)を計算します。各粒子と考えられるすべての時間間隔(Δt)は 、次の数式を使用して、平均二乗変位(MSD)を計算する。
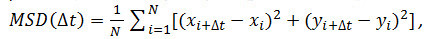
Δtはフレーム内の時間間隔であり、Nが分析ステップ数であり、xおよびyは、インデックスが示すフレームにおいて、粒子のx座標とy座標である。線形関係:指定された粒子の運動の種類を評価するために、時間のプロット上でMSDを使用監督の動きを示唆している自由拡散( すなわち 、ブラウン運動)、正の曲率( すなわち 、曲線は放物線のように見える)を示し、負の曲率は、限られた運動14の指標である。自由に拡散粒子の場合には、以下の式を用いて得られたMSDデータを当てはめることにより各粒子の拡散係数(D)を計算する。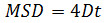
- ガウシアンフィッティング法と粒子サイズの計算
NOTE:キャリブレーションサンプル(ガラスおよび/ または蛍光標識された単量体の受容体上の単一のフルオロフォア)の強度分布がわかれば、受容体複合体のサイズを決定するために、画像シーケンスの開始時に、粒子強度の分布に混合ガウスフィットを実行する( すなわち 、粒子当たりの受容体の数)10。 Matlabのか、Sを使用して、これらの分析を実行imilarソフトウェア。- 強度の最初の変更が発生する前の(光退色に起因する減少がほとんどの場合)のフレームに最初のフレームからの粒子の強度を平均化することにより、各粒子の強度を計算します。
- 次の式に従って混合ガウスフィッティングを行います。
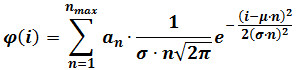
φ(i)は 、粒子がiの強度を有する周波数であり、nは成分の数であり、αnの成分nの高さに寄与するパラメータであり、μ及びσは、平均値と基準単一のフルオロフォアの強度の標準偏差である(ステップ3で説明したように計算される) 注 :Tの決定F検定により判断されるように、彼は徐々に成分を添加するまでのn maxが増加することによって、各画像シーケンスのためのコンポーネント(N 最大 )の最大数は、もはや、統計学的に優れたフィッティングを生じないない。 - オプション:(。 例えば 、400フレームシーケンスの最後の60フレーム)ムービーの最後のフレームに得られた強度分布に混合ガウスフィットを実行します。これらのパラメータのための洗練された推定値を提供する。このフィッティングの後に得られた値とμとσを交換して、ステップ4.3.2を繰り返します。
- 混合ガウスフィットの各成分の曲線下面積(AUC)を計算する。異なるサイズ分布全体のAUCで、各成分のAUC値を除算すること(すなわち、モノマー、ダイマー、トリマーなど)の受容体粒子の相対的な存在量を計算します。
- オプション:別の細胞と異なるサイズの粒子の分布は粒子密度10と相関してプロットを生成する(4.2.6で説明したように計算される)に対応する粒子密度から、使用データ。
- ステップフィッティング法と粒子サイズの計算
注:受容体複合体10のサイズを決定するための別の方法として、ステップフィッティング分析を使用してください。 n個の蛍光団を含むこのように粒子徐々に漂白nステップで強度プロファイルを生成することが期待される-この分析のための基礎は、その瞬間の消失、単一のフルオロフォアの結果の光誘起破壊(退色)があることである。- U-トラックまたは同様の検出/追跡ソフトウェア(4.2.6を参照)によって生成される。matファイルから各粒子の強度プロファイルを抽出します。
- このような提示1工程として、フィッティングアルゴリズムを使用REF IN。 10、各粒子のための漂白ステップの数をカウントする。
- オプション :(4.3参照)は、異なる大きさの受容体粒子の相対量を示す分布を生成し、予め混合ガウスフィッティングの結果を説明したように粒子密度とそれらを関連付けるために、結果を使用しています。
結果
記載されたプロトコルは、異なる膜タンパク質の種々に適用することができる。例として、β2 -アドレナリン作動性およびGABA B受容体を用いて得られた代表的な結果を10で報告されている。単一分子からの蛍光シグナルが弱いので、バックグラウンド蛍光の最小化が成功した結果の最初の重要なステップである。これにより、広範囲にカバースリップを洗浄( 図...
ディスカッション
記載されたプロトコルは、単一分子レベルでの細胞表面受容体複合体の空間的配置、移動度及びサイズの分析を可能にする。蛍光タンパク質の使用と比較して、より明るく光安定性である小さな有機フルオロフォアで標識化は、単一の受容体粒子の可視化を拡張できるという利点を有する。非常に低い発現レベルが達成されているので(<0.45レセプター個/μm2)は 、受容体および他...
開示事項
The authors declare that they have no competing financial interests.
謝辞
The development of this protocol was supported by grants from the European Research Council (Advanced Grant TOPAS to M.J.L.) and the Deutsche Forschungsgemeinschaft (Grants CA 1014/1-1 to D.C. and SFB487 to M.J.L.). T.S. was supported by the Alexander von Humboldt Foundation.
資料
| Name | Company | Catalog Number | Comments |
| Chloroform | AppliChem GmbH | A1585 | CAUTION: toxic and irritating substance as well as a possible carcinogen |
| NaOH | Sigma-Aldrich | S8045 | CAUTION: strong base and highly corrosive reagent |
| Absolute ethanol | Sigma-Aldrich | 32205 | |
| Glass coverslip | Marienfeld-Superior | 111640 | 24 mm diameter, 0.13-0.16 mm thickness |
| 0.2 mm sterile filter | Sarstedt | 83.1826.001 | |
| CHO cells | ATCC, USA | ATCC CCL-61 | Chinese hamster ovary cell line |
| 6-well cell culture plate | Nunc | 140675 | |
| DMEM/F-12 medium | GIBCO, Life Technologies | 11039-021 | Phenol-red free medium |
| Fetal bovine serum | Biochrom | S 0115 | |
| Penicillin - streptomycin | Pan Biotech GmbH | P06-07 100 | |
| Trypsin-EDTA | Pan Biotech GmbH | P10-23100 | |
| Lipofectamine 2000 | Invitrogen, Life Technologies | 11668-019 | |
| Opti-MEM I Reduced Serum Medium | Invitrogen, Life Technologies | 31985-047 | |
| Fluorophore-conjugated benzylguanine | New England BioLabs | S9136S | SNAP-Surface Alexa Fluor 647. Make a 1 mM stock solution in DMSO. Store at -20°C. |
| DMSO | AppliChem GmbH | A1584 | |
| Imaging buffer: | 137 mM NaCl, 5.4 mM KCl, 2 mM CaCl2, 1 mM MgCl2, 10 mM HEPES, pH 7.3, sterile-filtered | ||
| NaCl | AppliChem GmbH | A1371 | |
| KCl | AppliChem GmbH | A3582 | |
| CaCl2 | AppliChem GmbH | A2303 | |
| MgCl2 | AppliChem GmbH | A3618 | |
| HEPES | AppliChem GmbH | A3724 | |
| Imaging chamber | Molecular Probes, Life Technologies | A-7816 | Attofluor Cell Chamber, for microscopy |
| TIRF-M | Leica | Model: DMI6000B | |
| TIRF objective | Leica | 11 506 249 | HCX PL Apo 100x/1.46 Oil CORR |
| EM-CCD camera | Roper Scientific | Photometrics Cascade 512B | |
| Temperature controller | Pecon | Tempcontrol 37-2 digital | |
| ImageJ software | NIH, USA | http://rsbweb.nih.gov/ij | |
| u-track software | Laboratory for computational cell biology, Dept. of Cell Biology, Harvard Medical School, USA | http://lccb.hms.harvard.edu/software.html | |
| Matlab software | The MathWorks, USA |
参考文献
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Angers, S., Salahpour, A., Bouvier, M. Dimerization: an emerging concept for G protein-coupled receptor ontogeny and function. Annu Rev Pharmacol Toxicol. 42, 409-435 (2002).
- Ferré, S., et al. Building a new conceptual framework for receptor heteromers. Nat Chem Biol. 5, 131-134 (2009).
- Milligan, G. G. protein-coupled receptor hetero-dimerization: contribution to pharmacology and function. Br J Pharmacol. 158, 5-14 (2009).
- Lohse, M. J. Dimerization in GPCR mobility and signaling. Curr Opin Pharmacol. 10, 53-58 (2010).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nat Methods. 4, 319-321 (2007).
- Triller, A., Choquet, D. New concepts in synaptic biology derived from single-molecule imaging. Neuron. 59, 359-374 (2008).
- Hern, J. A., et al. Formation and dissociation of M1 muscarinic receptor dimers seen by total internal reflection fluorescence imaging of single molecules. Proc Natl Acad Sci U S A. 107, 2693-2698 (2010).
- Kasai, R. S., et al. Full characterization of GPCR monomer-dimer dynamic equilibrium by single molecule imaging. J Cell Biol. 192, 463-480 (2011).
- Calebiro, D., et al. Single-molecule analysis of fluorescently labeled G-protein-coupled receptors reveals complexes with distinct dynamics and organization. Proc Natl Acad Sci U S A. 110, 743-748 (2013).
- Keppler, A., et al. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat Biotechnol. 21, 86-89 (2003).
- Gautier, A., et al. An engineered protein tag for multiprotein labeling in living cells. Chem Biol. 15, 128-136 (2008).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nat Methods. 5, 695-702 (2008).
- Saxton, M. J., Jacobson, K. Single-particle tracking: applications to membrane dynamics. Annu Rev Biophys Biomol Struct. 26, 373-399 (1997).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved