Method Article
比色金ナノ粒子アッセイに適用される構造スイッチングアプタマーを選択するための方法
要約
A protocol is provided to select structure-switching aptamers for small molecule targets based on a tunable stringency magnetic bead selection method. Aptamers selected with structure-switching properties are beneficial for assays that require a conformational change to signal the presence of a target, such as the described gold nanoparticle assay.
要約
小分子は、人間の健康とパフォーマンスのさまざまな側面のバイオマーカーとしての生理的意義のためにバイオセンシングアプリケーションのための豊富な標的を提供する。核酸アプタマーは、ますますバイオセンサープラットフォーム上の認識要素として適用するが、小分子の目標に向かってアプタマーを選択すると、特別な設計上の考慮を必要とされてきた。この作品は、修正および小分子標的への構造スイッチングアプタマーを選択するように設計された方法の重要なステップについて説明します。磁気ビーズ上の相補的DNA捕捉プローブにハイブリダイズしたDNAライブラリーからの結合配列は、立体配座の標的に誘導される変化を介して、非結合体から分離される。支持マトリックス(ビーズ)に結合する配列をさらに増幅されないため、この方法は有利であり、それは、標的分子の固定化を必要としない。しかしながら、捕捉プローブおよびライブラリの融解温度がで保持されるか、またはわずかにRT上記、このような配列トンこと熱力学に基づいた帽子の脱ハイブリダイズも、上澄み溶液中に存在するであろう。これは効果的に分割効率(非結合体から結合配列を標的と分離する能力)を制限するため、多くの選択ラウンドは、背景配列を除去するために必要とされる。報告された方法は、負の選択工程を簡略化濃縮モニタリング、および強化されたストリンジェンシーを提供するために選択富化以下捕捉プローブの長さの延長の実装前構造スイッチングアプタマーの選択とは異なる。選択された構造スイッチングアプタマーは、アプタマーの構造変化により、標的分子の存在を報告して金ナノ粒子アッセイプラットフォームで有利である。それは臨床的または配備環境において有益で簡単、迅速な比色読み取りを提供するので、金ナノ粒子アッセイを適用した。設計と最適化の考慮は証明のprinciplとしてアッセイのために提示されている生理学的流体において、小分子バイオセンシングに向けて作業をさらに拡張するための基盤を提供するために、バッファ内の電子の仕事。
概要
小分子は、長い生理毒性または栄養、細胞シグナル伝達、および疾患の1医薬の治療などの多様な生物学的プロセスにおいて重要な役割を果たしていると認識されている。種々の小分子はまた、ストレス2,3、疲労4、及び5を含む疾患の生理学的状態を示すバイオマーカーとして提案されている。例えば、上昇したコルチゾールレベルが低下し、生理学的パフォーマンスと他の健康状態6-8になることがあり、ストレスと相関する。生理的な症状は、集中力欠如障害、反応時間、および還元認知機能4を含む場合も同様に、唾液中の特定のペプチドの比の変化は、疲労を予測する。したがって、小分子のレベルを監視するための標的特異的バイオセンサーの開発がINDIVの状態とパフォーマンス能力を評価するための貴重なメトリックを提供することができるidual。
歴史的に、小分子の検出は、労働集約的な分離技術によって、または抗体に基づく認識6,7によって行われている。より最近では、核酸アプタマー9,10は、特定の用途での抗体に比べて明らかな利点を有する認識要素として浮上している。金属イオン組織12〜11の大きさの変化の標的に結合するそれらの能力に、アプタマーは、広くバイオセンシングプラットフォーム13,14における認識要素として適用されている。抗体と比較して、アプタマーは化学的に合成され、そしてそれは、表面固定化または報告のための再現性のある化学修飾を導入することが簡単であるしている。抗体は、生理学的条件15,16に限定されるのに対し、アプタマーは、使用される選択条件を変更することにより、所望の条件下で高い標的特異性で選択することができる。また、アプタマーは番目に、より高いリガンド密度が可能であるEIR小型化バイオセンサープラットフォーム上で効率シグナリング高いターゲットが得られると、アプタマーの安定性の向上は、繰り返し使用や長期センサ記憶装置16を可能にする。
アプタマーは、配列の最初のライブラリーは、標的分子に結合する可能性のあるいくつかの(10 1 -10 2)配列を含む濃縮された最終的なプールに10 13 -10 15のユニークな分子から発生されるSELEXとして知られている手順を使用して生成される。最終的な濃縮されたプールは、次いで、配列決定され、そして解離定数(K d)は、標的分子と最高コピー数配列の結合アッセイから得られる。最終的な濃縮された集団のプールの進化は、最大の濃縮が得られるまで、各ラウンドにおける標的結合プールの割合を監視することによって追跡される。これは結合配列は、非結合体とAMPL仕切られている進化のラウンド数、を介して行われますポリメラーゼ連鎖反応(PCR)を使用してified。効果的な結合剤を分割して保持する能力は、選択の効率と直接影響を与える選択されたアプタマー15の特性の主な決定要因の一つである。それらはサイズまたはタンパク質標的1,15の分割処理を助ける様々な官能基を有していないので、SELEX区分化段階は、小分子のために、より困難である。例えば、多くのタンパク質のパーティションプラットフォームは、しかしながら、結合したDNA、小分子標的複合体の特性は、無効パーティション1で得られた、一般的に非結合配列のそれよりも有意に異なっていない、大きさまたは電荷に基づく分離に依存している。代替セレックスパーティション化メソッドは、潜在的に、小分子の結合特性を変化させるもの、対象の固定化または標識を含むことができる。その結果、選択手順の設計は、小分子ターゲットrのためのアプタマーを生成する特別な配慮をequires。
種々の修飾は、手順の区分化効率を高める最終プール中の配列の親和性または特異性を改善し、または異なるアプリケーション15,16に対してより従順にするために、元のSELEX法に適用されている。小分子を選択することができる一つSELEX修飾は、標的分子17,18と相互作用する際にコンホメーション変化を受ける構造スイッチングアプタマー、またはアプタマーを生成するように設計した。この方法の一例において、ライブラリーは、磁気ビーズ17上に固定非結合の相補的DNAの短い断片(cDNA)をハイブリダイズさせる。のcDNA /ビーズにハイブリダイズし、残りの非結合体を磁気的に分割され、廃棄されている間、ターゲットが結合すると、結合配列の構造変化は、上澄み液にそれらを解放し、cDNAから脱ハイブリダイズすることを可能にします。この方法は、アドバンですtageous小分子を、分離を達成するために、標的分子の固定化または標識化を必要としないからである。
構造スイッチングが可能であるアプタマーは、標的分子の存在を検出するためにアプタマー構造の変更を必要とする任意の潜在的なバイオセンサプラットフォームのための貴重な特徴を有している。具体的には、アプタマーは、塩チャレンジ19,20の後、標的分子の存在下での比色応答を生成するために、構造スイッチング原理に基づく機能アッセイを作成するために、金ナノ粒子(AuNPs)と組み合わせることができる。 AuNPアッセイでは、一本鎖DNA(ssDNA)アプタマーは、最初AuNP表面に吸着し、塩チャレンジから保護する。ターゲットの存在は、塩の添加後に赤色から青色に変化をもたらす、AuNP表面を露出させるアプタマーのコンフォメーション変化を誘導する。 AuNPアッセイはまた、信号、マイクロモルの親和性アプタマーを増幅することが示されているsは解離定数(K d)が21より低い大きさのレベルのオーダーで標的を検出することができる。この機能は、親和性は、通常、低マイクロモルからミリモル濃度1,15の範囲の小分子ターゲット、のために特に魅力的である。一般的に、アッセイは、バイオセンサの検出プラットフォームとしてアプタマーAuNPアッセイのさらなる研究を奨励する、実行することも迅速で比較的簡単である。
この研究の目的は、代表的な分子としてストレスマーカーのコルチゾールを用いたバイオセンシング用途のために小分子に構造スイッチングアプタマーを選択するための汎用のプロトコルを提供することにある。 AuNPバイオ検出スキームは、その動作の簡単さと比色読み取り値に関心があるが、構造スイッチングアプタマーはまた、アプタマーconformatioの変化を介して検出を提供するような電気化学22または蛍光23などの代替のセンシングプラットフォームの出力に適用される標的結合の際にn個。以前の方法と比較して、複数の負の選択ステップは、設計17,18に組み込まれた、簡単なUVベースの濃縮検出方式( 図1)を実施した。このプロトコルは、従来の方法17で使用される放射性リガンド濃縮検出スキームとは対照的である。提案される方法はまた、最大濃縮は24を観察した後、選択のストリンジェンシーを区分化効率を向上させ、効果的に調整するために、選択に用いたcDNA捕捉プローブの長さの増加を組み込んだ。調整されたストリンジェンシーの最終プール中の標的により溶出DNA低い総割合リードが、以前のラウンドから最高コピー数配列と比較して増強された親和性で結合した配列のより高いコピー数をもたらした。最終プールから最高コピー数配列は、配列が、プラットフォームに適したことを示すためにAuNPアッセイに適用したそれが標的結合を示すために構造変化が必要です。このバッファベースのアッセイは、表面に塗布AuNPアッセイの応答は、DNAの濃度を変えることによって変更することができることを示しており、小分子、アプタマーベースのAuNPバイオセンサーの開発に向けて今後の研究努力を注ぐための原理証明ワークとして役立つ生理液。
プロトコル
1.図書館とプライマー設計と合成
- 25,26(このトピックでは、以前の出版物で広く検討されている)最初のライブラリおよびプライマーを設計します。
- 標準的なホスホルアミダイト化学を使用して、この研究のために、以下の配列を合成する。
ライブラリ:GAATGGATCCACATCCATGG-N 40 -TTCACTGCAGACTTGACGAAGCTTGACGAA
フォワードプライマー:GGAATGGATCCACATCCATGG
リバースプライマー:ビオチンAAGCTTCGTCAAGTCTGCAGTGAA - ライブラリーの5 '領域に相補的であるようにするcDNA捕捉プローブを設計する。増加した融解温度を供給する後続の各塩基と、わずかにRT上記融解温度を提供するためにオンライン溶融温度ソフトウェアの分析による初期長さを選択してください。
マー捕捉プローブ:CCATTCCビオチン
マー捕捉プローブ:TCCATTCCビオチン
マー捕捉プローブ:ATCCATTCCビオチン
- 標準的なホスホルアミダイト化学を使用して、この研究のために、以下の配列を合成する。
- 標準HPLCによるライブラリを精製する。脱塩は基本仕様であるプライマーと捕捉プローブのためのcient。数ヶ月まで-20℃で所望の濃度と店舗でのヌクレアーゼを含まない水でオリゴヌクレオチドを再構成する。
2.バッファ、図書館、サンプル調製
- ミリポアまたは50 mMトリス、137のNaCl、5mMのMgCl 2、pHが7.4の濃度のヌクレアーゼフリーグレードの水でバッファの500ミリリットルを準備します。無菌0.2μmの膜を通してバッファをフィルタリング。ストレージヶ月間、周囲条件で可能である。
- あるいは、 等 PBS(リン酸緩衝生理食塩水)、HEPES(4-(2-ヒドロキシエチル)-1-ピペラジンエタンスルホン酸)などの他のバッファーを使用
- 2サーモを設定する。 95℃に1を設定し、周囲条件(この作品で〜20℃)で他を保つ。アイスバケットを準備します。
- 熱/サーモセットA(10 15 -10 16配列について〜2.5ナノモル)100μlのライブラリを配置することによって、DNAライブラリーを冷却スナップ5分間トン95°C。磁気ビーズを準備している間(セクション3.1から3.6)チューブを氷上に置きます。 λ= 260nmでのUV吸収によるDNAライブラリーの実際の濃度を決定し、ライブラリの適切な変換係数を適用する。
- ターゲット·溶解性のための媒体の適切な中で、目標のストック溶液を準備します。
- DMSO中100mMの分析物のストックを準備します。これらの株式は、周囲条件で保存され、約1カ月生存しているが、別のターゲットは異なる分解プロフィールのデモンストレーションを行います。
磁気ビーズと選択の初期ラウンドの調製
- ボルテックス6.7×10 8ビーズ/ mlのストレプトアビジン被覆磁気ビーズと新しいマイクロチューブに400μlのを削除します。 2分間磁石上にチューブを置き、上清を除去。
- 、結合緩衝液の400μlのを追加し、ボルテックスし、配置した後、上清を吸引することにより、ビーズを洗浄ビーズを分離するための磁石にチューブ。このプロセスを3回繰り返します。
- 50μM(最終)捕獲プローブと400μlの結合緩衝液中でビーズを再懸濁し、周囲条件でサーモミキサー上で穏やかに攪拌しながら10分間インキュベートすることによって、磁気ビーズ上の捕捉プローブを固定化する。
- 無料の捕捉プローブを除去するためのセクション3.2で説明した洗浄ステップを繰り返すことによって、結合緩衝液の400μlのビーズを3回洗浄します。
- 結合緩衝液400μlのビーズを再懸濁します。次のステップのためのビーズ溶液100μlを外し、3週間の最大のためのさらなる選択ラウンドに使用する4℃で、残りの300μlのを保持します。
- 3.2節で説明したように、結合緩衝液200μlで二回ビーズを洗ってください。
- スナップ100μlの洗浄したビーズにセクション2.3からDNAを冷却して追加し、周囲条件でサーモミキサーを使用して穏やかに撹拌しながら30分間インキュベートする。
NOTE:最初のラウンドでのDNA濃度は、特有の配列は、理論的に存在している以前のラウンドでの配列の損失を最小限にするその後のラウンドのものよりも高い。 - 2.3節のように、UV吸収を用いて上清中のDNAを定量し、ビーズに結合したDNAの量を評価するためにセクション3.7で追加された初期量からこれを差し引く。
- 周囲条件で5分間、サーモミキサーで穏やかに撹拌しながら、結合緩衝液200μlの二つの追加回洗浄し、結合緩衝液200μlでビーズを洗ってください。
- 結合緩衝液で希釈した100μMの標的200μlのビーズを再懸濁し、そして周囲条件で30分間、サーモミキサー上で回転する。ターゲットと制御が時間をかけて、水溶液から集約することが観察されたため、以前の選択に、毎日の初めに新鮮なワーキング溶液を調製する。ターゲット溶出配列を含む上澄み液を保持さらなる研究のための。
- セクション3.8.1で作成ビーズに1μMのプロゲステロンの200μlのを追加して1-2ラウンド終了後の負の選択を行います。周囲条件でサーモミキサー上で30分間穏やかに振とうする。
- ビーズを捕捉し、前のセクション3.9のように、コルチゾールのほかに結合バッファー200μlの二回ビーズを洗浄、上清を廃棄するように磁気スタンドを使用してください。
4.濃縮モニタリング、PCR、および一本鎖DNAの生成
- 選択濃縮を監視するために透析カセットに上澄み液を加える。透析は、コルチゾールが存在するため、DNAよりもはるかに高い濃度で溶出したDNAからコルチゾールを除去することが必要であり、また、標準的なDNA UVλ 最大 = 260nmのと重なるUV吸収ピークを有する。
- このステップを選択の初期のラウンド内の他のすべてのラウンド(ラウンド2、4、6、および8)を実行します。これは、サンプルロスを軽減sの各シーケンスの潜在少ないコピー数(最高多様性)が存在するとき。より速く結果を得るために、そのようなサイズ排除カラムのような別の方法も利用することができる。
- 二回、周囲条件で2時間の結合緩衝液新鮮250mlのサンプルを有するカセットを透析する。バッファーを交換し、4℃CO / Nでインキュベートする。
- (セクション3.8の結果と比較して)ターゲットによって溶出したDNAの割合を決定するために、UV分光法によって透析プールを分析します。
- エチジウムブロマイドw / vの1%と3%アガロースゲルを準備します。この作品では、アガロースの1.5グラム、TAE緩衝液(40 mMトリス酢酸、1mMのEDTA、pHは= 8.0)の50ミリリットル、エチジウムブロマイド10μlのからゲルを調製。
- 〜5-15サイクルを使用して小規模PCR(5×50μlのアリコート)を行うことによって透析したプールをサイクル·最適化する。 PCR構成要素については、1×PCR反応緩衝液(全て最終濃度)、200μMのdNTP、DNAテンプレートの10%の反応容積、500 nMの各プライマー、および2.5を使用してUポリメラーゼ(2.5 U /μLストック)。
- 2分間95℃で最適化された条件を用いてPCRを実行し、その後、72℃(10〜95℃(30秒)、55℃(30秒)、72℃(1分)の反復サイクル、続いて分)、最終伸長し、4℃で保持する。
- 比較のために25bpの標準ラダーを採用し、ゲルイメージャーで可視化する。不要な副産物ずに正しいサイズマーカーで最高強度のバンドを生産サイクルの数を選択します。
- 6Xブルー/オレンジローディング色素の2μlの各PCRサイクルの10μlのを組み合わせて、ステップ4.4(45分間125 V)で調製し、3%アガロースゲルの5つの別々のウェルにロードすることによって、サイクルの最適化の結果を分析します。
- セクション4.5で決定された条件と適切なサイクル数を利用し、大規模なPCRを行います。通常のPCR反応6-8 mLのこの選択で各ラウンドのために使用される1〜2.5ナノモルを得るために必要であった。簡素化するために、8ストリップチューブを使用して、PCR増幅のためにこのボリュームをアリコートするために必要な多くのチューブの取り扱い。
- 4.4節で説明したように、3%アガロースゲルを調製し、PCR産物のバンドはセクション4.5.2-4.5.3の手順に従って、正しい場所に表示されていることを検証する。
- 以下の手順により、一本鎖DNAにセクション4.7から全体の二本鎖DNA産物を変換します。
- フリットによってブロック小塔にストレプトアビジン被覆ビーズ(磁気ではない)の300μlを添加する。ルアーロックシリンジを取り付け、プランジャに穏やかな圧力を加えることにより、結合緩衝液5mlでビーズを洗浄する。列から注射器を取り外し、ビーズが、プランジャ吸引によって妨害されないように、各材料の添加後にオフラインシリンジからプランジャーを取り外します。
- PCR産物を追加し、プランジャーで穏やかな圧力を使用してカラムに通し。 PCR産物を超えない1-1.2よりmlの各カラムに添加されるように複数の列を使用する。最終FLを破棄DNAは、アンチセンス鎖上のビオチン部分によってカラム上に保持されるので、スルーOW。
- 結合バッファー5ミリリットルでカラムを洗浄します。
- 0.2 MのNaOHを0.5mlを加えることによってDNAを脱ハイブリダイズ。それは一本鎖センスDNAが含まれているように溶出液を保持します。
- ヌクレアーゼを含まない水で洗浄して調製脱塩カラムに0.5ミリリットルを追加することで、0.2M NaOH中のssDNAを脱塩。その後1ミリリットルヌクレアーゼを含まない水を追加し、この脱塩したssDNAサンプルを収集し、初期の0.5ミリリットル流体を捨てる。複数の脱塩カラムを使用すると、0.5mlずつの部分に必要とされる。
- λ= 260nmでのUV吸収により溶出したDNAの濃度を決定する。
- 真空は、サンプルを乾燥させ、結合緩衝液の適切な容量の再構成。選択の次のラウンドのために取得した十分なDNAがなかった場合は、セクション4.6-4.9.2を繰り返します。
選択とシーケンシング5.その後のラウンド
- 濃縮監視(4.1節)の結果に応じて選択条件のストリンジェンシーを調整します。一般的には、プールからの最も高い親和性バインダーを選択するために、連続した選択ラウンドで条件をより厳格にする。
注:これは、DNAの濃度を増加させる洗浄工程数、時間、または体積を増大させる、標的濃度が減少する負の選択化合物の濃度、または他の方法27の種々の増加との組み合わせを含む可能性がある。- 標的分子のための配列の特異性を確実にするために、必要に応じて適切な制御を適用する。この作品では、システム( 表1)3ラウンド開始し、選択を通じて濃度の増加に負の選択分子(プロゲステロン)を適用する。 (セクション3.7の前に)ビーズ上に固定化前に10〜20μMのプロゲステロンと30分間プレインキュベートDNAプール、ラウンド11で起動する。
- 最大の濃縮が観察された後、捕捉プローブの長さを増やします。
- 互換性のあるプライマーと高忠実度ポリメラーゼによるPCR増幅による配列決定のために最大限に濃縮された(ラウンド13)と最小限濃縮(ラウンド6)の条件を表す最終プール(ラウンド15)と他のプールを準備します。
- 1Xの最終濃度を用いて、50μlの反応容量(反応バッファー)、200μM(各dNTP)、400 nMの(各プライマー)、DNAプール、およびポリメラーゼ0.5μlの0.5μlのを設定します。サイクルは、最後の伸長のために72℃で7分間保持を除いて、同じPCR条件を使用して、セクション4.5に記載されている手順を使用して、ラウンドごとに最適化します。
- 徹底的に非接着ラップで密閉キャップでマイクロ遠心チューブにシークエンシング施設に準備されたプールの50μLを送信します。プチプチと船のO / Nシング施設へアイスパックとが充填された15ミリリットルコニカルチューブにチューブを置きます。
- シーケンスされたプール(複数可)の濃縮を評価するために、各配列のコピー数を決定します。
- シーケンシング機能によって提供されるすべてのプールのデータで繰り返さ各シーケンスの頻度/コピー数を決定するために、FASTA形式で次世代シーケンシングデータのための手順を(補助コードファイルを参照してください)ソート/コード·ライティングを使用します。
- 最高コピー数系列の特異性と親和性を分析します。相互作用(タンパク質または小分子)、結合時にアプタマーの構造特性、及び予想される親和性のタイプが適切である方法が決まります。このトピックでは、参考文献1,28-30の数にさらに詳細に検討されている。
6. AuNP合成と検出
- クエン酸還元法21を用いてAuNPsを合成する。沸騰への50mMのHAuCl 4と熱の2ミリリットルでミリポア水の98ミリリットルを混ぜる。
- として38.8 mMクエン酸ナトリウムの10ミリリットルを追加します。すぐに逆流が始まると。色は、数分後に赤色に変わります。熱をオフにして20分間溶液を攪拌し続けます。
- AuNP溶液は0.2μmのポリエステル膜でろ過後、室温まで冷却させる。暗い琥珀色の瓶の中にすべてのAuNP懸濁液に保管してください。
- 2.4×10 8 Lモル-1 cm -1で31の吸光係数を用いて、UV吸収によってAuNP濃度を計算する。濃度が21を動的光散乱によって決定される17±0.6程度の大きさで、この作業に10 nMであった。
- アルミホイルで保護され、周囲条件で1時間10 nMのAuNPでDNAをインキュベートします。 AuNPの体積は全ての所望の試験を実施することを可能にするのに十分な解決策を提供することが異なり(〜1〜2ミリリットル)。 73のローディング密度、120、及び200 D / NP(AuNPあたりDNA分子)本研究で検討した、体積および濃度に応じて変化するであろう。
- HEPを用いて、1:DNAを希釈/ AuNP溶液1ES緩衝液(20mM HEPES、2mMのMgCl 2を 、でpH = 7.4)。混合物をアルミホイルでカバーRTで平衡化することを許可する。
注:このステップに必要な時間は、研究者によって最適化される必要がある。この研究では、数分からO / Nの範囲の時間を調べた。 - DMSO中の100mMでの分析物(コルチゾール、コール酸、2-メトキシ)ストック溶液を準備し、アルミホイルでカバーしています。バッファー(2.1節)を結合コルチゾールの1/3希釈さ100μmに株式の希釈によって各実験の前に新鮮な初期解を行います。さらに1/3の結合緩衝液を用いて、初期溶液を希釈するように、目標の一定量(10μl)を、濃度範囲を生成するために添加される。
- 1/3の結合緩衝液10μlのDNA / AuNP溶液80μLを添加することにより、必要な塩濃度の最適化(「ブランク」と呼ばれる)。
- その後のNaCl solutの異なるボリュームを追加RTで20分間インキュベートイオン。塩の添加(この作品で使用されるの1M NaCl 13-40μL)の後に青の色相に向かってかろうじて視覚的に顕著な変化を誘導し、塩のボリュームを探します。これは良い出発点を提供しますが、ターゲット·追加を含めた結果、以下のさらなる調整が必要な場合があります。
- 対象溶液10μlのDNA / AuNP溶液を80μLを組み合わせて、室温で20分間インキュベートする。常にブランク(6.8節)を含め、同時に複数の標的濃度を分析するために、96ウェルプレートおよびマルチチャンネルピペットを使用しています。
- NaCl溶液の適切な量(1 M NaClを13μlのこの作業に適用された)を追加し、直ちにプレートリーダーを用いて650と530 nmの吸光度を分析する。
- ブランク測定の目標濃度に対して正規化対吸光度値の比(E 650 / E 530)として結果をプロットする。
結果
条件のストリンジェンシーは、最初に、最初の数ラウンドで低コピー数で存在する結合剤を保持するために低く維持した。最初のラウンドは、特に、一般的に高いコルチゾールとDNA濃度、及び負の選択手順の欠如によって実証ユニークな配列として提示バインダーを保存するために最低のストリンジェンシーを実装しました。このアプタマーの選択の成功は、ラウンド12-13で比較的一定のままでターゲット( 表1)によって溶出分画に標的(ラウンド2)により溶出割合が低いからプールの進化によって実証される。ターゲットによって溶出したDNAのこの高原は、伝統的に選択(「濃縮」)でエンドポイントです。しかし、この方法はさらに、cDNAの捕獲プローブ( 図1)の長さを増加させることによって濃縮した後、選択のストリンジェンシーを上げた。このプローブを長くすると、CDNとの間のハイブリダイゼーションの強度を増加させるAプール、複合体の融解温度(T m)を増加させると、溶液中の結合配列を解放するために、標的と配列との間の強力な相互作用を必要とする。選択の最初のラウンドは、ライブラリの5 '末端上の1未満のG塩基に弱いのcDNAとの対話を実施しました。これは、最初のラウンドで、一般に、より低いストリンジェンシー条件と一致している、著しく次の選択ラウンドに渡す複数の潜在的な結合剤、ならびに背景配列(熱力学に基づいて脱ハイブリダイズ)させることによって他の選択に影響を与えないと予想される。選択は、その後の選択ラウンドにおいてより高いストリンジェンシー条件を実施することにより、濃縮に向かって継続するように、これらのバックグラウンド配列は、最小化した。 cDNAは、ラウンド14で8量体、7量体より長くした場合に、T mは 26.7℃、19.2℃から増加するために、ターゲットによって溶出したDNAのパーセンテージは11.1%15.3%から減少している。 cDNAはWとしてさらに3.3%に溶出した合計を落とし、31.3°C の T mとのラウンド15で9マーに拡張。
濃縮に関するさらなる情報は、各ラウンドでのプールの進行を追跡するために、濃縮された非富化例とも、複数のプールを配列決定することによって得ることができる。次世代シークエンシング(NGS)は、より高い配列読み取り主な理由は、選択を配列決定のための強力な方法として、(報告された配列の総数)が出現したサンガー配列と比較して得られる。これらの高い配列は、プール中の配列の総数の存在も大きい範囲を読み込み配列の多様性のより完全な像を提供する。 NGSはまた、クローニングバイアス32を削除し、バーコード24,33を追加した単一のバッチで複数のプールのシーケンス制御することができます。 NGSは、このサンプルの選択( 図2)に3つの別々のプールからの濃縮の進行を分析した。プール6(若干の濃縮が溶出し%でラウンド2に比べて)、ラウンド13(最高濃縮)、ラウンド15(最高ジェンシー)ラウンドからの選択の代表点として選択した。シーケンスの総数の割合は、各プールに読み込むように、各固有の配列のコピー数をプロットした。ランクトップ(最高コピー数)シーケンスは、すべてのラウンド6で配列(33435合計シーケンス、22463ユニークな配列)の0.1%を構成した。プールが最大限ラウンド13で富化なると、トップランクのシーケンス増加はUVによって観察濃縮でのみ〜5倍の増加にもかかわらず、ラウンド6に比べて150倍高いプール全体の15.4%(17681合計系列、2987ユニークな配列)を含むように。 (28919全配列を読み取り、2,980ユニークな配列)15ラウンドは、UV濃縮監視がターゲットによって溶出量が(ラウンド13より〜5倍低かったことを示したにもかかわらず、単一のトップランク配列で表されるすべてのシーケンスの44.9%に跳ね上がった表1 )。濃縮はまたプールは潜在的な標的結合といくつかの配列に多くの進化に合わせてユニークな配列の低い割合(ラウンド15で10%ラウンド6で67%)によって実証される。 cDNAの長さが延長された、ラウンド13で第二位のシーケンスがラウンド15で最も高いコピー数の順序になった後にさらに、ラウンド13(15-3)でトップランクのシーケンスがラウンド15で三番目に高いコピー数のシーケンスであった(15 -1):
15-1 GGAATGGATCCACATCCATGGATGGGCAATGCGGGGTGGAGAATGGTTGCCGCACTTCGGCTT
CACTGCAGACTTGACGAAGCTT
15-3 GGAATGGATCCACATCCATGGGAGGGTTGGAAGGGAGGGGCCCGGGGTGGGCCATCGTTCGTT
CACTGCAGACTTGACGAAGCTT
これまでの研究では、マイクロスケール熱泳動と平衡透析24によりこれら二つの配列の結合を調べた。結果は、平衡透析によってシーケンス15-1(のK D = 6.9±2.8μMのバインディング重要なコルチゾールを示した。16.177。マイクロスケール熱泳動による0.6μm)を最小結合しながら、コルチゾールとシーケンス15-3との間で観察された。これは、ラウンド15で捕捉プローブの長さの増加は、より高い親和性結合を解析するために、より高いストリンジェンシー条件を提供する際に支援することを示す。さらに、いずれの配列が負の選択手順の追加、プロシージャに有益であったことを示し、負の選択分子に結合プロゲステロンを観察可能示した。
上昇したストリンジェンシー条件は、さらなる回の選択に適用された後、より高い親和性のバインダーが最大富化(標的によって溶出したDNAの割合を分析する従来の方法によって決定される)以下の出現するという事実は、cDNA捕捉の長さを拡張する方法を検証するポスト濃縮をプローブ。この目標のために配列決定プールとの親和性から番号をコピーし、結果のみを示して詐欺で、一定の条件の下で24,34に関連付けることができる直接的な相関関係35を意味する前にレポートとtrast。それはまた、より高いストリンジェンシー条件が適用されるように1ラウンドから次へ自然に変化し得る、というだけで、ほとんどの選択の中で一般的な%溶出した戦略によるよりも、NGSによる監視濃縮の利点を強調しています。しかし、溶出した%は、同様の条件はいくつかのラウンドの間に印加されると濃縮度をモニターするのに便利です。例えば、濃縮には有意な変化( 表1)、6-10すべての関係と同様の条件を丸めます。これは数回にわたって観察された場合、条件は濃縮を促進する厳しすぎる可能性があり、研究者は、次のラウンドでのストリンジェンシーを減少させるはずである。例えば、コルチゾール濃度は、ラウンド11-13で100μMまで35μMから増加し、そして%が調整するためのガイドとして役立つことができるので、ラウンド12に14.5%へ、ラウンド10で2.5%から増加%溶出した溶出した選択の過程ジェンシーsimilアル条件はラウンドのラウンドから比較され、選択エンドポイントを決定するためのNGSに相補的な情報を提供することができる。
配列15-1は、構造スイッチングアプタマーを生成するような方法で選択された配列は、応答を生成するために、標的結合以下の構造変化に依存するアッセイにおいて機能するかどうかを決定するためにAuNPアッセイに適用した。以前の初期の研究では、15-1とAuNPアッセイはストレスバイオマーカーコルチゾールのではなく、ストレス、エピネフリンおよびノルエピネフリン、またはコール酸、コルチゾール24と構造的に類似した肝臓疾患のマーカーの二つの他のマーカーに反応したことを示した。応答は、コルチゾールの結合の際に構造変化のために選択されたアプタマーは、AuNPアッセイにおける応答を生成することを検証するヒト血清(〜150〜600 nM)を6に報告遊離コルチゾールの正常範囲で観察された。この現在の研究では、システムの特徴付けを調整することによって、さらに一歩を取ったAuNP表面上の15-1の報道(密度)。条件の同じセットを使用して、DNAカバレッジが120 D / NP及び200 D / NP( 図3)、73 D / NP(DNA分子/ AuNP)まで増加した。コール酸は200 D / NPで10μMを除いて、最小限の応答を生成した。検出限界(LOD)を行ったように報道として減少したアッセイの応答が増加した。 LODは120 D / NPのための73のD / NP、145.2 nMのための29.5 nMで、及び200 D / NPのための27.3であった。この結果は、アプタマーカバレッジが300 D / NP 36 60 D / NPから増加させたときにコカインアプタマーを利用AuNPアッセイの応答が減少したことを示したSmith らの仕事と一致する。これは、目的の標的の検出範囲は、AuNPアッセイにおいてDNA表面被覆率を最適化することに基づいて調整することができることを意味する。ヒト唾液(〜5-25 nMで)対ヒト血清(〜150〜600 nM)の中遊離コルチゾールの、例えば、範囲を比較する場合に有益であり得る 6,37。生理的な流体は、このバッファアッセイよりはるかに複雑ですが、結果はアプリケーションに応じてAuNP条件を調整することができることを実証する証明の原則の仕事上の拡張機能、および生物学的流体中〜を拡大することになるに向けて、その更なる作業を提供バイオセンサーコミュニティへの関心の。

構造スイッチング選択方法図1.概要小ビオチン化cDNAの捕捉プローブを、ストレプトアビジン被覆磁気ビーズ上に固定化される。 cDNAをビーズにライブラリをハイブリ、ライブラリーの5 '領域に相補的である。ターゲットが追加されると、コンフォメーション変化は、磁石を適用することによって容易にパーティショニングを可能にする、ビーズから結合配列を放出するように誘導される。上清を目により溶出し、濃縮(%のDNAを監視するために保持されているDNAからターゲットを透析後にUVによるEターゲット)。標的分子がDNAと同一のUV範囲で吸収する場合は、このステップは必要である。次いで、この配列をPCR増幅し、選択の次のラウンドのために準備される。選択が進行すると、負の選択、負の選択分子によって溶出シーケンスが破棄される場合、適用され、潜在的な標的結合剤を有するビーズは、その後、標的と共にインキュベートされる。 cDNAプローブの長さは、選択のストリンジェンシーを増加させる最大の濃縮(%溶出)後に増加される。この図は、24から許可を得て転載してきた。 この図の拡大版をご覧になるにはこちらをクリックしてください。
| ラウンド | [コルチゾール](μM) | [プロゲステロン](μM) | 結合したDNA(ピコモル) | ターゲットによって溶出% | cDNAの長さ |
| 1 | 100 | 0 | 357.57 | N / A | 7量体 |
| 2 | 50 | 0 | 145.49 | 1.4 | 7量体 |
| 3 | 50 | 1 | 188.74 | N / A | 7量体 |
| 4 | 50 | 5 | 336.4 | 2.3 | 7量体 |
| 5 | 35 | 5 | 88.05 | N / A | 7量体 |
| 6 | 35 | 10 | 303.89 | 3.1 | 7量体 |
| 7 | 35 | 10 | 255.01 | N / A | 7量体 |
| 8 | 35 | 10 | 236.81 | 2.1 | 7量体 |
| 9 | 35 | 10 | 318.95 | 2.6 | 7量体 |
| 10 | 35 | 10 | 297.18 | 2.5 | 7量体 |
| 11 | 100 | 10 | 147.61 | N / A | 7量体 |
| 12 | 100 | 10 | 154.36 | 14.5 | 7量体 |
| 13 | 100 | 10 | 150.39 | 15.3 | 7量体 |
| 14 | 75 | 20 | 247.71 | 11.1 | 8量体 |
| 15 | 50 | 40 | 409.63 | 3.3 | 9マー |
%溶出24によって、表1の選択の進行および濃縮 < (該当なし)strong>の。N / Aは、透析/ UV濃縮の監視は、低コピー数系列の損失を軽減するために、早期の選択ラウンドで行われていなかったラウンドのために報告されます。この表は、24から許可を得て転載してきた。
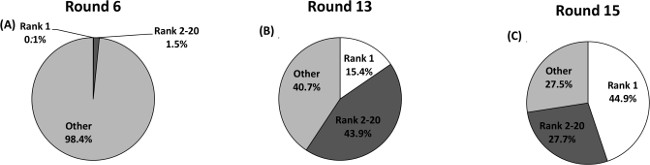
次世代シークエンシングによって決定図2セレクション濃縮(A)ラウンド6。 (B)丸13。 (C)ラウンド15配列は、コピー数に応じてランク付けした。最高コピー数シーケンスは、プールがコルチゾール結合配列について濃縮されたように、ラウンド15(44.9%)で高い割合にラウンド6(0.1%)で全体の配列決定され、プールの低い割合を構成しているから増加。この図は、24の許可を得て転載してきた。ら= "_空白">この図の拡大版をご覧になるにはこちらをクリックしてください。

アプタマー15-1カバレッジを変えることから図3. AuNPアッセイ応答。AuNPsとインキュベートアプタマーの密度は120 D / NP(緑色の四角形)と200 D / NP(に、73 D / NP(赤い三角形)から増加した青丸)。コルチゾール応答が大胆な色である、コール酸応答は、明るい色である。アッセイ反応は、このようにLODとターゲット内で検出可能な範囲を下げ、より低い密度でコルチゾールのために改善される。最高コルチゾール応答(73 D / NP)との条件がないヒト血清(〜150〜500 nMで)で報告コルチゾールと唾液(5-25 nMでの予想される正常範囲内でより多くのポイントを提供するために、線形範囲でさらに特徴づけた)6,37。同じ73のD / NP条件を適用コール酸の最小限の応答がなけれあります前作24で詳述EN。すべてのプロットは、重複または3回の測定のための平均±SEMを表す。

。PCRサイクルの最適化から図4.代表的な結果から、井戸を右に左に表す:4サイクル、6サイクル、8サイクル、10サイクル、12サイクル、負(無DNAテンプレート)、25 bpのDNAラダー標準を。最適条件は、単一の生成物のバンドは、より高い周期で存在過増幅産物なしで高強度である8サイクルで観察される。
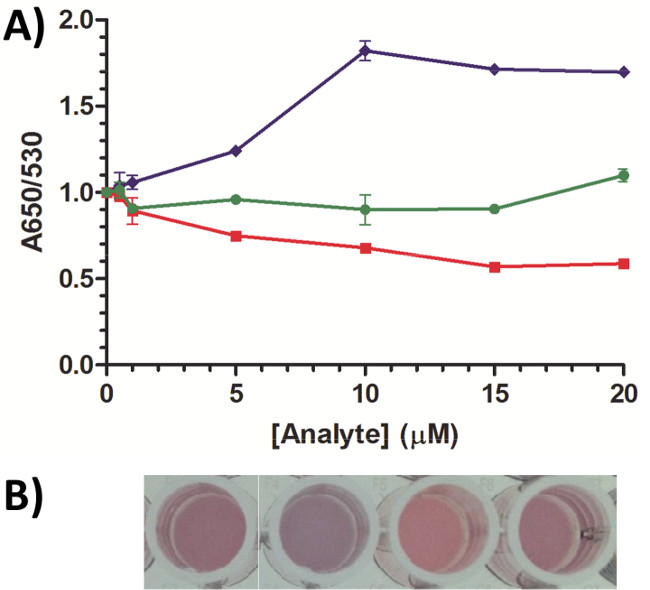
図5 AuNPアッセイインキュベーション時間の最適化。73 D / NP 30分O / N( 図3)、および標的incuから減少した時AuNP / DNAステップのインキュベーション時間bationはすぐに塩を添加することによってではなく、20分( 図3)の後に行った。 (A)応答はコルチゾール(青色菱形)のためではなく、コール酸(緑の円)または2MNPについて観察された(2-メトキシナフタレン;赤四角)。すべてのプロットは、重複または3回の測定のための平均±SEMを表す。ブランク、10μMのコルチゾール、10μMのコール酸、10μMの2MNP:左から右へ(B)。視覚の変化は、コルチゾールのために肉眼で観察することができる。 この図の拡大版をご覧になるにはこちらをクリックしてください。
ディスカッション
小分子は生物学的な関心があるが、唯一報告されている全てのアプタマーの19%を構成しているという事実は、大きな意義の小分子に適用可能なアプタマーを選択するために設計された方法をレンダリングします。より少ない官能基が、構造モチーフ、および表面積は、タンパク質1と比較した配列との相互作用のために利用可能であるような小分子のSELEXは特に困難であり、それは試みられ、すべてのターゲットの選択の30%未満がアプタマーをもたらしたと推定されている38。したがって、1は正常に小分子標的に対するアプタマーを選択するために実験計画と実行の非常に注意する必要があります。
それは、それらの結合特性を変化させることができる任意の化学修飾を必要とせずに小分子に適用可能であるので、この研究に記載のアプタマー選択方法が有利である。それはまた、T以来のことを意味ベースの溶出、ある彼DNAのではなく、ターゲットは、最初にターゲットによって磁気ビーズから溶出後、結合している目的の分子に結合剤上澄み液中に放出されるので、ビーズマトリックスとの相互作用DNAは次回の選択のために増幅されることはありません非特異的なマトリックス結合剤は、結合したまま。逆に、マトリックスに対するネガティブ選択法は、マトリックスと相互作用するDNAは、標的結合剤1に加えて、増幅されるため、固体支持体に各ラウンドの標的を固定化方法のために必要とされる。現在の方法は、T mの限られた選択戦略として設計されました。これは、cDNA /プールハイブリダイゼーションの T mを RTに近く維持したことを意味します。モースは、同様の戦略を用いたが、4°Cで17選択条件での T m <10℃で6-merのcDNAプローブを適用した。私たちのアプリケーションは、室温で行わバイオアッセイのためにあったので、cDNAの長さは応じて調整したLY。標的結合相互作用は結合するcDNAを破壊することができるコンフォメーション変化を誘導しない遠隔地で発生するため、潜在的な結合剤が失われないようにするために十分に低く選択のストリンジェンシーを保つ。いくつかの配列が、熱力学に基づいてもっぱら脱ハイブリダイズしますので、提案手法のパーティショニング効率が低い。対照的に、Nutiu らは、15マーのT mは 、小分子標的による放出を可能にするために高すぎた可能性があるため、15マーのcDNAを適用し、4つだけのターゲットのうち2アプタマーを同定することができた18。したがって、現在の選択戦略は、選択のストリンジェンシーを制御し、成功の可能性が増加する可能性のある結合剤の損失を最小化することによって、バックグラウンド配列を除去するために多くのラウンドを必要とする一方。
アプタマー選択に新しい多くの研究者はのPCRに関連する重要な側面を知らない潜在的なバインダー。 DNAライブラリーの過剰増幅は副産物高サイクル数での(サイズは一般的に大きい)不要な生成し、所望の生成物が完全にのみ5サイクル39の過剰で消える可能性があるため、サイクルの最適化(セクション4.5)が必要です。過剰増幅の場合には、PCR産物は、もはやターゲットによって溶出、これらの配列を表していない、選択成功の可能性が大幅に減少している。 図4は、本研究で5ラウンドからサ イクルの最適化の一例を示す図である。最低のサイクルは、8サイクルを通じて量のバンドの増加を最小限の産物バンドを生成する。 10サイクルのバンドは、より高い大きさを超える増幅産物への移行を開始し、ほぼ完全に12サイクル以内に過増幅産物から構成されている。 SELEXに不慣れな多くの研究者が見落とすことがもう一つの詳細は、プール全体がモミで大規模PCR増幅(セクション4.6)で増幅されなければならないことであるバインダーの損失を軽減するためのSTラウンド。オリゴヌクレオチドからの可能なユニークな配列の数は4は、4つの核酸塩基を表す4 Nであり、そしてNは、ライブラリーのランダム領域の塩基数である。 1.2×10 24の可能な独特の配列のシーケンススペースで、その結果、この作品のために、N = 40。選択の最初のラウンドで使用されるライブラリの2.5 nmolのは、DNAの各コピーが可能性が高いユニークなシーケンスとして初期プールで表現されていることを意味する、〜10 15分子に対応している。そのため、ターゲットによってビーズから溶出したDNAの全体積は、次回の選択のために、各潜在的なバインダーのコピーを保持するために増幅されなければならない。この最初の増幅が発生すると、複数のコピーは、均質な溶液がサンプリングのために想定され、次のラウンドで選択できます。
標的の濃度はまた、慎重に各ラウンドにおいて考慮されるべきである。 Nutiu ら使用ダ選択プロセスを通して1mMの標的の濃度、およびK dは = 600μM18とATPアプタマーを選択した。現在の仕事とモールス17の研究の両方が、選択の過程を通じて減少、100μMのターゲットで始まり、低マイクロモルの親和性を有するアプタマーをもたらした。正確にどのラウンドのストリンジェンシーを増加させる必要がある(低い標的濃度)の濃縮が観察された場合に依存する。別の重要なステップは、アプタマーは、標的分子に特異性を示すように適切な負の選択手順を適用することである。コントロールが使用される、選択されたアプタマーの意図された用途次第である。例えば、アプタマー15-1は、コルチゾールのためのバイオアッセイにおいて、認識要素として機能するように設計されたので、それは生理的流体40に見出されるコルチゾールの代謝前駆体であるため、プロゲステロンは陰性選択分子として使用した。 Nutiu らによって選択されたATPアプタマー。</ EM>は負の選択工程が含まれ、ADP、AMP、アデノシン、およびdATP 18を含む構造的に類似の分子と相互作用しませんでした。
検討のための最後の注意は、AuNPアッセイは、多くの場合、各アプタマー/ターゲットのペアのためにかなりの最適化を必要とすることである。塩添加後に青の色相に向かってかろうじて視覚的に顕著な変化を誘導する塩の量をお探しすることは良い出発点ですが、出発点の調整が応答を観察するために必要になる場合があります。我々はまた、アッセイ緩衝液濃度(緩衝液の塩濃度が高いが、多くの場合、凝集させるための選択緩衝液は希釈を必要とする場合がある)及び組成、DNA被覆度( 図3)、試料調製(いくつかの有機によっては大幅に異なる応答を生成することを見出した標的を溶解するために使用される溶媒マスク応答を標的とする高いバックグラウンドを引き起こすこと)、温度、塩の種類と濃度、及びincuba(ターゲットとAuNP及びDNA / AuNPとDNAの両方の)る時間。完全に最適化された条件下で、その結果は、典型的には、AuNPバイオセンシングプラットフォームの迅速なターゲットレスポンスの利点を実証する、標的インキュベーション時間<5分で観察されている。以降( 図3例えば、 図5アプタマーのインキュベーション時間で/ AuNP工程は30分にO / N( 図3)から減少し、塩が目標添加ではなく、20分後にすぐに(<10秒)を添加した)73、D / NPの積載密度で。これは、前の条件( 図3)を使用して、約40%に対して10μMの標的濃度で( 図5A)、ブランクより〜82パーセント高いコルチゾール応答に増加した。この応答は、肉眼( 図5B)と区別することができる。コルチゾール検出の直線範囲は、上記の条件を使用して、これらの条件は、所望のために最適化することができることを示唆しているとは異なることに注意してください検出範囲。コール酸及び2-メトキシナフタレン(2MNP)コントロールは、有意な応答( 図5A-B)を生成しなかった。研究者は、これらのインキュベーション時間を減らすことが、全体的な強化された信号(ターゲット)またはバックグラウンド(非標的分子)に貢献できるAuNP表面と分析物の増加応答を容易にすることができることに注意する必要があります。そのため、慎重なAuNPアッセイデザインと性能特性評価は、各アプタマー/ターゲットのペアのために必要である。
この手順は、ターゲットの存在を検出するために、DNAの構造変化を必要とするような説明したAuNPアッセイのようなバイオセンシングプラットフォームで機能の小分子構造スイッチングアプタマーを、選択するためのプロトコルについて説明します。しかし、この方法は、実質的に任意のサイズのターゲットに対して同じ前提に機能するような電気化学または蛍光のような他のバイオセンサーシステムに適用することができる。プロトコルの力をさらにによって実験的に開発することができるいくつかのアプローチ。まず、選択自体は、潜在的にそのような小分子標的と相互作用するのに十分に弱い相互作用を提供するcDNA /ライブラリーのハイブリダイゼーションの理想的なT mを決定するような最適化の方法を調査することによって改善することができる、まだ量を減少させるのに十分な強背景の配列は、熱力学のみから脱ハイブリダイズ。これは、労働時間を節約し、試薬の消費量を低減する、選択に必要なサイクル数を凝縮する。選択への変更へのさらなる調査は、特に最初のラウンドで、配列の多様な集団がターゲットに露出するように、ビーズとDNAの最も好ましい濃度を決定することである。これらの証明の原理実験の成功は、生理的な流体にAuNPバッファアッセイを最適化し、適応に向けて更なる資源を投資する基盤を提供する。缶FU急速な、堅牢なバイオセンシングプラットフォームのための正当な必要性がある個々の生理状態、及びヒト血清中のAuNPアッセイのさらなる発展、汗、または唾液の診断ツールとしてnction、この電流のギャップを満たすだろう。
開示事項
配布ステートメントA:一般公開のために承認された。分布は(88 ABW-2014から4103)に制限はありません。 著者は、彼らが競合する経済的利益を持っていないことを宣言。
謝辞
This research was performed while the author (JAM) held a National Research Council Research Associateship Award at Air Force Research Laboratories. Co-author MW held a Wright Scholar Fellowship supported by the Air Force Office of Scientific Research. This work was supported by Air Force Office of Scientific Research, Air Force Research Laboratory, and Bio-X Strategic Technology Thrust.
資料
| Name | Company | Catalog Number | Comments |
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| Dynabeads M280 Streptavidin | Invitrogen | 112.06D | Compatible with DynaMag-2 Magnet |
| DynaMag-2 Magnet | Invitrogen | 12321D | Compatible with Dynabeads M280 Streptavidin |
| Streptavidin Sepharose High Performance | GE Healthcare | 17-5113-01 | This is a high density streptavidin product for ssDNA preparation |
| Hydrocortisone | Sigma | H4001 | ≥98% |
| Progesterone | Sigma | P0130 | ≥99% |
| Cholic Acid | Sigma | C1129 | ≥98% |
| NanoDrop ND-1000 | NanoDrop | ND-1000 | Replaced by ND-2000 model |
| Tris Base | Promega | H5135 | ≥99.9% |
| Sodium Chloride | Sigma | S9888 | ≥99.5% |
| Magnesium Chloride Hexahydrate | Fluka | 63068 | ≥98% |
| 500 mL Filter System | Corning | 430769 | 0.22 µm cellulose acetate membrane |
| DNA Library | Operon | Custom | HPLC purified |
| All other DNA | IDT | Custom | Capture probes and primers- desalted; Aptamers- HPLC |
| Thermomixer | Eppendorf | R | Any model with temperature and mixing speed control will work |
| PfuUltra II Fusion HotStart DNA Polymerase, 400 rxn | Agilent | 600674 | Taq polymerase can be used as well |
| Deoxynucleotide Mix, PCR-Grade | Agilent | 200415 | Other brands will work here |
| 10x Cloned Pfu DNA Polymerase Buffer | Agilent | 200532 | This buffer was optimized for PfuUltra II polymerase |
| GeneAmp PCR System | Applied Biosystems | 9700 | Any model that allows researcher to open lid for cycle optimization will work |
| Agarose-LE | Ambion | AM9040 | ≥99.9% |
| Ethidium Bromide | Sigma | E-1510 | Acute toxicity, inhalation; suspected carcinogen |
| Empty Synthesis Columns, 1 µm Expedite Style | Glen Research | 20-0021-01 | This model is ideal because of the Luer-Lok fitting to couple with syringe |
| Replacement Filters Expedite | Glen Research | 20-0021-0F | 1 µm size |
| Illustra NAP-25 Columns | GE Healthcare | 17-0852-01 | Any brand with DNA grade resin gel filtration column will work |
| Slide-A-Lyzer Dialysis Cassette | ThermoFisher Scientific | 66205 | 2k MWCO used |
| Gold(III) chloride hydrate | Sigma | 254169 | 99.999% purity is important |
| Sodium Citrate Dihydrate | SAFC | W302600 | We have found that the compound produced from different manufacturers greatly affects AuNP assays |
| SpectraMax | Molecular Devices | M5 | Results were similar to Bio-TEK system |
| Synergy | Bio-TEK | HT | Results were similar to Molecular Devices system |
| HEPES Buffer | Amresco | J848 | Any brand that makes a sterilized product will work |
| 250 mL Filter System | Corning | 430767 | 0.22 µm cellulose acetate membrane |
| UV Spectrophophotometer | Varian | Cary 300 | Other brands will work here |
| 5 mL Syringe | Becton-Dickinson | 309646 | This model is ideal because of the Luer-Lok fitting to couple with Expedite column |
| ThermoEC Minicell Primo | ThermoFisher Scientific | EC320 | Compatible with Power Supply |
| Power Supply | Fisher Scientific | FB300 | Compatible with ThermoEC Minicell Primo |
| Dimethyl Sulfoxide | Sigma | D8418 | Flammable liquid |
| 2 Methoxynaphthalene | Sigma | 148245 | 99% |
| Assay Plate | Corning | 3370 | 96-well Flat Bottom, Sterile |
参考文献
- McKeague, M., DeRosa, M. C. Challenges and opportunities for small molecule aptamer development. J. Nucleic Acids. 2012, 1-20 (2012).
- Gatti, R., Antonelli, G., Prearo, M., Spinella, P., Cappellin, E., De Palo, E. F. Cortisol assays and diagnostic laboratory procedures in human biological fluids. Clin. Biochem. 42, 1205-1217 (2009).
- Morgan, C. A., Wang, S., Rasmusson, A., Hazlett, G., Anderson, G., Charney, D. S. Relationship among plasma cortisol, catecholamines, neuropeptide Y, and human performance during exposure to uncontrollable stress. Psychosom. Med. 63 (3), 412-422 (2001).
- Michael, D. J., Valle, B., Cox, J., Lalns, J. E., Fogt, D. L. Salivary Biomarkers of Physical Fatigue as Markers of Sleep Deprivation. J. Clin. Sleep Med. 9 (12), 1325-1331 (2013).
- Kwak, J., et al. Volatile biomarkers from human melanoma cells. J. Chrom. B. 931, 90-96 (2013).
- Stevens, R. C., Soelberg, S. D., Near, S., Furlong, C. E. Detection of Cortisol in Saliva with a Flow-Filtered, Portable Surface Plasmon Resonance Biosensor System. Anal. Chem. 80 (17), 6747-6751 (2008).
- Arya, S. K., Ghornokur, G., Venugopal, M., Bhansali, S. Antibody functionalized interdigitated μ-electrode (IDμE) based impedimetric cortisol biosensor. Analyst. 135 (8), 1941-1946 (2010).
- Kapczinski, F., et al. Allostatic load in bipolar disorder: Implications for pathophysiology and treatment. Neurosci. Biobehav. Rev. 32 (4), 675-692 (2008).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249 (4968), 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346 (6287), 818-822 (1990).
- Ciesiolka, J., Gorski, J., Yarus, M. Selection of an RNA domain that binds Zn2. RNA. 1 (5), 538-550 (1995).
- Liu, J., et al. Selection of Aptamers Specific for Adipose Tissue. PlosOne. 7, e37789 (2012).
- McCauley, T. G., Hamaguchi, N., Stanton, M. Aptamer-based biosensor arrays for detection and quantification of biological macromolecules. Anal. Biochem. 319 (2), 244-250 (2003).
- Huang, L., et al. A label-free electrochemical biosensor based on a DNA aptamer against codeine. Anal. Chim. Acta. 787, 203-210 (2013).
- Stoltenburg, R., Reinemann, C., Strehlitz, B. SELEX—A (r)evolutionary method to generate high-affinity nucleic acid ligands. Biomol. Eng. 24 (4), 381-403 (2007).
- Jayasena, S. D. Aptamers: An Emerging Class of Molecules That Rival Antibodies in Diagnostics. Clin Chem. 45 (9), 1628-1650 (1999).
- Morse, D. P. Direct selection of RNA beacon aptamers. Biochem. Biophys. Res. Commun. 359, 94-101 (2007).
- Nutiu, R., Li, Y. Structure-Switching Signaling Aptamers. J. Am. Chem. Soc. 125 (16), 4771-4778 (2003).
- Luo, F., Zheng, L., Chen, S., Cai, Q., Lin, Z., Qiu, B., Chen, G. An aptamer-based fluorescence biosensor for multiplex detection using unmodified gold nanoparticles Chem. Commun. 48 (51), 6387-6389 (2012).
- Li, H., Rothberg, L. J. Label-Free Colorimetric Detection of Specific Sequences in Genomic DNA Amplified by the Polymerase Chain Reaction. J. Am. Chem. Soc. 126 (35), 10958-10961 (2004).
- Chávez, J. L., MacCuspie, R. I., Stone, M. O., Kelley-Loughnane, N. Colorimetric detection with aptamer–gold nanoparticle conjugates: effect of aptamer length on response. J. Nanopart. Res. 14 (9), 1166-1177 (2012).
- Hagen, J. A., et al. Biofunctionalized Zinc Oxide Field Effect Transistors for Selective Sensing of Riboflavin with Current Modulation. Sensors. 11 (7), 6645-6655 (2011).
- Han, K., Liang, Z., Zhou, N. Design Strategies for Aptamer-Based Biosensors. Sensors. 10 (5), 4541-4557 (2010).
- Martin, J. A., Chávez, J. L., Chushak, Y., Chapleau, R. R., Hagen, J., Kelley-Loughnane, N. Tunable stringency aptamer selection and gold nanoparticle assay for detection of cortisol. Anal. Bioanal. Chem. 406 (19), 4637-4647 (2014).
- Hall, B., et al. Synthesis, and Amplification of DNA Pools for In Vitro Selection. Curr. Protoc. Nucleic Acid Chem. 39, 9.2.1-9.2.28 (2009).
- Sefah, K., Shangguan, D., Xiong, X., O’Donoghue, M. B., Tan, W. Development of DNA aptamers using Cell-SELEX. Nat. Protoc. 5 (6), 1169-1185 (2010).
- Djordjevic, M. SELEX experiments: New prospects, applications and data analysis in inferring regulatory pathways. Biomol. Eng. 24 (2), 179-189 (2007).
- Thomas, J. R., Hergenrother, P. J. Targeting RNA with Small Molecules. Chem. Rev. 108 (4), 1171-1224 (2008).
- Jing, M., Boswer, M. T. A Review of Methods for Measuring Aptamer-Protein Equilibria. Anal. Chim. Acta. 686 (1-2), 9-12 (2011).
- Sachs, E. -. F., Diederichsen, U. . Binding of Triostin Analogues to DNA. , (2011).
- Liu, J., Lu, Y. Preparation of aptamer-linked gold nanoparticle purple aggregates for colorimetric sensing of analytes. Nat. Protoc. 1 (1), 246-252 (2006).
- Cho, M., et al. Quantitative selection of DNA aptamers through microfluidic selection and high-throughput sequencing. Proc. Natl. Acad. Sci. USA. 107 (35), 15373-15378 (2010).
- Martin, J. A., et al. Selection of an Aptamer Antidote to the Anticoagulant Drug Bivalirudin. Plos One. 8 (3), e57341 (2013).
- Schütze, T., et al. Probing the SELEX Process with Next-Generation Sequencing. Plos One. 6 (12), e29604 (2011).
- Kupakuwana, G. V., Crill, J. E., McPike, M. P., Borer, P. N. Acyclic identification of aptamers for human alpha-thrombin using over-represented libraries and deep sequencing. Plos One. 6 (5), (2011).
- Smith, J. E., Griffin, D. K., Leny, J. K., Hagen, J. A., Chávez, J. L., Kelley-Loughnane, N. Colorimetric detection with aptamer-gold nanoparticle conjugates coupled to an android-based color analysis application for use in the field. Talanta. 121, 247-255 (2014).
- Chatterton Jr, R. T., Vogelsong, K. M., Lu, Y. -. C., Hudgens, G. A. Hormonal Responses to Psychological Stress in Men Preparing for Skydiving. J. Clin. Endocrinol. Metab. 82 (8), 2503-2509 (1997).
- Gold, L., et al. Aptamer-Based Multiplexed Proteomic Technology for Biomarker Discovery. Plos One. 5 (12), (2010).
- Musheev, M. U., Krylov, S. N. Selection of aptamers by systematic evolution of ligands by exponential enrichment: Addressing the polymerase chain reaction issue. Anal. Chim. Acta. 564 (1), 91-96 (2006).
- Wiebe, J. P. Progesterone metabolites in breast cancer. Endocr.-Relat. Cancer. 13 (3), 717-738 (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved