Method Article
のレア細胞集団からのTRAP-RC、翻訳リボソームアフィニティー精製
要約
Translating Ribosome Affinity Purification (TRAP) is able to capture cell-type-specific translation of mRNA. Here we report the first TRAP protocol dedicated to isolation of mRNA in rare cell populations of Drosophila embryos.
要約
Measuring levels of mRNAs in the process of translation in individual cells provides information on the proteins involved in cellular functions at a given point in time. The protocol dubbed Translating Ribosome Affinity Purification (TRAP) is able to capture this mRNA translation process in a cell-type-specific manner. Based on the affinity purification of polysomes carrying a tagged ribosomal subunit, TRAP can be applied to translatome analyses in individual cells, making it possible to compare cell types during the course of developmental processes or to track disease development progress and the impact of potential therapies at molecular level. Here we report an optimized version of the TRAP protocol, called TRAP-rc (rare cells), dedicated to identifying engaged-in-translation RNAs from rare cell populations. TRAP-rc was validated using the Gal4/UAS targeting system in a restricted population of muscle cells in Drosophila embryos. This novel protocol allows the recovery of cell-type-specific RNA in sufficient quantities for global gene expression analytics such as microarrays or RNA-seq. The robustness of the protocol and the large collections of Gal4 drivers make TRAP-rc a highly versatile approach with potential applications in cell-specific genome-wide studies.
概要
細胞は、開発中に特定のプロパティを取得する方法を理解することは、臓器の複雑さと病理学的状態に対するそれらの潜在的な進化を理解するために非常に重要です。 unrevealing全体的な遺伝子発現プロファイルによってセルの仕様と分化を根底遺伝子調節ネットワークを解読するための大きな研究プッシュがあり、ポルII結合状態、調節配列またはヒストンの翻訳後修飾の転写因子占有。
グローバルレベルマイクロアレイでの遺伝子発現を評価し、RNA-seqのアプローチが使用されています。マイクロアレイは、RNA-seqは、コードを含むとマイクロRNA、lncRNAsかのsnRNAのような非コードRNAのRNAの異なる種類の通知に良いギヤードであるのに対し、mRNA量を検出することを可能にします。しかしながら、これらの技術は、活発に転写、定常状態のmRNAを区別能動的mRNAの翻訳、そしてmRNAが分解プロセスに入ることができません。着実-STの測定mRNAレベルは、プロテオーム組成1-3の悪い推定であることが知られている食べました。逆に、積極的に、翻訳のmRNAを同定することはタンパク質生産のより正確な画像を提供します。
しかし、トランスクリプトーム研究は主に解釈が困難な遺伝子発現プロファイルを作成する、野生型条件4-7または解剖組織上の特定の因子の機能の損益を比較することにより、全生物レベルでつながってきました。細胞標的ツール、アフィニティ精製および感度の改善の最近の進歩は、現在ゲノムワイドな生体内の小さな細胞集団または単一の細胞に分析を行うことができます。
ショウジョウバエでは、トランスジェニック系統の大規模なコレクションは、細胞機能のために必要な分子機構のより正確な定量化をもたらすGAL4 / UASシステム8-10を経由して異なる組織および細胞亜集団の特定の時間的、空間的ターゲティングを可能にします。
分別によってプロファイリングをポリソームを積極-翻訳RNA 11をキャプチャする方法として説明しました。ショ糖勾配遠心分離に基づいて、この方法は、マイクロアレイまたは深いシーケンシングで分析すると、mRNAのポリソーム画分と決意の選択はゲノムワイドな翻訳ができます。ポリソーム分離は、翻訳プロセス12の間にリボソームによって保護されたRNA断片に対応するリボソームフットプリントを識別するために、ヌクレアーゼ消化と結合することができます。リボソームフットプリントは、RNAの翻訳およびRNA配列レベルの解像度およびリボソーム位置決めの定量化の精度を増加させる、翻訳の速度を測定することができます。しかし、今日まで、それは、主としてリボソームフットプリンティングプロセスの最後に得られたRNAの収量の強い減少に、translatomesの細胞特異的単離に適用されていません。 RNAのサイズ選択は、潜在的な偽Pを生成することができることを考えるとまた、同様にRNAを保護することができ、異なるのRNPからositivesは、さらなる開発がリボソームフットプリントを改善し、細胞特異的なアプローチに適合させるために必要とされます。
このような方法の一つは、翻訳リボソーム親和性精製(TRAP)は、ハイマンらによって 2008年に記載されていると呼ばれます。マウスのニューロンのサブセットからtranslatomeを単離するために適用されます。エピトープタグ付け細胞型特異的にリボソームタンパク質をすることによって、ポリソームを選択的細胞解離および選別の手間のかかる工程を経ずに精製することができます。この場合は、リボソームの表面の60Sリボソームタンパク質L10Aは、eGFPを13でタグ付けされました。 2008年以降、いくつかの研究は、Xに 14〜19匹のマウスから、異なる種でこのメソッドを使用しています24シロイヌナズナ20,21、ゼブラフィッシュ22,23とシロイヌナズナをアフリカツメガエル 。 TRAP方法は、 ショウジョウバエモデルに適合し、プリするために使用されています神経細胞25とアストロサイト26からFY細胞型特異的mRNA。多目的バイナリGAL4 / UASシステムは、組織特異的にGFPタグ付きRpL10Aを発現するために使用しました。大人のハエの頭を配列決定した(汎神経ELAV-Gal4ドライバーによってターゲット)神経細胞および単離したRNAからポリソーム選択を実行するために解剖しました。アップレギュレートされる遺伝子の多数は、 ショウジョウバエの胚の開発に(1未満の細胞の%)現在のアプローチは、優れた特異性を有することを示す、希少細胞集団に適合させるために、私たちを促し、神経系で発現することが知られているものに相当します。
分子俳優や形態形成の動きが進化的に保存されているようショウジョウバエモデルの中では、胚段階では、発達過程の研究のための選択のモデルです。このモデルでは、これまで行っのみ組織特異的トランスクリプトームアプローチはそう細胞または核を使用して行われてきましたrtingだけハエ胚の組織/細胞特異的translatomeプロファイリング専用の方法の必要性を作成し、定常状態のトランスクリプトーム27-31を研究することができました。ここでは、 ショウジョウバエの胚に捧げ最初のトラッププロトコルを報告しています。この方法では、胚当たり約100筋細胞の非常に制限された細胞集団における従事イン翻訳mRNAは、正常に高品質と特異性で単離しました。以前に25示すように、バイナリGAL4 / UASシステムは、明らかな表現型や発育遅延、任意の毒性を伴わずに筋肉の部分集団におけるGFPタグ付きRpL10Aの発現を駆動するために使用されていません。 ショウジョウバエの胚は筋肉組織に上昇を与えるhemisegmentあたり30筋肉を持っています幼虫の。前かがみのアイデンティティ遺伝子の近くで発見された調節領域を使用することにより、30多核筋のうち6が対象としています。このアッセイでは、リボソームが翻訳伸長を使用して、mRNAの上に固定化しますシクロヘキシミドを阻害。サイトゾル抽出物は、その後、GFP抗体でコートした磁気ビーズを用いてアフィニティー精製のために使用されます。精製したRNAの品質は、バイオアナライザーを使用して検証しました。定量的逆転写PCR(RT-qPCRには)mRNA単離の特異性および感度を決定するために使用され、我々の最適化プロトコルは非常に効率的であることが実証されました。
プロトコル
1.フライラインの生成
- 2異なる構築物を生成します。最初の構築物では、RpL10AのcDNA(リボソームタンパク質サブユニットL10A)はpUASP-PL-GFP-NTERプラスミド(32から修正版)にGFPの下流にクローニングされます。生殖細胞系列のプロモーターの使用は、トランスジェニックラインのより多くの多価利用することができます。
注:pPTGAL4プラスミドを使用して、GAL4(TF)の上流に前かがみ遺伝子の組織特異的発現を駆動する調節配列をクローニングすることにより、第2の構造体を取得します。 - (標準的な手順に従って)33 ワット1118ハエに別々のプラスミドを注入し、Pエレメントの挿入によってハエのゲノムに構築物を挿入します。
- 二つの異なる染色体上の構築物でフライラインを回復するために、形質転換体を選択します。最終TRAPラインは一つの二重トランスジェニック安定ラインを得るために以下の2行(遺伝的交雑)を組み合わせて作成されます。 F0:W-。 CYO、UAS EGFP :: RpL10A。 MKRS / TM6BX W-; CYO / SCU;前かがみGAL4 / TM6B。 F1:W-。 CYO、UAS EGFP :: RpL10A。前かがみGAL4 / TM6B。超この行を増幅し、所望のステージの胚を収集します。
2.胚コレクション
- (18万ハエ/ケージの周り)ケージあたり若いハエの約40グラムを含む8大型円筒人口ケージを準備します。
- そうハエが彼らの卵管から胚を開発クリアすることができ、その前産むと呼ばれるに進みます。そのため、固化した寒天とグレープジュースの混合物を含有する2 11センチメートル径のペトリ皿に1時間ハエを置き、新たに作られた酵母ペーストで表面の⅓をカバーしています。 3後グレープジュースプレートの交換は同様のたてプレート上の実胚を収集します。
注:インキュベーション時間は研究発達時間窓に応じて異なります。 - ケージからプレートを取り外して、正しい発達段階を得るために必要な時間同温度でそれらを残す(例えば、ADDITの産卵+ 10時間の3時間ionalのインキュベーションは、10〜13時間の産卵(AEL))後の段階から胚を提供します。ブラシを使用して50mlの水でプレート内容(胚、酵母ペースト、死んだハエ)を再懸濁します。
- 死んだハエの胚および残りの体の部分を分離し、小径ふるい上に残った胚を収集し、次いで脱イオン水で簡単に洗浄するための篩(700ミクロン、355ミクロン、112ミクロン)のシリーズを通過します。それはふるいを詰まらせることができますようにコレクションごとに18以上のプレートを使用しないでください。
- 2分間の脱イオン水中の4.5%の漂白剤でDechorionate胚および30〜60秒間、脱イオン水で十分に洗い流してください。攪拌しながら室温で5〜10分間、PBS、0.01%トゥイーン20、100μg/ mlのシクロヘキシミドで胚を培養します。
- セルロース吸収性シートの乾燥胚およびマイクロ遠心チューブに移します。チューブの重量を測定し、液体窒素に浸漬することによって胚をフラッシュ凍結。この段階では、乾燥した胚を、-80℃で数ヶ月間保存することができます。
- 徹底的に穏やかに撹拌して、元の瓶の中にプロテインG磁気ビーズを再懸濁します。
- 転送90 RNaseフリーのチューブにビーズμlのマグネット(30〜60秒)でそれらを収集。ビーズ90μlを60〜80 mg / mlのタンパク質を含む溶解物1mlの免疫沈降(IP)を行う必要があります。ビーズの飽和を防止するための比率を尊重。タンパク質の量に応じて、いくつかのチューブを使用してください。
- 1×PBS、0.01%トゥイーン20(500μL)でビーズを洗浄し、上清を廃棄し、磁石にビーズを収集します。ビーズに0.01%のTween 20のPBS 350μlのGFP抗体30μgのを追加し、室温で30分間混合端の上にゆっくりと端とインキュベートします。
- 、磁石(30〜60秒)にビーズを集め、上清を破棄し、ポリソーム抽出緩衝液500μlですすぐ(ヘペス10mMの、のKCl 150 mMの、のMgCl 2 5mMの、DTT、0.5mMの、トリトン1%、シクロヘキシミド100μg/ mlの広報otease 1×阻害剤、RNaseインヒビター100 U)。
- 磁石にビーズを収集し、バッファ(BSA 0.1μgの/μL、酵母tRNAは0.1μg/μL、ポリソーム抽出緩衝液中のグリコーゲンは0.1μg/μl)をブロックする500μlを添加します。
- 室温で30分間インキュベートし、新鮮なブロッキング緩衝液で1回、この手順を繰り返します。
- その後、ビーズを思い出すと免疫精製ステップ(セクション6)にすぐに進み、磁石にビーズを収集し、ポリソーム抽出緩衝液500μlですすぎます。
4.溶解物の準備
- 開始する前に、多方向高速ビーズグラインダーのプログラムを設定:2×10秒間5000rpmで、15秒のポーズで。ポリソーム抽出バッファーを含む氷の上に予備冷却15ミリリットルチューブに乾燥させた胚(1.5 g)を移し、すぐにホモジナイズします。ポリソーム抽出緩衝液4ml中の胚を1.5gを均一化。
- 2で均質化された胚を広げ2 mLの試験管を予冷し、エンブリーを挽きますホモジナイザープログラムを実行して、OS。氷の上に新鮮な予冷マイクロ遠心チューブにライセートを移し、2000グラムで10分間、4℃で遠心分離して核後上清を準備します。
- 氷の上に新鮮な予冷マイクロ遠心チューブに上清を移し、上澄み液(最終濃度= 0.1%)に10%のNonidet P-40の1/100サンプル容量を追加し、チューブを反転させて軽く混ぜます。
- 300mMの1,2-ジヘプタノイル-sn-グリセロ-3-ホスホコリン(DHPC)(最終濃度= 30mmの1/9サンプルボリュームを追加、チューブの底に液体を収集するためのパルス遠心minifugeのサンプル)、(インキュベーションの間に数回転倒混和)チューブを反転させて静かに混合し、氷上で5分間混合物をインキュベートします。
- 20,000gで10分間4℃で遠心分離することにより、ポストミトコンドリア上清を準備します。ブラッドフォード法によりタンパク質濃度を測定します:溶解液中の濃度は60〜80 mg / mlの間であるべきです。 T新鮮な新しい予冷マイクロチューブに上清AKEと前吸収工程(第5節)にすぐに進みます。
5.前の吸収
- 徹底的に穏やかに撹拌して磁気ビーズを再懸濁し、室温でマイクロ遠心チューブに(ビーズ/ 1ミリリットル胚溶解物の30μl)をビーズ30μlのを転送します。
- 、磁石のビーズを集め上清オフピペット、およびポリソーム抽出バッファー(500μlの)中でビーズを再懸濁します。 、磁石上にビーズを収集し、胚溶解液を追加し、回転装置上で4℃で1時間インキュベートします。
- ビーズを除去し、免疫精製工程のために氷上で上澄みを保ちます。免疫精製前に、例えば、RT-qPCRにより、免疫精製後に回収したRNAサンプルとグローバルなRNAサンプルを比較するために、上清(入力)の100μLを保ちます。
6.免疫精製
- 1ミリリットル(60〜80 mg / mlのタンパク質に対応する)のを加えますGFP抗体に結合されたブロックされたビーズの90μLを含むRNaseフリーのチューブに、溶解物を事前に吸収しました。チューブ回転で穏やか転倒混合しながら2時間、4℃でサンプルをインキュベートします。また、4℃でのインキュベーションのO / Nを実行します。
- インキュベーション後、磁石にビーズを収集します。その後、0.5 mMの、シクロヘキシミド100μg/ mlのRNアーゼ阻害剤100 U洗浄緩衝液500μl(ヘペス10mMの、KClの350ミリメートル、のMgCl 2 5mMの、ノニデットP-40 1%、DTTで再懸濁し、ビーズをスピンダウンするminifugeを使用して、 )と磁石の上にそれらを収集。このステップをさらに2回繰り返します。
- 新しい予冷RNaseフリーのチューブにビーズを変更し、二回洗います。磁石にビーズを収集し、ビーズに直接1ミリリットルのTRIzolを追加することにより、RNA抽出に進み、製造元の指示に従ってください。
7. RNAのクリーンアップと品質評価
- 製造業者の指示に従ってのRNeasyマイクロキットを使用してください。完了時に、ELUT(x)のμlのRNaseフリー水を用いて電子のRNA。 2 60℃分、-80℃で保存スナップ凍結サンプルのために暖かいです。バイオアナライザーを用いてRNAの品質をチェックします(製造元の指示に従ってください)。
- TRAPed RNAの特異性をテストするために、製造業者の説明書(上付き文字III)に記載の精製されたRNA(入力およびIP)の3 ngの上で逆転写反応を行います。遺伝子特異的プライマーセットを用いて、定量PCRのために得られたcDNAを使用してください。
結果
ショウジョウバエの胚の体細胞の筋システムがhemisegmentあたり30の筋肉で構成され、各筋肉は、プロパティの特定のセットを持っています。アイデンティティ遺伝子、位置、融合イベント、付着部位と神経支配の数のコード。特定の筋肉のサブセットに翻訳されたmRNAを同定することは、これらの特定の筋肉の特性を形成するために必要なタンパク質の情報を提供します。 TRAPベースの方法を使用して、同一遺伝子前かがみを発現する筋肉の亜集団からのRNAは、( 図1A-B)で精製しました。このアプローチの主要な関心事は、単離されたRNAの品質と特異性です。ここで報告された最適化されたプロトコルは、バイオアナライザー分析を用いて評価した高品質のRNAの体系的な回復を可能にしました。得られたプロファイルは、RNA( 図1C)の劣化を示さありません。
トラップ法を中心に開発された他の研究では、質問は、データの特異性を調達しました。ここで私はビーズまたはチューブにRNAの非特異的結合および洗浄緩衝液の最適化によって結合されたバックグラウンドを抑制するための方法をmprove。前かがみ発現細胞を単離するバックグラウンドレベルと効率を評価するために、我々は同じ胚段階の3反復のRT-qPCRの試験を主導しました。 4つの異なる遺伝子の-富化をフォールド(MEF2、前かがみ、プロスペロー、soxNeuro)はRpL32遺伝子( 図1D)に対する入力と正規化と比較して算出しました。これらの結果は、汎筋肉遺伝子MEF2と前かがみ遺伝子の5.6倍の濃縮の2.3倍の濃縮を示しました。対照的に、神経系で表される2つの遺伝子が枯渇入力と比較しました。非常に類似した倍数変化値は、プロトコルが堅牢であることを実証し、3生物学的反復に観察されました。 150×10 ^ 6の正の(合計100 GFP細胞/胚を含んで得られた1.5×10 ^ 6胚を中心に、1.5グラムで始まる前かがみ-Gal4ドライバーを持つと細胞)。
TRAP実験を実行した後、収量は、ステージ・オブ・関心に応じて、特定のRNAの約25〜45 ngのです。この材料は、マイクロアレイ解析またはRNA-seqのライブラリーを構築するために増幅プロトコルを用いてRNA-seqのを実行するのに十分です。効率がRpL10A-EGFPを発現するために使用されるドライバの強さに強く依存します。
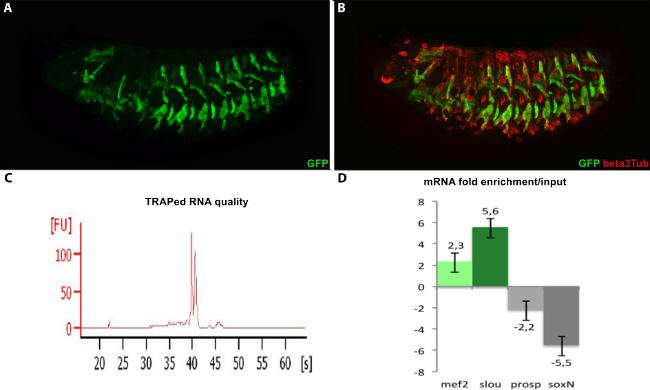
図1.品質と前かがみ-GAL4> UASRpL10A-EGFP胚からのTRAP-単離されたRNAの特異性評価。
(AB)hemisegment(A)ごとに6つの前かがみの筋肉細胞に特異的にRpL10A-EGFP発現を示すステージ16胚の共焦点画像。一般的な筋肉のマーカーBeta3tubulinとRpL10A-EGFPの共局在は、マージ画像(B)に観察されます。 (C)TRAPed RNA raの品質管理nはバイオアナライザー上のrRNA 18Sおよび28Sの完璧な整合性を示します。 (D)ステージ16胚の3生物学的複製のTRAP-単離されたmRNA実験の高い特異性を示す、RT-qPCR分析。倍の変化は、入力とRpL32遺伝子に対して正規に比べて計算されます。すべての筋肉系統に存在するMEF2転写物は、より制限された前かがみの転写物が5.6倍に富むあるのに対し、入力に比べて2.3倍に富むです。 プロスペローとsoxN遺伝子を発現する神経細胞が2.2倍に枯渇し、それぞれ5.5倍されています。エラーバーは標準偏差を表す(n = 3)であった。 この図の拡大版をご覧になるにはこちらをクリックしてください。
ディスカッション
本稿では、ショウジョウバエの胚では珍しい細胞集団の研究に専念修正翻訳リボソームのアフィニティー精製プロトコル(吹き替え「TRAP-RC ')を記述します。情報は、胚の溶解およびポリソーム抽出のためのマイクロアレイまたはRNA-seqの分析、 すなわち 、1)必要な生物学的物質の量と最適化された手順に適した収率で特定のRNAの正常な分離のための重要なステップに設けられています。最適な免疫沈降のための2)ビーズ/抗体への溶解物の比率; 3)高い特異性および感度を有するTRAP-RC実験を実行するように、洗浄緩衝液組成を含むバックグラウンドの低減を可能にするステップ。
このプロトコルは、容易に任意の他の細胞型に適用作る再現可能なデータが得られます。この技術は、積極的に、mRNAの翻訳レベルで遺伝子発現差を分析し、従ってスペックでタンパク質の発現を理解する道を開くことを可能にします特定の時間窓でのIFICセルタイプ。タンパク質存在量が翻訳及び分解プロセスの速度に依存することに注意してください。 TRAP法の主な制限は、正確な定量的にタンパク質含有量を測定するための、または翻訳後修飾を検出することができないことです。 mRNAの本体に沿ってリボソーム密度を測定し、そうすることで、細胞型特異的に非常に強力なツールをフットプリントリボソームと結合したTRAPを作ることができることにより、定量化を改善します。最近の論文34において、この組み合わせは、ヒト胚性腎臓293細胞で行いました。著者らは、ストレプトアビジンビーズを使用してRpL10Aの誘導ビオチン化形態の親和性精製したヌクレアーゼフットプリントを実行しました。これは、制限を目的とするために必要な生物学的物質の量であり、特定の細胞型を標的にしながら、生物全体にリボソームフットプリントを行うことが可能であるという原理の証明です。
">グローバルトランスクリプトーム解析と並行してTRAP実験を行うことにより、新たに転写の間の割合を追跡することを可能にし、RNAを翻訳actively-。これは、特定の発達の状況や特定の細胞集団で行われる潜在的な転写後のメカニズム深く有益になります例えば-byマイクロRNA。結論として、TRAPは、細胞特異的に能動的に翻訳するリボソームに結合したRNAを識別するための非常に効率的で特異的かつ高感度な方法です。この方法は、生物および組織型の多種多様に使用することができます。ここで説明する新しいTRAP-RCプロトコルは希少細胞集団(総細胞数の1%未満)および全ゲノムレベルでのその後の分析のために十分な最終材料の量のために最適化しました。
この方法の可能な制限は、コンプするのに十分な量のタグ付きリボソームを製造するための強力なドライバの必要条件であります標的細胞型における内因性タグなしのものとETE。最近の研究25では 、著者は10〜30%にタグなしリボソームに対するタグ付けの割合を推定しました。
UAS-RpL10A-EGFPのコピー数を増加させるこの問題を克服するためには、かなりこのバランスを改善するはずです。あるいは、記載の遺伝的バックグラウンドにUAS-GAL4導入遺伝子を追加するGAL4タンパク質の産生を増幅し、間接的にRpL10A-EGFPの発現を増加させます。これは、mRNA上のタグ付けされたリボソームのよりよい占有を優先すべきです。
このすでに効率的なアプローチは、より強力な、より定量的な側面を持ってたり発見し、遺伝子発現の制御のために必須である分子機構を解明するために補完的な方法を適合させることにより行うことができることに注意してください。
開示事項
著者らは、開示することは何もありません。
謝辞
The authors thank Nicolas Allegre, Maud Peyny and Jean-Philippe Da Ponte for their excellent technical assistance. This work was supported by the Agence Nationale de la Recherche (ANR-JC-CARDIAC-SPE), the INSERM starting grant, ANR ID-CELL-SPE grant, Equipe FRM and the AFM 15845 grant.
資料
| Name | Company | Catalog Number | Comments |
| HEPES | Sigma | H4034-1006 | |
| 1,2-diheptanoyl-sn-glycero-3-phosphocholine | Avanti Polar Lipids | 850306P | |
| Glycogen | thermo scientific | R0561 | |
| Purified BSA 100X | Biolabs | B9001 | |
| Yeast tRNA | Sigma | R5636 | |
| Super RNase In | Ambion | AM2694 | |
| Antibody GFP clone N86/38 | UC DAVIS/NIH Neuromab Faculty Antibodies Incorporated | 75-132 | |
| Nonidet P40 | Roche | 11754595001 | |
| Precellys 24 Lysis homogenisation | Bertin Technology | ||
| Nuclease free water | Nalgene | ||
| Dynabeads ProteinG | Invitrogen | 10004D | |
| Potassium chloride (KCl) | Applied Biosystem | AM96406 | |
| Magnesium Chloride (MgCl2) | Applied Biosystem | AM95306 | |
| Cellulose waddin unbleached | VWR | 115-2597 | |
| Sieves 112, 355, 710 um | Retsch | ||
| TRiZol | Invitrogen | 15596-018 | |
| MagRack 6 | GE HealthCare | 28948964 | |
| Low binding filter tips | ClearLine | ||
| Rnase free tube 1.5 ml | ClearLine | 390689DD | |
| Tween 20 | Fischer Bioreagent | BP337-500 | |
| Cycloheximide | Sigma | C1985 | |
| Protease inhibitor tablets, Mini-Complete, EDTA-free | Roche | 11836170001 |
参考文献
- Sousa Abreu, R., Penalva, L. O., Marcotte, E. M., Vogel, C. Global signatures of protein and mRNA expression levels. Molecular bioSystems. 5, 1512-1526 (2009).
- Maier, T., Guell, M., Serrano, L. Correlation of mRNA and protein in complex biological samples. FEBS letters. 583, 3966-3973 (2009).
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473, 337-342 (1038).
- Junion, G., et al. Genome-wide view of cell fate specification: ladybird acts at multiple levels during diversification of muscle and heart precursors. Genes. 21, 3163-3180 (2007).
- Jakobsen, J. S., et al. Temporal ChIP-on-chip reveals Biniou as a universal regulator of the visceral muscle transcriptional network. Genes. 21, 2448-2460 (2007).
- Cunha, P. M., et al. Combinatorial binding leads to diverse regulatory responses: Lmd is a tissue-specific modulator of Mef2 activity. PLoS genetics. 6, e1001014 (2010).
- Sandmann, T., et al. A core transcriptional network for early mesoderm development in Drosophila melanogaster. Genes, & development. 21, 436-449 (2007).
- Kvon, E. Z., et al. Genome-scale functional characterization of Drosophila developmental enhancers in vivo. Nature. 512, 91-95 (2014).
- Pfeiffer, B. D., et al. Tools for neuroanatomy and neurogenetics in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 105, 9715-9720 (2008).
- Gallo, S. M., et al. REDfly v3.0: toward a comprehensive database of transcriptional regulatory elements in Drosophila. Nucleic acids research. 39, D118-D123 (2011).
- Gandin, V., et al. Polysome fractionation and analysis of mammalian translatomes on a genome-wide scale. Journal of visualized experiments : JoVE. , (2014).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7, 1534-1550 (2012).
- Heiman, M., et al. A translational profiling approach for the molecular characterization of CNS cell types. Cell. 135, 738-748 (2008).
- Doyle, J. P., et al. Application of a translational profiling approach for the comparative analysis of CNS cell types. Cell. 135, 749-762 (2008).
- Dougherty, J. D., Schmidt, E. F., Nakajima, M., Heintz, N. Analytical approaches to RNA profiling data for the identification of genes enriched in specific cells. Nucleic acids research. 38, 4218-4230 (1093).
- Fomchenko, E. I., et al. Recruited cells can become transformed and overtake PDGF-induced murine gliomas in vivo during tumor progression. PloS one. 6, e20605 (2011).
- Helmy, K., et al. Identification of global alteration of translational regulation glioma in vivo. PloS one. 7, e46965 (2012).
- Zhou, P., et al. Interrogating translational efficiency and lineage-specific transcriptomes using ribosome affinity purification. Proceedings of the National Academy of Sciences of the United States of America. 110, 15395-15400 (2013).
- Drane, L., Ainsley, J. A., Mayford, M. R., Reijmers, L. G. A transgenic mouse line for collecting ribosome-bound mRNA using the tetracycline transactivator system. Frontiers in molecular neuroscience. 7, 82 (2014).
- Yoon, B. C., et al. Local translation of extranuclear lamin B promotes axon maintenance. Cell. 148, 752-764 (2012).
- Watson, F. L., et al. Cell type-specific translational profiling in the Xenopus laevis retina. Developmental dynamics : an official publication of the American Association of Anatomists. 241, 1960-1972 (2012).
- Fang, Y., et al. Translational profiling of cardiomyocytes identifies an early Jak1/Stat3 injury response required for zebrafish heart regeneration. Proceedings of the National Academy of Sciences of the United States of America. 110, 13416-13421 (2013).
- Tryon, R. C., Pisat, N., Johnson, S. L., Dougherty, J. D. Development of translating ribosome affinity purification for zebrafish. Genesis. 51, 187-192 (2013).
- Jiao, Y., Meyerowitz, E. M. Cell-type specific analysis of translating RNAs in developing flowers reveals new levels of control. Molecular systems biology. 6, 419 (2010).
- Thomas, A., et al. A versatile method for cell-specific profiling of translated mRNAs in Drosophila. PloS one. 7, e40276 (2012).
- Huang, Y., Ng, F. S., Jackson, F. R. Comparison of Larval and Adult Drosophila Astrocytes Reveals Stage-Specific Gene Expression Profiles. G3. , (2015).
- Steiner, F. A., Talbert, P. B., Kasinathan, S., Deal, R. B., Henikoff, S. Cell-type-specific nuclei purification from whole animals for genome-wide expression and chromatin profiling. Genome Res. 22, 766-777 (2012).
- Henry, G. L., Davis, F. P., Picard, S., Eddy, S. R. Cell type-specific genomics of Drosophila neurons. Nucleic acids research. 40, 9691-9704 (2012).
- Dobi, K. C., Halfon, M. S., Baylies, M. K. Whole-genome analysis of muscle founder cells implicates the chromatin regulator Sin3A in muscle identity. Cell reports. 8, 858-870 (2014).
- Salmand, P. A., Iche-Torres, M., Perrin, L. Tissue-specific cell sorting from Drosophila embryos: application to gene expression analysis. Fly. 5, 261-265 (2011).
- Busser, B. W., et al. Molecular mechanism underlying the regulatory specificity of a Drosophila homeodomain protein that specifies myoblast identity. Development. 139, 1164-1174 (2012).
- Rorth, P. Gal4 in the Drosophila female germline. Mechanisms of development. 78, 113-118 (1998).
- Rubin, G. M., Spradling, A. C. Genetic transformation of Drosophila with transposable element vectors. Science. 218, 348-353 (1982).
- Ingolia, N. T., et al. Ribosome profiling reveals pervasive translation outside of annotated protein-coding genes. Cell reports. 8, 1365-1379 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved