Method Article
クロマニョンを用いた超解像生物学的イメージング時代における3次元色素の高精度補正
要約
3次元(3D)多色蛍光顕微鏡画像における色分けの補正は、定量的データ分析において重要です。このプロトコルは、適切な参照画像の取得とオープンソースソフトウェアChromagnonでの処理を通じて、生物学的サンプルの色調変化を測定し、修正するために開発されています。
要約
定量的多色蛍光顕微鏡は、異なる波長で獲得したカラーチャンネルの慎重な空間マッチングに依存しています。色収差とカメラの不完全な位置合わせにより、各チャンネルで取得した画像がシフトされ、拡大され、3次元のいずれかで互いに回転する可能性があります。従来のキャリブレーション法では、色のずれはカバースリップの表面に取り付けられた多色ビーズによって測定され、そのようなキャリブレーションサンプルからの色の変化を測定するためのソフトウェアが多数用意されています。しかし、色収差は深度によって変化し、観察条件によって変化し、生物学的サンプル自体によって誘導され、したがって、目的のサンプルおよび体積にわたって、真の量の色分けの決定を妨げる。色調シフトを高精度に補正することは、微少な色のシフトだけが定量的な分析に影響を与え、多色画像の解釈を変える超解像顕微鏡に特に関連しています。我々は、オープンソースソフトウェアクロマニョンと生物学的サンプルの3D色の変化を測定し、修正するためのそれに付随する方法を開発しました。ここでは、目的の生物学的サンプルの色素シフトを測定するためのサンプル調製、データ取得、およびソフトウェア処理のための特別な要件を含む詳細なアプリケーションプロトコルを提供します。
概要
多色イメージングは、生物学的蛍光顕微鏡の基本的な側面の1つであり、異なる分子または構造の空間的関係が大きな関心事である場合です。色収差は、分散によって引き起こされる多色光の光学収差であり、対象とする着色物の見かけの位置を変化させる。同様に、各色の獲得に専念する複数のカメラを搭載した顕微鏡は、光学要素の違いとチャンネル間の不完全なアライメントにより、より複雑な色調シフトを有する。したがって、このような色の変化は、ユーザーによって明示的に修正されない限り、誤った結論につながる可能性があります。顕微鏡の解像度が古典的な解像度限界によって制限されている限り、色彩学的変化は大きな問題とはならなかったが、近年の超解像顕微鏡1の開発は、色素シフトのより正確な補正の必要性を促している。
多色ビーズキャリブレーションスライド2を用いて顕微鏡システムの色調シフトを測定することは一般的な習慣でした。ビーズベースのキャリブレーション方法は、顕微鏡の光学系全体からカバースリップ2の表面に向かって色分けシフトを測定するのに適しています。しかし、この方法は、対象となる生物学的サンプルの色の変化を測定することができない。多くの生物学的サンプルは3次元(3D)であり、そのようなサンプルの色調シフトはカバースリップの表面のものとは異なることを注意することが重要です。さらに、光の変化は、画像化条件22,33と共に変化する。我々は、3D生物学的サンプルにおける色調変化を測定し、色度シフトの不確実性が古典的な多色ビーズ較正方法3によって350nmと多いことがわかった。したがって、色相シフトは、対象深度および使用されているイメージング条件下で生物学的サンプルで測定する必要があります。
ここでは、生物学的サンプルの色の変化を測定し、当社のソフトウェア、クロマニョン3を使用してこれらのシフトを修正する手順を説明します。生物学的サンプルの色素シフトを測定するために、我々の方法は「ターゲット」画像と「参照」画像の2種類のデータセットを使用します。「標的」画像は、目的とする多色画像であり、例えば、DNA、核エンベロープ、および微小管について染色された画像である。このような画像の色の変化を測定することはしばしば不可能です。したがって、サンプルの色の変化を測定するために専用の「参照」画像が必要です。「参照」画像の唯一の定義は、同じオブジェクトの多色画像です。この意味で、多色ビーズ画像は参照画像の一種でもある。ここでは、生体サンプルにおける色の変化を測定するために使用される3種類の参照画像について、「クロストーク参照画像」、「明視野参照画像」、「生物学的較正参照画像」について説明します。参照画像の種類は、使用されている顕微鏡の種類や、表1に要約した補正精度によって異なります。
| クロストーク | 明視野 | 生物学的キャリブレーション(別のスライド上) | 生物学的キャリブレーション(同じスライド上) | |
| 精度a | +++ | + | ++b | +++ |
| シンプル さ | ++ | +++ | ++ | ++ |
| 適用可能な顕微鏡 | 広視野 | 広視野 | すべての | すべての |
| ローカルアライメントの可用性 | + | + | -c | -c |
|
a: 「+」の数は、評価の増加を示します。シングルプラスは約50nm、3プラスは3Dで約15nmである。 b: 精度は、可変イメージング条件をどの程度一定に保つのかによって異なります。 c:多色ビーズサンプルで測定されたローカルキャリブレーションは、プロトコルセクション4で説明されているように組み合わせることができる。 | ||||
表1:参照画像のタイプを選択する際のパラメータ
「クロストーク参照画像」は、補正精度が最も高く3、3,4(4表1)を達成するのは比較的簡単です。欠点は、励起経路の色の変化を測定する能力がないため、顕微鏡アプリケーションの制限です。また、このような画像を得るためには、顕微鏡には、マルチバンドの二色性ミラー、および励起フィルタまたは光源から独立して制御される発光フィルタを装備する必要があります。適切な顕微鏡検査には、従来の広視野顕微鏡、光活性化局在化顕微鏡/確率的光学再構成顕微鏡(PALM/STORM)5、6、広視野顕微鏡検査で観察された拡張顕微鏡7などの一分子局在顕微鏡5,6(SMLM)が含まれる。クロストーク参照画像は、ターゲットサンプル自体から取得されます。必要なすべてのチャネルで得られた色素のクロストーク(ブリードスルー)蛍光の画像です。蛍光発光は常に長波長に向かって膨張するため、最短の発光波長を有する色素は、より長い波長のチャネルでクロストーク蛍光を得るために励起される。例えば、試料が青、緑、赤で染色されると、青染料だけが励起され、発光灯は青、緑、赤の各チャンネルで得られます。このプロトコルでは、4',6-ジミジノ-2-フェニリンドール(DAPI)で染色されたDNAを使用して、蛍光クロストークを得た。
「明視野参照画像」は、「クロストークリファレンス画像」に代わる、より簡単で光毒性の低い画像ですが、最も正確でない3(表1)です。これらはターゲットサンプルの明るいフィールド画像であり、ターゲット画像で使用されるすべてのカラーチャンネルで取得される。
「生物学的較正基準画像」は、励起および放出経路3の両方で色分けシフトを測定する能力のために顕微鏡の任意のタイプに適用可能であるという利点を有する3、8(8表1)。適切な顕微鏡検査には、広視野顕微鏡、共焦点顕微鏡、光シート顕微鏡、刺激放出破壊(STED)9、構造化照明顕微鏡(SIM)10、Airyscan/SORA1011、12、SMLMが11,12全内部反射蛍光(TIRF)モード、オリンパス超分解能(OSR)13、および第2回を含む。13生物学的較正基準画像は、ターゲットサンプルと同様に調製されたキャリブレーションサンプルから取得されますが、複数の色を持つ単一の構造を染色します。補正精度は、ほとんどの超解像度顕微鏡の分解能に優れ、生物学的較正サンプルの調製は比較的簡単です。もう 1 つの利点は、複数の参照イメージを「平均」する可用性です。したがって、個々の画像にクロマティックシフトの測定に関する情報が乏しくても、複数の画像を平均化することで情報内容を増やすことができます。精度は、イメージング条件を一定に保つ量によって異なります。この点で、ターゲットサンプルとリファレンスサンプルの両方が同じスライド上にある場合、例えば8ウェルチャンバーカバーグラス(表1、右)を使用すると、最良の性能が得られる。このプロトコルでは、3色のファロイドンで染色されたアクチンを生体較正として用いた。
参照画像が得られたら、クロマチックシフトを測定し、当社のソフトウェアクロマニョンによって補正されます。クロマニョンがクロマチックシフトを測定して修正できるチャンネル、Zセクション、タイムフレームの数に制限はありません。クロマニョンは2段階でクロマチックシフトを測定します。最初のステップでは、X、Y、Z 軸、X、Y、Z 軸に沿った拡大、Z 軸の周りの回転における平行移動の「グローバル」または「アフィン」の位置合わせパラメータを取得します。グローバルアライメントの計算精度は、3Dで~16 nm、2Dで約8 nmです。2 番目のステップは、高い精度を得るために投影された画像に対するオプションの 2D 反復「ローカルアライメント」です。局所アライメントプロセスでは、画像は複数の領域に細分化され、これらの局所領域の色調シフトが測定されます。その後、領域がさらに分割され、サブ領域の色調シフトは、領域内のピクセル数が最小ピクセル数(通常は 60 x 60 ピクセル)に達するまで反復的に測定されます。結果として得られるローカル位置合わせマップは、グローバル位置合わせパラメータと結合され、弾性変換によってターゲット イメージに適用されます。このステップに続いて、計算精度は3Dで~14nm、2Dでは~6nmに改善される。局所的な位置合わせは、参照中の生物学的構造が標的の構造と異なるため、生体較正基準画像には適さない(表1)。したがって、生物学的較正参照画像にはグローバルアライメントのみが使用されます。
局所的な色の変化は2つのソースから始まる。顕微鏡器械のローカルな歪みおよび生物学的構造の不均一性。顕微鏡器用局所歪みは一定であるため、多色ビーズの基準試料から測定し、固定パラメータとして補正することができます。クロマニョンは、顕微鏡器による局所歪みマップと、生物学的較正からグローバルアライメントパラメータを組み合わせることができる(表1)。この方法を用いることで、生物学的較正の平均精度が1~2nm向上することが期待されます。
ここでは、最も簡単なローエンドから最高精度まで、当社のソフトウェアクロマニョンを用いて3D蛍光画像の色調シフトを補正するプロトコルについて説明します。HeLa細胞の免疫染色を例に挙げ、3次元広視野顕微鏡と3D-SIMを用いて観察します。第1のセクションでは、標的サンプルと生物学的較正サンプルの調製方法について説明する。プロトコルのこの部分は、研究の特定のターゲットに合わせて最適化する必要があります。第2部では、顕微鏡による3種類の参照画像の取得方法について述べています。想定は青、緑、赤のチャネルを取得することであったが、チャネルの組成は、研究の特定のターゲットと顕微鏡のセットアップによって変更する必要があります。顕微鏡が単一のカメラまたは複数のカメラを搭載しているかどうかは関係ありません。第3節では、当社のソフトウェアを使用して、参照画像を使用してターゲット画像の色調シフトを測定し、修正する方法について説明します。最後に、第4のセクションでは、顕微鏡の器械ローカル較正を用いて生体較正基準画像を補う方法について述べている。
プロトコル
1. サンプル準備
- ターゲットサンプルの調製
- 種子2.5 x 105 HeLa細胞を35mmガラス底皿に入れ、2mLの成長培地(L-グルタミンとピルビン酸ナトリウムを37°Cで10%の牛血清[FBS])で添加した酸化ワシ培地で37°C、CO2濃度5%で2増殖させます。あるいは、8ウェルチャンバーカバーグラス上の種子6 x 104 HeLa細胞を、37°Cで0.5mLの成長培地で、CO2濃度5%で増殖させます。高解像度の顕微鏡検査には、#1.5カバーグラス(厚さ0.170mm)を使用してください。
注:8ウェルチャンバーカバーグラスには、コンテナの種類に関係なく、ボリュームが100μlのステップ1.1.5、1.1.6、1.1.8、1.1.11、1.2.5を除くステップで1/4ボリュームを使用してください。 - 約24時間のインキュベーションの後、リン酸緩衝生理食塩水(PBS)中の3.7%ホルムアルデヒドの2mLで溶液を交換してください。穏やかな混合の後、回転プラットフォーム上の室温(RT)で15分間細胞を固定し続けます。
注意:ヒュームフードで作業してください。 - 2 mLのPBSで細胞を洗浄し、回転プラットフォームで5〜10分間それぞれ3倍にします。
注:ホルムアルデヒド廃棄物を処分するには、現地の規制に従ってください。 - 2 mL の 0.1% トリトン X-100 を持つ細胞を回転プラットフォームで 5 分間 PBS で 5 分間透過し、続いて 2 mL の PBS を使用して 3 回のスキを行い、それぞれ回転プラットフォームで 5~10 分間回転します。
- 細胞を1%のウシ血清アルブミン(BSA)の100 μLで1%回転プラットフォーム上のRTで1時間培養する。
- 溶液を100μLの一次抗体(抗エメリンポリクローナル抗体14および抗尿細管モノクローナル抗体15)をPBS中の1%BSAで適切な希釈(抗エメリンの場合は1/500、抗チューブリンの場合は1/100)で一次抗体の混合物に置き換え、4°Cで一晩インキュベートする。
- 2 mLのPBSで細胞を洗浄し、回転プラットフォームで10分間、それぞれ3〜5倍の洗浄を行います。
- 溶液を1/500希釈でPBSの2次抗体(アレクサフルオール488と抗マウスIgGとアレクサフルーオール555と抗マウスIgG)の混合物の100 μLに置き換え、RTで3-4時間インキュベートします。
- 2 mLのPBSで細胞を洗浄し、回転プラットフォームで5分間、それぞれ3~4倍の洗浄を行います。
- 溶液をPBSの0.5 μg/mL DAPIの2 mLに交換し、回転プラットフォーム上のRTで30分間DNAを染色します。
注: DAPI の代わりに、同じ濃度の Hoechst 33342 も使用できます。 - 溶液を取り付け媒体(材料表)の約100 μLに交換してください。
- 種子2.5 x 105 HeLa細胞を35mmガラス底皿に入れ、2mLの成長培地(L-グルタミンとピルビン酸ナトリウムを37°Cで10%の牛血清[FBS])で添加した酸化ワシ培地で37°C、CO2濃度5%で2増殖させます。あるいは、8ウェルチャンバーカバーグラス上の種子6 x 104 HeLa細胞を、37°Cで0.5mLの成長培地で、CO2濃度5%で増殖させます。高解像度の顕微鏡検査には、#1.5カバーグラス(厚さ0.170mm)を使用してください。
- 生物学的較正サンプルの調製
- ステップ 1.1.1-1.1.4 の説明に従って、固定セルを準備します。
注:好ましくは、サンプルはチャンバーカバーグラス上に用意され、ターゲットサンプルとリファレンスサンプルの両方を同じカバースリップ上の別々のチャンバーに配置します(表1、右端)。この調製は、実験が最高の補正精度を必要とする場合に好ましい。サンプルを通常のカバースリップで準備する必要がある場合は、厚さの変動が低い精密カバーリップ(「No. 1.5H」)を使用して再現性を保証します。 - 1.5 mLのメタノールで蛍光色素共役ファロイジンのストック溶液を調製し、-20°Cで保管します。以下の希釈でPBSにファロイジンストック溶液を混合する:アレクサフルオール405とファロイジンの場合は1/100、アレクサフルオール488でファロイジンの場合は1/1,000、アレクサフルオール594でファロイジンの場合は1/200。
- ステップ1.2.2で調製したファロイジン混合物の溶液を1 mLに交換し、回転プラットフォーム上のRTで30分間インキュベートします。
- 2 mLのPBSで細胞を洗浄し、回転プラットフォームで10分間、それぞれ3〜5倍の洗浄を行います。
- 溶液を約100μLの取り付け媒体に交換してください。
注:キャリブレーションサンプルは、4°Cまたは-20°Cで繰り返し使用するために保存することができます。
- ステップ 1.1.1-1.1.4 の説明に従って、固定セルを準備します。
2. 参考画像の取得
- クロストークリファレンス画像
- ステップ1.1で調製したターゲットサンプルを広視野顕微鏡に配置します。
- 青、緑、赤のチャネルでターゲットの蛍光画像を取得します。
注: 3D 画像の場合、イメージファイルにステップサイズの情報が含まれている場合、参照画像の Z ステップサイズはターゲットイメージの Z ステップサイズと異なる場合があります。基準画像およびターゲット画像のZステップサイズは、Z軸の光学解像度の半分以下であることが好ましく、これは回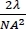 折限定顕微鏡で計算され、λはナノメートルの波長、NAは対物レンズの数値開口である。たとえば、軸方向の解像度が 550 nm の場合は、Z ステップを 275 nm より小さくします。参照イメージに時系列は必要ありません。
折限定顕微鏡で計算され、λはナノメートルの波長、NAは対物レンズの数値開口である。たとえば、軸方向の解像度が 550 nm の場合は、Z ステップを 275 nm より小さくします。参照イメージに時系列は必要ありません。 - 続いて、参照の蛍光画像を取得するために、DAPIのみの励起光を選択し、青、緑、および赤色のチャネルで取得することを選択します。3D スタックの場合は、同じステージ位置と Z 高さで参照画像を正確に取得します。
注: 長い発光波長の場合は、照明強度と露光時間の増加が必要になります。
- 明視野参照画像
- ステップ1.1で調製したターゲットサンプルを広視野顕微鏡に配置します。
- 青、緑、赤のチャネルでターゲットの蛍光画像を取得します。
注: Z ステップ サイズは、手順 2.1.2 で説明されているように、ナイキスト基準を満たすことが望ましいです。 - 2.2.2 と同じステージ位置で、3D スタックの場合は同じ Z 高さで、青、緑、および赤のチャネルでターゲットの明るいフィールド イメージを取得します。
- 生物学的較正参照画像
- ステップ1.1で作成したターゲットサンプルを3D-SIM顕微鏡に配置します。
- 3D-SIMで、青、緑、赤のチャネルでターゲットの蛍光画像を取得します。超解決イメージを再構築します。
メモ:顕微鏡の種類は、3D-SIM、共焦点、STEDなどの任意のタイプにすることができます。Zステップサイズは、ステップ2.1.2で説明したように、ナイキスト基準を満たすことが好ましい。 - 3D-SIMにより、目標画像と同様の異なる段階位置(ステップ2.3.2)でステップ1.2で調製した参照の複数の蛍光画像を取得する。超解決イメージを再構築します。
注: XY の合計ピクセル数は、すべての参照イメージで同じである必要があります。Z のステップ サイズは、すべての参照イメージで同じである必要があります。Z セクションの総数は、ターゲット イメージと同じであることが望まれますが、これは絶対要件ではありません。これらの基準画像のステージ上のXY位置は、位置またはカバースリップがターゲットサンプルの位置と既に異なっており、さらに単一のカバースリップ上の色度シフトの差が15nm3未満であるため、重要3ではない。対物レンズ、観察温度、浸漬油、共焦点顕微鏡におけるピンホールサイズ、高傾斜照明顕微鏡16における傾斜角を含む画像化条件は、すべて最良の性能を得るための基準と一致するべきである。顕微鏡が複数のカメラを装備して複数のチャンネルを同時に取得する場合、参照画像は週に限り取得して、インストゥルメンタルドリフトを補正する必要があります。
クロマニョンソフトウェアを用いたクロマチックシフトの補正
- Web ブラウザを使用して、 https://github.com/macronucleus/Chromagnon/releasesに進み、クロマニョンの最新バイナリ リリースをダウンロードします。
注: バイナリリリースは、Windows、Mac、および一部の Linux バージョンで利用可能です。 - プログラムを抽出し、実行可能ファイルを便利な場所に置きます。Windows または Mac でファイルをダブルクリックして開くか、Linux システムのコマンドラインからバイナリファイルを実行します。
注:図 1のようなグラフィカル ユーザー インターフェイスが開きます。セキュリティ上の理由から、プログラムをダブルクリックして初めて開けないようにすることができます。この場合、プラットフォームに依存する方法を使用して、インターネットからダウンロードしたプログラムを開きます。
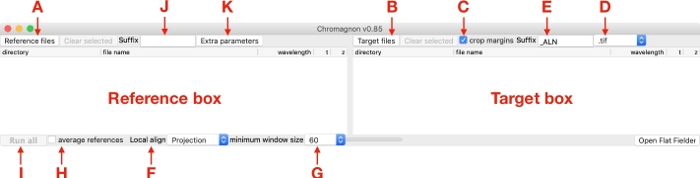
図1:クロマニョングラフィカルユーザーインターフェースのスクリーンショットこの図の大きなバージョンを表示するには、ここをクリックしてください。
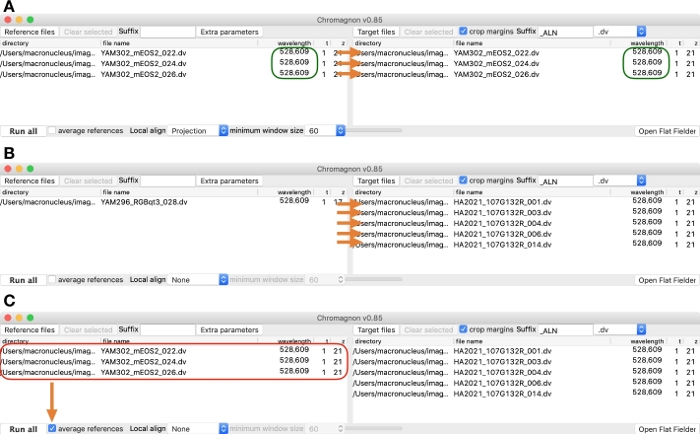
図 2: 複数のファイルを読み込むスクリーンショットの例(A) すべての参照画像に対応するターゲット画像が含まれる場合。チャネル名は、この例で使用するイメージ ファイルの波長 (緑色のボックスで示されます) によって正しく識別されます。(B) 複数のターゲット画像を修正するために単一の参照画像を使用する場合。(C)複数の参照画像(赤いボックスで示される)が平均化され、平均化後の参照画像を使用して複数のターゲット画像を補正する場合。この図の大きなバージョンを表示するには、ここをクリックしてください。
- [ターゲット]ボックス(図1)内のターゲットファイルをドラッグアンドドロップするか(図1B)をクリックしてファイルセレクタダイアログを開きます。
注: 複数のターゲット イメージがあり、それぞれのイメージに対応する参照イメージがある場合、対応する参照イメージとターゲット イメージは、それぞれの参照ボックスとターゲット ボックスの同じ行に配置する必要があります (図 2A)。単一の参照イメージを使用して、複数のターゲットイメージを整列させることもできます (図 2B)。 - オフの場合は、トリミングマージンチェックボックスをオンにします (図1C)。
注: このチェックボックスをオンにすると、配置の結果として生じる余白が切り取られます。このオプションをオンにして一般的に使用しますが、配置の前後に画像間でピクセルレベルの比較が必要な場合はオフにします。 - 選択リストから出力イメージ形式を選択します (図 1D)。
注: 利用可能な形式は 'tif' (ImageJ17形式)、'dv'、および 'ome.tif' です。'ome.tif' 形式は JDK をインストールした後にのみ使用できます。 - 出力ファイル名のサフィックスを指定します (図 1E、デフォルト値は '_ALN' です)。
- 信号対雑音比の高いクロストークリファレンス画像の場合は、ローカル位置合わせ(図1F)の選択リストからローカルアライメントを使用し、ローカルアライメントを使用するには[投影]を、無効にするには[なし]を選択します。最小ウィンドウ サイズ 60 (図 1G) を使用します。
注: ローカルアライメントを使用する場合、すべてのターゲット イメージに対応する参照イメージが必要です (図 2A)。局所的な色のシフトがサンプルによって異なるため、生物学的較正基準画像では局所的な位置合わせは推奨されない(表1)。同じ理由で、単一のローカル位置合わせは、多くのターゲットイメージに適用できません(図2B)。例外として、目的のサンプルがカバースリップの表面にのみ、多色蛍光ビーズサンプルのように明るい物体で充填された場合に使用できます。 - 複数の生物学的較正基準画像の場合、平均参照オプション(図1H)をチェックして、平均画像から単一のアライメントパラメータを測定し、その後、単一のアライメントパラメータがすべてのターゲット画像に適用されます(図2C)。Cローカル位置合わせの場合は[なし]を選択します(図1F)。
- 測定を開始するには、[すべて実行] (図 1I) をクリックします。整列パラメータがターゲットイメージに適用されます。
注:参照ファイルからクロマティックシフトを測定した後、プログラムは拡張子"chromagnon.csv"または"chromagnon.tif"のファイルを作ります。出力ファイルのタイプは、ローカル・アライメントが有効かどうかによって異なります (図 1F)。ローカルアライメントなしで整列を行う場合、出力は "chromagnon.csv" であり、ローカルアライメントが使用されている場合、出力は "chromagnon.tif" になります。ファイル名は、指定されたサフィックス (図 1J、デフォルトはサフィックスなし) と拡張子 (「chromagnon.csv」または「クロマニョン.tif」) を除き、参照ファイルと同じです。アライメントプロセスの詳細な説明は、同じフォルダに作成された"Chromagnon.log"ファイルにあります。 - 修正した画像がビューアに表示されるまで待ちます (図 3)。
メモ:マウスで画像をドラッグすると、画像が移動し、マウスホイールを動かすとズームが変わります。スライダを動かす (図 3A)を動かして、表示する Z (および/または T) セクションを変更します。ビューアがイメージを更新するのに時間がかかりすぎる場合は、[データ全体をメモリに読み込む] ボタン (図 3B) をクリックして、ハード ディスク上のデータへのアクセスを停止します。カラーボックスの左端または右端をドラッグCすると、表示の最小値または最大値を制御できます。各チャンネルのボタンをクリックすると(図3D)、ビューアで選択したチャンネルの表示/非表示を切り替えることができます。カラーボックス (図 3D)を右クリックすると、表示するカラーを選択したり、カラーバーの表示オプションを変更したりすることが可能で、表示倍率の正確な最小値と最大値を指定することもできます。[直交ビュー ] ボタン (図 3E)をクリックすると、ZY ビューと XZ ビューのイメージが表示されます。クロスラインを移動すると、サイドビューに表示される位置が変わります。
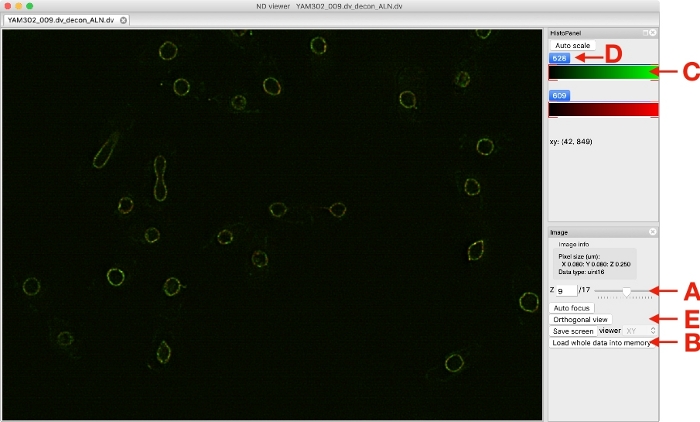
図 3: 画像ビューアのスクリーンショット。この図の大きなバージョンを表示するには、ここをクリックしてください。
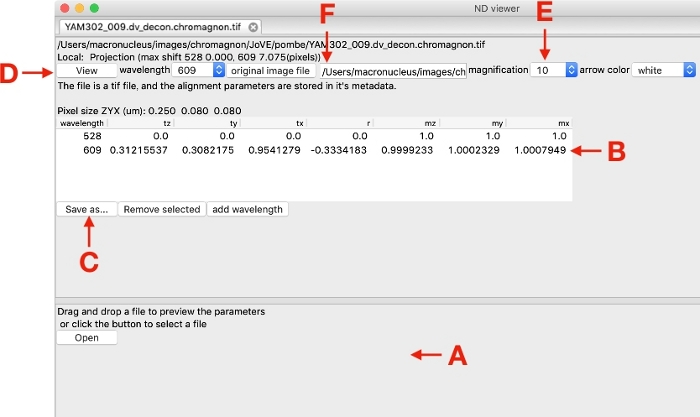
図 4: 配置パラメーター エディターのスクリーンショット。この図の大きなバージョンを表示するには、ここをクリックしてください。
- ローカルアライメントマップを確認するには、クロマニョンのリファレンスまたはターゲットボックス(図1)から"chromagnon.tif"をロードし、ダブルクリックしてアラインメントエディタを開きます(図4)。[ビュー ] (図 4D) をクリックすると、拡大表示の選択肢リストで指定した係数で長さが拡大された線で示されるローカル シフトが表示されます (図 4E)。
メモ: 「元の画像ファイル」(図4F)を指定すると、シフトが元の画像と一緒にマッピングされます。
4. 顕微鏡固有のローカルアライメントマップの生成
結果
クロストーク参照画像を用いた色度シフト補正の例を図5に示す。画像は、単一のカメラを搭載した広視野顕微鏡で得られた。DAPIからの蛍光発光を基準として使用し、青色、緑、および赤色のチャネルを補正します(図5A,B)を参照しました。画像は60 Zスライスの3チャンネルで構成され、それぞれ256 x 256ピクセルで構成されています。画像は、色の変化を測定する前にデコンボルブされました。クロマニョンを使用してローカルの色のシフトを測定すると、Intel Core i7(クアッドコア、8スレッド、2.7 GHz)、16 GB RAM、1 TBフラッシュストレージを搭載したMacで51秒を取りました。アライメント パラメータは、参照画像とまったく同じ数のボクセルを持つターゲットイメージ (図 5C,D)に適用されました。整列ファイルの準備には 3 s が必要でした。配置プロセス中にエッジピクセルをトリミングした結果 (図 1Cのトリミングマージンチェックボックス)、位置合わせ後にボクセルの数が減少しました (図 5B、51Z スライス、252 x 251 ピクセル)。アナフェーズブリッジ内のDNA(矢印で示される)は、アライメントの前に核エンベロープの外で誤って見られます(図5C、XZビューを示す下のパネルでは明らかです)。しかし、アライメント後にエンベロープ内で期待通り(図5D)。
生体較正基準画像を用いた色度シフト補正の例を図6に示す。画像は3台のカメラを搭載したSIM顕微鏡で得られた。青色、緑色、赤色の色素に結合したファロイジンで染色されたHeLa細胞の3つの画像を平均化した(図6A)。参照画像は、76 Zスライスの3チャンネルで構成され、それぞれ1,024 x 1,024ピクセルで構成されています。上述のMacシステム上で194s必要なローカルアライメントなしでクロマニョンを用いてクロマチックシフトを測定する。このパラメータは、それぞれ 1,024 x 1,024 ピクセルで構成される 73 Z スライスの 3 チャンネルからなるターゲット イメージに適用されました。整列ファイルの生成には 25 s がかかった。XZ ビューは、Z 方向に沿ってチャネルの位置が正しくないことを示し、X 方向 (図 6A,C)に沿って少しだけ表示されますが、この誤った登録はアライメント後に修正されました (図 6B,D)。
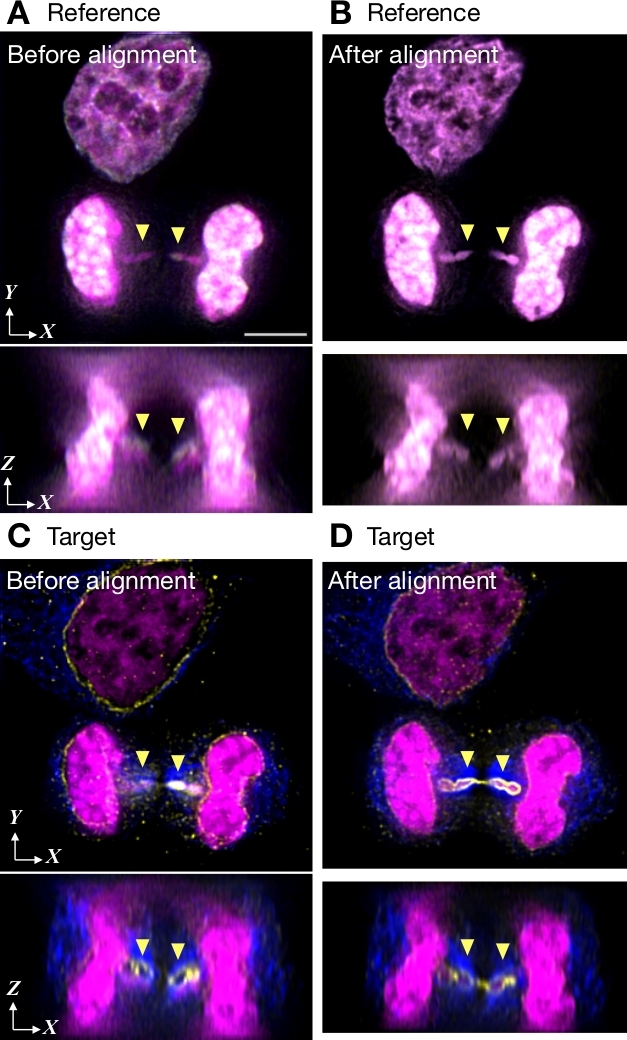
図5:クロストーク参照画像によるアライメントの例。HeLa細胞は、DNA(マゼンタで示されている)、核エンベロープのアレクサフルオール488(黄色で示す)、微小管のアレクサフルオール555(青色で示される)でDAPIで染色された。画像は、単一のカメラで3D広視野顕微鏡によって取得され、デコンボルブされました。(A,B)DAPI放出を使用した代表的なクロストーク参照画像で、クロマニョンによるアライメントの前後に.3 つのカラー チャネルがオーバーレイとして表示されます。(C,D)アライメント前後の 3 つのチャネルで 3D スタックの光学部分。矢印が示すアナフェーズブリッジでは、軸色収差が明らかです。パネルAのスケールバーは、すべてのパネルに対して5μmを示します。この図の大きなバージョンを表示するには、ここをクリックしてください。
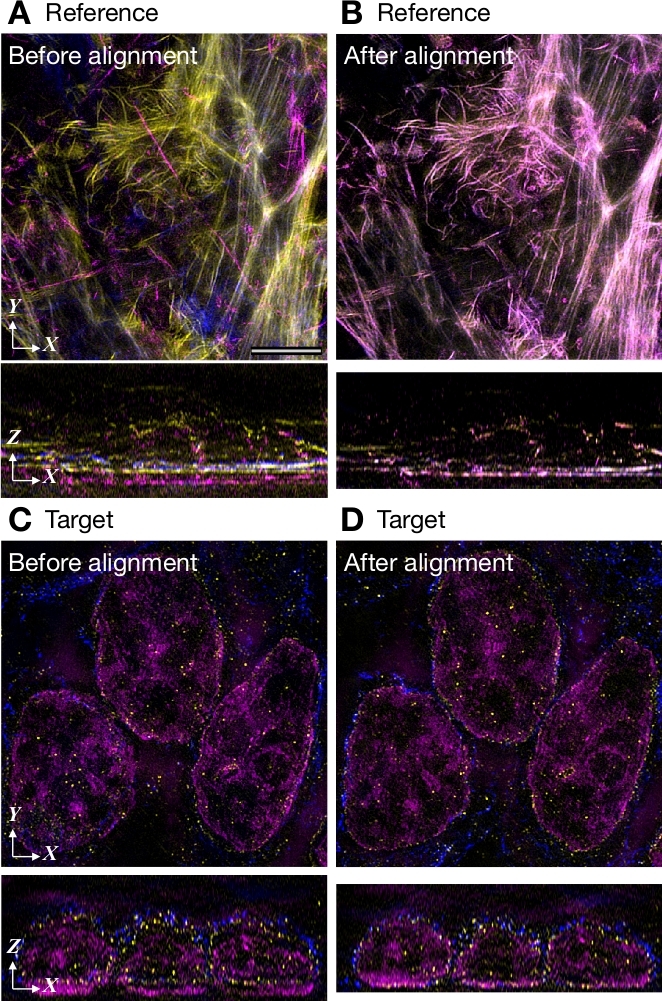
図6:生物学的キャリブレーション基準画像とのアライメントの例3台のカメラを搭載した3D-SIMで撮影した画像です。(A,B)(A) と (B) の配置の前の 3 つの画像から平均化された参照画像。HeLa細胞をアレクサフルオール405、488または594と共役したファロイジンで染色した。(C,D)(C)の前と後の (D) の配置のターゲット イメージ。HeLa細胞は、DNA(マゼンタで示されている)、核エンベロープのアレクサフルオール488(黄色で示す)、微小管のアレクサフルオール594(青色で示される)のDAPIで染色された。パネルAのスケールバーは、すべてのパネルに対して5μmを示します。この図の大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
色補正の手順は、精度と労力のトレードオフです。不必要な努力を省くために、あなたの研究に必要な正確さの程度を知る方がよいでしょう。従来の広視野(ライブ)イメージングでは最高の精度が必要ない場合があり、したがって、明視野参照画像は多くの場合、色調シフトを補正するのに十分です。同様に、イメージング条件と環境が一定の場合、生物学的較正を繰り返し使用すると時間を節約できます。一方、高精度な登録が望まれる場合は、高品質のクロストークや生体校正基準画像が必要です。最適なパフォーマンスを得るため、参照画像は、可能な限りターゲットイメージと同様の条件とタイミングで取得する必要があります。参照画像とターゲット画像の両方が同じ顕微鏡で取得されている限り、空間解像度が高いほど補正精度が向上します。参照イメージとターゲット イメージの両方にデコンボリューションが使用可能な場合、修正前にこれを実装すると、補正精度が向上する可能性があります。また、最適なパフォーマンスを得るため、正確なサブピクセル補間を行うため、参照ファイルとターゲットファイルの両方で光(Z)軸のサンプリング定理を満たす必要があります(プロトコルステップ2.1.3)。
色のシフトを修正しないと、誤った結論につながります。さらに、間違ったキャリブレーションを使用すると、クロマティックシフトを補正するのではなく、さらに悪化させる可能性があるため、これを避ける必要があります。失敗の考えられる原因と、その共通の解決策を表 2に要約しました。故障の原因を調べるには、まず、基準画像の色ずれが正確に補正されているかを視覚的に確認する必要がある(プロトコルステップ3.12)。ほとんどの障害は、参照イメージの品質によるものであり、表 2の説明に従って簡単に改善できます。参照画像の品質に関しては、全体の視野がサンプルで満たされていない場合、グローバル配置の精度が低下することに注意することが重要です (図7、表2)。図7Aに示す良い例と比較すると、図7Bに示されている悪い例は左上領域に3つの核エンベロープしか含まれず、クロマニョンはこの画像の一部を整列させることができませんでした。これは、クロマニョンのグローバルアライメント方法が、高い精度で回転と倍率の差を測定するために視野を4つの領域(図7C)に分割するためである。この方法は、正しく動作すれば、対数極変換やシンプレックス法3などの他の線形法よりも1つの順序で正確である。4つの領域のいずれかが利用できない場合、クロマニョンは効果の低い線形法に切り替えます。したがって、最適なパフォーマンスを得るため、図7Bおよび図 7Cに示す例は望ましくありません。ユーザーは、ログファイル(「Chromagnon.log」;プロトコルステップ3.10を参照)を見て、視野の二次領域が測定に利用できないかどうかを確認できます。幸いにも、この問題は、複数の生物学的較正画像を平均化するか、クロストークまたは明視野参照画像に局所的なアライメントを使用することによって簡単に克服できる(表2)。参照画像の修正に失敗した場合とは対照的に、ターゲット画像の修正に失敗すると識別が困難になります。このような障害は、ファイル形式、画像化条件、イメージングのタイミング、参照画像とターゲット画像のイメージング/アライメント方法の違いにより生じるため(表2)、ターゲット画像とは異なる条件/タイミングで取得した参照画像を使用する場合は常に注意が必要です。いくつかの例の画像は、良い例と悪い例のイメージの具体的なアイデアを得るために(https://github.com/macronucleus/Chromagnon)テストのために利用可能です。
| 問題 | 原因 | ソリューション |
| 参照イメージを修正できませんでした | 低コントラスト | 可能であれば、より高いコントラスト画像を取得します。明視野参照画像を使用する場合は、水ベースの溶液で画像を再取得し、セルのコントラストを高くします。あるいは、計算ノイズリダクション(ガウスフィルタリングなど)を適用してみてください。ノイズに対する感度が高いローカルアライメントをオフにします。 |
| 無関係画像の汚染 | 可能な場合は、サンプル内の無関係の画像のソースを削除します。クロストーク参照画像の場合、ターゲット画像に使用される染料の励起スペクトルを確認します。クロストーク画像の取得中に色素が励起される場合(例えば、Alexa Fluor 568または594)、他の色素(例えばAlexa Fluor 555)を検討してください。カメラチップのほこりが明らかなチャネルの違いを生み出す場合は、カメラチップをクリーニングするか、計算的なフラットフィールディング方法を使用します。 | |
| 宇宙線で作られた非常に明るいスポット | 可能であれば、画像を再度取得します。あるいは、計算ノイズリダクション(例えば、中央値またはガウスフィルタリング)を適用してみてください。 | |
| デコンボリューションアーティファクト(軸辺と横辺の人工信号) | デコンボルション後にエッジピクセルまたはZセクションをトリムします。一方の側をトリムする場合は、もう一方の側もトリミングしてイメージの中心を維持する必要があります。 | |
| Z ステップ サイズがまばらすぎる | Z スタックは、プロトコル 2.1.3 に記述されているようにナイキスト基準を満たすために取得する必要があります。 | |
| 光学収差 | 球面収差は、ユーザーによって引き起こされる主要な収差です。サンプルに適した対物レンズを選択し、カバースリップ厚さ170μmを使用します。対物レンズに補正リングが付いている場合は、焦点から蛍光数が最も高い位置を見つけるために調整します。補正リングのない油浸性目的の場合、焦点で蛍光数を増加させる浸漬油の屈折率を調整します。 | |
| 視野が未入力(図7) | 生物学的較正基準画像の場合、多くの画像を平均する。クロストークまたは明視野参照画像の場合は、ローカルアライメントを使用します。 | |
| ソフトウェアの未確認バグ | GitHub を通じて問題を報告する (https://github.com/macronucleus/Chromagnon/issues) | |
| ターゲット イメージを修正できませんでした | イメージ ファイルのメタデータが失われる | 完全なメタデータを含むオリジナルの顕微鏡ファイル形式を使用し、処理前にマルチページのtiffファイルに変換しないようにしてください。プロトコル 3.3 で書かれているチャネルの順序と同じ順序を使用します。 |
| 与えられた顕微鏡のための間違ったアライメント方法 | 生体キャリブレーション基準画像からターゲット画像まで測定する場合は、局所アライメント法を適用しないでください。広視野顕微鏡以外のクロストーク参照画像は使用しないでください。 | |
| イメージング条件の違い | プロトコル 2.3.3 に記述されているように、参照イメージとターゲット イメージの間で一定のイメージング条件を維持します。 | |
| サンプルの違い (カバースリップを含む) | 常に同じ取り付け媒体、カバースリップ(例えばNo.1.5H)および同様の焦点深度を使用してください。 | |
| キャリブレーションが最後に行われたので顕微鏡のドリフト | 2週間ごとにキャリブレーションを行います。温度を一定に保ち、浮動テーブルを使用して、顕微鏡のハードウェアドリフトを避けます。 |
表2:色補正のトラブルシューティング
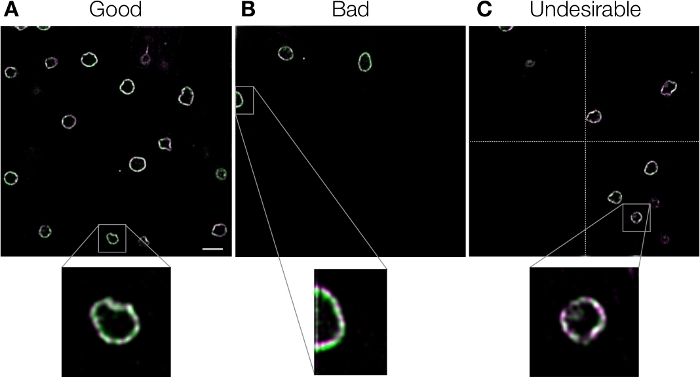
図7:参照画像の例核分裂酵母細胞にGFPとmCherryで標識された核エンベロープ。従来の広視野顕微鏡で画像を取得した。クロマティックシフトは、画像自体を基準画像として使用して、局所的な位置合わせなしにクロマニョンを使用して補正しました。その後、画像をデコンボルブして詳細を表示しました。(A) 視野に多数のオブジェクトがある良い例。(B) オブジェクトが左上隅にある場合にだけ、不適切な例です。イメージの特定の領域では、ずさみが明らかです。(C) 四重節の 1 つ (点線で区切られた) が空の望ましくない例。パネルAのスケールバーは、全視野ビューでは5μm、拡大表示では1.25μmと示し、すべてのパネルに適用可能です。この図の大きなバージョンを表示するには、ここをクリックしてください。
このプロトコルでは、3 つの異なる参照タイプについて説明しました (表 1)。その中で、クロストークリファレンス画像と生物学的較正基準画像は、さらに慎重な議論が必要です。クロストーク基準画像の場合、サンプルはDAPIまたはHoechst 33342で染色され、グリセロールまたは市販の取り付け媒体に取り付けられ、ブルー、グリーン、および赤色のチャネルを整列させるために効率的に使用できます。同様に、アレクサのフルーラ 488 は、緑と赤のチャンネルを整列するために使用できます。しかし、DAPIとHoechstを除く多くの青色染料は、ほとんどの緑と赤の染料よりも薄暗く、腐敗するので、クロストーク蛍光を得ることはしばしば困難です。さらに、現代の色素の発光スペクトルは狭く、この方法によって3チャンネル以上のアライメントが困難になります。また、青色の色素からコントラストの高いクロストーク画像を得ることを防ぐ紫色光で励起できる一般的な赤色色素(例えば、Alexa Flour 568と594、Alexa Fluor 555ではない)にも注意が払われるべきです。もう一つの欠点は、この方法が励起に単一の励起波長しか使用されていないので、多色励起における励起光経路の色収差を測定できないことである(表1)。最も高度な顕微鏡は、変更された照明光学を使用するので、この方法の適用は限られている。それでも、その高い補正精度は、このプロトコルに記載されるには十分に有利である。一般に、クロストーク画像は、漂白や光毒性の影響を防ぐために、ターゲット画像の後に撮影する必要があります。広視野モードで観察されたSMLMの場合、蛍光色素がイメージング中に漂白することができるので、ターゲット画像を取得する前に基準画像を取得する必要があります。
生物学的キャリブレーション基準画像により、ユーザーは追加サンプル調製のコストで任意の数のチャネルを容易に整列させることができます。生物学的較正基準画像のもう1つの利点は、すべての視野を埋めるのに役立つ複数の参照を「平均」する可能性です。この方法は、キャリブレーションサンプルが別のスライドで調製された場合、イメージング条件の違いに苦しむ可能性があります。この問題のほとんどは、市販のチャンバーカバーグラス(表1)を使用して同じスライド上にターゲットと参照の両方を準備し、他のイメージング条件がプロトコルステップ2.3.3のように一定に保たれている場合に解決できます。この場合、クロストーク参照画像と同様の補正精度が期待できる3.ここに示すようにファロイジンを使用するプロトコルは、複数の色で単一の細胞構造を染色する最も簡単な方法の一つです。生物学的較正サンプルを準備するシナリオは数多くあります。免疫染色の場合、サンプルは単一の一次抗体で標識され、その後に複数色の二次抗体で染色することができます。このようにして、1 つのターゲット構造に複数の色でラベルを付けることができます。あるいは、5-エチニル-2'-デオキシウリジンは、高密度で複数色で新たに合成されたDNAを「クリック」化学ラベルで検出し、詳細に前述した8。生細胞の場合、GFPまたはmCherryに融合した遺伝子の2つのコピーを含むトランスジェニック株を調製して、同じ構造を2色でラベル付けすることが有用である。膜タンパク質に対してしばしば観察されるほど、遺伝子のコピー数が重要である場合、遺伝子の単一のコピーをGFPとmCherryとタンデム的に融合させることができる(図7)。mEOS218のような光変換可能な蛍光タンパク質は、適度なレベルの紫色光を照明することによって使用して、光変換の有無にかかわらず両方のタンパク質種を得ることができる。低酸素条件下では、GFPは、緑から赤19、20,20までの光変換可能なタンパク質としても使用できます。適切なキャリブレーションサンプルを選択すると、実験がより堅牢になります。
開示事項
著者らは開示するものは何もない。
謝辞
この研究は、JSPS KAKENHIグラント番号JP19H03202からA.M.に支援されました。 JP18H05528およびJP17H003636からT.H.、JP17H01444およびJP18H05533からH.Y.L.S.は、ウェルカム・トラスト・ストラテジック・アワード091911および107457/Z/15/Z資金調達のシニアイメージングを認めています。
資料
| Name | Company | Catalog Number | Comments |
| 16% formaldehyde solution | Polyscience | 18814-10 | |
| 35 mm glass-bottom dish | MatTek | P35G-1.5-10-C | |
| Alexa Fluor 405 phalloidin | Thermo Fisher Scientific | A30104 | |
| Alexa Fluor 488 phalloidin | Thermo Fisher Scientific | A12379 | |
| Alexa Fluor 594 phalloidin | Thermo Fisher Scientific | A12381 | |
| Bovine Serum | Thermo Fisher Scientific | 16170078 | |
| Coverslip | Matsunami | No. 1S HT | |
| DAPI (4',6-Diamidino-2-Phenylindole, Dihydrochloride) | Thermo Fisher Scientific | D1306 | |
| Dulbecco’s Modified Eagle Medium with L-Gln and sodium pyruvate | Nacalai Tesque | 08458-16 | |
| Mounting medium (VECTASHIELD) | Vector Laboratories | H-1000 | |
| Mouse anti-tubulin monoclonal antibody (TAT1) | Described in Ref 15. | ||
| Nunc Lab-Tek II chambered coverglass (8 well) | Thermo Fisher Scientific | 155409 | |
| Rabbit anti-emerin polyclonal antibody (ED1) | A gift from Hiroshi Yorifuji, Gunma University, Gunma, Japan and Kiichi Arahata, National Center of Neurology and Psychiatry, Tokyo, Japan; deceased. | ||
| Secondary antibody with Alexa Fluor 488 | Thermo Fisher Scientific | A-11034 | |
| Secondary antibody with Alexa Fluor 555 | Thermo Fisher Scientific | A-21424 | |
| Secondary antibody with Alexa Fluor 594 | Thermo Fisher Scientific | A-11032 | |
| TetraSpeck Microspheres, 0.2 µm | Thermo Fisher Scientific | T7280 |
参考文献
- Schermelleh, L., et al. Super-resolution microscopy demystified. Nature Cell Biology. 21 (1), 72-84 (2019).
- Manders, E. M. M. Chromatic shift in multicolour confocal microscopy. Journal of Microscopy. 185 (3), 321-328 (1997).
- Matsuda, A., Schermelleh, L., Hirano, Y., Haraguchi, T., Hiraoka, Y. Accurate and fiducial-marker-free correction for three-dimensional chromatic shift in biological fluorescence microscopy. Scientific Reports. 8 (1), 7583(2018).
- Grünwald, D., Singer, R. H. In vivo imaging of labelled endogenous β-actin mRNA during nucleocytoplasmic transport. Nature. 467 (7315), 604-607 (2010).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6221), 543-548 (2015).
- Kraus, F., et al. Quantitative 3D structured illumination microscopy of nuclear structures. Nature Protocols. 2, 1011-1028 (2017).
- Hell, S. W. Far-Field Optical Nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. Journal of Microscopy. 198 (2), 82-87 (2000).
- Schulz, O., et al. Resolution doubling in fluorescence microscopy with confocal spinning-disk image scanning microscopy. Proceedings of the National Academy of Sciences of United States of America. 110 (52), 21000-21005 (2013).
- Müller, C. B., Enderlein, J. Image Scanning Microscopy. Physical Review Letters. 104 (19), 198101(1981).
- Hayashi, S., Okada, Y. Ultrafast superresolution fluorescence imaging with spinning disk confocal microscope optics. Molecular Biology of the Cell. 26 (9), 1743-1751 (2015).
- Yorifuji, H., et al. Emerin, deficiency of which causes Emery-Dreifuss muscular dystrophy, is localized at the inner nuclear membrane. Neurogenetics. 1 (2), 135-140 (1997).
- Woods, A., Sherwin, T., Sasse, R., MacRae, T. H., Baines, A. J., Gull, K. Definition of individual components within the cytoskeleton of Trypanosoma brucei by a library of monoclonal antibodies. Journal of Cell Science. 93 (3), 491-500 (1989).
- Tokunaga, M., Imamoto, N., Sakata-Sogawa, K. Highly inclined thin illumination enables clear single-molecule imaging in cells. Nature Methods. 5 (2), 159-161 (2008).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529(2017).
- McKinney, S. A., Murphy, C. S., Hazelwood, K. L., Davidson, M. W., Looger, L. L. A bright and photostable photoconvertible fluorescent protein for fusion tags. Nature Methods. 6 (2), 131-133 (2009).
- Sawin, K. E., Nurse, P. Photoactivation of green fluorescent protein. Current Biology. 7 (10), 606-607 (1997).
- Elowitz, M. B., Surette, M. G., Wolf, P. E., Stock, J., Leibler, S. Photoactivation turns green fluorescent protein red. Current Biology. 7 (10), 809-812 (1997).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved