Method Article
腫瘍微小環境のマイクロスケールプロテオミクス分析のための工業化された人工知能誘導レーザーマイクロダイセクション
* これらの著者は同等に貢献しました
要約
このプロトコルは、レーザーマイクロダイセクションを使用して組織学分解細胞集団を濃縮するための、染色された薄い組織切片画像から病理学的に確認された関心領域を人工知能駆動でセグメンテーションするためのハイスループットワークフローを記述します。この戦略には、関心のある細胞集団を示す境界をレーザー顕微鏡に直接転送することを可能にする新しいアルゴリズムが含まれる。
要約
腫瘍微小環境(TME)は、腫瘍、間質、免疫細胞集団を含む数十種類の異なる細胞型で構成される複雑な生態系を表しています。プロテオームレベルの変動と腫瘍の不均一性を大規模に特徴付けるためには、固形腫瘍悪性腫瘍中の離散細胞集団を選択的に単離するためのハイスループット法が必要である。このプロトコルは、人工知能(AI)によって可能になるハイスループットワークフローを記述し、ヘマトキシリンとエオジン(H&E)染色された薄い組織切片の画像を病理学的に確認された関心領域にセグメント化し、レーザーマイクロダイセクション(LMD)を使用して組織学的に解決された細胞集団を選択的に採取します。この戦略には、デジタル画像ソフトウェアを使用して注釈を付けた目的の細胞集団を示す領域をレーザー顕微鏡に直接転送することを可能にする新しいアルゴリズムが含まれており、より簡単な収集を可能にします。このワークフローの実装が成功裏に実施され、高分解能質量分析による定量的多重化プロテオーム分析のためにTMEから腫瘍細胞集団を選択的に採取するこの調和法の有用性が実証されました。この戦略は、日常的な病理組織学レビューと完全に統合され、デジタル画像解析を活用して目的の細胞集団の濃縮をサポートし、完全に一般化可能であり、多体異系分析のためにTMEから細胞集団を調和のとれた収穫を可能にします。
概要
TMEは、複雑な細胞外マトリックス1とともに、腫瘍細胞、間質細胞、免疫細胞、内皮細胞、他の間葉系細胞型、脂肪細胞などの非常に多様な細胞型が住む複雑な生態系を表しています。この細胞生態系は、異なる疾患器官部位内および異なる疾患部位間で変化し、複雑な腫瘍の不均一性をもたらす2,3。最近の研究では、不均一な腫瘍および腫瘍細胞性が低い(純度が低い)腫瘍は、しばしば疾患予後不良と相関することが示されている2,3。
TME内の腫瘍細胞集団と非腫瘍細胞集団の間の分子相互作用を大規模に理解するためには、下流のマルチオミクス分析のために目的の個別の細胞集団を選択的に採取するために、標準化されたハイスループット戦略が必要です。定量的プロテオミクスは、がん生物学の理解を深めるために急速に進化し、ますます重要な技術です。今日まで、プロテオミクスを用いた研究の優勢は、腫瘍組織全体の調製物から抽出されたタンパク質(例えば、凍結凍結)を用いてそうしており、TMEにおけるプロテオームレベルの不均一性の理解において不足をもたらしている4,5,6。
臨床病理学ワークフローとシームレスに統合し、臨床病理学ワークフローからの情報を活用するサンプル収集戦略の開発により、ゴールドスタンダードの診断病理学ワークフローを高度に補完する新世代の組織学解決プロテオミクスが可能になります。LMDは、組織学的に染色された組織薄切片7の顕微鏡検査を通じて、細胞亜集団または関心領域(ROI)の直接的かつ選択的な収集を可能にする。デジタル病理学とAI対応解析における最近の大きな進歩により、TME内のユニークな組成特徴とROIを自動化された方法で特定する能力が実証されており、その多くは分子変化や治療に対する耐性や疾患予後などの臨床的疾患の特徴と相関しています8。
ここで紹介するプロトコルで説明するワークフローは、商用ソフトウェアソリューションを活用してデジタル組織病理学画像内の腫瘍ROIに選択的に注釈を付け、社内で開発されたソフトウェアツールを使用してこれらの腫瘍ROIをレーザー顕微鏡に転送し、下流のマルチオミック解析ワークフローとシームレスに統合する目的の離散細胞集団の自動収集を行います。この統合戦略により、LMDオペレータの時間が大幅に短縮され、組織が周囲温度にある必要がある期間が最小限に抑えられます。自動特徴選択とLMD収穫とハイスループット定量プロテオミクスの統合は、2つの代表的な上皮性卵巣癌組織学的サブタイプ、高悪性度漿液性卵巣癌(HGSOC)および卵巣明細胞癌(OCCC)からのTMEの差動解析によって実証される。
プロトコル
すべての研究プロトコルは、米国連邦規則45 CFR 46.102(f)の下で免除されるとみなされた、西部IRB承認プロトコル「臨床的に有益なバイオマーカーを同定および検証するための子宮内膜癌および卵巣癌の統合分子分析」の下での使用が承認された。この研究におけるヒトデータを含むすべての実験プロトコールは、ヘルシンキ宣言に準拠していた。インフォームドコンセントは、研究に関与するすべての被験者から得られた。
警告: プロトコール全体を通して使用される以下の試薬は、発がん性物質が既知または疑われる試薬、および/または有害物質を含んでいます:エタノール、DEPC水、メイヤーズヘマトキシリン溶液、エオシンY溶液、メタノール、アセトニトリル、およびギ酸。各安全データシート(SDS)に記載されているように、適切な取り扱い、および適切な個人用保護具(PPE)の使用が必須です。
1. キャリブレータ基準を含むデフォルトのシェイプリストデータ(.sld)ファイルの生成
メモ: このセクションで説明するプロトコル手順は、倒立レーザー顕微鏡および関連ソフトウェアで使用するために固有の ものです(材料表を参照)。デフォルトの.sldファイルの作成は、レーザー顕微鏡ごとに1回だけ必要です。結果のファイルは、基準をその後に使用されるすべてのPENスライドにカットするために使用できます。おおよその時間:5分(1回のみ)。
- LMD ソフトウェアを開き、ポリエチレンナフタレート (PEN) メンブレンをスライドさせて LMD ステージに下向きにロードし、ラベルをユーザーに近づけます。プログラムウィンドウの右側にある [線を閉じる]ボックスの選択を解除します。
- 高倍率(63倍)下で PtoP (ポイントツーポイント)関数を使用して、3つの「V」矢印を描画してキャリブレーション基準として機能します。V上の1つの外部点から始めて、Vの中点に線を引き、シングルクリックします。次に、V の中心点から 2 番目の外部 V 点の端まで 2 番目の線を引き、ダブルクリックして、2 本の線から閉じていない単一の V 字型を作成します。
メモ:これらのキャリブレーション基準は、スライドの3つのコーナー(フロント右、リア右、リア左)に配置する必要があります。 - カット前にAF(オートフォーカス)オプションを選択します。スライドを位置1にカットし、残りのスライド位置のそれぞれに移動し、キャリブレーションカットを正確にトレースします。
- .sld ファイルを保存し、ポップアップダイアログボックスから「キャリブレーション なしで保存」オプションを選択して、キャリブレーション 基準を膜にカットしないようにします。
注: 4 つのスライド位置の標準キャリブレータ基準を含む代表的な .sld ファイルは、 補足ファイル 1 に記載されています。
2. LMD スライドの準備
メモ: このセクションで説明するプロトコル手順は、倒立レーザー顕微鏡および関連ソフトウェアで使用するために固有の ものです(材料表を参照)。おおよその時間:5分。
- 基準校正基準を切断する前に、スライドが完全に乾いていることを確認してください。LMD ソフトウェアを開き、[ 図形のインポート] オプションで既定のキャリブレーション .sld ファイルを開きます。
- カット前にAF(オートフォーカス)オプションを選択します。ティッシュが下を向いた状態でスライドをロードし、ラベルをオペレータの近くにスライドさせてLMDステージのスライドホルダーに挿入します。
- レーザー顕微鏡とデフォルトのキャリブレーション.sldファイルを使用して、キャリブレーション基準をPEN膜にカットします。
- オプション:組織切片がスライド上に置かれる前または後に、校正基準をPEN膜に切断します。組織配置前にキャリブレーション基準を切断する場合は、ステップ2.5で組織をスライド上に配置したときに、組織および/または固定液がキャリブレータと重ならないことを確認してください。組織配置後に較正基準が切断される場合は、ステップ2.4の完了後に停止し、セクション3に進みます。
- すべてのキャリブレータを個別に見直して、各カットが完全で見えることを確認します。
メモ: 移動およびカット 機能を使用して、PEN膜を完全に切断しなかったキャリブレーション基準にレーザーを手動で向けます。 - 凍結またはホルマリン固定パラフィン包埋(FFPE)組織切片を、較正基準を含むスライドの上に置きます。
3. 組織染色
注:おおよその時間:30分。
- 凍結LMD組織スライドをホスファターゼ阻害剤カクテル試薬を含む70%エタノール(EtOH)中で5分間固定する。
- ホスファターゼ阻害剤カクテル試薬を含むジエチルピロカルボメート(DEPC)水でスライドを1分間洗浄する。
- スライドをDEPC水で1分間洗う。
- スライドをメイヤーのヘマトキシリン溶液中で3分間インキュベートする。
- スライドをDEPC水で3分間すすいでください。
- DEPC水を1分間交換したばかりの状態でスライドをすすいでください。
- スライドをEosin Y水溶液中で1秒間インキュベートする。
- スライドを95%EtOH中で2 x 5秒すすぎます。
- スライドを100%EtOH中で3 x 10秒すすぎます。
- スライドの背面から余分なEtOHを拭き取り、スライドを風乾させます。
- LMDがすぐに実行されない場合は、スライドを-80°Cで保管してください。
4. スライドイメージング
メモ: このセクションで説明するプロトコル手順は、スキャンしたスライド( 材料表を参照)と、.svsファイルとして保存された結果のイメージに固有のものです。画像解析ソフトウェア( 材料表を参照)が開くことができる形式で画像ファイルを生成するスキャナおよび関連するソフトウェアを使用します。サポートされているピラミッド型 TIFF を使用するファイルの種類には、JPG、TIF、MRXS、QPTIFF、コンポーネント TIFF、SVS、AFI、SCN、LIF、DCM、OME などがあります。TIFF、ND2、VSI、NDPI、NDPIS、CZI、BIF、KFB、および ISYNTAX です。おおよその時間:5分。
- スキャナーの電源を入れ、スライドスキャナーソフトウェアを開きます。ティッシュを上に向けてスライドをスキャナーの 1 つのスライドステージにセットします。スライドが完全に乾いていることを確認し、カバースリップをティッシュの上にそっと置きます。カバースリップの下にエタノールや浸漬油を使用しないでください。
- メーカーの指示に従って、ガラスの背景の代わりにPEN膜を調整し、背景膜染色を無視するように較正された設定を使用して顕微鏡写真画像をキャプチャします。
- 必要に応じて、内側の緑色の周囲をドラッグしてサイズ変更し、PEN膜領域全体をキャプチャして、イメージング領域を調整します。スナップショットの概要画像をダブルクリックして組織に4つのフォーカスポイントを追加し、キャリブレーション基準の近くの膜に3つのフォーカスポイントを追加します(3つのキャリブレーション基準のそれぞれに1つのフォーカスポイント)。
メモ:4つのフォーカスポイントは、組織断面のほぼどこにでも配置できますが、暗すぎる染色で黒く見える組織に配置すると、スキャンが失敗する可能性があります。 - 表示メニューで、ビデオモニターを選択します。必要に応じて、ファインフォーカススライダーおよび/またはマクロフォーカススライダーを使用して、LMD組織の周りの各ポイントに対して手動でフォーカスを調整します。高倍率(20倍)で画像スキャンをキャプチャします。保存した画像にすべてのキャリブレーション基準が透明で明瞭であることを確認します。
5. 画像解析ソフトウェアによる自動特徴選択
- 腫瘍コレクション全体(おおよその時間:5分、症例依存):
- 画像解析ソフトウェアを開きます( 材料表を参照)。 [画像を開く] を選択し、ポップアップ ウィンドウから、AT2 スキャナーでのスライドのスキャンから生成された .svs 画像ファイルを選択します。
注: 代表的な .svs イメージ ファイルは、 補足ファイル 2 で提供されています。 - 「 アノテーション」 タブに移動します。アノテーションツールバーの ペン ツールを選択し、ティッシュの周囲にシェイプを描画します。
- 図形を選択し、画像を右クリックします。 [詳細設定] ドロップダウン メニューから、[ パーティショニング (タイル)] を選択します。 [タイル サイズ ] と [スペースの範囲 ] をそれぞれ 500 と 40 に設定し、[ OK ] を選択してタイルを生成します。手順 5.1.2 でタイルの生成に使用した境界図形を選択して削除します。
- [レイヤーアクション]ドロップダウンメニュー|選択します。エクスポートして、タイル状の注釈を .annotation ファイルとして保存します。
注:腫瘍組織コレクション全体に対する代表的な.annotationファイルは、 補足ファイル3に提供されています。 - セッションまたはプロジェクト用のフォルダーを作成し、スライドの一意の識別子でラベル付けされたサブフォルダー内に .annotation ファイルを保存します。
- 「 アノテーション」 タブに移動します。 [レイヤーアクション] ドロップダウン |選択します。[すべてのレイヤーを削除] : 画像からすべての注釈を削除します。 ペン ツールを選択し、キャリブレーション基準ごとに矢印の内側の先端から短い線を引きます。マークから、左上、右上、右下の順序で線を描画します。
- [レイヤーアクション]ドロップダウンメニューを選択|エクスポートして、行注釈を .annotation ファイルとして保存します。ファイル名に_calibを追加し、タイル図形の座標を含むサブフォルダーにファイルを配置します。
注: 代表的な _calib.annotation ファイルは、 補足ファイル 4 に用意されています。 - メインプロジェクトまたはセッションフォルダのアドレスをコピーします。IDLE統合開発環境を使用して、XMLインポート生成スクリプト「Malleator」(https://github.com/GYNCOE/Mitchell.et.al.2022 から入手可能)を開き、スクリプトの下部にある引用符の間にプロジェクトフォルダアドレスを貼り付けます。
- [ 実行 ] ドロップダウンメニュー |選択します。モジュールを実行して スクリプトを実行します。
メモ: .xml LMD インポートファイルは、画像/スライド用に作成されたサブフォルダ内に生成されます。代表的な.xmlファイルは、 補足ファイル5に提供されています。
- 画像解析ソフトウェアを開きます( 材料表を参照)。 [画像を開く] を選択し、ポップアップ ウィンドウから、AT2 スキャナーでのスライドのスキャンから生成された .svs 画像ファイルを選択します。
- LMD が豊富なコレクションのみ (おおよその時間: 15 分、大文字と小文字が異なります):
- 画像解析ソフトウェアを開きます( 材料表を参照)。 [画像を開く] を選択し、ポップアップ ウィンドウから、スライドのスキャンによって生成された .svs 画像ファイルを選択します。
- 「 アノテーション」 タブに移動します。 [長方形] 注釈ツールを選択して使用し、組織の周囲にボックスを描画します。
- ボックスの注釈を選択し、画像を右クリックします。[ 詳細設定 ]ドロップダウンメニュー |選択します。パーティショニング(タイル) オプション。 [タイル サイズ ] と [スペースの範囲 ] をそれぞれ 500 と 40 に設定し、[ OK ] を選択してタイルを生成します。手順 5.2.2 でタイルの生成に使用した境界ボックスの注釈を選択して削除します。
- [レイヤーアクション]ドロップダウンメニュー|選択します。エクスポートして、タイル状の注釈を .annotation ファイルとして保存します。
- AI 分類アノテーションレイヤーをマージするために開発された Python "Dapọ" (https://github.com/GYNCOE/Mitchell.et.al.2022 経由で利用可能) アルゴリズムの保存済みコピーを、タイルアノテーションファイルと同じフォルダーに配置します。
- タイルアノテーションファイルの名前をコピーします。IDLE 統合開発環境を使用して Python プログラムを開き、プログラムの下部にある引用符の間にタイル状の注釈ファイルの名前を貼り付けます。
- [ファイル名を指定して 実行 ]ドロップダウンメニュー |モジュールを実行します。すべてのタイルアノテーションが単一のレイヤーの下にマージされる新しいファイルが生成されるのを待ちます。
- 画像解析ソフトウェアを開き、[ 注釈] タブに移動します。 [レイヤーアクション] ドロップダウンメニュー |選択します。[すべてのレイヤーを削除] : 画像からすべての注釈を削除します。
- [レイヤーアクション]ドロップダウンメニュー|選択します。ローカルアノテーションファイルをインポートします。ポップアップウィンドウで、スクリプトによって生成されたマージされた .annotation ファイルを選択します。読み込まれたすべてのタイルが同じアノテーション レイヤーの下にあることを確認します。
- [分類子] タブに移動し、製造元の指示に従って ROI の図形を生成します。分類子を実行する前に、[高度な分類子オプション] の下にある [注釈] タブの [ROI ボックス] をオンにして、目的の注釈レイヤー (腫瘍または間質層) を選択します。分類子アクションメニューの「アノテーションレイヤー」オプションを使用して、分類子を実行します。
- 分類子解析が完了したら、「 アノテーション」(Annotations) タブに移動し、解析から生成されたアノテーションレイヤーを選択します。 [レイヤーアクション] ドロップダウンメニュー |選択します。「現在のレイヤーを除くすべてのレイヤーを削除」 を選択すると、他のすべてのアノテーションレイヤーが画像から削除されます。
- [レイヤーアクション]ドロップダウンメニューを選択|エクスポートして、注釈を .annotation ファイルとして保存します。セッションまたはプロジェクト用のフォルダーを作成し、スライドの一意の識別子でラベル付けされたサブフォルダー内に .annotation ファイルを保存します。
注: 分類された LMD 濃縮組織コレクションの代表的な .annotation ファイルは、 補足ファイル 6 に用意されています。 - [ 注釈] タブに移動し、[ レイヤー アクション] ドロップダウン |[すべてのレイヤーを削除] : 画像からすべての注釈を削除します。 ペン ツールを選択し、各キャリブレーション基準から短い線を引きます。マークから、左上、右上、右下の順序で線を描画します。
- [レイヤーアクション]ドロップダウンメニュー|選択します。エクスポートして、行注釈を .annotation ファイルとして保存します。ファイル名に_calibを追加し、タイル図形の座標を含むサブフォルダーにファイルを配置します。
- メインプロジェクトまたはセッションフォルダのアドレスをコピーします。IDLE統合開発環境を使用してXMLインポート生成スクリプト「Malleator」(https://github.com/GYNCOE/Mitchell.et.al.2022 から入手可能)を開き、スクリプトの下部にある引用符の間にプロジェクトフォルダアドレスを貼り付けます。
- [ファイル名を指定して 実行 ]ドロップダウンメニュー |モジュールを実行して スクリプトを実行します。
メモ: .xml LMD インポートファイルは、イメージ/スライド用に作成されたサブフォルダ内に生成されます。
6. レーザーマイクロダイセクション
メモ: このセクションで説明するプロトコル手順は、倒立レーザー顕微鏡および関連ソフトウェアで使用するために固有の ものです(材料表を参照)。おおよその時間:2時間。ケースによって異なります。
- 組織を下向きにし、ラベル側をオペレータに近づけた状態で、マークされたメンブレンスライド(校正基準を含む)をレーザー顕微鏡ステージのスライドホルダーにロードします。
- [ファイル] ドロップダウン メニューから [図形のインポート] を選択します。スライド用に生成された.xml LMD インポートファイルを選択します。ポップアップウィンドウで「いいえ」(No) を選択すると、ファイルから参照点が読み込まれず、2 番目のポップアップウィンドウで「いいえ」を選択すると、以前に保存した基準点がキャリブレーションに使用されなくなります。
- LMDアプリケーションのプロンプトに従って、キャリブレーションクロスをスライド上の3つのキャリブレーション基準のそれぞれに合わせます。画像解析ソフトウェアで、スライド画像の左上、右上、右下に表示されるキャリブレーション基準を探します。これは、顕微鏡ステージ上の反転 LMD スライドの前面右隅、後部右隅、および後部左隅の基準点に対応します。5x対物レンズを使用して位置を特定し、63x対物レンズを使用して各キャリブレーション基準を整列させます。ポップアップ ウィンドウで [ いいえ ] を選択して参照ポイントをファイルに保存しないようにし、2 番目のポップアップ ウィンドウで [ OK ] を選択してスライドが挿入されていることを確認します。
- 5倍対物レンズを所定の位置に移動し、ポップアップウィンドウで[ はい ]を選択して実際の倍率を使用します。読み込んだ図形が表示されたら、カメラをティッシュに合わせます。
- 「シェイプ リスト」(Shapes List) ウィンドウですべてのシェイプをハイライト表示して選択し、視野内の 1 つまたは 2 つのアノテーションを参照として使用して所定の位置にドラッグし、レーザーで切断するために垂直 Z 軸を揃えます。
- 読み込まれた図形を確認し、コレクションの適切なチューブ位置に割り当てます。「 カット開始」 を押してレーザーを開始します。
注: .xml ファイルで読み込まれた図形は、[ 図形リスト ] ウィンドウの [キャップなし] の位置に自動的に割り当てられます。組織を採取するには、インポートした形状を、装填されたチューブを含む位置に再割り当てする必要があります。
7. 圧力サイクル技術(PCT)によるタンパク質消化
注:おおよその時間:4時間(真空遠心分離機乾燥時間なしで3時間)。
- LMD採取組織を含むキャップ付きPCTマイクロチューブを20 μLの100 mM TEAB/10%アセトニトリルに収めた0.5 mLチューブをサーモサイクラーに入れ、99°Cで30分間加熱した後、50°Cまで10分間冷却します。
- チューブを 4,000 × g で 30 秒間回転させ、0.5 mL チューブからマイクロチューブを取り外します。マイクロキャップツールを使用して、PCTマイクロチューブからマイクロキャップを取り外して廃棄します。トリプシン( 材料表を参照)を30 mm 2組織あたり1 μgの割合で加え、MicroCapツールを使用して MicroTubeにMicroPestreを挿入します。
- マイクロチューブをバロサイクラーカートリッジに移し、カートリッジ全体を組み立てます。カートリッジをバロサイクラー圧力チャンバーに入れ、蓋を固定します。バロサイクルは45,000 psiで50秒間、大気圧は50°Cで10秒間、60サイクルです。
- バロサイクルが完了したら、マイクロチューブを0.5 mLの微量遠心チューブに移し、4,000 × gで2分間遠心分離 します。
- キャップツールを使用して、0.5 mL の微量遠心チューブから MicroTube を取り外します。キャップツールを使用して乳棒を慎重に取り外し、乳棒の下半分を20 μLの液体クロマトグラフィー質量分析(LC-MS)グレードの水ですすぎ、洗浄液を清潔な0.5 mLの微量遠心チューブに集めます。
- ベンチ上部のMicroTubeを静かに叩いて液体を底に移動し、すべての溶液をMicroTubeから0.5mLの微量遠心チューブに移します。
- 20 μL の LC-MS グレードの水を MicroTube に加え、ベンチトップで軽く叩きます。洗浄液を0.5mLチューブに移し、この洗浄工程をもう一度繰り返す。
- 真空遠心分離機でサンプルを〜2 μLまで乾燥させ、100 μLの100 mM TEAB、pH 8.0を加えます。
- 比色アッセイ(ビシンコニン酸(BCA)アッセイ; 材料表を参照)を用いてペプチド濃度を決定し、製造業者のプロトコールに従っている。
8. タンデムマスタグ(TMT)ラベリングとイージーペップクリーンアップ
注:おおよその時間:7時間20分(真空遠心分離機乾燥時間なしで2時間20分)。
- アイソバリックTMT標識試薬を周囲温度にしてから開封してください。各TMTバイアル(5mg)に500μLの100%アセトニトリルを加える。時折ボルテックスで10分間インキュベートします。
- 5 μg のペプチドサンプルを 100 μL の 100 mM TEAB (pH 8.0) に溶解し、所定の TMT 試薬を 10 μL 加えます。複数のTMT多重化9にわたる試料の定量化を容易にするために、実験中の各TMT多重化セットにおける各個々の試料を表す参照プールを構築し、含める。周囲温度で反応を1時間インキュベートし、時折振盪/タッピングします。
- 5%ヒドロキシルアミンを10μL添加してTMT標識反応をクエンチし、時折タッピングしながら周囲温度で30分間インキュベートする。クエンチ後、TMT標識サンプルを1本のチューブに結合し、約200μLまで乾燥させます。
- 1,800 μLの0.1%ギ酸を加える。pHペーパーでpHを確認してください:pH〜3の場合は、1mLの0.1%ギ酸を加えます。pHが>3の場合は、pH〜3になるまで5%ギ酸を10〜20μL添加する。0.1%ギ酸を加えて、3mLの最終容量にする。
- ペプチドクリーンアップカラムの下部にあるタブを取り外し、キャップを外して、15 mL の円錐形チューブに入れます。TMT 標識サンプルをカラムに移し、製造元のプロトコルに従ってクリーンアップを続行します。
- 真空遠心分離機で溶出したペプチドを〜20 μLまでドライダウンし、最終容量80 μLの25 mM炭酸水素アンモニウムを使用してLCバイアルに移し、オフライン分画に進みます。
9. TMTマルチプレックスサンプル分画とプーリング
注:おおよその時間:3時間30分(真空遠心分離機乾燥時間なしで1時間30分)。
- 移動相B(アセトニトリル)の直線勾配(0.69% min-1)を移動相A(10 mMNH4HCO3、pH 8.0)に展開することにより、塩基性逆相クロマトグラフィーによりTMT標識ペプチド多重化を96画分に分画します。
- サンプルウェルをプールして36個の連結画分を生成します。真空遠心分離機で乾燥画分を約2 μLにし、25 mM 炭酸水素アンモニウム (終濃度 1.5 μg/10 μL) に再懸濁し、15,000 × g で 10 ~ 15 分間遠心分離し、MS 分析のために LC バイアルに移します。
10. 液体クロマトグラフィータンデム質量分析(LC-MS/MS)
注:おおよその時間:機器の方法と実験計画によって異なります。
- 質量分析計は、製造元の指示/プロトコルに従って校正します。
- 新鮮な移動相および標準を準備し、適切なLCプレラン調製(参照された機器の溶媒のパージ、空気のフラッシュ、およびリークテストスクリプトを含むがこれらに限定されない)を実行します[ 材料表を参照])。分析を開始する前に、分析前および分析前のカラムとサンプルループを平衡化します。
- シリアルTMTマルチプレックス分析の前および間に、LC-MSシステムが品質保証/品質管理(QA/QC)TMTM標識ペプチドダイジェストおよび(例:MSPE( 材料表を参照)、HeLa)を使用して、以前にベンチマークされたパフォーマンスメトリックを満たしていることを検証します。
- オートサンプラーバイアルをLCオートサンプラーの適切な位置にロードします。適切な勾配/ MS法で個々の画分を分析します。ペプチド標準物質(例えば、ペプチド保持時間較正[PRTC])で「洗浄」ランを散りばめ、クロマトグラフィーおよびマススペクトル性能を評価するために、約1日1回行う。各TMTマルチプレックスサンプルフラクションシリーズの分析に続いて、QA/QC TMTベンチマーク標準を実行してシステム性能を評価します。
- QA/QC TMTベンチマーク標準の後に質量分析計の評価ルーチンを実行してサンプル後の性能を評価し、次のサンプルセットのステップ10.1のようにシステムを校正します。
11. バイオインフォマティクスデータ解析
注: おおよその時間: 実験計画によって異なります。
- すべてのサンプルデータ(.rawファイルなど)を適切なネットワークストレージ/コンピュータドライブに転送します。
- 種特異的タンパク質参照データベースに対して適切なパラメータ9 を使用して、所望のデータ分析アプリケーション(例えば、プロテオームディスカバー、マスコット)を使用してすべての画分を一緒に検索し、ペプチドスペクトルマッチ(PSM)を生成し、TMTレポーターイオンシグナル強度を抽出する。適切な品質管理メトリックに基づいてPSMをフィルタリングし、前述のように正規化された中央値log2変換TMTレポーターイオン比存在量をグローバルタンパク質レベル存在量に集約する3、9。
- 目的の差動分析ソフトウェアを使用して、目的の条件下でのタンパク質の変化を比較します。
結果
2人のHGSOC患者と2人のOCCC患者からの新鮮凍結組織薄切片を、この統合されたAI駆動組織ROI識別、セグメンテーション、LMD、および定量的プロテオーム分析ワークフローを使用して分析しました(図1)。各腫瘍の代表的なH&E染色組織切片を、理事会認定病理学者によってレビューした。腫瘍細胞性は70%〜99%の範囲であった。組織はPENメンブレンスライド(補足ファイル2)に薄切片化され、キャリブレータ基準(補足ファイル1)でプレカットされ、画像解析ソフトウェア( 材料表を参照)で生成された注釈からの位置方向データをLMDソフトウェアのデカルト座標方向と統合することができました。H&E染色に続いて、組織とキャリブレーターを含むPENスライドの高解像度画像(20x)をキャプチャしました。
顕微鏡写真中の腫瘍および間質細胞集団を、LMDによる選択的収穫のために画像解析ソフトウェア(材料表を参照)を用いてセグメント化し、組織薄切片全体(例えば、腫瘍組織全体)を表す収穫物と共にセグメント化した(図1)。組織切片全体を500μm2のタイルで区画し、タイル間に40μmの隙間を残してPEN膜の完全性を維持し、LMD中の膜のカールを防止することによって、腫瘍組織コレクション全体に対する無差別注釈を生成した。組織学で解決されたLMD濃縮のためのスライドでは、画像解析ソフトウェアのAI分類器(材料表を参照)が、空白のスライドガラスの背景とともに、腫瘍細胞と間質細胞を区別するように訓練されました。代表的な腫瘍、間質、およびブランクガラス領域を手動で強調表示し、分類器ツールを使用して、組織切片全体にわたってこれらのROIをセグメント化しました。組織全体、腫瘍上皮、および間質を表すセグメント化された層は、個々の.annotationファイル(補足ファイル3および補足ファイル6)として別々に保存された。イメージ ファイルの別のコピー (パーティション分割された ROI 注釈なし) では、3 つの基準キャリブレータのそれぞれの中心端からの短い行に注釈が付けられ、各 LMD 注釈レイヤー ファイルと同じファイル名を使用して .annotation ファイルとして保存されますが、サフィックス "_calib" が付加されました (補足ファイル 4)。これらの線を使用して、PEN膜校正器の位置を画像解析ソフトウェアで描画されたアノテーション形状リストデータと共登録しました。
本研究は、このAI駆動型のLMDワークフローをサポートするために、Pythonの「Malleator」と「Dapọ」の2つのアルゴリズムを提供しており、https://github.com/GYNCOE/Mitchell.et.al.2022 で利用可能である。Malleator アルゴリズムは、ペアの .annotation ファイルからすべての個々のアノテーション (組織の ROI とキャリブレーター) の特定のデカルト座標を抽出し、これらを 1 つの拡張マークアップ言語 (XML) インポート ファイル (補足ファイル 5) にマージします。具体的には、Malleator アルゴリズムは、親フォルダーのディレクトリ名を入力として使用してすべてのサブディレクトリフォルダーを検索し、.xmlマージされたファイルをまだ持たないサブフォルダーの.xmlファイルを生成します。Malleatorアルゴリズムは、画像解析ソフトウェア( 材料表を参照)内のすべてのアノテーションレイヤーを単一のレイヤーにマージし、独自の.annotationファイルタイプとして保存されるAI生成の形状リストデータをLMDソフトウェアと互換性のある.xml形式に変換します。アノテーションファイルとキャリブレータファイルをマージした後、アルゴリズムによって生成された.xmlファイルが保存され、LMDソフトウェアにインポートされます。アノテーションのアライメントを手動で調整するにはわずかな調整が必要であり、レーザー顕微鏡のスライドステージの垂直(z平面)位置を登録するのにも役立ちます。Dapọ アルゴリズムは、LMD で強化されたコレクションに特に使用されます。パーティション分割されたタイルは、画像解析ソフトウェアによって個々のアノテーション レイヤーに自動的に割り当てられます。Dapọ アルゴリズムは、分類子ツールを使用する前に、パーティション分割されたすべてのタイルを 1 つの注釈レイヤーにマージするため、LMD エンリッチド コレクションの分類子分析の実行時間が短縮されます。
腫瘍全体およびLMD富化組織サンプルを消化し、TMT試薬で標識し、多重化し、オフラインで分画し、前述のように定量的MSベースのプロテオミクスを介して分析した9。このAI駆動ワークフローを使用して採取したサンプルの平均ペプチド収量(43-60 μg)および回収率(0.46-0.59 μg/mm2)は、以前の報告9,10と同等でした。合計5,971個のタンパク質を全サンプルにわたって共定量した(補足表S1)。100の最も可変的なタンパク質を用いた教師なし階層クラスタリングは、前述のものと同様に、LMD富化および腫瘍全体サンプルからのHGSOCおよびOCCCヒストタイプの分離をもたらした(図2A)。対照的に、HGSOCおよびOCCCの両方からのLMD富化間質サンプルは、LMD富化腫瘍および腫瘍全体サンプルから独立してクラスタリングされた。5,971の定量タンパク質のうち、215はHGSOC検体からの腫瘍コレクション全体とOCCC標本の間で有意に変化(LIMMA調整後p<0.05)した(補足表S2)。これらの改変されたタンパク質を、Hughes et al.11によってHGSOCおよびOCCC腫瘍組織を区別するために同定されたタンパク質と比較した。Hughesらによって定量された76のシグネチャタンパク質のうち、57は、このデータセットで共定量され、高度に相関していた(Spearman Rho = 0.644, p < 0.001)(図2B)。
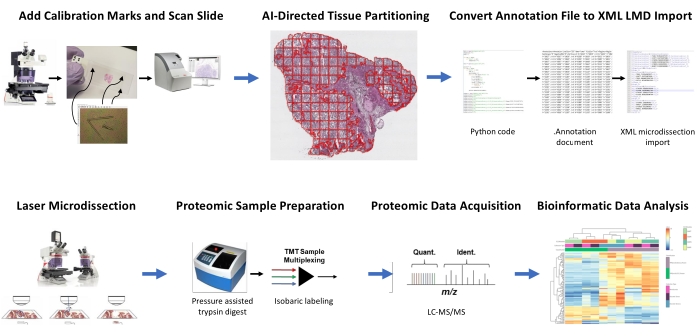
図1:下流の定量的プロテオミクスのためのレーザーマイクロダイセクションのための自動組織関心領域選択のための統合ワークフローの要約。 校正基準はPENメンブレンスライドに切断され、組織ROIのAI由来セグメントからの位置配向データを画像解析ソフトウェアHALOに同時登録し、LMD顕微鏡上の水平位置決めを行います。Malleator アルゴリズムは、スライドのすべてのアノテーションレイヤーにわたるアノテーションセグメンテーションデータを_calib参照ファイルとマージし、LMD ソフトウェアと互換性のある.xmlファイルに変換するために使用されます。プロテオーム分析のためにLMD採取された組織は、先に記載したようにハイスループット定量的プロテオミクスによって消化および分析される9。略語:LMD =レーザーマイクロダイセクション;ROI = 関心領域;TMT = タンデム質量タグ;定量 = 定量化;識別する。= 識別;LC-MS/MS = 液体クロマトグラフィー - タンデム質量分析。 この図の拡大版を表示するには、ここをクリックしてください。
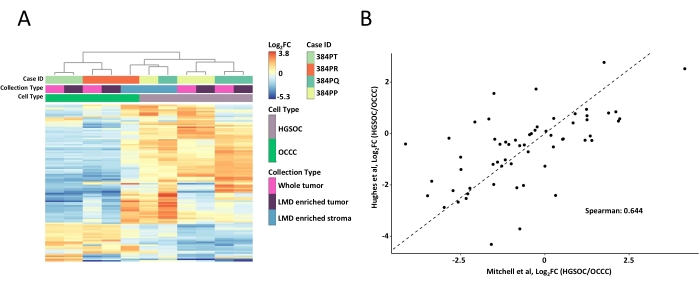
(A) HGSOCおよびOCCC LMD濃縮および腫瘍全体サンプル中の最も変動的に豊富な100のタンパク質の教師なし階層クラスター分析。(B)本研究におけるHGSOCとOCCC全腫瘍収穫物との間のlog2フォールド変化タンパク質存在量の相関(Mitchell et al.、x軸)およびHughesらによる同様の研究(y軸)11。略語:LMD =レーザーマイクロダイセクション;HGSOC = 高悪性度漿液性卵巣癌;OCCC = 卵巣明細胞癌;log2 FC = log2 形質転換プロテオミクス存在量。この図の拡大版を表示するには、ここをクリックしてください。
補足表S1:HGSOCおよびOCCC組織標本からのすべてのLMD濃縮および腫瘍全体サンプルにわたって同時定量された5,971タンパク質の存在量。 略語:LMD =レーザーマイクロダイセクション;HGSOC = 高悪性度漿液性卵巣癌;OCCC = 卵巣明細胞癌。 この表をダウンロードするには、ここをクリックしてください。
補足表S2:HGSOC対OCCCからの腫瘍コレクション全体における発現差のあるタンパク質(215)(LIMMA調整後p<0.05)。 略語: HGSOC = 高悪性度漿液性卵巣癌;OCCC = 卵巣明細胞癌。 この表をダウンロードするには、ここをクリックしてください。
補足ファイル 1: 4 つのスライド位置の標準キャリブレータ基準を含む代表的な形状リスト データ (.sld) ファイル。 このファイルはLMDソフトウェアにインポートできます。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル2:H&E染色された高解像度(20x)組織切片の代表的な.svs画像ファイル。 ファイルは、画像解析ソフトウェアまたはLMDソフトウェアを使用して開いて表示することができます。略語:H&E=ヘマトキシリンおよびエオジン;LMD = レーザーマイクロダイセクション。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル3:パーティション分割された腫瘍セグメント全体の代表的な.annotationファイル。 このファイルは、画像解析ソフトウェアにインポートできます。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 4: キャリブレータ基準セグメントの代表的な _calib.annotation ファイル。 座標情報は、各矢印基準から引かれた短いキャリブレータ線の方向位置を表す。このファイルは、画像解析ソフトウェアにインポートできます。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 5: Malleator アルゴリズムによって生成された代表的な拡張マークアップ言語 (.xml) ファイル。 このファイルは、レーザーマイクロダイセクションソフトウェアにインポートできます。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 6: LMD 強化コレクションのパーティション分割された AI 分類セグメントの代表的な .annotation ファイル。 このファイルは、画像解析ソフトウェアにインポートできます。略語: AI = 人工知能;LMD = レーザーマイクロダイセクション。 このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
FFPEおよび/または新鮮凍結組織からの標的細胞亜集団の濃縮のためのワークフローおよび9,12,13,14,15の処理中にサンプル品質を維持するための方法論を開発および/または改善することを目的とした複数の研究先例がありましたが、変動性を低減するために分子分析用の臨床組織標本を調製するための自動化された戦略を開発することがかなり必要であり、 再現性を高めます。このワークフローでは、LMDによる臨床組織標本からの離散的な細胞集団の組織学的解決回収のための既存の画像解析ソフトウェアツール(材料表を参照)を統合する標準化された半自動化プロトコルについて説明します。
個別の細胞集団を捕捉するROIの空間分解LMD濃縮は、分子特性評価と同定を改善し、細胞選択的バイオマーカーの発見を容易にするためのマルチオミック解析前の次世代組織処理ステップを表します。このプロトコルは、組織学者によるROIの手動セグメンテーションに関連する周囲環境への組織切片のしばしば長時間の曝露を低減することによって、既存の方法論を改善する(LMD収集の前に>1〜2時間かかることがある)。このワークフローにより、AIガイドによる分類とセグメンテーションによってROIを事前に識別できます。組織の滞留時間を制限することで、リンペプチドやmRNAなどの非常に不安定な分子標的の評価や、検出のために標的タンパク質が天然の立体構造にあることに依存する抗体ベースの分析技術の評価における偽の変動が減少します。
スキャンしたスライド画像にはっきりと見えるきれいなキャリブレータ基準をPENメンブレンスライドに切断することは、画像解析ソフトウェア( 材料表を参照)をLMDワークフローと統合することを可能にする重要な要素の1つです。キャリブレータが「V」形状の下部に正確な(「クリーン」)点を持つようにすることで、ステップ5.1.6および5.2.13で説明されているように、キャリブレータの線を描画する正確な点を画像解析ソフトウェアで選択できます。LMDソフトウェアへのインポート中にこれらの点をアライメントすることは、アノテーション(「マルレータ」および/または「Dapọ」アルゴリズムを使用して互換性のある.xmlファイルを生成することによって促進される)を物理的なLMDスライド上の関連する組織ROIに適切にオーバーレイするために重要です。レーザー顕微鏡上のスライドステージの垂直(z面)位置を登録するには、LMDソフトウェアへのインポート時にアライメントが正確であっても、すべての形状を強調表示し、集合的に所定の位置に「ドラッグアンドドロップ」する必要があります。必要に応じて、組織ROIに対するアノテーションの位置決めを、このステップ中に行うこともできます。
Malleatorアルゴリズムの現在のバージョンの制限は、画像解析ソフトウェアによって提供される事前定義された注釈形状ツール( 材料表を参照)と互換性がないことですが、アルゴリズムの将来の更新/バージョンは、この互換性を向上させることを目的としています。これらのツールを使用して描画された図形の .annotation ファイルには、各アノテーションのペアの x 座標と y 座標が 2 セットしか含まれておらず、これらのポイントの周囲の完全な空間方向はありません。これらのツールを現在使用していると、インポート処理中にアノテーションが 2 点のみで定義された直線に変換されます。組織ROIセグメントの手動定義は、XML形式への変換とLMDインポートを成功させるために必要です。これは、ターゲット領域に固有の個々のフリーハンドポリゴンアノテーションを使用して各ROIを手動で定義するか、または必要に応じて、すべての組織ROIセグメントにわたって近似円形または長方形アノテーションを適用することによって実行することができ、このワークフローと互換性があります。
ここで紹介したワークフローは、新鮮凍結ヒトがん組織標本のプロテオーム解析のために実証されましたが、このAI駆動LMDワークフローは、FFPE組織、非がん組織タイプ、および非ヒトソースからの組織と同等に使用できます。また、トランスクリプトーム解析、ゲノム解析、ホスホプロテオミクス解析など、他の下流の分子プロファイリングワークフローもサポートできます。このワークフローは、細胞計数に関連する機能や、「マルチプレックスIHC」モジュールや「組織マイクロアレイ(TMA)アドオン」などの分析モジュールに関連する機能を含む、画像解析ソフトウェアの他の用途( 材料表を参照)を活用することもできます。このワークフローの将来のアプリケーションは、ROIセグメントあたりの細胞数を事前に定義して、それによって複数のコレクションにわたって同等の細胞入力を保証すること、または免疫組織化学または細胞社会学などによって、関心のある細胞ROIを定義するために代替方法を使用することによっても利益を得る可能性がある。
開示事項
T.P.C. はサーモフィッシャーサイエンティフィック社の SAB メンバーであり、AbbVie から研究資金を受けています。
謝辞
このプロジェクトの資金の一部は、防衛保健プログラム(HU0001-16-2-0006およびHU0001-16-2-00014)によって、婦人科がんセンターオブエクセレンスのためのユニフォームドサービス大学に提供されました。スポンサーは、研究の設計、実行、解釈、または執筆において何の役割も持っていませんでした。 免責事項: ここに表明された見解は著者のものであり、陸軍/海軍/空軍、国防総省、または米国政府の公式方針を反映していません。
資料
| Name | Company | Catalog Number | Comments |
| 1260 Infinity II System | Agilent Technologies Inc | Offline LC system | |
| 96 MicroCaps (150uL) in bulk | Pressure Biosciences Inc | MC150-96 | |
| 96 MicroPestles in bulk | Pressure Biosciences Inc | MP-96 | |
| 96 MicroTubes in bulk (no caps) | Pressure Biosciences Inc | MT-96 | |
| 9mm MS Certified Clear Screw Thread Kits | Fisher Scientific | 03-060-058 | Sample vial for offline LC frationation and mass spectrometry |
| Acetonitrile, Optima LC/MS Grade | Fisher Chemical | A995-4 | Mobile phase solvent |
| Aperio AT2 | Leica Microsystems | 23AT2100 | Slide scanner |
| Axygen PCR Tubes with 0.5 mL Flat Cap | Fisher Scientific | 14-222-292 | Sample tubes; size fits PCT tubes and thermocycler |
| Barocycler 2320EXT | Pressure Biosciences Inc | 2320-EXT | Barocycler |
| BCA Protein Assay Kit | Fisher Scientific | P123225 | |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | |
| Easy-nLC 1200 | Thermo Fisher Scientific | Liquid Chromatography | |
| EasyPep Maxi Sample Prep Kit | Thermo Fisher Scientific | NCI5734 | Post-label sample clean up column |
| EASY-SPRAY C18 2UM 50CM X 75 | Fisher Scientific | ES903 | Analytical column |
| Eosin Y Solution Aqueous | Sigma Aldrich | HT110216 | |
| Formic Acid, 99+ % | Thermo Fisher Scientific | 28905 | Mobile phase additive |
| ggplot2 version 3.3.5 | CRAN | https://cran.r-project.org/web/packages/ggplot2/ | |
| HALO | Indica Labs | Image analysis software | |
| IDLE (Integrated Development and Learning Environment) | Python Software Foundation | ||
| iheatmapr version 0.5.1 | CRAN | https://cran.r-project.org/web/packages/iheatmapr/ | |
| iRT Kit | Biognosys | Ki-3002-1 | LC-MS QAQC Standard |
| limma version 3.42.2 | Bioconductor | https://bioconductor.org/packages/release/bioc/html/limma.html | |
| LMD Scanning stage Ultra LMT350 | Leica Microsystems | 11888453 | LMD stage model outfitted with PCT tube holder |
| LMD7 (software version 8.2.3.7603) | Leica Microsystems | LMD apparatus (microscope, laser, camera, PC, tablet) | |
| Mascot Server | Matrix Science | Data analysis software | |
| Mass Spec-Compatible Human Protein Extract, Digest | Promega | V6951 | LC-MS QAQC Standard |
| Mayer’s Hematoxylin Solution | Sigma Aldrich | MHS32 | |
| PEN Membrane Glass Slides | Leica Microsystems | 11532918 | |
| Peptide Retention Time Calibration Mixture | Thermo Fisher Scientific | 88321 | LC-MS QAQC Standard |
| Phosphatase Inhibitor Cocktail 2 | Sigma Aldrich | P5726 | |
| Phosphatase Inhibitor Cocktail 3 | Sigma Aldrich | P0044 | |
| Pierce LTQ Velos ESI Positive Ion Calibration Solution | Thermo Fisher Scientific | 88323 | Instrument calibration solution |
| PM100 C18 3UM 75UMX20MM NV 2PK | Fisher Scientific | 164535 | Pre-column |
| Proteome Discoverer | Thermo Fisher Scientific | OPTON-31040 | Data analysis software |
| Python | Python Software Foundation | ||
| Q Exactive HF-X | Thermo Fisher Scientific | Mass spectrometer | |
| R version 3.6.0 | CRAN | https://cran-archive.r-project.org/bin/windows/base/old/2.6.2/ | |
| RColorBrewer version 1.1-2 | CRAN | https://cran.r-project.org/web/packages/RColorBrewer/ | |
| Soluble Smart Digest Kit | Thermo Fisher Scientific | 3251711 | Digestion reagent |
| TMTpro 16plex Label Reagent Set | Thermo Fisher Scientific | A44520 | isobaric TMT labeling reagents |
| Veriti 60 well thermal cycler | Applied Biosystems | 4384638 | Thermocycler |
| Water, Optima LC/MS Grade | Fisher Chemical | W6-4 | Mobile phase solvent |
| ZORBAX Extend 300 C18, 2.1 x 12.5 mm, 5 µm, guard cartridge (ZGC) | Agilent Technologies Inc | 821125-932 | Offline LC trap column |
| ZORBAX Extend 300 C18, 2.1 x 150 mm, 3.5 µm | Agilent Technologies Inc | 763750-902 | Offline LC analytical column |
参考文献
- Motohara, T., et al. An evolving story of the metastatic voyage of ovarian cancer cells: cellular and molecular orchestration of the adipose-rich metastatic microenvironment. Oncogene. 38 (16), 2885-2898 (2019).
- Aran, D., Sirota, M., Butte, A. J. Systematic pan-cancer analysis of tumour purity. Nature Communications. 6, 8971 (2015).
- Hunt, A. L., et al. Extensive three-dimensional intratumor proteomic heterogeneity revealed by multiregion sampling in high-grade serous ovarian tumor specimens. iScience. 24 (7), 102757 (2021).
- Dou, Y., et al. Proteogenomic characterization of endometrial carcinoma. Cell. 180 (4), 729-748 (2020).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Gillette, M. A., et al. Proteogenomic characterization reveals therapeutic vulnerabilities in lung adenocarcinoma. Cell. 182 (1), 200-225 (2020).
- Silvestri, A., et al. Protein pathway biomarker analysis of human cancer reveals requirement for upfront cellular-enrichment processing. Laboratory Investigation. 90 (5), 787-796 (2010).
- Echle, A., et al. Deep learning in cancer pathology: a new generation of clinical biomarkers. British Journal of Cancer. 124 (4), 686-696 (2021).
- Lee, S., et al. Molecular analysis of clinically defined subsets of high-grade serous ovarian cancer. Cell Reports. 31 (2), 107502 (2020).
- Xuan, Y., et al. Standardization and harmonization of distributed multi-center proteotype analysis supporting precision medicine studies. Nature Communications. 11 (1), 5248 (2020).
- Hughes, C. S., et al. Quantitative profiling of single formalin fixed tumour sections: proteomics for translational research. Scientific Reports. 6 (1), 34949 (2016).
- Espina, V., et al. A portrait of tissue phosphoprotein stability in the clinical tissue procurement process. Molecular & Cellular Proteomics. 7 (10), 1998-2018 (2008).
- Espina, V., Heiby, M., Pierobon, M., Liotta, L. A. Laser capture microdissection technology. Expert Review of Molecular Diagnostics. 7 (5), 647-657 (2007).
- Havnar, C. A., et al. Automated dissection protocol for tumor enrichment in low tumor content tissues. Journal of Visualized Experiments. (169), e62394 (2021).
- Mueller, C., et al. One-step preservation of phosphoproteins and tissue morphology at room temperature for diagnostic and research specimens. PLoS One. 6 (8), (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved