このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
アデノ随伴ウイルス(AAV)キャプシド変異体のエンジニアリング、バーコード化、スクリーニングによる次世代遺伝子治療ベクターの単離(英語)
要約
次世代AAVの作成のための新規特性を持つ候補のバーコード化によるAAVペプチドディスプレイライブラリの生成とその後の検証。
要約
アデノ随伴ウイルス(AAV)に由来する遺伝子送達ベクターは、遺伝病の治療のための最も有望なツールの1つであり、有望な臨床データといくつかのAAV遺伝子治療の承認によって証明されています。AAVベクターの成功の2つの主な理由は、(i)異なる特性を持つさまざまな天然に存在するウイルス血清型の事前分離、および(ii)その後の分子工学とハイスループットでの転用のための強力な技術の確立です。これらの技術の可能性をさらに高めるために、最近、選択されたAAVカプシドをDNAおよびRNAレベルでバーコード化するための戦略が実装され、単一の動物のすべての主要な臓器および細胞タイプで包括的かつ並行したin vivo層別化が可能になります。ここでは、AAVペプチドディスプレイを使用して、利用可能なカプシドエンジニアリング技術の多様な武器を表す、この一連の補完的な手段を網羅する基本的なパイプラインを紹介します。したがって、まず、所望の特性を有する候補のin vivo選択のためのAAVペプチドディスプレイライブラリを生成するための極めて重要なステップを説明し、続いて、二次in vivoスクリーニングのために最も興味深いカプシド変異体をバーコード化する方法のデモンストレーションを行います。次に、バーコード増幅やアダプターライゲーションを含む次世代シーケンシング(NGS)用のライブラリ作成の方法論を例示し、NGSデータ解析中の最も重要なステップの概要で締めくくります。ここで報告されているプロトコルは汎用性と適応性があるため、研究者はそれらを簡単に利用して、お気に入りの疾患モデルや遺伝子治療アプリケーションに最適なAAVキャプシドバリアントを濃縮できます。
概要
遺伝子導入療法は、細胞に遺伝物質を導入して、細胞の遺伝物質を修復、置換、または変更して、病気を予防、治療、治癒、または改善することです。遺伝子導入は、インビボとエクスビボの両方で、非ウイルス性とウイルス性の異なる送達システムに依存しています。ウイルスは、標的細胞を効率的に形質導入するために自然に進化しており、送達ベクターとして使用できます。遺伝子治療に使用されるさまざまな種類のウイルスベクターの中で、アデノ随伴ウイルスは、病原性の欠如、安全性、免疫原性の低下、そして最も重要なことに、長期の非組み込み型発現を維持する能力のためにますます使用されています1,2,3。AAV遺伝子治療は、過去10年間でかなりの成果を上げてきました。3つの治療法が、欧州医薬品庁と米国食品医薬品局によってヒトでの使用が承認されています3,4。他の場所でレビューされているように、血友病、筋肉、心臓、神経疾患などのさまざまな疾患を治療するためのいくつかの臨床試験も進行中です3。何十年にもわたる進歩にもかかわらず、遺伝子治療の分野は近年一連の挫折を経験しており4、最も重要なのは、特に筋肉などの巨大な組織、または脳などの到達が困難な組織の場合、用量制限毒性のために保留された臨床試験5での死亡6です。
現在臨床試験で使用されているAAVベクターは、いくつかの例外を除いて天然血清型に属しています1。AAVエンジニアリングは、優れた臓器または細胞特異性と効率を備えたベクターを開発する機会を提供します。過去20年間で、ペプチドディスプレイ、ループスワップ、カプシドDNAシャッフリング、エラー発生型PCR、ターゲットデザインなど、いくつかのアプローチが成功裏に適用され、多様な特性を持つ個々のAAVバリアントまたはそのライブラリが生成されました7。次に、これらは、他の場所でレビューされているように、目的の特性を持つバリアントを選択するために、指向性進化の複数のラウンドにかけられます1,3。すべてのカプシド進化戦略の中で、ペプチドディスプレイAAVライブラリは、比較的簡単に生成でき、高い多様性とハイスループットシーケンシングを達成できるため、進化を追跡できるといういくつかのユニークな特性により、最も広く使用されています。
最初に成功したペプチド挿入AAVライブラリは、ほぼ20年前に記述されました。最初の1つでは、Peraboら8 は、ランダムに生成されたオリゴヌクレオチドのプールが、カプシドから突き出た3倍軸のVP1キャプシドタンパク質のアミノ酸587に対応する位置のプラスミドに挿入された修飾AAV2キャプシドのライブラリを構築しました。アデノウイルスの共感染を用いて、AAVライブラリーを複数回の選択を通じて進化させ、最終的な再標的変異体は、親のAAV28に難治性の細胞株を形質導入できることが示された。その後まもなく、Müllerら9 はライブラリ作成のための2段階システムを導入し、プロトコルを大幅に改善しました。最初に、プラスミドライブラリは、アデノウイルスヘルパープラスミドとともに、キメラカプシドを含むAAVライブラリを生成するために使用されます。このAAVシャトルライブラリは、細胞ごとに1つのウイルスゲノムを導入することを目的として、感染多重度(MOI)の低い細胞に感染するために使用されます。アデノウイルスとの同時感染により、ゲノムとキャプシド9が一致するAAVの産生が保証されます。約10年後、ダルカラ10 は in vivo 指向進化を使用して7m8バリアントを作成しました。この変異体は、10アミノ酸挿入(LALGETTRPA)を有し、そのうち3つはリンカーとして作用し、硝子体内注射後の外網膜を効率的に標的とする10。このエンジニアリングキャプシドは、これまでにクリニックにたどり着いた数少ないエンジニアリングキャプシドの1つであるため、並外れたサクセスストーリーです11。
この分野は、次世代シーケンシング(NGS)技術の導入により、2番目の後押しを経験しました。2014年のAdachi et al.12と2015年のMarsic et al.13の2つの出版物は、バーコード化されたAAVキャプシドライブラリの分布を高精度で追跡するNGSの力を紹介しました。数年後、バーコード領域のNGSは、カプシドの進化に従うようにペプチド挿入領域に適応されました。Körbelinら14は、肺を標的とするAAV2ベースのキャプシドを同定するためにNGSガイド下スクリーニングを実施した。NGS分析は、選択ラウンド間の濃縮スコア、組織特異性を決定するための一般的な特異性スコア、そして最後に合計スコア14の3つの評価スコアを計算するのに役立ちました。Gradinaruラボ15は、細胞タイプ特異的な選択を容易にするCre組換えベースのAAVターゲット進化(CREATE)システムを同年に発表しました。このシステムでは、ポリA信号が2つのloxPサイトに隣接しているため、カプシドライブラリはCre反転可能スイッチを備えています。次に、AAVライブラリをCreマウスに注入し、ポリAシグナルがCre+細胞でのみ反転し、リバースPCRプライマーとキャプシド遺伝子内のフォワードプライマーを結合させるためのテンプレートを提供します。この非常に特異的なPCRレスキューにより、AAV-PHPの同定が可能になりました。血液脳関門15を通過できるB変異体。このシステムはさらにM-CREATE(Multiplexed-CREATE)に進化し、NGSと合成ライブラリ生成がパイプライン16に統合されました。
マグワイアラボ17のこのシステムの改良されたRNAベースのバージョンであるiTransduceは、細胞を機能的に形質導入し、そのゲノムを発現するカプシドのDNAレベルでの選択を可能にします。ペプチドディスプレイライブラリーのウイルスゲノムは、ユビキタスプロモーターの制御下にあるCre遺伝子と、p41プロモーターの制御下にあるカプシド遺伝子とを含む。このライブラリーは、tdTomatoの上流にloxP-STOP-loxPカセットを有するマウスに注入される。ウイルスゲノムを発現し、したがってCreがtdTomatoを発現するAAV変異体で形質導入された細胞は、細胞マーカーと組み合わせて、選別および選択することができる17。同様に、Nonnenmacher et al.18およびTabebordbarら19は、カプシド遺伝子ライブラリーを組織特異的プロモーターの制御下に置いた。異なる動物モデルに注射した後、ウイルスRNAを使用してカプシド変異体を単離した。
別の方法は、バーコードを使用してcapsidライブラリにタグを付けることです。ビョルクルンドラボ20 は、このアプローチを使用してペプチド挿入キャプシドライブラリをバーコード化し、バーコード化された合理的なAAVベクトル進化(BRAVE)を開発しました。1つのプラスミドでは、Rep2Capカセットを、倒立末端リピート(ITR)に隣接する黄色蛍光タンパク質(YFP)発現、バーコードタグ付き導入遺伝子の隣にクローニングします。 キャップ の端とバーコードの先頭の間のloxPサイトを使用して、 in vitro Cre組換えにより、NGSに十分小さいフラグメントが生成され、それによってペプチド挿入と固有のバーコード(ルックアップテーブル、LUT)との関連付けが可能になります。AAV産生はプラスミドライブラリを用いて行われ、mRNAで発現されたバーコードは、 in vivo 適用後に再びNGS20でスクリーニングされる。カプシドライブラリがキャプシド遺伝子全体のバリアント(すなわち、シャッフルされたライブラリ)を含む場合、ロングリードシーケンシングを使用する必要があります。いくつかのグループは、バーコードを使用してこれらの多様なライブラリにタグを付け、より高い読み取り深度でNGSを可能にしています。Kay lab21 は、 キャップ ポリA信号の下流にバーコードを使用して、非常に多様なキャプシドシャッフルライブラリにタグを付けました。最初のステップでは、バーコード化されたプラスミドライブラリが生成され、シャッフルされたカプシド遺伝子ライブラリがその中にクローニングされました。次に、MiSeq(ショートリード、より高い読み取り深度)とPacBio(ロングリード、より低い読み取り深度)のNGSとサンガーシーケンシングの組み合わせを使用して、LUT21を生成しました。2019年、チャーチラボ22 のオグデンと同僚は、すべての位置に一点変異、挿入、欠失を持つライブラリを使用して、複数の機能に対するAAV2キャプシド適合性を描写し、最終的に機械誘導設計を可能にしました。ライブラリの生成のために、カプシド遺伝子のより小さな断片を合成し、バーコードでタグ付けし、次世代配列を決定し、次に完全なカプシド遺伝子にクローニングしました。NGSデータはLUTを生成するために使用されました。次に、バーコードとショートリードシーケンスのみを使用してライブラリをスクリーニングし、より高い読み取り深度22を可能にしました。
バーコードライブラリは、カプシドライブラリを数回選択した後、またはカプシド進化研究とは無関係に、既知の天然および工学的バリアントのプールをスクリーニングするために主に使用されてきました。このようなライブラリの利点は、動物の数を減らし、動物間の変動を最小限に抑えながら、複数のカプシドをスクリーニングする機会です。この技術をAAV分野に導入した最初の研究は、ほぼ10年前に発表されました。中井研究室12は、AAV9のVP1上のアミノ酸356〜736をカバーする191個の二重アラニン変異体を12 ヌクレオチドのバーコードでタグ付けした。NGSを用いて、ライブラリーを、ガラクトース結合および他の特性について インビボ でスクリーニングした12。Marsicらは、1年後の二重バーコード分析も使用して、AAV変異体の生体分布を描写した13。非ヒト霊長類を対象としたより最近の研究では、異なる送達経路を使用して29個のカプシドの中枢神経系における生体分布を比較しました23。私たちの研究室は最近、天然および人工AAVを含む183のバリアントのバーコードAAVライブラリ画面を公開しました。DNAおよびRNAレベルでのこれらのスクリーニングは、マウスにおける高筋向性AAV変異体24ならびにマウス脳において高い細胞型特異性を示す他のもの25の同定につながった。
ここでは、この研究で使用された方法論について説明し、AAVペプチドディスプレイライブラリのスクリーニングを含むように拡張します。これには、AAV2ペプチドディスプレイライブラリの生成、定量のためのデジタルドロップレットPCR(dd-PCR)法、そして最後にAAV変異体を分析するためのNGSパイプラインが含まれ、Weinmannらの研究に一部基づいています24。最後に、バーコードAAVライブラリの生成と、同じ出版物で使用されるNGSパイプラインの説明が提供されます。
プロトコル
1. AAV2ランダム7量体ペプチドディスプレイライブラリーの調製
注:AAV2ランダムペプチドディスプレイライブラリの調製では、縮重オリゴヌクレオチドを一本鎖DNAとして合成し、それを二本鎖DNAに変換し、消化し、アクセプタープラスミドにライゲートし、エレクトロポレートします。
- 縮重オリゴヌクレオチドの設計
- 縮重オリゴヌクレオチドを注文し、コドンバイアスを回避します。オリゴヌクレオチド5'CAGTCGGCCAG AG W GGC(X01)7 GCCCAGGCGGCTGACGAG 3'において、X01は20個のコドンに対応し、それぞれが20個のアミノ酸のうちの1つをコードする。WはAまたはTであり得、アミノ酸アルギニン(R)またはセリン(S)をコードするコドンAGAまたはAGTを生じる。
- 増幅プライマーを注文する:5' CTCGTCAGCCGCCTGG 3' (詳細については 図1 を参照)。これにより、次のタンパク質インサートが生成されます: R/S GX 7。理論上の多様性は次のように計算されます:1 x 2 x 207 = 2.56 x 109 つの一意のバリアント。
注:この多様性は、変換効率によって制限される可能性があることに注意してください。
- セカンドストランド合成
- 両方のオリゴヌクレオチド(縮重オリゴヌクレオチドおよび増幅プライマー)をTEバッファーで終濃度100 μMまで再懸濁します。
- PCR反応では、各プライマー1 μL、バッファー10 μL、DMSO1.5 μL、dNTP(10 mM)0.5 μL、ハイフィデリティホットスタートポリメラーゼII0.5 μL、ヌクレアーゼフリー水35.5 μLで50 μLの反応を設定します。
- 反応物をサーモサイクラーに移し、98°Cで10秒間のプレインキュベーションステップを実行し、続いて98°Cで10秒間、59°Cで30秒間、72°Cで10秒間の3サイクルを実行し、次に72°Cで5分間、最終冷却ステップを実行します。
- ヌクレオチド除去キットを使用して反応を精製し、100 μLのヌクレアーゼフリー水で溶出します。
- バイオアナライザーでの分析により、セカンドストランド合成の効率を確認します( 図2を参照)。製造元の指示に従って、DNA 1000試薬キットからマイクロ流体チップに1 μLの反応液をロードすることにより、二本鎖インサートのサイズと純度を分析します。このキットは、25〜1,000 bpsの二本鎖DNAフラグメントのサイズと濃度を測定するように最適化されています。
- 両方のオリゴヌクレオチド(縮重オリゴヌクレオチドおよび増幅プライマー)をTEバッファーで終濃度100 μMまで再懸濁します。
- インサートおよびプラスミドベクターの消化
- 精製インサート85 μLを、10 μLの10xバッファーと5 μLのBglI酵素で最終100 μLの反応容量で消化します(詳細については 図1 を参照)。37°Cで一晩インキュベートします。ヌクレオチド除去キットを使用して精製し、50 μLのヌクレアーゼフリー水で溶出し、分光光度計でタイプ「オリゴDNA」を使用して定量します。
- 複製コンピテントAAVプラスミド(pRep2Cap2_PIS)26 (ITRに隣接するウイルスゲノム)10 μgを、20 μLの10xバッファーと10 μLのSfiI酵素で最終200 μLの反応容量で消化します(詳細については 図1 を参照)。50°Cで一晩インキュベートします。ゲル抽出キットを使用して1%アガロースゲル上のベクターを精製した後、DNA精製キットを使用して追加の精製ステップを行います。分光光度計で濃度を定量します。
- ベクターへのインサートのライゲーション
- 20 μLのライゲーション反応で、2 μLのバッファーと2 μLのリガーゼを含む45 ngのインサートを含む955 ngのプラスミドベクターをリゲートします。16°Cで一晩インキュベートし、続いて70°Cで10分間インキュベートしてリガーゼを熱失活させます。
- 形質転換、複雑性計算、プラスミドライブラリの調製
- 製造元の指示に従って、DNA精製キットで反応を精製します。ヌクレアーゼフリー水の開始量の約80%で反応を溶出し、その後の形質転換のために氷上に保存します。
- エレクトロコンピテントセルの形質転換:1バイアルのエレクトロコンピテントセルを氷上で10分間解凍します。次に、精製ライゲーション反応液1〜2 μLを30 μL(1バイアル)のエレクトロコンピテントセルに加え、軽くたたくことで混合します。次に、気泡を導入せずに、細胞/DNA混合物を事前に冷却した1 mmギャップのエレクトロポレーションキュベットに注意深くピペットで固定します。
- 次の設定を使用してエレクトロポレーションします:1800 V、600 Ω、および10 μF。エレクトロポレーションパルスから10秒以内に、970 μLの予熱した回収培地(エレクトロコンピテントセルに付属)をキュベットに加え、ピペッティングで混合します。最後に、細胞をマイクロ遠心チューブに移し、37°C、250rpmで1時間インキュベートします。所望の多様性を達成するために、10〜100回の反応を行い、そしてインキュベーション後、全ての反応を1本のチューブにプールする。
- プールされた形質転換の10 μLをPBSで10倍、100倍、または1,000倍に希釈し、適切な抗生物質(75 mg/mLのアンピシリン)を含む栄養寒天プレートに100 μLを広げて、多様性を計算します。寒天プレートを37°Cで一晩インキュベートし、寒天プレート上のコロニーをカウントします。
- 理論上の多様性を次のように計算します。
理論最大多様性= 10 x 希釈係数 x コロニー数 x エレクトロポレーション反応数
注:ライブラリの品質を確認するには、サンガーシーケンシングによって少なくとも20コロニーをシーケンスします。ほとんどのクローンには挿入が含まれている必要があり、すべてが一意である必要があります。 - 適切な抗生物質を含む400〜1,000 mLのLB培地に残りのプールされた形質転換を接種し、37°C、180 rpmで一晩インキュベートします。
- プラスミドライブラリーの作製
- 一晩培養してからグリセロールストックを調製し(ヌクレアーゼフリーの水に等量の細菌培養液と50%グリセロール溶液を混合し、-80°Cで凍結)、プラスミドマキシキットを使用してプラスミドライブラリーを精製します。
- AAVウイルスライブラリーの作製
- 前述のようにウイルスライブラリーを準備する27。プラスミドライブラリ(pRep2Cap2_PI、ペプチドインサート)をアデノヘルパープラスミドとともに、ポリエチレンイミン(PEI)などのトランスフェクション試薬を用いてHEK293T細胞にトランスフェクトします。
- 3日後に細胞を収集し、それらを凍結融解の3サイクルにかける。塩化セシウムグラジエント超遠心分離を使用してウイルスライセートを精製し、続いてPBSにバッファー交換を行い、最後にウイルス粒子を濃縮します。
- dd-PCRを用いたAAVベクター滴定
- 2 μLのAAVベクターストックを198 μLのヌクレアーゼフリー水で連続希釈し、1:106 の最終希釈液を得ます。200 μLのピペットを使用して毎回完全に混合します。テンプレートなしコントロール (NTC) をネガティブ コントロールとして 1 つ追加します。
注:追加の低希釈または高希釈をアッセイできます(1:105-1:107)。 - 20xプライマー-プローブミックスを準備します。100 μM プライマー (順方向および逆方向、Rep2、および ITR) をそれぞれ 3.6 μL、100 μM dd-PCR プローブ (Rep2 および ITR) をそれぞれ 1 μL、およびヌクレアーゼフリー水 3.6 μL を 1.5 mL 遠沈管に追加します。
注:AAVライブラリは、FAM標識プローブで検出された導入遺伝子標的プライマープローブセット(Rep2)と、HEX標識プローブで検出されたITR標的プライマープローブセットを使用して測定されます。 - 5.5 μLのサンプル、1.1 μLの20xプライマー-プローブミックス、11 μLのプローブ用dd-PCRスーパーミックス(dUTPなし)、および4.4 μLのヌクレアーゼフリー水を加えて、22 μLのPCR反応を準備します。これにより、プライマーとプローブでそれぞれ900 nMと250 nMの濃度が得られます。
- 液滴発生器を使用して液滴を生成し、反応液を96ウェルプレートに移し、プレートをサーモサイクラーに入れ、94°Cで10分間の変性ステップを実行し、続いて94°Cで30秒、58°Cで1分間の変性ステップを実行します。 次に、ポリメラーゼを98°Cで10分間加熱失活させ、最後の冷却ステップを追加します。液滴リーダーで反応を読み取り、分析28に進みます。
- 保存したdd-PCRプレートファイルを解析ソフトで開きます。[1D 振幅]タブの閾値ツール(蛍光振幅対イベント番号)を使用して、NTCをガイドとして使用して各チャンネルの負液滴と正液滴を分離し、データをcsvファイルにエクスポートします。
- ベクトル濃度を計算するには、まず次の式を使用して補正係数CFを計算します。
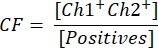
CFは、導入遺伝子とITR[Ch1+ Ch2+]の両方に対して陽性である導入遺伝子[陽性]に陽性の液滴の割合を決定し、機能性ベクター粒子の検出を確実にします。最終的なベクトル濃度cは、次の式を使用して計算できます。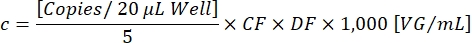
DFは希釈係数です(1:105-1:107 は以前に決定されました)。20 μL/ウェル反応あたりのコピー数は、希釈サンプルの5 μLに相当します。係数1,000は、スケールをVG / mL(ウイルスゲノム/ mL)に補正します。例示的な滴定結果を 表1 および 図3に示す。
- 2 μLのAAVベクターストックを198 μLのヌクレアーゼフリー水で連続希釈し、1:106 の最終希釈液を得ます。200 μLのピペットを使用して毎回完全に混合します。テンプレートなしコントロール (NTC) をネガティブ コントロールとして 1 つ追加します。
- NGSによるAAVウイルスライブラリーの解析
- プループリーディングポリメラーゼキットを使用して20 μLのPCR反応を設定し、96ヌクレオチドペプチド挿入フラグメントを増幅します(2x; 図4を参照)。1 x 108 vgを含むAAVストック1 μL、100 μMプライマー(NGS_forwardおよびNGS_reverse)それぞれ0.5 μL、および酵素ミックス10 μLを反応に加えます。ヌクレアーゼフリー水で最終容量を20 μLに調整します。
- 反応物をサーモサイクラーに移し、98°Cで3分間の変性ステップを実行し、続いて98°Cで10秒、59°Cで10秒、72°Cで20秒の30〜35サイクルを実行し、続いて72°Cで5分間、最終冷却ステップを実行します。
- PCR精製キットを使用してサンプルを精製します。分光光度計で濃度を定量し、3%アガロースゲルを実行して、純度とフラグメントサイズを検証します。
- NGSライブラリの調製に関する製造元の指示に従って、複雑度の低いサンプルキット用のライブラリシステムを使用してPCRフラグメントを処理します。30 ngのPCRフラグメントでエンドリペア反応を行い、その後アダプターライゲーションとPCR増幅を10サイクル行います。反応の精製にはPCR精製キットを使用してください。
- 最終製品をバイオアナライザーで処理し、製造元の指示に従ってDNA試薬キットを使用してサイズと純度を検証します。
- 蛍光光度計を使用してアンプリコンを定量し、プールします。最終的にプールされたNGSライブラリを蛍光光度計で再度定量し(製造元の指示に従って)、バイオアナライザーで品質を検証します。
- 読み取り長が84、インデックスが8の75サイクル高出力キットを使用して、シングルエンド(SE)モードでNGSライブラリをシーケンスします。
注:この記事の例のシーケンスは、EMBLハイデルベルク(http://www.genecore.embl.de/)のGeneCore施設で実行されました。 - Python 3 と biopython で NGS シーケンシングデータを解析します。ファイルは https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022 にあります(または https://doi.org/10.5281/zenodo.7032215 にあります)。NGS分析は2つのステップで構成されています。
- 最初のステップでは、特定の基準(挿入部位に隣接する認識配列の存在)を満たす配列を配列ファイルで検索します( 図4、ステップ1.9.8.5を参照)。これは、スクリプト (Script#1) と必要な情報を提供する構成ファイルを使用して行われます。正しいシーケンスが特定されると、プログラムはシーケンスを抽出して出力ファイル (シーケンス ファイルと同じ名前の txt ファイル) に保存します。
- 2番目のステップは、出力ファイルの分析です。ライブラリ内の配列は、9つのアミノ酸挿入物の6つのヌクレオチド(AGWggc、W = A / T)のいずれかから始まります。この開始配列に基づいて、ペプチドが翻訳される。これにより、ペプチドバリアント(PV)を含む出力ファイルが生成されます。
- スクリプトとデータの2つのフォルダーを準備します。Data フォルダーに、シーケンス処理の結果の gzip 圧縮ファイルをコピーします。スクリプト フォルダーに、次のファイル Python ファイルをコピーします: スクリプト #1_DetectionExtraction_JoVE_Py3.py;Python ファイル: スクリプト #2_PV_extraction_and_ranking_Py3.py;構成ファイル: Barcode_Script_JoVE.conf;およびルックアップテーブル(LUT)ファイル:Zuordnung.txt。
- スクリプトを実行する前に、[スクリプト] フォルダー内の次のファイルを編集します。"Zuordnung.txt" ファイルを開き、タブ区切りの 2 つの列、gzip ファイルの名前 (列 1)、および目的の最終名 (列 2、タブ区切りの値) を追加します。
注: サンプル txt ファイルは、GitHub フォルダー "PV_analysis_script" にあります。GitHub フォルダーで提供されるファイルは、上記のライブラリの 3 つのサンプル データ (xaa.txt.gz、xab.txt.gz、xac.txt.gz) の分析用に用意されています。出力ファイルも提供されます。 - 構成ファイル "Barcode_Script_JoVE.conf" の次の変数を変更します。
my_dir = "~/データ/"
filename_sample_file = "~/Script/Zuordnung.txt"
シーケンス固有の変数: BCV_size = 27、BCVleft = TCCAGGCCAG、BCVright = GCCCAGG、BCVloc = 30、BCV マージン = 8、BCVleft_revcomp = GCCGCCTGGGC、BCVright_revcomp = CTGGCCCC、および BCVloc_revcomp = 41 (詳細については 、図 4 を参照)。 - 次のコマンドを使用して、バリアント配列の検出と抽出を呼び出します。
>python3 ~/Script#1_DetectionExtraction_JoVE_Py3.py ~/Barcode_Script_JoVE.conf
注:出力は、抽出されたDNA配列とそのリード数を含むtxtファイルです。このファイルのヘッダーには、統計データ(つまり、読み取りの総数と抽出された読み取り)が含まれています。これらのデータは次のファイルに転送されます。これらのtxtデータは、DNA配列が翻訳、ランク付け、および分析されるScript#2の入力ファイルです。 - 次のコマンドを使用して、PV の抽出と分析を実行します。
>python3 ~/Script#2_PV_extraction_and_ranking_Py3.py ~/Barcode_Script_JoVE.conf - スクリプト #2 のテキスト出力ファイルを分析します。スクリプト #2 の出力ファイルには、"Zuordnung.txt" の LUT の 2 番目の列を使用して、分析の種類に基づいた拡張子を付けた名前が付けられます。
注:3つの出力ファイルの最初の行(「有効なPV読み取りの#」、「無効なPV読み取りの#」、および「一意のPV読み取りの#」)、入力txtファイルの各DNA配列のインデックスを含む最初の列(スクリプト#1の出力)、および次の列に統計データが含まれていることを確認してください:(1)「...analyzed_all.csv": "サンプル:"(DNA配列)、"#"(リード数)、"FrwまたはRev"(正リードまたは逆リード)、および"PV"(翻訳ペプチド配列)。無効なシーケンスの最後の 2 つの列に "NA" と "無効" があります。(2) "...analyzed_validSeq.csv": 前のファイルと同じで、有効なシーケンスでフィルタリングされています。(3) "...analyzed_PV.csv": "PVs"(翻訳されたペプチド配列)、"#"(リード数)、および"カウント"(前のファイルのfrwカウントとrevカウントがマージされ、カウントが1または2になります)。 - ユーザーのニーズに基づいて、利用可能なソフトウェアを使用して出力ファイルを視覚化します。
2. AAV2ランダム7量体ペプチドディスプレイライブラリの選択
- 定量化と品質管理(セクション1)の後にAAVライブラリを使用して、目的の特性を持つ候補を繰り返し選択します(図5を参照)16、18、21。
注:これらの候補は、以下のセクション3で説明するように、バーコードライブラリの生成に使用されます。
3. バーコード付きAAVキャプシドライブラリの調製と分析
注:ペプチド表示画面で潜在的に特異的で効率的なAAVキャプシドのセットを同定した後、同定されたペプチド配列の機能を検証し、それらを一般的に使用される、またはよく説明されている参照AAVカプシドバリアントのセットと比較します。これを行うには、キャプシド シーケンスを ITR なしで Rep/Cap ヘルパー コンストラクトに挿入します。
- バーコードAAVライブラリの制作
- 前述のように、3プラスミドシステムを用いて各カプシド変異体について組換えAAV産生を行う24。
注:異なるカプシド変異体を区別するために、ITRに隣接するレポーター導入遺伝子プラスミドは、長さ15ヌクレオチドの一意のバーコードを保持しています。バーコードは、増強された黄色蛍光タンパク質(EYFP)とpolyAシグナルの間の3' UTR(非翻訳領域)にあります( 図6Aを参照)。EYFP発現は、十分なレベルのRNA転写物を提供する強力なユビキタスサイトメガロウイルス(CMV)プロモーターによって駆動されます。 - 3ヌクレオチド未満のホモポリマー、<65%29のGC含量、および4ヌクレオチドを超えるハミング距離を持つ15ヌクレオチド長のバーコードを設計する24。
- 各カプシドを、固有のバーコードを持つ導入遺伝子プラスミドと組み合わせて個別に製造します。このようにして、各キャプシドバリアントは、特定の追跡を可能にする個別のバーコードでタグ付けされます( 図6Bを参照)。
- 前述のように、3プラスミドシステムを用いて各カプシド変異体について組換えAAV産生を行う24。
- dd-PCRを用いたAAVベクター滴定
- セクション1.8で前述したように、Rep2プライマーペアをYFPプライマーペアに置き換えてAAV滴定を実行します。
- 個々のAAV生産を定量化し、各生産を同量プールして、最終的なバーコードライブラリを生成します。
- 最終ライブラリを再度定量して、最終濃度と品質を確認します ( 図7を参照)。
- バーコード付きAAVライブラリ in vivo アプリケーション
- バーコードAAVライブラリを、選択したモデルシステムに全身的に適用する(例えば、マウス24において全身的に)。
- 実験に基づいて、ONおよびOFFターゲット組織(すなわち、肝臓、肺、心臓、横隔膜、平滑筋、十二指腸、膵臓、結腸、上腕二頭筋、卵巣、胃、内耳、腎臓、腹部大動脈、胸部大動脈、脳、褐色および白色脂肪、および脾臓)または細胞タイプを収集します。次のセクションで説明するように、それらを-80°Cで凍結し、DNA/RNAを抽出し、NGS定量分析を適用します。
- DNA/RNA抽出
- DNA/RNAミニキットを使用して、目的の組織からDNAとRNAを抽出します。
- 目的の組織の小片(1 mm3、約5 mg)を2 mLの反応チューブに入れます。
- β-メルカプトエタノール(1%)および5 mmスチールビーズを混合した350 μLの溶解バッファーを組織に加えます(ドラフトフードの下でβ-メルカプトエタノールを含むサンプルを処理します)。
- 組織を組織に均質化します40Hzで45秒間ライザー。
- プロテイナーゼK(10 mg/mL)を10 μL加え、400 rpmで振とうしながら55°Cで15分間インキュベートします。
- 室温で20,000 x g で3分間遠心分離し、上清を回収して、DNA/RNAキットの製造元のプロトコルに進みます。
- 洗浄ステップを2つのステップに分割し、各ステップで350 μLの洗浄バッファーを使用します。これらの洗浄ステップの間に、RNaseフリーのDNase Iでカラム上の残りのDNAを消化し、製造元の指示に従って調製した80 μLのDNase I溶液をカラムに加え、室温で15分間インキュベートします。
- ヌクレアーゼフリーの水でカラムからRNA/DNAを溶出します。単離したRNAを-80°Cで保存し、gDNAを-20°Cで保存します。
- cDNA合成
- 逆転写反応の前に、RNAサンプルを15〜30分のDNase I処理の別のラウンドにかけます(RNAサンプルから汚染されたDNAを完全に除去するため)。1 μLのDNase I溶液、4 μLのバッファー(キットに付属)、およびヌクレアーゼフリー水を最終容量40 μLから212 ngのRNAに加えます。室温で30分間インキュベートし、70°Cで10分間加熱失活します。
- 製造元の指示に従ってキットを使用して、150 ngのRNAを使用してcDNAを合成します。逆転写酵素を含まないコントロールを含め、サンプルからの混入ウイルスDNAの不在を確実にする。cDNAは-20°Cで保存されます。
注:最適な逆転写のための入力RNAの量は、組織の種類とそれぞれの組織で予想される形質導入効率によって異なります。
- AAVウイルスライブラリーの解析(in-vivo) by NGS
- 低コストで高いシーケンシング深度を実現するには、前述のようにイルミナシーケンシング を介して NGSを実行します(セクション1.9)。バーコード配列を増幅し、シーケンシングアダプターをアンプリコンにライゲーションします。
- リード長が短く、アンプリコンの両側にあるシーケンシングアダプターのライゲーションのため、設計時には、アンプリコンがNGSリード内にバーコードシーケンスが存在することを確認するのに十分なほど小さいことを確認してください。ウイルスゲノム内のバーコードとウイルス転写物のシーケンシングのために、PCRアンプリコンは113 bpの長さになるように設計されています( 図8を参照)。
- バーコード領域を、BC-seq前方およびBC-seq逆方向のプライマーで増幅します。次のPCR反応を準備します:0.5 μLのハイフィデリティDNAポリメラーゼ、10 μLの5xバッファー、0.25 μLの各100 μMプライマー(BC-seq fw/BC-seq rv)、および1 μlの10 mM dNTPs。25 ngのcDNAまたはDNA/反応を鋳型として使用し、ヌクレアーゼフリー水で最終容量を50 μLに調整します。
- 汚染を避けるために、清潔なPCRフードの下でPCRマスターミックスを準備します。次のサイクル条件を使用します:98°Cで30秒、続いて98°Cで10秒、72°Cで20秒間40サイクル、72°Cで最後の5分間のステップ。
- PCRマスターミックスに汚染DNAがないことを確認するためのPCRコントロールを含めます。cDNAサンプルには、逆転写酵素を含まないコントロールを含めます。最後に、AAV 入力ライブラリにサンプルを含めます。この情報は、解析で使用されるNormalization_Variant.txtファイルの生成に使用されます。
- PCR精製前にゲル電気泳動により各サンプルのPCRフラグメントのサイズを確認します。後者は、市販の磁気ビーズまたはカラムベースのDNA精製システムのいずれかを使用することによって達成されます( 材料の表を参照)。
- セクション1.9で前述したように、製造元の指示に従って、複雑度の低いサンプル用のライブラリシステムを使用してNGSライブラリを準備します。
- dsDNA HSキット を介して DNA濃度を決定し、前述のようにライブラリの品質を分析し(セクション1.9.6)、続いてプーリングします。プールされたライブラリを蛍光光度計で定量し、バイオアナライザーで品質を評価します。
- セクション 1.9.7 で説明されているように NGS シーケンスを実行します。
- 導入遺伝子(ウイルスゲノム)とハウスキーピング遺伝子のコピー数をqPCRで定量し、DNA上の組織または臓器間のプールされたライブラリの分布を評価します。
- 以下のように30 μLのqPCR反応を設定し、EYFP(導入遺伝子)およびGAPDH(グリセルアルデヒド3-リン酸デヒドロゲナーゼ、ハウスキーピング遺伝子)のコピー数を決定しました。
- EYFP用の60倍のプライマー/プローブミックス(1.5 μM YFP_fw、1.5 μM YFP_rv、および0.6 μM YFP_probe;材料表を参照)を準備します。GAPDHプライマー/プローブミックス( 材料の表を参照)を使用して、ハウスキーパー遺伝子のコピー数を決定します。氷上で反応を設定します。
- PCRマスターミックス(15 μL、 材料表を参照)を調製し、すべてのサンプルと標準に対して60倍のプライマー/プローブミックス(0.5 μL)を追加します(標準のコピー数を計算するには、次のリンクを使用します:http://cels.uri.edu/gsc/cndna.html)。氷上で反応を設定します。
- 15.5 μLのマスターミックスを96ウェルプレートに移し、14.5 μLのサンプル(総DNA濃度75 ng)または標準物質をそれぞれのウェルに加えます。96ウェルプレートをホイル、ボルテックス、およびスピンで短時間密封します。
- 各サンプル10 μLを384ウェルプレートに2回に分けて移します。プレートをホイルで密封し、800 x g で4°Cで5分間回転させます。
- 反応混合物をサーモサイクラーで初期温度50°Cで2分間インキュベートし、続いて95°Cで10分間の初期活性化ステップを行います。 95°Cで15秒間40サイクルの変性を行い、60°Cで1分間24のアニーリング/伸長を行います。
- 二倍体ゲノムの数(dg)を取得するには、GAPDHコピー数を使用して2で割ります。次に、EYFPコピー数の値を取り、dgの数で割ると、二倍体ゲノムあたりのベクターゲノム(vg / dg)が得られます。この値を使用して、バイオインフォマティクス分析用のNormalization_Organ.txtファイルを生成します。
- NGSシーケンシングデータの解析をWeinmannらと同様に実行する。24、Python3(https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022)のカスタムコードを使用します。ワークフローは、隣接する配列、その長さと位置によって導かれるバーコード配列の検出(Script#1_BarcodeDetection.py)、および一連の組織に対するバーコードの濃縮と分布の分析(Script#2_BarcodeAnalysis.py)で構成されます。
- バーコードを検出し、AAVバリアントに割り当てます。シーケンスデータをアーカイブされたfastqファイルとして1つのディレクトリに配置します(例:「Data_to_analyze」)。入力ライブラリのシーケンスデータファイルはこのディレクトリに含まれており、入力ライブラリのcapsid比率を計算するためにのみ使用されます。
- スクリプトを実行する前に、AAV capsidバリアント名に割り当てられたバーコードシーケンスを含むキャプシドバリアントファイル(「バリアント.txtの例」を参照)と、汚染の可能性があるバーコードシーケンスを含むコンタミネーションファイル(「コンタミネーション.txt」を参照)の2つのタブ区切りテキストファイルを作成します(ラボで入手可能な他のバーコードは、汚染の一因となります)。
- 最後に、構成ファイル「Barcode_Script.conf」を編集して、シーケンスデータを含むフォルダーへのパス(「Data_to_analyze」など)、バーコードの隣接領域のシーケンス、それらの位置、バーコード検出用のウィンドウサイズ(1.9.8.5と同様、 図8を参照)の情報を含めます。
- 次のコマンドを使用して、Script#1_BarcodeDetection.py および構成ファイルへの指定されたパスを使用してバーコード検出を呼び出します。
>python3 ~/Script#1_BarcodeDetection.py ~/Barcode_Script.conf
注: Script#1_BarcodeDetection.py 実行の出力は、capsid バリアントごとの読み取りカウントと、生データから回復された読み取りの合計数を含むテキスト ファイルです。 - 次のtxtファイルとともにScript#2_BarcodeAnalysis.pyを実行して、組織または臓器間のバーコードAAVキャプシドの分布を評価します。
- 「Zuordnung.txt」ファイルで、バーコード検出実行から取得した各txtファイルに組織/臓器名(最初の列のtxtファイルの名前とタブ区切りの割り当ての対応する組織/臓器名)に名前を割り当てます。
注:例については、「例」フォルダ(https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022)を確認してください。注目すべきことに、組織/臓器名には、cDNAまたはgDNA測定値および生物学的複製数(M1、M2など)を定義する文字を含めることができます。 - 割り当て "Zuordnung.txt" ファイルで指定された名前に対応する、ON-および OFF ターゲット臓器の名前のリストを含む "organ.txt" テキスト ファイルを作成します ("Example" フォルダー: https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022 を参照)。
- すべてのキャプシドバリアントとすべての臓器/組織の正規化された値を持つ「Normalization_Organ.txt」および「Normalization_Variant.txt」タブ区切りのテキストファイルを作成します。「Normalization_Organ.txt」ファイルの最初の列に、各臓器に与えられた名前(割り当てファイル「Zuordnung.txtのように)を書き、2番目の列にセクション3.6.11で生成された対応する組織の正規化値を書き込みます。
- "Normalization_Variant.txt" ファイルの最初の列に capsid 名のリストを入力し、2 番目の列にプールされたライブラリ内の各 capsid の読み取りカウントの正規化された値を入力します (正規化は、最初のスクリプトから得られた入力ライブラリの txt 出力ファイルに基づいて計算できます)。
- 上記のすべての追加ファイルへのフルパスを指定して、構成ファイルを編集します。スクリプト #2_BarcodeAnalysis.py を次のように実行します。
>python3 /Script#2_BarcodeAnalysis.py ~/Barcode_Script.conf
注:バーコード分析スクリプトは、前述の複数の正規化ステップに基づく異なる組織内のカプシド分布の相対濃度(RC)値を持つテキストファイルと、テキストファイルデータをマージされたマトリックスデータに結合するスプレッドシートファイルなど、いくつかのファイルを出力します。後者は、クラスター分析と視覚化に使用できます。 - データを視覚化し、マトリックスデータのクラスター分析を実行して、キャプシド特性を区別し、組織全体のRCプロファイルに基づいてそれらの類似性を評価します。追加のスクリプトPCA_heatmap_plotを使用します。リポジトリに配置されたR:
>スクリプト --バニラ ~/PCA.R~/相対濃度.xls
注:このスクリプトは、相対濃度.xlsファイルを入力として受け取り、階層クラスターヒートマップと主成分分析(PCA)の2つのプロットを生成します。 - プロット(ヒートマップの軸、PCAの主成分)またはpngパラメータ(色、サイズ、ラベル)を変更するには、Rスクリプトを開き、コメントセクションの指示に従います。
- 「Zuordnung.txt」ファイルで、バーコード検出実行から取得した各txtファイルに組織/臓器名(最初の列のtxtファイルの名前とタブ区切りの割り当ての対応する組織/臓器名)に名前を割り当てます。
結果
AAV2ペプチドディスプレイライブラリの作製。 改変されたAAVの選択に向けた最初のステップとして、プラスミドライブラリの生成について説明します。ペプチドインサートは、縮重プライマーを使用して製造されます。コドンの組み合わせを64から20に減らすと、タンパク質レベルではなくDNAのライブラリの多様性を減らすことで、終止コドンを排除し、NGS分析を容易にするとい?...
ディスカッション
このプロトコルでは、ペプチドディスプレイAAVキャプシドエンジニアリングとバーコードAAVライブラリスクリーニング、およびライブラリ組成とキャプシド性能のバイオインフォマティクス分析に必要な手順が概説されています。このプロトコルは、ほとんどのウイルス学研究所が分子生物学技術の習熟度に匹敵するプログラミングスキルに遅れをとっているため、これらのタイプのライブ?...
開示事項
D.G.はAaviGen GmbHの共同創設者であり、D.G.とK.R.は、免疫回避AAVキャプシド変異体の生成に関連する出願中の特許出願の発明者です。残りの著者は開示するものは何もありません。
謝辞
D.G.は、DFG共同研究センターSFB1129(Projektnummer 240245660)およびTRR179(Projektnummer 272983813)を通じたドイツ研究財団(DFG)、ならびにドイツ感染研究センター(DZIF、BMBF;TTU-HIV 04.819)。
資料
| Name | Company | Catalog Number | Comments |
| Amplification primer | ELLA Biotech (Munich, Germany) | - | Second-strand synthesis of oligonucleotide insert |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | DNA fragment validation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | DNA fragment validation |
| AllPrep DNA/RNA Mini Kit | Qiagen (Venlo, Netherlands) | 80204 | DNA/RNA extraction |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | NGS Library preparation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | NGS Library preparation |
| BC-seq fw: | IDT (San Joce, CA, CA, USA) | ATCACTCTCGGCATGGACGAGC | NGS Library preparation |
| BC-seq rv: | IDT (San Joce, CA, CA, USA) | GGCTGGCAACTAGAAGGCACA | NGS Library preparation |
| β-Mercaptoethanol | Millipore Sigma (Burlington, MA, USA) | 44-420-3250ML | DNA/RNA extraction |
| BglI | New England Biolabs (Ipswich, MA, USA) | R0143 | Digestion of double-stranded insert |
| C1000 Touch Thermal Cycler | Bio-Rad (Hercules, CA, USA) | 1851196 | dd-PCR cycler |
| dNTPS | New England Biolabs (Ipswich, MA, USA) | N0447S | NGS Library preparation |
| ddPCR Supermix for probes (no dUTP) | Bio-Rad (Hercules, CA, USA) | 1863024 | dd-PCR supermix |
| Droplet Generation Oil for Probes | Bio-Rad (Hercules, CA, USA) | 1863005 | dd-PCR droplet generation oil |
| DG8 Cartridges for QX100 / QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864008 | dd-PCR droplet generation cartridge |
| DG8 Cartridge Holder | Bio-Rad (Hercules, CA, USA) | 1863051 | dd-PCR cartridge holder |
| Droplet Generator DG8 Gasket | Bio-Rad (Hercules, CA, USA) | 1863009 | dd-PCR cover for cartridge |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad (Hercules, CA, USA) | 12001925 | dd-PCR 96-well plate |
| E.cloni 10G SUPREME Electrocompetent Cells | Lucigen (Middleton, WI, USA) | 60081-1 | Electrocompetent cells |
| Electroporation cuvettes, 1mm | Biozym Scientific (Oldendorf, Germany) | 748050 | Electroporation |
| GAPDH primer/probe mix | Thermo Fischer Scientific (Waltham, MA, USA) | Mm00186825_cn | Taqman qPCR primer |
| Genepulser Xcell | Bio-Rad (Hercules, CA, USA) | 1652660 | Electroporation |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystems (Waltham, MA, USA) | 4368814 | cDNA reverse transcription |
| ITR_fw | IDT (San Joce, CA, USA) | GGAACCCCTAGTGATGGAGTT (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_rv | IDT (San Joce, CA, USA) | CGGCCTCAGTGAGCGA (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_probe | IDT (San Joce, CA, USA) | HEX-CACTCCCTCTCTGCGCGCTCG-BHQ1 (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR probe |
| Illumina NextSeq 500 system | Illumina Inc (San Diego, CA, USA) | SY-415-1001 | NGS Library sequencing |
| KAPA HiFi HotStart ReadyMix (2X)* | Roche AG (Basel, Switzerland) | KK2600 07958919001 | NGS sample prepration |
| MagnaBot 96 Magnetic Separation Device | Promega GmbH (Madison, WI, USA) | V8151 | Sample prepration for NGS library |
| NanoDrop 2000 spectrophotometer | Thermo Fischer Scientific (Waltham, MA, USA) | ND-2000 | Digestion of double-stranded insert |
| NGS_frw | Sigma-Aldrich (Burlinght, MA, USA) | GTT CTG TAT CTA CCA ACC TC | NGS primer |
| NGS_rev | Sigma-Aldrich (Burlinght, MA, USA) | CGC CTT GTG TGT TGA CAT C | NGS primer |
| NextSeq 500/550 High Output Kit (75 cycles) | Illumina Inc (San Diego, CA, USA) | FC-404-2005 | NGS Library sequencing |
| Ovation Library System for Low Complexity Samples Kit | NuGEN Technologies, Inc. (San Carlos, CA, USA) | 9092-256 | NGS Library preparation |
| PX1 Plate Sealer | Bio-Rad (Hercules, CA, USA) | 1814000 | dd-PCR plate sealer |
| Pierceable Foil Heat Seal | Bio-Rad (Hercules, CA, USA) | 1814040 | dd-PCR sealing foil |
| Phusion High-Fidelity DNA-Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F530S | Second-strand synthesis of oligonucleotide insert |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences, Inc. (Warrington, PA, USA) | 24765-1G | AAV library preparation |
| ProNex Size-Selective Purification System | Promega GmbH (Madison, WI, USA) | NG2002 | Sample prepration for NGS library |
| Phusion Hot Start II Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F549L | NGS Library preparation |
| Proteinase K | Roche AG (Basel, Switzerland) | 5963117103 | DNA/RNA extraction |
| pRep2Cap2_PIS | ITR-Rep2Cap2-ITR vector. Peptide insertion site within the Cap2 ORF, manufactured/prepared in the lab | ||
| QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864002 | dd-PCR droplet generator |
| QX200 Droplet Reader | Bio-Rad (Hercules, CA, USA) | 1864003 | dd-PCR droplet analysis |
| QIAquick Nucleotide Removal Kit | Qiagen (Venlo, Netherlands) | 28306 | Second-strand synthesis of oligonucleotide insert purification |
| QIAquick Gel Extraction Kit | Qiagen (Venlo, Netherlands) | 28704 | Plasmid vector purification |
| QIAGEN Plasmid Maxi Kit | Qiagen (Venlo, Netherlands) | 12162 | Plasmid library DNA preparation |
| Qiaquick PCR Purification kit | Qiagen (Venlo, Netherlands) | 28104 | Sample prepration for NGS library |
| Qubit fluorometer | Invitrogen (Waltham, MA, USA) | Q32857 | NGS Library preparation |
| Qubit dsDNA HS | Thermo Fischer Scientific (Waltham, MA, USA) | Q32851 | NGS Library preparation |
| QuantiFast PCR Master Mix | Qiagen (Venlo, Netherlands) | 1044234 | Taqman qPCR |
| rep_fw | IDT (San Joce, CA, USA) | AAGTCCTCGGCCCAGATAGAC | dd-PCR primer |
| rep_rv | IDT (San Joce, CA, USA) | CAATCACGGCGCACATGT | dd-PCR primer |
| rep_probe | IDT (San Joce, CA, USA) | FAM-TGATCGTCACCTCCAACA-BHQ1 | dd-PCR probe |
| RNase-free DNase | Qiagen (Venlo, Netherlands) | 79254 | DNA/RNA extraction |
| SfiI | New England Biolabs (Ipswich, MA, USA) | R0123 | Digestion of vector |
| 5 mm, steel Beads | Qiagen (Venlo, Netherlands) | 69989 | DNA/RNA extraction |
| TRIMER-oligonucleotides | ELLA Biotech (Munich, Germany) | - | Degenerate oligonucleotide |
| T4 Ligase | New England Biolabs (Ipswich, MA, USA) | M0202L | Plasmid library ligation |
| TissueLyserLT | Qiagen (Venlo, Netherlands) | 85600 | DNA/RNA extraction |
| YFP_fw | IDT (San Joce, CA, USA) | GAGCGCACCATCTTCTTCAAG | dd-PCR primer |
| YFP_rv | IDT (San Joce, CA, USA) | TGTCGCCCTCGAACTTCAC | dd-PCR primer |
| YFP_probe | IDT (San Joce, CA, USA) | FAM-ACGACGGCAACTACA-BHQ1 | dd-PCR probe |
| Zymo DNA Clean & Concentrator-5 (Capped) | Zymo research (Irvine, CA, USA) | D4013 | Vector and Ligation purification |
参考文献
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Muhuri, M., Levy, D. I., Schulz, M., McCarty, D., Gao, G. Durability of transgene expression after rAAV gene therapy. Molecular Therapy. 30 (4), 1364-1380 (2022).
- Li, C., Samulski, R. J. Engineering adeno-associated virus vectors for gene therapy. Nature Reviews Genetics. 21 (4), 255-272 (2020).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Mullard, A. Gene therapy community grapples with toxicity issues, as pipeline matures. Nature Reviews Drug Discovery. 20 (11), 804-805 (2021).
- Nature Biotechnology. Gene therapy at the crossroads. Nature Biotechnology. 40 (5), 621 (2022).
- Becker, P., et al. Fantastic AAV Gene Therapy Vectors and How to Find Them-Random Diversification, Rational Design and Machine Learning. Pathogens. 11 (7), 756 (2022).
- Perabo, L., et al. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Molecular Therapy. 8 (1), 151-157 (2003).
- Muller, O. J., et al. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nature Biotechnology. 21 (9), 1040-1046 (2003).
- Dalkara, D., et al. In vivo-directed evolution of a new adeno-associated virus for therapeutic outer retinal gene delivery from the vitreous. Science Translational Medicine. 5 (189), (2013).
- Sahel, J. A., et al. Partial recovery of visual function in a blind patient after optogenetic therapy. Nature Medicine. 27 (7), 1223-1229 (2021).
- Adachi, K., Enoki, T., Kawano, Y., Veraz, M., Nakai, H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nature Communications. 5, 3075 (2014).
- Marsic, D., Mendez-Gomez, H. R., Zolotukhin, S. High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Molecular Therapy-Methods & Clinical Development. 2, 15041 (2015).
- Korbelin, J., et al. Pulmonary targeting of adeno-associated viral vectors by next-generation sequencing-guided screening of random capsid displayed peptide libraries. Molecular Therapy. 24 (6), 1050-1061 (2016).
- Deverman, B. E., et al. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 34 (2), 204-209 (2016).
- Ravindra Kumar, S., et al. Multiplexed Cre-dependent selection yields systemic AAVs for targeting distinct brain cell types. Nature Methods. 17 (5), 541-550 (2020).
- Hanlon, K. S., et al. Selection of an efficient AAV vector for robust CNS transgene expression. Molecular Therapy-Methods & Clinical Development. 15, 320-332 (2019).
- Nonnenmacher, M., et al. Rapid evolution of blood-brain-barrier-penetrating AAV capsids by RNA-driven biopanning. Molecular Therapy-Methods & Clinical Development. 20, 366-378 (2021).
- Tabebordbar, M., et al. Directed evolution of a family of AAV capsid variants enabling potent muscle-directed gene delivery across species. Cell. 184 (19), 4919-4938 (2021).
- Davidsson, M., et al. A systematic capsid evolution approach performed in vivo for the design of AAV vectors with tailored properties and tropism. Proceedings of the National Academy of Sciences. 116 (52), 27053-27062 (2019).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. Journal of Clinical Investigation Insight. 4 (22), (2019).
- Ogden, P. J., Kelsic, E. D., Sinai, S., Church, G. M. Comprehensive AAV capsid fitness landscape reveals a viral gene and enables machine-guided design. Science. 366 (6469), 1139-1143 (2019).
- Kondratov, O., et al. A comprehensive study of a 29-capsid AAV library in a non-human primate central nervous system. Molecular Therapy. 29 (9), 2806-2820 (2021).
- Weinmann, J., et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nature Communications. 11 (1), 5432 (2020).
- Kremer, L. P. M., et al. High throughput screening of novel AAV capsids identifies variants for transduction of adult NSCs within the subventricular zone. Molecular Therapy-Methods & Clinical Development. 23, 33-50 (2021).
- Borner, K., et al. Pre-arrayed pan-AAV peptide display libraries for rapid single-round screening. Molecular Therapy. 28 (4), 1016-1032 (2020).
- Kienle, E., et al. Engineering and evolution of synthetic adeno-associated virus (AAV) gene therapy vectors via DNA family shuffling. Journal of Visualized Experiments. (62), e3819 (2012).
- Furuta-Hanawa, B., Yamaguchi, T., Uchida, E. Two-dimensional droplet digital PCR as a tool for titration and integrity evaluation of recombinant adeno-associated viral vectors. Human Gene Therapy Methods. 30 (4), 127-136 (2019).
- Lyons, E., Sheridan, P., Tremmel, G., Miyano, S., Sugano, S. Large-scale DNA barcode library generation for biomolecule identification in high-throughput screens. Scientific Reports. 7 (1), 13899 (2017).
- Korbelin, J., et al. Optimization of design and production strategies for novel adeno-associated viral display peptide libraries. Gene Therapy. 24 (8), 470-481 (2017).
- Korbelin, J., Trepel, M. How to successfully screen random adeno-associated virus display peptide libraries in vivo. Human Gene Therapy Methods. 28 (3), 109-123 (2017).
- Herrmann, A. K., et al. A robust and all-inclusive pipeline for shuffling of adeno-associated viruses. American Chemical Society Synthetic Biology. 8 (1), 194-206 (2019).
- Choudhury, S. R., et al. In vivo selection yields AAV-B1 Capsid for central nervous system and muscle gene therapy. Molecular Therapy. 24 (7), 1247-1257 (2016).
- Buschmann, T., Bystrykh, L. V. Levenshtein error-correcting barcodes for multiplexed DNA sequencing. BMC Bioinformatics. 14, 272 (2013).
- Buschmann, T. DNABarcodes: an R package for the systematic construction of DNA sample tags. Bioinformatics. 33 (6), 920-922 (2017).
- Li, B., et al. A comprehensive mouse transcriptomic BodyMap across 17 tissues by RNA-seq. Scientific Reports. 7 (1), 4200 (2017).
- Clarner, P., et al. Development of a one-step RT-ddPCR method to determine the expression and potency of AAV vectors. Molecular Therapy-Methods & Clinical Development. 23, 68-77 (2021).
- Zolotukhin, S., Vandenberghe, L. H. AAV capsid design: A Goldilocks challenge. Trends in Molecular Medicine. 28 (3), 183-193 (2022).
- Brown, D., et al. deep parallel characterization of AAV tropism and AAV-mediated transcriptional changes via single-cell RNA sequencing. Frontiers in Immunology. 12, 730825 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved