デジタル液滴ポリメラーゼ連鎖反応による精製ベクターサンプル中のアデノ随伴ウイルスゲノムの定量(英語)
要約
アデノ随伴ウイルス(AAV)ベクターゲノムコピーの正確な定量は重要ですが、標準化されたプロトコルはまだ確立されていません。このプロトコールでは、精製されたAAVサンプルを調製し、デジタル液滴ポリメラーゼ連鎖反応(dd_PCR)を実施してウイルスゲノムの力価を確実に定量するための検証済みの方法について説明します。
要約
アデノ随伴ウイルス(AAV)は、治療遺伝子を患者に導入するための送達媒体として使用される非病原性ウイルスです。ベクター調製物におけるAAVゲノムコピー数の正確な定量化は、AAVベースの遺伝子治療製品の前臨床研究と臨床研究の両方におけるバイオプロセスの最適化と投与量計算に不可欠です。現在、AAV ウイルスゲノム滴定のコンセンサスプロトコルは不足しています。ここでは、精製されたベクターサンプル中のウイルスゲノムを定量するためのデジタルドロップレットPCR(dd_PCR)プロトコルを紹介します。サンプルはDNase Iで処理され、パッケージ化されていない汚染物質DNAが除去されます。次に、DNase処理したサンプルを適切なプライマープローブセット(標的AAVゲノムに従って設計)およびPCR試薬と混合し、液滴発生器にロードします。調製した液滴をPCRプレートに移し、PCR増幅を行い、分析します。ウイルスゲノムの力価は、サンプルの希釈分を考慮した濃度(コピー数/μL)に基づいて計算されます。測定が成功すると、正と負の液滴雲が明確に分離され、少なくとも 10,000 個の液滴が受け入れられ、10 コピー/μL から 10,000 コピー/μL の間の値が示され、繰り返し間の変動係数 (CV) が 20% 未満であることが示されました。信頼性の高いウイルスゲノム滴定は、安全で効果的なAAVベースの遺伝子治療製品の開発に役立ちます。
概要
遺伝子治療は、遺伝性疾患の治療に一般的に使用される治療法です。任意の遺伝子治療のデザインは、標的適応症の関連する病理に特異的ですが、すべての遺伝子治療は、治療効果を引き出すために、標的細胞への遺伝物質の細胞内送達を伴います1。遺伝子治療は、機能喪失型突然変異に対する遺伝子置換、機能獲得異常に対する遺伝子サイレンシング、遺伝子編集技術など、さらにいくつかのカテゴリーに分類できます。採用する特定の戦略に関係なく、標的細胞内送達を達成するためには、治療用核酸材料(導入遺伝子と呼ばれる)をベクター内にパッケージ化する必要があります2。
遺伝子治療の開発にはさまざまなウイルスおよび非ウイルスベクターシステムが利用可能ですが、このウイルス群に関連する広範なウイルス指向性と免疫原性が低いため、アデノ随伴ウイルス(AAV)が頻繁に選択されます1,2。現在までに、AAVを治療用遺伝子送達に利用する7つの遺伝子治療薬が、血友病(ロクタビアンなど)から脊髄性筋萎縮症(ゾルゲンスマなど)までの疾患を対象として、欧州医薬品庁(EMA)または食品医薬品局(FDA)から承認を得ています3。
AAVベースの遺伝子治療の作製は、野生型AAV自体の理解から生まれます。AAVは、13の主要な血清型(AAV1-13)3で構成されるパルボウイルス科の小さなDNAウイルスです。AAVゲノムは、ゲノム複製、キャプシドアセンブリ、およびパッケージング(rep、 cap)に必要な必須ウイルス遺伝子をコードする2つの主要なオープンリーディングフレーム(ORF)を含む~4.7 kbの一本鎖DNA分子で構成されています。ウイルスゲノムは、5'末端と3'末端の両方に隣接しており、逆末端反復配列(ITR)と呼ばれる回文塩基配列があります。これらのITRは、ゲノム複製や de novo ウイルスゲノムの新規合成ウイルスカプシドへのパッケージングに重要な役割を果たすヘアピンのような構造を形成します。AAVはヘルパー依存性ウイルスであるため、複製能力を持つようになるためには、単純ヘルペスウイルス(HSV)やアデノウイルス(AdV)などの他のウイルス由来の補助遺伝子を発現させる必要があります1。
AAVを作製するためには、適切な細胞ベースの発現系を用いて、ウイルスカプシドタンパク質の発現を促進し、その後のde novoウイルス粒子への集合を促進し、続いてITRに隣接する選択導入遺伝子(ベクターゲノムとも呼ばれる)のカプセル化を促進します。このプロセスでは、一般に、(1)ヘルパーウイルスに由来するヘルパー遺伝子を保有するプラスミド、(2)必須ウイルス要素をコードするプラスミド(rep/cap)、および(3)治療用発現カセットを運ぶプラスミド(一般にトランスファープラスミドと呼ばれる)4を含むトリプルプラスミドシステムを利用します。トランスファープラスミドの治療用発現カセットに隣接する逆末端反復(ITR)パッケージングシグナルのユニークな存在により、導入遺伝子の特異的パッケージングが保証され、他のプラスミドに存在するウイルス遺伝子はほとんど除外されます。この3プラスミドシステムを細胞ベースの発現プラットフォーム(通常はHEK293T細胞)に同時トランスフェクションすると、遺伝子治療アプリケーションでの使用に適した形質導入能力があり、複製欠損のあるウイルス粒子が産生されます3,4。
AAVベースの遺伝子治療の製造には、意図した医薬品の効力、純度、および安全性を確保するために評価しなければならない多くの重要な品質特性(CQA)があります4。これらのCQAには、ウイルス力価、キャプシド含有量、および凝集が含まれます。ウイルス力価自体は、任意の調製物に存在するウイルス粒子の数(キャプシド力価)とベクターゲノムの数(ベクターゲノム力価)の組み合わせです。理想的には、各ウイルス粒子が1つのベクターゲノムを含む必要があるため、これら2つの力価の比率は1:1であるべきですが、生合成中のベクターゲノムパッケージングの非効率性により、空または部分的に充填されたカプシド(部分的なベクターゲノム配列および/または非ベクターゲノム配列を含むもの)が共産生されます5。このような不純物の存在は、不当な免疫応答を引き起こし、ベクター結合部位をめぐって競合する可能性があり、それによって免疫毒性のリスクを高め、フルカプシドの形質導入速度を低下させる可能性がある6。したがって、AAVゲノムの正確な定量は、ウイルスの力価とキャプシド含有量を確立するために不可欠です。これは、基礎研究だけでなく、医薬品の安全性と有効性を維持するために正確な投与量を必要とする遺伝子治療業界にも影響を及ぼします。
デジタル液滴ポリメラーゼ連鎖反応(dd_PCR)は、任意の調製物中に存在するベクターゲノムの数を決定するために利用できるため、ウイルス力価の定量と密接に関連しています7。デジタルPCR自体は1990年代に初めて導入されました8,9 dd_PCR、この技術を強化してハイスループットのサンプル処理を可能にしました10,11。dd_PCRでは、20 μL のリアルタイム PCR 反応液滴が約 20,000 個の油で包まれた液滴に分割され、標準的な 96 ウェルプレートに収容すると最大 96 回の反応が得られます。従来の定量PCR(qPCR)と比較して、dd_PCRには、感度の向上、精度の向上、標準曲線を必要とせずにターゲット配列のより直接的かつ絶対的な定量化など、いくつかの利点があります。さらに、dd_PCRでの高レベルのパーテーションにより、PCR阻害剤の影響が軽減され、特定のテンプレートの優先的増幅によるバイアスの可能性が最小限に抑えられるため、ベクターゲノム滴定の分析測定に魅力的なオプションとなっています。
プロトコル
ここで説明するプロトコールは、導入遺伝子12 として緑色蛍光タンパク質(GFP)を含む精製された自社製AAV9ベクターのウイルスゲノム力価を高い精度で定量するように設計されています(表1)。ただし、このプロトコルは、プライマー/プローブセットが目的の特定のベクターゲノムを標的とするように設計されている場合、任意のAAV血清型および任意のベクターゲノム設計に適用できます。この研究で使用した試薬、プライマー、プローブ、および機器の詳細は、 材料表に記載されています。
1. 原液の調製
注:外来DNAによる汚染を避けるために、PCRワークステーションでサンプル希釈に必要なすべてのストック溶液を調製してください。
- 500 mMのKCl、100 mMのTris-HCl、15 mMのMgCl2 、および0.01 %(w/v)BSAからなる10x PCRアッセイバッファーを調製します。溶液のpHを測定し、8.3に調整します。
注:この溶液は、室温で最大1か月間保存できます。 - 1x PCR assay buffer、0.2 ng/μL salmon sperm sheared (sss) DNA、および 0.1 % pluronic F-68 からなる AAV 希釈バッファーを調製します。この溶液は各アッセイで新たに調製し、4°Cに保ちます。
注:プルロン酸F-68が利用できない場合は、Poloxamer 188非イオン性界面活性剤などの代替品を使用できます。
2. サンプルの調製 - DNase I処理と段階希釈
注:ベクターゲノム滴定の精度を低下させる可能性のあるDNA汚染物質を除去するために、AAVサンプル内に存在する遊離DNA(残りのプラスミドDNAまたはカプセル化されていないウイルスゲノムを含む)を、DNaseを使用してPCR増幅する前に除去することができます。これが可能なのは、ベクターゲノムがAAVキャプシド内にカプセル化されているため、PCR反応自体の変性ステップまでアクセスできないためです(ステップ5を参照)。また、どのサンプルのベクターゲノム番号も不明なため、検出可能な上限と下限の範囲内に収まるように、サンプルを段階希釈する必要があります。すべてのサンプル操作はPCRワークステーションで行います。特に指定がない限り、すべてのサンプルを氷の上に置いてください。
- 分析用に選択したAAVサンプルを短時間ボルテックスして混合し、次に遠心分離して、サンプルを含むチューブの底にすべての液体が残るようにします。
- DNase処理では、DNaseフリー水に1x DNase Iバッファー、0.1%プルロン酸F-68、および0.04 U/μL DNase Iを含む溶液45 μLを0.2 mL PCR 8チューブストリップチューブに加えます。
- 5 μLのサンプルをDNAse溶液の入ったチューブに移します。サンプルをボルテックスし、短時間遠心分離して、すべての液体が底に残るようにします。
- サーモサイクラーで、サンプルを37°Cで1時間インキュベートした後、4°Cまで冷却します。 4°Cに達したらできるだけ早く氷の上に置きます。
- 新しい0.2 mL PCR 8チューブストリップを使用して、DNase処理したサンプルをAAV希釈バッファーで適切に希釈します。推奨されるサンプル希釈は、予想されるvg力価によって異なりますが(表2)、サンプルごとに少なくとも2つの異なる希釈を目指し、各希釈を重複して実行します。ボリュームを取り出す前に、サンプルを厳密に(2,000〜3,000 rpm)5秒間ボルテックスしてください。
- 段階希釈後、サンプルをボルテックスして遠心分離し、すべての液体がチューブの底に残っていることを確認します。
- vg力価が既知であるポジティブコントロールと、テンプレートなしコントロール(NTC)とも呼ばれるネガティブコントロール(AAV希釈バッファーのみ)が含まれます。
注:ポジティブコントロールは、プライマー/プローブの標的配列を含む、自社製のAAVまたは市販のAAV標準物質にすることができます。
3. マスターミックスdd_PCR調製
注:ベクターゲノム内の適切な標的配列を選択し、公開されたガイドライン13に従ってFAMまたはHEXレポーター蛍光色素で標識されたフォワードプライマー、リバースプライマー、および加水分解プローブを設計します。導入遺伝子はベクターゲノムデザインに特異的であり、ITRのヘアピン形成に関連する二次構造が効率的なプライマー結合を妨げる可能性があるため、ITRを標的とするプライマーよりも導入遺伝子を標的とするプライマーが好まれます。さらに、ITRプライマーは、切断されたベクターゲノム断片を含む部分的に充填されたカプシドが存在し、ITR配列のいずれかをまだ保持している場合、AAV力価を過大評価する可能性があります14。dd_PCRマスターミックスを専用の独立したワークステーション(PCR前室)で調製します。この部屋は、相互汚染を避けるために、サンプルが準備される部屋とは分離する必要があります。
- プライマー、プローブ、およびdd_PCRスーパーミックス試薬を室温(20〜25°C)まで待ちます。その後、すべての試薬をボルテックスし、チューブを短時間遠心分離して、チューブの底にすべての液体を収集します。
- 表 3 に記載されている容量と濃度に従って、フォワードプライマーとリバースプライマー、プローブ、dd_PCRスーパーミックス、および DNase-free 水を組み合わせて、必要な反応数に応じて、別のマイクロ遠心チューブに dd_PCR マスターミックスの量を調製します。
- マスターミックスをボルテックスし、チューブを短時間遠心分離して、すべての液体がチューブの底に残っていることを確認します。
注:ピペッティング容量の損失を考慮するために、常に必要量(n + 1)よりも余分なdd_PCRマスターミックス容量を準備してください。
- マスターミックスをボルテックスし、チューブを短時間遠心分離して、すべての液体がチューブの底に残っていることを確認します。
- 19.8 μLのマスターミックスを、新鮮な0.2 mL PCR 8チューブストリップ上の各チューブに移します。
4.液滴の生成
注:サンプルとdd_PCRマスターミックスは、液滴発生カートリッジにロードする前に別々に混合されます。アンプリコンの汚染を避けるために、PCRワークステーション、できればサンプルが調製された場所とは異なるPCRワークステーションですべての操作を実行します。または、サンプルを調製した後、PCRワークステーションを完全に洗浄します。
- 希釈したサンプル2.2 μLを、PCR 8チューブストリップの各チューブ内の19.8 μLの容量のdd_PCRマスターミックスに移し、よく混合します。ボルテックスし、短時間遠心分離して、すべての液体が底に留まるようにします。
- この溶液20μLを、液滴発生カートリッジの中央の「サンプル」列に含まれるウェルに移します。液滴発生カートリッジ内の気泡を避けてください。これにより、機器がエラーを発し、液滴の生成に失敗する可能性があります。
注:カートリッジはPCRチューブストリップと同様に8ウェル形式であるため、8チャンネルピペットを使用することをお勧めします。 - 液滴発生カートリッジの下部の「油」列に含まれるウェルに60μLの液滴発生油を移します。
注意: 液滴発生カートリッジのウェルを空のままにしないでください。空のサンプルウェルにプローブ用のdd_PCRバッファーコントロールを充填します(上記の対応する容量を使用)。 - 液滴発生カートリッジにゴム製のガスケットをかぶせ、液滴発生器に入れます。
- 液滴が生成されたら、8チャンネルピペットを使用して、dd_PCRチップから42.5μL溶液をマルチウェルPCRプレートにゆっくりと移します。
注:ピペッティング中に液体中の液滴がせん断しないように、ゆっくりと45度の角度でピペットします。プレートを目視検査して、すべてのウェルに同じ量の液体が充填され、液滴がウェル内の不透明な層として見えるようにします。 - ヒートシール機のアルミホイルカバーでプレートを180°Cで5秒間シールします。
注:すべてのウェルが密封されていることを目視で確認します。そうでない場合は、180°Cでさらに5秒繰り返します。 密閉されたプレートは、サーマルサイクラーでdd_PCR増幅する前に、4°Cで最大4時間保持できます。
5. dd_PCR増幅
注:サーマルサイクラーは、PCR前の活動をPCR増幅から空間的に分離し、アンプリコン汚染による偽陽性の結果を避けるために、PCR前の部屋とは別の部屋に配置する必要があります。
- プレートをサーマルサイクラー(96ディープウェル反応モジュール付き)に置き、PCR増幅を行い、しっかりと閉じます。
- 表4に記載されている条件で、反応量40 μLで動作するようにサーモサイクラーを設定します。プログラム設定に予熱ステップを含め、蓋が99°Cに加熱されていることを確認します。
- PCRプログラムが終了したら、プレートを4°Cに少なくとも15分間保持して、プレートが完全に冷却されていることを確認します。
注:プレートは、液滴を読み取る前に4°Cで48〜72時間保持できます。
6.液滴の読み取り
- プレートを液滴リーダーにセットし、システムソフトウェアに次の情報を入力して、読み取りを続けます。
注:実験タイプ=直接定量;Supermix = プローブのdd_PCRスーパーミックス(dUTPなし)。アッセイタイプ = チャネルごとに 1 つのターゲット。ターゲット情報、信号チャネル1 = FAMチャネルまたはHEXチャネルは、使用するプローブによって異なります。 - サンプルに注釈を付けて、各サンプルの位置をデータファイルから効果的に特定できるようにします。
7. データ分析
- 液滴リーダーの製造元が提供する互換性のあるソフトウェアを使用してデータ分析を実施します。
- システム適合性試験を実施して、アッセイの全体的な性能(したがって信頼性)を評価します。
- PCRプレート上の各ウェルについて測定されたイベントカウント(液滴数)を確認します。イベント数は 15,000 から 20,000 の間が理想的です。イベント数が <10,000 の場合、この値は分析から除外する必要があります。
- 1Dまたは2Dの振幅プロットを評価し、液滴の雨の存在を確認します。
- [データ テーブル] タブに移動し、データを Excel ファイルにエクスポートします。
- 以下の手順に従ってvg力価を計算します。
- dd_PCRの作業範囲は10-10,000コピー/μLで、<10コピー/μLの値を分析から除外します。ここで使用するdd_PCR機器の推奨範囲は、25〜5000コピー/μLです。
注:データ解析ソフトウェアは、PCR反応量(40μL)に応じたベクターゲノム番号を提供しますが、これはサンプル調製中に適用される希釈係数を考慮していません。 - 以下の関係を使用して、サンプル調製全体のさまざまな希釈係数を考慮に入れることにより、容量あたりのサンプルvg力価(通常はvg/mLで表されます)を計算します。
注:濃度(データ解析から得られた値)×10(DNase I処理前希釈)×10(PCRマスターミックス希釈)×1000(μL〜mL)選択したサンプル希釈(AAV希釈バッファー内 - 表2など)。 - 反復間および同じサンプルの異なる希釈間の変動係数(%CV)を計算します。
注:%CVは、標準偏差を平均で割って計算されます。異なる希釈間の%CVが>20%の場合、その値は正確であると見なすべきではありません。 - ネガティブコントロールとポジティブコントロールの値を確認します。ネガティブコントロールは5コピー/μL未満である必要があります。
- dd_PCRの作業範囲は10-10,000コピー/μLで、<10コピー/μLの値を分析から除外します。ここで使用するdd_PCR機器の推奨範囲は、25〜5000コピー/μLです。
代表的な結果
結果は、適切なソフトウェアを使用して視覚化できます。各ウェルについて、1D振幅プロットには、その振幅に対するすべての液滴が表示されます。正の液滴と負の液滴が明確に分離されることが期待されます。プラスとマイナスの液滴雲の間に全液滴の10%以上が見つかった場合(液滴雨と呼ばれる現象)、サンプルを再測定する必要があります( 図1を参照)。水滴雨に関する追加情報は、ディスカッションで入手できます。
サンプル名、許容された液滴の数(イベントカウント)、濃度(コピー数/μL)など、記録されたすべての情報を要約するデータテーブルを作成できます。理想的には、イベント数は 15,000 から 20,000 の許容された液滴の間に収まる必要があります。井戸のイベント数が 10,000 未満の場合は、データ ポイントを分析から除外する必要があります。出力データの例を 表 5 に示します。
vg力価は、サンプルの希釈率を考慮して、濃度(コピー/μL)に基づいて計算できます。dd_PCRの作業範囲は10〜10,000コピー/μLです。10コピー/μL未満の値は分析から除外する必要があります。サンプルのvg力価、および陽性コントロールとネガティブコントロールを計算する必要があります。ポジティブコントロールの測定値は、理論値に対して20%未満の変動係数(%CV)を持つ必要があります。ネガティブコントロールは5コピー/μL未満である必要があります。さらに、各サンプルのリピート間および異なる希釈間の%CVを計算する必要があります。異なる希釈液間の%CVが20%を超えると、値は不正確であると見なされ、サンプルの再測定が必要になる場合があります。
測定が成功するとは、正と負の液滴雲が明確に分離されていること、少なくとも10,000個の液滴が受け入れられていること、10コピー/μLから10,000コピー/μLの間の値、および繰り返し間の%CVが20%未満であることが特徴です。
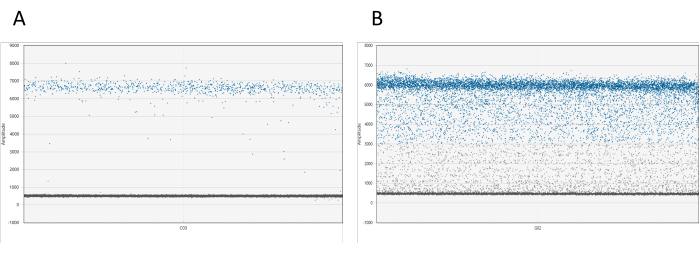
図1:dd_PCR液滴の可視化 (A)1D振幅プロットは、正と負の液滴が明確に分離されていることを示しており、液滴の分配が成功したことを示しています。(B)プロットは、液滴雨として知られる正と負の液滴の分離が不十分であることを示しており、これは最適でない分割またはアッセイの潜在的な問題を示唆しています。 この図の拡大版を表示するには、ここをクリックしてください。
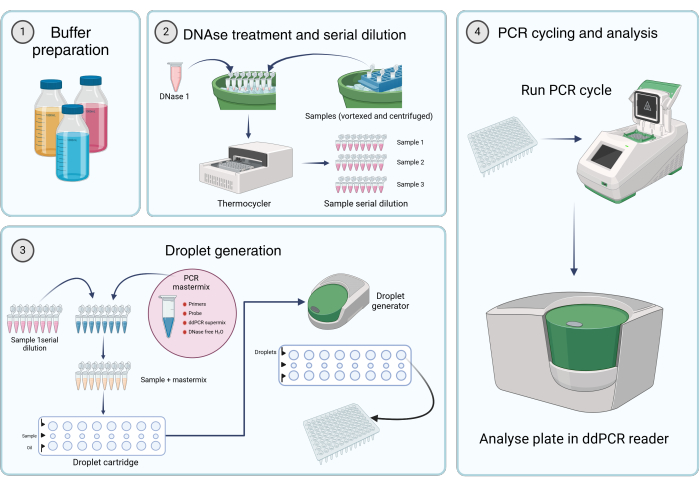
図2:デジタル液滴PCRを使用してウイルスゲノムを定量するためのワークフロー (1)バッファー、試薬、および溶液は、製造元の指示に従って調製するか、プロトコルのステップ1(ストック溶液の調製)に従って調製します。(2)45 μLのDNase I溶液を8チューブPCRストリップの各チューブに分注します。サンプルをボルテックスして短時間遠心分離した後、5 μLのサンプルをチューブの1つに加えます。DNase Iとサンプル含有8チューブPCRストリップをサーモサイクラーで37°Cで1時間インキュベートした後、サンプルを段階希釈します。(3)PCRマスターミックスを記載通りに調製し、19.8μLを8チューブPCRストリップの各チューブに分注します。ステップ2のサンプル連続希釈液をマスターミックスに加えます。20 μL のマスターミックスプラス
サンプル溶液をカートリッジの中央列にロードし、60μLの液滴発生オイルを液滴発生カートリッジの下部ウェルに移します。次に、カートリッジを液滴発生器に入れ、指定された条件に従って実行します。液滴生成後、カートリッジの最上列から42.5 μLをマルチウェルPCRプレートに移します。(4)PCRプレートをPCRサーモサイクラーにロードし、所定の条件に従って運転します。プレートはdd_PCRリーダーを使用して分析されます。 この図の拡大版を表示するには、ここをクリックしてください。
表 1: 精度データ。 この表は、4セットの品質管理(QC)サンプルから得られた精度データを示しており、それぞれに5つの濃度(QC1-QC5)があります。各QCを5回測定しました。異なる実行間の変動係数(%CV)は繰り返し性(A)を表し、異なるサンプルセット間の変動係数(%CV)は中間精度(B)を表します。 この表をダウンロードするには、ここをクリックしてください。
表2:サンプル希釈。 この表は、予想されるウイルスゲノム力価(vg/mL)に基づく推奨サンプル希釈を示しています。少なくとも2つの値がアッセイの作業範囲内に収まるように、合計3回の希釈が推奨されます。 この表をダウンロードするには、ここをクリックしてください。
表3:dd_PCRマスターミックスの構成。 この表は、フォワードプライマーとリバースプライマー(909 nM)、プローブ(227 nM)、プローブ用のdd_PCRスーパーミックス(dUTPなし、1x)を含むdd_PCRマスターミックスの組成を示しています。 この表をダウンロードするには、ここをクリックしてください。
表4:熱サイクル条件。 この表には、推奨されるPCRプログラムの詳細が示されており、これには、(1)95°Cで10分間のインキュベーションによるキャプシドの破壊と酵素の活性化、(2)DNA変性のための94°Cでの30秒のサイクル40サイクル、アニーリングと伸長のための60°Cでの1分間、(3)酵素の不活性化のための98°Cでの10分間のインキュベーション、 (4)4°Cで保持します。 アニーリング温度は、使用するプライマー/プローブセットに基づいて最適化が必要な場合があります。 この表をダウンロードするには、ここをクリックしてください。
表 5: 実際の AAV 実行からの出力データの例。 この表は、1 つのサンプルが 2 つの異なる希釈率で、それぞれが重複して測定された実際の AAV 実行からの出力データの例を示しています。ウイルスゲノム(vg)力価(vg/mL)は、10(DNase I前処理)×10(マスターミックスでの希釈)×1,000(μLからmL)×AAV希釈バッファーでの希釈という式で計算されます。STDEVは標準偏差を表し、CVは変動係数を示します。 この表をダウンロードするには、ここをクリックしてください。
ディスカッション
ベクター調製物におけるAAVベクターゲノムコピーの正確な定量は、AAVベースの遺伝子治療製品の開発に不可欠です。vg力価を決定するためのいくつかの方法が存在し、定量的PCR(qPCR)とdd_PCRが最も広く使用され受け入れられている2つの技術です。dd_PCRは、増幅効率からの独立性、より高い精度、およびより高い堅牢性のために、qPCRよりもしばしば好まれます15。dd_PCRによるベクターゲノム滴定のための多数のプロトコルが文献で利用可能であり、それぞれが独自のサンプル調製方法15,16,17を有する。しかし、適格なコンセンサスプロトコルが不足しています。この記事では、精製されたベクターサンプル中のAAVベクターゲノムを定量するための、目的に適合した検証済みのdd_PCRプロトコルを紹介します(図2)。
このプロトコルを実行する際には、サンプルの取り扱いに細心の注意を払うことが重要です。クロスコンタミネーションは、vg力価を正確に評価する上で大きな課題となる可能性があります。したがって、サンプルはPCRワークステーションで処理するのが最適です。これは、外来DNAによる汚染を避けるためです。さらに、DNaseインキュベーションを適切に実行することは、キャプシドを破壊したり、パッケージ化されたベクターゲノムを消化したりすることなく、パッケージ化されていない汚染物質DNAを排除するために重要です。多くのプロトコルには、熱不活性化とプロテイナーゼK治療16,17,18が含まれます。しかし、社内でのメソッド開発中に、過度の加熱はvg滴定に有害であり、プロテイナーゼKの処理は不要であることがわかりました(データは示されていません)。
ウイルスゲノムの定量化dd_PCRますます人気が高まる中、メーカーはdd_PCRアッセイの設計と最適化に関するガイドラインを含む詳細なアプリケーションガイドを公開しています19。トラブルシューティング情報もご利用いただけます。実際には、アッセイが正しく設計されている場合、最も頻繁に発生する問題には、液滴の雨と液滴数の減少が含まれます。液滴の雨は、プライマー/プローブがアンプリコンにアクセスできないことが原因であることがよくありますが、いくつかの戦略でこの問題に対処できます。たとえば、温度勾配実験は、最適なアニーリング温度を確立するのに役立ちます。さらに、サンプルを希釈してDNA量を減らす(66 ng未満を推奨)か、アンプリコン領域の外側を切り取る特定の酵素で制限消化を行うと、テンプレートのアクセシビリティを向上させることができます。通常、DNAのμgあたり10Uの制限酵素の推奨が効果的です。液滴数が少ない(10,000液滴未満)という問題は、多くの場合、サンプルとカートリッジ内のオイルのピペッティングが不十分であることが原因です。液滴のせん断を避けるために、適切なチップを使用してゆっくりとピペッティングすることをお勧めします。dd_PCRアッセイは堅牢ですが、1つの制限は結果が出るまでの時間が長いことです。サンプル処理から液滴読み取りまでのプロセスには数時間かかるため、迅速に結果が必要な場合に欠点となる可能性があります。
ベクター調製におけるAAVゲノムコピーの正確な定量は、AAV遺伝子治療のライフサイクルのすべての段階で必要です。具体的には、AAVベースの遺伝子治療製品の生産および精製プロセスの最適化、前臨床試験の実施、および臨床投与量の決定に重要です。ここで紹介するdd_PCRプロトコルは広く適用でき、さまざまな血清型や導入遺伝子を持つ精製AAV製品に使用できます。
開示事項
IVH、BM、EHはTavira Therapeuticsの共同設立者です。他の著者は、利益相反を宣言しません。
謝辞
この研究は、「欧州復興・レジリエンス・ファシリティ」(RRF)(VV021/13)に端を発するフランダース政府からの「フランダース・レジリエンス」補助金によって可能になった。 図 2 は Biorender.com を使用して作成されています。
資料
| Name | Company | Catalog Number | Comments |
| 8-channel pipette 10 µL | Eppendorf | 3,12,50,00,010 | |
| 8-channel pipette 200 µL | Eppendorf | 3,12,50,00,036 | |
| 8-channel pipette 300 µL | Eppendorf | 3,12,50,00,052 | |
| 8-well PCR strip | Sarstedt | 72.991.002 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A9418 | |
| C1000 Touch Thermal Cycler with 96–Deep Well Reaction Module | Bio-Rad | 1851197 | |
| ddPCR Buffer Control for Probes 9 mL (2 x 4.5 mL) | Bio-Rad | 1863052 | |
| ddPCR Supermix for Probes (No dUTP) (2 x 1 mL) | Bio-Rad | 1863023 | |
| ddPCR 96-Well Plates (pkg of 25) | Bio-Rad | 12001925 | |
| ddPCR Droplet Reader Oil (2 x 1L) | Bio-Rad | 1863004 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges for QX200/QX100 (pkg of 24) | Bio-Rad | 1864008 | |
| DG8 Gaskets for QX200/QX100 (pkg of 24) | Bio-Rad | 1863009 | |
| DNase I (10U/µL) + buffer | Roche | 4716728001 | |
| Droplet Generation Oil for Probes (10 x 7 mL) | Bio-Rad | 1863005 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 0.1-10 μL, 34 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078500 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 20-300 μL, 55 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078560 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 2-100 μL, 53 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078543 | |
| Forward lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Forward primer: 5'-GAACGGCATCAAGGTGAACT-3' | |
| Lyopohilized probe and master stock at 100 µM | IDT | GFP as target sequence. PrimeTime Eco Probe: /56-FAM/CAAGATCCG/ZEN/CCACAACATCGAGGA/3IABkFQ/ | |
| Magnesium Chloride (MgCl2) | Chemlab Analytical | CL00.1381 | |
| Nuclease free water | IDT | 11-04-02-01 | |
| PCR Plate Heat Seal, foil, pierceable (pkg of 100) | Bio-Rad | 1814040 | |
| Pluronic F-68 non-ionic surfactant (100x) | Thermo Fisher Scientific | 24040032 | |
| Potassium Chloride (KCl) | Honeywell research chemicals | 31248 | |
| QX manager software | Bio-Rad | Software to analyse ddPCR data | |
| QX200 Droplet Generator | Bio-Rad | 1864002 | |
| QX200 Droplet Reader | Bio-Rad | 1864003 | |
| Reagent reservoir | VWR | 613-1181 | |
| Reverse lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Reverse primer: 5'-TGCTCAGGTAGTGGTTGTCG-3' | |
| SafeSeal reaction tube, 1.5 mL | Sarstedt | 72.706.200 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Thermo Fisher Scientific | AM9680 | |
| TE buffer | IDT | Accompanied by primers when ordering | |
| Tris hydrochloride (Tris-HCl) | Roche | 10812846001 |
参考文献
- Balakrishnan, B., Jayandharan, G. R. Basic biology of adeno-associated virus (AAV) vectors used in gene therapy. Curr Gene Ther. 14 (2), 86-100 (2014).
- Srivastava, A. In vivo tissue-tropism of adeno-associated viral vectors. Curr Opin Virol. 21, 75-80 (2016).
- Pupo, A., et al. AAV vectors: The Rubik's cube of human gene therapy. Mol Ther. 30 (12), 3515-3541 (2022).
- Gimpel, A. L., et al. Analytical methods for process and product characterization of recombinant adeno-associated virus-based gene therapies. Mol Ther Methods Clin Dev. 20, 740-754 (2021).
- Brimble, M. A., et al. Preventing packaging of translatable P5-associated DNA contaminants in recombinant AAV vector preps. Mol Ther Methods Clin Dev. 24, 280-291 (2022).
- Wright, J. F. Product-related impurities in clinical-grade recombinant AAV vectors: Characterization and risk assessment. Biomedicines. 2 (1), 80-97 (2014).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, (2019).
- Sidransky, D., et al. Identification of ras oncogene mutations in the stool of patients with curable colorectal tumors. Science. 256 (5053), 102-105 (1992).
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proc Natl Acad Sci USA. 96 (16), 9236-9241 (1999).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Anal Chem. 83 (22), 8604-8610 (2011).
- Pinheiro, L. B., et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Anal Chem. 84 (2), 1003-1011 (2012).
- Sanmiguel, J., Gao, G., Vandenberghe, L. H. Quantitative and digital droplet-based AAV genome titration. Methods Mol Biol. 1950, 51-83 (2019).
- . Planning droplet digital PCR experiments Available from: https://www.bio-rad.com/en-in/life-science/learning-center/introduction-to-digital-pcr/planning-ddpcr-experiments (2024)
- Dorange, F., Le Bec, C. Analytical approaches to characterize AAV vector production & purification: Advances and challenges. Cell Gene Ther Insights. 4 (2), 119-129 (2018).
- Lock, M., et al. Absolute determination of single-stranded and self-complementary adeno-associated viral vector genome titers by droplet digital PCR. Hum Gene Ther Methods. 25 (2), 115-125 (2014).
- Prantner, A., Maar, D. Genome concentration, characterization, and integrity analysis of recombinant adeno-associated viral vectors using droplet digital PCR. PLoS One. 18 (1), e0280242 (2023).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, 1570 (2019).
- Suoranta, T., Laham-Karam, N., Yla-Herttuala, S. Optimized protocol for accurate titration of adeno-associated virus vectors. Hum Gene Ther. 32 (19-20), 1270-1279 (2021).
- Droplet digital PCR applications guide. Bulletin 6407 Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6407.pdf (2024)
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
JoVEについて
Copyright © 2023 MyJoVE Corporation. All rights reserved