모델 멤브레인에서의 Dynamic light-induced protein pattern
요약
여기에서는 인공 지질막에서 높은 시공간 정밀도로 광 조절 및 가역성 단백질 패턴을 생성하기 위한 프로토콜에 대해 설명합니다. 이 방법은 청색광 아래에서 파트너 단백질 Nano(야생형 SspB)에 결합하는 모델 멤브레인에 고정된 단백질 iLID(Improved Light-Inducible Dimer)의 국부적인 광활성화로 구성됩니다.
초록
특정 시간에 세포막에서 단백질의 정확한 위치 파악과 활성화는 세포 분극, 이동 및 분열을 포함한 많은 세포 과정을 일으킵니다. 따라서, 세포 내 해상도와 높은 시간 제어를 가진 막을 모델링하기 위해 단백질을 모집하는 방법은 합성 세포에서 이러한 과정을 재현하고 제어할 때 필수적입니다. 여기에서는 높은 시공간 정밀도로 지질막에서 광 조절 가역적 단백질 패턴을 제작하는 방법에 대해 설명합니다. 이를 위해 당사는 지지된 지질 이중층(SLBs)과 거대 단층 소포(GUV)의 외막에 광전환 가능한 단백질 iLID(개선된 광 유도 이량체)를 고정합니다. 국소 청색광 조명 시 iLID는 파트너 Nano(야생형 SspB)에 결합하고 용액에서 Nano에 융합된 관심 단백질(POI)을 멤브레인의 조명 영역으로 모집할 수 있습니다. 이 바인딩은 어둠 속에서 되돌릴 수 있으며, POI의 동적 바인딩 및 해제를 제공합니다. 전반적으로, 이것은 청색광을 사용하여 공간과 시간에서 높은 정밀도로 단백질의 국소화를 조절하는 유연하고 다재다능한 방법입니다.
서문
세포 내 영역 내 세포막에 단백질 패턴이 형성되면 이동, 분열 및 국소적인 세포 간 통신을 포함한 수많은 생물학적 과정이 발생합니다 1,2. 이러한 단백질 패턴은 공간과 시간에 따라 조절되며 매우 동적입니다. 합성 세포에서 이러한 단백질 패턴을 복제하는 것은 그들로부터 발생하는 세포 과정을 모방하고 이러한 조절이 분자 수준에서 어떻게 작용하는지 더 잘 이해하는 데 필수적입니다. 살아있는 세포의 막에서 관찰되는 것과 유사하게, 인공 막에서 단백질 패턴을 생성하는 방법은 그 역학을 포착하고 정확한 시공간 제어를 제공해야 합니다.
다양한 자극 중에서 빛은 최고의 시공간 제어와 몇 가지 추가 이점을 제공하는 것으로 두드러집니다3. 빛으로 조절을 통해 원하는 시간에 타의 추종을 불허하는 정밀도로 원하는 영역을 간단하게 비출 수 있습니다. 또한 빛은 빛의 강도와 펄스 지속 시간을 모두 조정할 수 있으므로 높은 조정 가능성을 제공합니다. 또한 가시광선은 단백질을 포함한 생체 분자에 무해하며 파장이 다른 여러 기능을 처리할 수도 있습니다. 따라서, 가시광선에 기초한 빛-반응성 접근법은 공간과 시간에서 단백질 패턴의 제어되고 이중직교적인 조절을 위한 유망한 방법으로 부상하고 있다 4,5,6. 광유전학에서 광전환 가능한 단백질 쌍을 활용하여 광 유도성 이합체화 역할을 하는 것은 특정 단백질을 막에 모집하는 간단한 방법을 제공합니다. 특히, iLID(Avena sativa의 광전환 가능한 LOV2 도메인을 기반으로 하는 개선된 광 유도 이량체)와 Nano(야생형 SspB)7,8 간의 청색광 유발 상호작용, 청색광 유도 SpyTag 시스템(BLISS)9, 청광 반응 단백질 사량체 CarH10 및 PhyB와 PIF611 사이의 적색광 유도 상호작용을 사용하여 인공 막에서 단백질 패턴을 성공적으로 형성했습니다.
iLID와 Nano5 사이의 광전환 가능한 상호 작용은 청색광7을 사용하여 모델 멤브레인의 단백질을 광 패터닝하는 데 사용할 수 있음이 입증되었습니다. iLID/Nano 상호 작용은 어두운 곳에서 가역적이고 매우 특이적이며 생리학적 조건에서 작동합니다. GUV(Giant Unilamellar vesicles) 또는 SLB(Supported Lipid Bilayers)와 같은 지질막 모델에 iLID를 고정하면 이러한 막에 나노를 광 조절식으로 모집할 수 있으며, 이는 어둠 속에서 가역적입니다. 특히, 모델 지질막에 대한 테더로 iLID의 N-말단에 무질서한 도메인을 도입하여(disiLID라는 단백질이 생성됨) 나노 모집 효율성과 회귀 역학을 향상시키는 것을 관찰했습니다8.
disiLID/Nano 상호 작용을 사용하여 SLB 및 GUV의 외부 멤브레인에서 나노 융합 관심 단백질(POI)의 고대비 패턴을 생성하는 방법을 개발했습니다. 이 방법을 사용하면 몇 분 내에 놀라운 공간 및 시간 분해능과 높은 가역성을 가진 단백질 패턴을 생성할 수 있습니다. 상세 프로토콜은 인공 멤브레인에 단백질을 국소적으로 모집하는 과정을 간략하게 설명합니다. 특히, 이는 비오틴-스트렙타비딘(SAv) 상호작용을 통해 SLB 및 GUV에서 disiLID의 비오틴화된 버전을 고정함으로써 달성됩니다. 그 후, 형광 표지된 나노(mOrange-Nano)가 청색광 조명 하에서 이러한 disiLID 기능성 멤브레인에 모집됩니다. 당사의 실험 프로토콜은 멤브레인에 국소적인 단백질 모집을 달성하기 위한 간단하고 적응 가능한 접근 방식을 제공합니다. 중요한 것은 이 방법론이 보고된 SLB 및 GUV 인터페이스 또는 mOrange-Nano에만 국한되지 않는다는 것입니다. 그것은 Nano에 융합된 다른 disiLID 기능화된 물자 및 단백질로 확장될 수 있습니다.
프로토콜
1. 실험 준비
- 이전에 보고된 절차 7,8에 따라 biotinylated-disiLID(b-disiLID) 및 mOrange-Nano(재료 표 참조)를 발현하고 정제합니다.
- 선택한 지질 조성 및 농도의 유리 바이알에 지질 혼합물을 준비합니다. 먼저 클로로포름에 지질을 용해시켜 1mg/mL 농도의 최종 지질 용액을 얻습니다.
- 94.9 mol % 2- dioleoyl-sn- glycero-3- 포스 포 콜린 (DOPC), 5 mol % 1,2- 디오 레오일 -sn - 글리세로 -3- 포스포에탄올 아민 N- (캡 비오티닐) 나트륨 염 (DOPE-biotin) 및 0.1 mol % 1,1'- 디옥타 데실 -3,3,3 ', 3'- 테트라 메틸 인도 디카르보시아닌 (DiD) ( 재료 표 참조).
참고: 지질 혼합물은 다양한 비율과 DOPE-비오틴 농도 및/또는 다른 멤브레인 염료로 조정할 수 있습니다. 권장되는 DOPE-비오틴 농도(5 mol%)는 다음 단계에서 고밀도 스트렙타비딘(SAv) 층을 형성할 수 있습니다.
- 94.9 mol % 2- dioleoyl-sn- glycero-3- 포스 포 콜린 (DOPC), 5 mol % 1,2- 디오 레오일 -sn - 글리세로 -3- 포스포에탄올 아민 N- (캡 비오티닐) 나트륨 염 (DOPE-biotin) 및 0.1 mol % 1,1'- 디옥타 데실 -3,3,3 ', 3'- 테트라 메틸 인도 디카르보시아닌 (DiD) ( 재료 표 참조).
- 이전에 보고된 방법 7,8,12에 따라 작은 단층 소포(SUV)를 준비합니다. 이 단계에서는 직경이 ≤100nm인 SUV를 준비하는 것이 좋습니다.
- 이 연구에서는 초음파 처리 방법이 사용됩니다. 먼저 유리 바이알의 클로로포름 용액을 질소 흐름으로 증발시키면서 바이알을 회전시켜 얇은 지질막을 형성합니다. 다음으로, 진공 상태에서 최소 1시간 동안 잔류 클로로포름을 제거합니다.
- 소용돌이를 통해 최종 농도 1mg/mL 지질의 최종 농도인 초순수에 건조된 필름을 재수화합니다. 마지막으로, 불투명 용액이 투명해질 때까지 얻은 용액을 10분 동안 초음파 처리합니다.
알림: 지질 혼합물을 미세 원심분리기 튜브에 담아 냉장고에 최대 2주 동안 보관하십시오. 최종 SUV의 크기가 100nm ≤인 경우 다양한 SUV 준비 방법(예: 압출 방법)도 사용할 수 있습니다.
- 소용돌이를 통해 최종 농도 1mg/mL 지질의 최종 농도인 초순수에 건조된 필름을 재수화합니다. 마지막으로, 불투명 용액이 투명해질 때까지 얻은 용액을 10분 동안 초음파 처리합니다.
2. disiLID 기능화 SLB에 대한 mOrange-Nano 모집
- μ-슬라이드 18웰 유리 바닥 챔버( 재료 표 참조)의 각 웰에 150μL의 2M NaOH를 추가하고 실온에서 1시간 동안 배양합니다. 그 후, NaOH를 제거하고 우물을 3-5 번 세척하십시오 먼저 150 μL 초순수로 세척 한 다음 10 μL 완충액 (10 mM Tris pH 7.4, 100 mM NaCl)으로 10 mM CaCl2 를 함유합니다.
- 갓 준비된 SUV 15μL(스톡 농도 1mg/mL)를 10mM CaCl2 와 함께 150μL 버퍼가 들어 있는 웰에 추가하여 버퍼에서 SUV의 약 10배 희석을 합니다. SUV를 실온에서 30분 동안 배양합니다. 배양 시간 후, 비오틴화 SLB가 형성됩니다.
- 먼저 용액을 제거하고 각 단계에서 새로운 완충액을 추가하여 CaCl 7.4 없이 완충액(10mM Tris pH 7.4, 100mM NaCl)으로 SLB를 최소 2 회 세척합니다. 각 세척 단계에 대해 80μL의 버퍼를 사용하는 것이 좋습니다.
알림: SLB의 최적 세척은 표면을 건드리지 않고 새 용액을 위아래로 여러 번 피펫팅하여 얻을 수 있습니다. 새로 형성된 SLB를 포함하는 웰에서 용액의 피펫팅은 형성된 SLB를 손상시킬 수 있는 작은 기포의 형성을 줄이기 위해 부드러워야 합니다. 이 순간부터 웰은 SLB가 건조되는 것을 방지하기 위해 충분한 양의 완충액을 포함해야 합니다. - SAv를 사용한 비오틴화 SLB의 추가 기능화를 위해 SAv 용액을 최종 농도 250nM까지 추가하고 실온에서 30분 동안 배양합니다. 그 후, 완충액 (10 mM Tris pH 7.4, 100 mM NaCl)으로 5 회 이상 세척하여 여분의 SAv를 제거하십시오.
- 이 순간부터는 광전환 가능한 단백질의 원치 않는 광활성화를 방지하기 위해 샘플을 보호용 적색광 아래에 두십시오. b-disiILD( 재료 표 참조)를 웰의 최종 농도 1μM에 추가합니다. 실온에서 30분 배양 후 완충액으로 5회 이상 세척하여 여분의 단백질을 제거합니다.
- mOrange-Nano( 재료 표 참조)를 최종 농도 200nM에 추가하고 알루미늄 호일로 덮어 샘플을 어두운 곳에 보관합니다.
- μ-슬라이드를 형광 현미경 아래에 놓고 이미징 설정을 조정합니다. mOrange-Nano의 여기(excitation)를 위해 552nm 레이저를 설정합니다. 방출 범위를 조정하여 mOrange 신호를 최적화합니다. disiLID의 광활성화는 2.58초 간격의 광 펄스를 사용하여 488nm 레이저로 이루어집니다.
3. GUV의 준비
- 폴리비닐 알코올(PVA, 재료 표 참조)(MW: 145,000g/mol)의 5%(w/v) 용액을 초순수에 100mM 자당과 함께 준비하고, 400rpm에서 80°C에서 밤새 혼합합니다.
- 원하는 조성(최종 농도 10mg/mL)의 클로로포름으로 지질 용액을 준비합니다. 이 방법의 경우 10mg/mL POPC, 10mol% 1-palmitoyl-2-oleoyl-sn-glycero-3-phospho-(1'-rac-glycerol)(POPG), 2 mol% DOPE-biotin 및 1 mol% DiD로 구성된 조성물을 권장합니다( 재료 표 참조).
- 수화 기술 7,8로 GUV를 준비합니다. 먼저 준비된 PVA 용액 40μL를 60mm x 24mm 유리 슬라이드 위에 균일한 얇은 층으로 펴 바르는 것이 좋으며, 가급적이면 피펫 팁을 사용합니다. 그런 다음 얇은 층을 50°C에서 30분 동안 건조시킵니다.
- PVA 층에 바늘로 5μL의 지질 용액을 펴 바르고 30 ° C에서 1 시간 동안 건조시킵니다.
- 스페이서(~40mm × 24mm × 2mm, 재료 표 참조)와 두 번째 유리 슬라이드를 사용하여 기능성 유리 슬라이드에 챔버를 조립합니다.
- 1mL의 재수화 완충액(10mM Tris pH 7.4, 100mM NaCl)을 실온에서 1시간 동안 챔버에 추가하여 GUV를 형성합니다. 1시간 후 챔버를 뒤집고 피펫 팁으로 유리 표면을 가볍게 두드립니다.
- 한쪽의 유리 슬라이드를 조심스럽게 제거하여 내장 챔버를 열고 피펫으로 GUV를 수확합니다.
- 용액을 플라스틱 튜브에 넣고 GUV를 2시간 동안 가라앉힙니다.
4. disiLID 기능화 GUV에 대한 mOrange-Nano 모집
- 갓 수확한 GUV에 SAv 용액을 넣고 실온에서 30분 동안 그대로 두십시오.
알림: disiLID의 광활성화를 방지하기 위해 보호용 적색광으로 다음 단계를 수행해야 합니다. - GUVs 용액에 1μM의 b-disiLID를 추가하고 샘플을 알루미늄 호일로 덮고 어두운 곳에서 30분 동안 둡니다.
- 150μL BSA 용액(물에서 3% w/v)으로 μ슬라이드 18웰 유리 바닥 챔버를 10분 동안 전처리합니다. 다음으로 BSA 용액을 제거하고 150μL 초순수로 우물을 3회 세척합니다.
- 완충액 (10mM Tris pH 7.4, 100mM NaCl)에 145 μL의 200 nM mOrange-Nano를 추가합니다.
- 다음으로, b-disiLID로 데코레이팅된 5μL의 GUV를 용액에 추가하고 GUV가 가라앉을 때까지 ~15분 동안 기다립니다.
- μ-슬라이드를 컨포칼 현미경 아래에 놓습니다. 552nm에서 샘플을 excitation하여 GUV 멤브레인에서 mOrange(λex = 557nm; λem = 576nm) 형광을 시각화하고 638nm에서 GUV 멤브레인의 DiD(λex = 644nm, λem = 665nm)를 시각화합니다. mOrange-Nano 모집은 원치 않는 광표백 효과를 최소화하기 위해 5.3초마다 청색광 펄스(488nm, 강도 1%)로 트리거됩니다.
참고: 여기 파장은 사용 중인 현미경의 유형에 따라 조정할 수 있습니다. 현미경에 사용할 수 있는 다른 일반적인 여기 파장도 532nm 또는 561nm 및 633nm, 647nm, 639nm 또는 640nm 레이저입니다.
결과
설명된 절차를 통해 SLB의 형성이 합성 멤브레인에서 mOrange-Nano를 모집할 수 있습니다. b-disiLID로 기능화된 SLB에서 패터닝된 정의된 mOrange-Nano의 형성은 그림 1A에 나와 있습니다. SLB의 제곱(24μm × 24μm) 관심 영역(ROI)이 488nm 청색광으로 조명되면 200초 이내에 ROI의 mOrange 채널(빨간색으로 표시)에서 형광 신호의 급격한 증가가 관찰됩니다. 이 패턴은 매우 명확하고 날카로운 모서리를 보여주며(그림 1B), 이는 광활성화 영역에 대한 높은 공간 제어를 나타냅니다. 상호 작용은 빠르고 청색광 조명이 중단될 때 완전히 되돌릴 수 있습니다. 또한 이 방법을 사용하면 여러 조명 주기에 걸쳐 패턴을 형성할 수 있습니다(그림 2). ~200초의 청색광과 200초의 어두운 빛을 번갈아 가며 주기를 사용하면 패턴에서 형광의 Δ 강도가 비슷한 값으로 선택한 영역에서 mOrange-Nano가 여러 번 가역적으로 모집됩니다.
그림 3 은 GUV 준비의 개략도를 보여줍니다. mOrange-Nano의 모집은 GUV에서도 관찰됩니다. 어두운 곳에 배치된 GUV는 mOrange 형광을 나타내지 않는 것으로 나타났습니다(그림 4A). GUV가 청색광으로 전 세계적으로 조명됨에 따라 mOrange 형광이 관찰되어 GUV 멤브레인 염료(DiD)와 함께 국소화됩니다. 상호 작용은 조명이 종료됨에 따라 매우 가역적입니다. 시간 경과에 따른 GUV 멤브레인의 mOrange 강도를 정량화하면 단백질의 빠르고 효과적인 모집과 완전한 가역성을 보여줍니다(그림 4B).
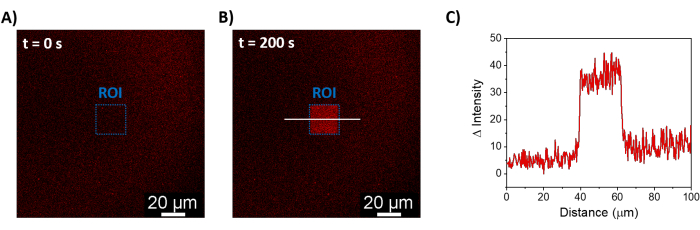
그림 1: b-disiLID로 기능화된 SLB의 형광 현미경 이미지. ROI에서 국부 청색광(488nm) 조명 전(A) 및 도중(B) mOrange-Nano가 있는 형광 이미지. 눈금 막대 = 20 μm. (C) b-disiLID로 기능화된 SLB의 ROI에서 측정된 mOrange의 형광 강도(= 200초에서). 이 그림은 Di Iorio et al.8에서 발췌한 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
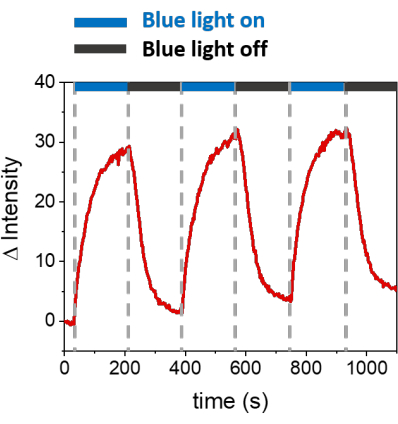
그림 2: 세 번의 모집 주기 동안 b-disiLID로 데코레이팅된 SLB의 ROI에서 모집된 mOrange의 형광 강도. 각 광활성화 단계 후에 mOrange-Nano 형광은 ROI 내에서 증가했습니다. 패턴은 120초 이내에 포화에 도달하고 형광은 120초 이내에 거의 배경 수준까지 감소합니다. 다양한 청색광/암흑 주기에서 패턴 품질의 손실이 관찰되지 않습니다. 이 그림은 Di Iorio et al.8에서 발췌한 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
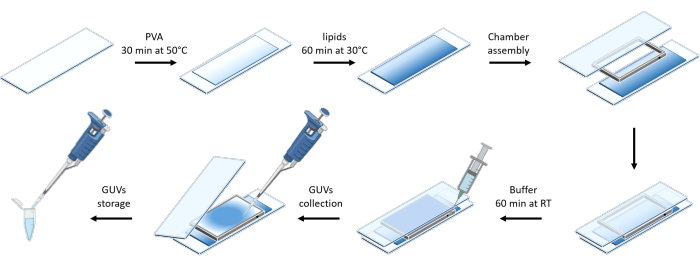
그림 3: 부드러운 수화 방법을 사용한 GUV 준비의 개략도. 이 계획은 여러 단계와 두 개의 유리 슬라이드와 스페이서를 사용하여 만들어진 챔버의 시각적 표현을 제공합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
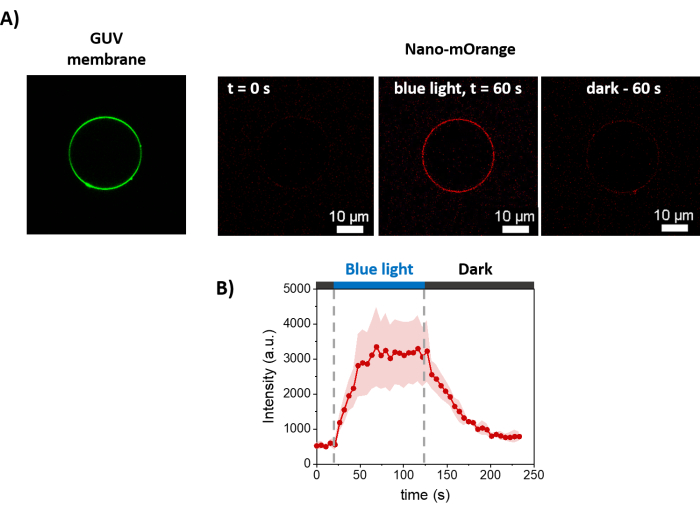
그림 4: GUV 멤브레인에서 mOrange-Nano의 빛 의존적 모집에 대한 형광 현미경 측정. (A) mOrange-Nano가 있는 disiLID 기능성 GUV의 형광 이미지. 녹색은 GUV의 멤브레인 염료이고 빨간색은 청색광 조명 전, 중, 후의 mOrange 형광입니다. 스케일 바 = 10 μm. (B) 시간 경과에 따른 GUV에 국한된 mOrange의 형광 강도. 조명 시 지질막의 mOrange 형광(빨간색으로 표시)은 60초 이내에 최대 강도에 도달하며 형광 강도는 5.9배 증가합니다. 조명이 중지되면 mOrange 형광은 60초 이내에 거의 사전 조명 값으로 감소합니다(90% 회수). 이 그림은 Di Iorio et al.8에서 발췌한 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
우리는 photoswitchable protein disiLID8을 사용하여 supported lipid bilayer 및 giant unilamellar vesicles와 같은 모델 막에서 mOrange-Nano 단백질의 국부적인 모집을 위한 방법을 설명했습니다. 패턴의 품질에 기여하는 측면에는 단백질의 품질뿐만 아니라 SLB 및 GUV의 우수한 품질이 포함됩니다.
발현 및 정제 후 우수한 단백질 품질을 보장하려면 먼저 disiLID의 광전환 가능 특성을 평가하는 것이 중요합니다. 이를 위해 FMN 보조 인자의 흡수는 어두운 곳과 청색광 조명 후에 측정해야 합니다. disiLID의 UV-Vis 스펙트럼은 어둠 속에서 보조인자 FMN의 특징적인 삼중 피크를 나타낼 것으로 예상되며, 이는 청색광 조명 시 현저히 감소하고 어두운 곳에서 회복된다13. 이 광전환 가능한 동작은 다음 단계에서 재현 가능하고 가역적인 모집을 얻는 데 중요합니다. 시료 준비 중 보호용 적색광으로 작업하고 disiLID를 최소한의 외부 조명에 노출시키면 실험 성능이 향상됩니다.
또 다른 중요한 단계이자 아마도 가장 중요한 단계는 적절한 SLB를 형성하는 것입니다. 멤브레인의 결함 및/또는 불균일한 SLB의 형성(즉, 다층 또는 패치된 SLB의 존재)은 단백질 패터닝의 품질에 영향을 미칩니다. 따라서 경험이 없는 사용자의 경우 형광 라벨링된 SLB를 형성하기 위해 SUV에 DiD 및 DiO와 같은 일부 멤브레인 염료를 라벨링하여 프로토콜을 재현하는 것이 좋습니다. 이러한 방식으로 SLB의 특성과 품질은 형광 현미경으로 잘 특성화할 수 있습니다. FRAP 측정은 멤브레인의 유동성을 평가하여 SLB의 품질을 평가하는 일반적인 접근 방식을 나타냅니다. 대안적으로, 이 프로토콜에 기술된 것과 같은 비오틴화된 SLB의 경우, 형광 표지된 SAv(예: Atto 488-SAv)를 사용하여 SLB의 품질을 시각화하고 평가할 수 있습니다.
프로토콜의 첫 번째 부분에서는 SLB의 패턴 형성에 대해 설명합니다. 최적의 결과를 얻으려면 SLB에 mOrange-Nano를 첨가하고 샘플을 15분 동안 어두운 곳에서 배양하는 것이 중요합니다. 광활성화 중에 ROI 선택은 특정 크기로 제한되지 않습니다. 그러나 형광 단백질의 원치 않는 광표백을 줄이기 위해 레이저 강도와 노출 시간을 조절해야 합니다.
이 방법은 비오틴화 단백질에만 국한되지 않으며, disiLID를 SLB에 고정하기 위해 다른 접근법을 사용할 수 있습니다. 예를 들어, His-태그된 disiLID는 Ni-NTA 함유 SLB에 발현되고 고정될 수 있습니다. 그러나 SLB의 단백질이 교체되는 것을 피하기 위해 Nano와 disiLID를 다른 태그로 발현하는 것이 중요합니다. 이 방법은 또한 단백질의 순서를 반전시킬 수 있는 가능성을 허용하여 SLB를 Nano로 기능화하고 청색광 조명 시 disiLID(또는 disiLID-융합 단백질)를 모집합니다.
단백질 국소화의 동적 제어를 위해서는 선택한 영역에 대한 단백질의 가역적 국소화가 반복적으로 가능해야 합니다. 이를 달성하기 위해 용액의 나노(200nM) 농도는 높은 가역성을 얻기 위한 중요한 매개변수입니다.
또 다른 우려 사항은 disiLID 기능성 GUV 표면에 Nano를 모집하는 것입니다. SLB의 단백질 패터닝의 경우와 마찬가지로 이 방법은 다양한 멤브레인 기능화 전략으로 확장될 수 있습니다. 이 프로토콜에서는 전체 GUV 표면에서 mOrange-Nano를 모집하기 위해 전체 GUV를 청색광으로 비췄습니다. 그러나 GUV 멤브레인에 국한된 작은 ROI를 선택하면 보다 제한된 영역에서 단백질의 정확한 국소화가 가능해야 합니다.
이 방법은 SUV 또는 GUV의 멤브레인에서 나노 모집의 이미징에 사용되는 형광단의 선택과 관련된 제한 사항만을 제시합니다. 특히, 청색광 범위의 여기 스펙트럼을 가진 형광단은 (dis)iLID의 광활성화를 방해하므로 피해야 합니다. 따라서 이러한 유형의 실험에는 녹색 또는 적색광 범위(예: mOrange 또는 Cy5)의 형광단을 선택하는 것이 좋습니다.
disiLID 설계는 멤브레인에 대한 국소 단백질 모집을 개선하고 광유전학에서 iLID 및 Nano의 동적 범위를 넓히는 간단하고 적응 가능한 방법을 제공합니다4. 이러한 방법은 지질 이중층 및 GUV와 같은 모방 막에 Nano를 모집하는 데 중점을 둡니다. 그럼에도 불구하고 이 접근 방식은 (dis)iLID 또는 Nano가 막에 연결된 세포의 수많은 광유전학 도구로 확장할 수 있습니다.
공개
저자는 밝힐 것이 없습니다.
감사의 말
이 작품은 유럽 연구 위원회(European Research Council)의 ERC 스타팅 그랜트 아티스트(# 757593 S.V.W.)의 지원을 받았습니다. DDI는 알렉산더 폰 훔볼트 재단(Alexander von Humboldt Foundation)의 박사후 연구원 과정에 대한 지원에 감사를 표합니다.
자료
| Name | Company | Catalog Number | Comments |
| µ-Slide 18 Well | Ibidi | 81817 | For SLB preparation |
| 25 µL Microliter Syringe | Hamilton | Model 702 N | For the preparation of lipid mixture and spreading the lipid solution on the PVA layer |
| Biotinyl Cap PE (1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-(cap biotinyl)) (sodium salt) | Avanti Polar Lipids | 870273C | For SUVs preparation |
| CaCl2 (Calcium chloride) | Sigma-Aldrich | C5670 | For SLB formation |
| Cover Slips 24 mm x 60 mm | Engelbrecht | K12460 | For GUVs formation |
| DiD (1,1'-Dioctadecyl-3,3,3',3'- Tetramethylindodicarbocyanine) | Thermo Fisher Scientific | D7757 | Membrane dye |
| disiLID | Sequence: MGGSGLNDIFEAQKIEWHEGGSH HHHHHGSMAATELRGVVGPGPAA IAALGGGGAGPPVGGGGGRGDA GPGSGAASGTVVAAAAGGPGPG AGGVAAAGPPAPPTGGSGGSGA GGSGSAGEFLATTLERIEKNFVIT DPRLPDNPIIFASDSFLQLTEYSR EEILGRNCRFLQGPETDRATVRK IRDAIDNQTEVTVQLINYTKSGKK FWNVFHLQPMRDYKGDVQYFIG VQLDGTERLHGAAEREAVMLIKK TAFQIAEAANDENYF | ||
| DOPC (1,2-di-(9Z-octadecenoyl)-sn-glycero-3-phosphocholine) | Avanti Polar Lipids | 850375P | For SUVs lipid composition |
| Eppendorf Protein LoBind microcentrifuge tubes | Merk | EP0030108116-100EA | For collecting freshly made GUVs |
| mOrange-Nano | Sequence: MRGSHHHHHHGSKIEEGKLVI WINGDKGYNGLAEVGKKFEKDT GIKVTVEHPDKLEEKFPQVAATG DGPDIIFWAHDRFGGYAQSGLLA EITPDKAFQDKLYPFTWDAVRYN GKLIAYPIAVEALSLIYNKDLLPNP PKTWEEIPALDKELKAKGKSALM FNLQEPYFTWPLIAADGGYAFKY ENGKYDIKDVGVDNAGAKAGLTF LVDLIKNKHMNADTDYSIAEAAFN KGETAMTINGPWAWSNIDTSKVN YGVTVLPTFKGQPSKPFVGVLSA GINAASPNKELAKEFLENYLLTDE GLEAVNKDKPLGAVALKSYEEELA KDPRIAATMENAQKGEIMPNIPQM SAFWYAVRTAVINAASGRQTVDEA LKDAQTNSSSNNNNNNNNNNLGI EGTTENLYFQGSVSKGEENNMAI IKEFMRFKVRMEGSVNGHEFEIE GEGEGRPYEGFQTAKLKVTKGG PLPFAWDILSPQFTYGSKAYVKH PADIPDYFKLSFPEGFKWERVMN FEDGGVVTVTQDSSLQDGEFIYK VKLRGTNFPSDGPVMQKKTMG WEASSERMYPEDGALKGEIKMR LKLKDGGHYTSEVKTTYKAKKPV QLPGAYIVGIKLDITSHNEDYTIVE QYERAEGRHSTGGMDELYKGG SGTSSPKRPKLLREYYDWLVDN SFTPYLVVDATYLGVNVPVEYVK DGQIVLNLSASATGNLQLTNDFIQ FNARFKGVSRELYIPMGAALAIYA RENGDGVMFEPEEIYDELNIG | ||
| NaCl (Sodium chloride) | Sigma-Aldrich | S9888 | For buffer |
| NaOH (Sodium hydroxide) | Sigma-Aldrich | 1064980500 | For surface activation in SLB formation |
| POPC (1-Palmitoyl-2- oleoylphosphatidylcholine) | Avanti Polar Lipids | 850457C | For GUVs lipid composition |
| POPG (1-palmitoyl-2-oleoyl-sn-glycero-3-phospho-(1'-racglycerol)) | Avanti Polar Lipids | 840457C | For GUVs lipid composition |
| PVA (Polyvinyl alcohol) fully hydrolyzed | Sigma-Aldrich | 8148940101 | For GUVs formation |
| SP8 confocal laser scanning microscope | Leica | ||
| Streptavidin | TermoFisher | 434301 | |
| Sucrose | Sigma-Aldrich | 84097 | For GUVs formation |
| Tris hydrochloride | Sigma-Aldrich | 10812846001 | For buffer |
참고문헌
- Kretschmer, S., Schwille, P. Pattern formation on membranes and its role in bacterial cell division. Curr Opin Cell Biol. 38, 52-59 (2016).
- Yang, H. W., Collins, S. R., Meyer, T. Locally excitable Cdc42 signals steer cells during chemotaxis. Nat Cell Biol. 18 (2), 191-201 (2016).
- Caldwell, R. M., et al. Optochemical control of protein localization and activity within cell-like compartments. Biochem. 57 (18), 2590-2596 (2018).
- Kennedy, M. J., et al. Rapid blue-light-mediated induction of protein interactions in living cells. Nat Methods. 7 (12), 973-975 (2010).
- Guntas, G., et al. Engineering an improved light-induced dimer (iLID) for controlling the localization and activity of signaling proteins. Proc Natl Acad Sci. 112 (1), 112-117 (2015).
- Levskaya, A., Weiner, O. D., Lim, W. A., Voigt, C. A. Spatiotemporal control of cell signalling using a light-switchable protein interaction. Nature. 461 (7266), 997-1001 (2009).
- Bartelt, S. M., et al. Dynamic blue light-switchable protein patterns on giant unilamellar vesicles. Chem Commun. 54 (8), 948-951 (2018).
- Di Iorio, D., Bergmann, J., Higashi, S. L., Hoffmann, A., Wegner, S. V. A disordered tether to iLID improves photoswitchable protein patterning on model membranes. Chem. Commun. 59 (29), 4380-4383 (2023).
- Hartzell, E. J., Terr, J., Chen, W. Engineering a blue light inducible Spytag system (BLISS). J Am Chem Soc. 143 (23), 8572-8577 (2021).
- Xu, D., Bartelt, S. M., Rasoulinejad, S., Chen, F., Wegner, S. V. Green light lithography: a general strategy to create active protein and cell micropatterns. Mater Horiz. 6 (6), 1222-1229 (2019).
- Jia, H., et al. Light-induced printing of protein structures on membranes in vitro. Nano Lett. 18 (11), 7133-7140 (2018).
- Di Iorio, D., Verheijden, M. L., vander Vries, E., Jonkheijm, P., Huskens, J. Weak Multivalent Binding of influenza hemagglutinin nanoparticles at a sialoglycan-functionalized supported lipid bilayer. ACS Nano. 13 (3), 3413-3423 (2019).
- Kasahara, M., Torii, M., Fujita, A., Tainaka, K. FMN binding and photochemical properties of plant putative photoreceptors containing two LOV domains, LOV/LOV proteins. J Biol Chem. 285 (45), 34765-34772 (2010).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유