Method Article
Curation of Computational Chemical Libraries Demonstrated with Alpha-Amino Acids
W tym Artykule
Podsumowanie
The purpose of this protocol is to efficiently generate and curate small-molecule structure libraries using open-source software.
Streszczenie
Exhaustive generation of molecular structures has numerous chemical and biochemical applications such as drug design, molecular database construction, exploration of alternative biochemistries, and many more. Mathematically speaking, these are graph generators with chemical constraints. In the field, the most efficient generator currently (MOLGEN) is a commercial product, limiting its use. Alternative to that, another molecular structure generator, MAYGEN, is a recent open-source tool with efficiency comparable to MOLGEN and the capacity for users to increase its performance by adding new features. One of the research fields that can benefit from this development is astrobiology; structure generators allow researchers to supplement experimental data with computational possibilities for alternative biochemistry. This protocol details one use case for structure generation in astrobiology, namely the generation and curation of alpha-amino acid libraries. Using open-source structure generators and cheminformatics tools, the practices described here can be implemented beyond astrobiology for the low-cost creation and curation of chemical structure libraries for any research question.
Wprowadzenie
Molecular structure generation serves as a practical application of the general problem of exhaustive graph generation; given several nodes (atoms) and constraints on their connectivity (e.g., valences, bond multiplicities, desired/undesired substructures), how many connected graphs (molecules) are possible? Structure generators have seen extensive application in drug discovery and pharmaceutical development, where they can create vast libraries of novel structures for in silico screening1.
The first structure generator, CONGEN, was developed for the first artificial intelligence project in organic chemistry, DENDRAL2 (short for DENDRitic ALgorithm). Several software successors of DENDRAL were reported in the literature; however, not all of them were maintained or efficient. Currently, MOLGEN3 is the state-of-the-art molecular structure generator. Unfortunately for most potential users, it is closed-source and requires a licensing fee. Thus, there has been the need for an efficient open-source structure generator that can easily adapt to specific applications. One challenge for an efficient structure generator is managing combinatorial explosion; as the size of a molecular formula increases, the size of the chemical search space increases exponentially. A recent review further explores the history and challenges of molecular structure generation4.
Prior to 2021, the Parallel Molecule Generator (PMG)5 was the fastest open-source structure generator, but it was still slower than MOLGEN by orders of magnitude. MAYGEN6 is approximately 47 times faster than PMG and around 3 times slower than MOLGEN, making MAYGEN the fastest and most efficient open-source structure generator available. More detailed comparisons and benchmarking tests can be found in the paper introducing MAYGEN6. A key feature of the program is its lexicographical ordering-based test for canonical structures, an orderly graph-generation method based on the Schreier-Sims7 algorithm. The software can be easily integrated into other projects and enhanced for the needs of the users.
Like MOLGEN and PMG, MAYGEN takes a user-defined molecular formula and generates all structures possible for that formula. For example, if a user runs MAYGEN with the formula C5H12, MAYGEN will generate all possible structures containing five carbon atoms and twelve hydrogen atoms. Unlike its open-source counterpart PMG, MAYGEN can also accommodate "fuzzy" molecular formulae that use intervals instead of discrete numbers for the count of each element. For example, if a user runs MAYGEN with the formula C5-7H12-15, MAYGEN will generate all possible structures that contain between five and seven carbon atoms and twelve and fifteen hydrogen atoms, allowing for simple generation of structures with a wide range of atomic compositions.
Astrobiology is one such field that can benefit from molecular structure generators. A popular topic in astrobiology is the evolution of the amino acid alphabet shared by all extant life on Earth. One of the defining features of the Last Universal Common Ancestor (LUCA) is its use of twenty genetically coded amino acids for protein construction8,9. Based on meta-analyses of work in multiple fields10,11,12, approximately 10 of these amino acids (Gly, Ala, Val, Asp, Glu, Ser, Thr, Leu, Ile, Pro) readily form under abiotic conditions and likely made up the amino acid alphabet of pre-LUCA organisms. Over time, this "early" alphabet was expanded in response to different structural and functional needs. For example, a recent review from Moosmann13 claims that the addition of more recent members of the genetically coded amino acids (namely Met, Tyr, and Trp) allowed for survival in oxygen-rich environments by preventing the intracellular proliferation of reactive oxygen species.
An ever-growing suite of analytical chemistry techniques allows insight into the amino acid structures that can form under abiotic conditions. A recent review14 by Simkus and others details the methods used to detect numerous organic compounds in meteorites, as well as organic compounds from in vitro simulations of early Earth environments15,16,17. Systematic generation of chemical structures allows researchers to explore beyond the organic compounds detected via instrumentation, populating the structural space around structural "islands" identified by analytical chemistry. In the case of the "early" amino acids, this systematic structure generation shows possible protein chemistries available to early life without limiting exploration to structures that have been experimentally detected under abiotic synthesis conditions. With open-source cheminformatics toolkits and efficient structure generators such as MAYGEN, creating and exploring novel chemical structure libraries is now easier than ever before and can guide more detailed investigations into alternative chemistries of life.
Protokół
NOTE: See Figure 1 for a summary of the protocol and the Table of Materials for details about the software used.

Figure 1: Summary flowchart of the protocol. Please click here to view a larger version of this figure.
1. Software and file downloads
NOTE: All programs are free for individual use and can be run on a personal computer.
- Create a new directory for this project. Place the files and executables here for easy access.
- Download and install the necessary software packages.
- Download the latest version of MAYGEN as a .jar file.
NOTE: MAYGEN is freely available as a .jar file from https://github.com/MehmetAzizYirik/MAYGEN/releases - Download and install the package management software Conda and the cheminformatics toolkit RDKit18.
NOTE: RDKit will filter the molecular structures produced by MAYGEN and runs best in a Conda environment. Instructions for downloading the Conda platform can be found at https://conda.io/projects/conda/en/latest/user-guide/install/index.html. RDKit installation and environment setup instructions can be found at https://www.rdkit.org/docs/Install.html.- Install RDKit in the main Conda environment instead of a separate RDKit environment via the Anaconda prompt. On Windows systems, search for "Anaconda prompt" and click on the resulting shortcut to run. On MacOS and Linux systems, interact with Conda through the terminal without running any additional programs. Next, type the following command and press Enter to run, and answer yes to any questions that come up during the installation:
conda install -c rdkit rdkit.
While there are many freely available descriptor calculation programs, this example uses PaDEL-Descriptor19, a free and fast calculator for molecular descriptors and fingerprints.
- Install RDKit in the main Conda environment instead of a separate RDKit environment via the Anaconda prompt. On Windows systems, search for "Anaconda prompt" and click on the resulting shortcut to run. On MacOS and Linux systems, interact with Conda through the terminal without running any additional programs. Next, type the following command and press Enter to run, and answer yes to any questions that come up during the installation:
- Download and save the .jar file in the project folder.
NOTE: PaDEL-Descriptor can be downloaded for free from http://www.yapcwsoft.com/dd/padeldescriptor/.
- Download the latest version of MAYGEN as a .jar file.
- Download the Jupyter notebooks and text files of substructure patterns from Supplemental Files 1-5.
NOTE: Jupyter notebooks can also be downloaded from the following GitHub page: https://github.com/cmayerb1/AA-structure-manip.
2. Structure generation using MAYGEN
- In a command prompt, navigate to the directory containing the MAYGEN .jar executable file.
- For each chemical formula of interest, run MAYGEN using the following command:
java -jar [MAYGEN .jar file name] -f [chemical formula] -v -o [folder for MAYGEN output] -m -sdf.
NOTE: This will save a .sdf file in the designated folder, named after the formula used.- If the formula is a fuzzy formula instead of a discrete formula, replace the -f flag with a -fuzzy flag, and enclose any element intervals in brackets (e.g., use C[5-7]H[12-15] to ensure that all structures generated have between 5 and 7 carbon atoms and between 12 and 15 hydrogen atoms).
3. Filter compounds with undesired substructures
- Open an Anaconda prompt (see step 1.2.2.1) and navigate to the folder containing the Jupyter notebooks downloaded from Supplemental File 1.
- Open the Jupyter notebook for substructure filtering using the following command:
jupyter notebook [notebook file name] - In the designated cell at the start of the notebook, enter the full file path of the input .sdf file (generated by MAYGEN), full file path of the desired .sdf output file, and file path of the "badlist" file as strings (within quotes). See Supplemental File 2 for an example of a badlist.
- If some substructures in the filtered library (a goodlist) are to be retained, create a .txt file of SMARTS patterns20 for those substructures (a goodlist) and put the goodlist file path in the designated line at the start of the notebook. See Supplemental File 3 for an example of a goodlist.
- Restart the notebook kernel and run all cells (from the menu at the top, select Kernel, Restart & Run All) to get a .sdf file with the desired name in the specified output folder.
- Repeat the previous two steps for each structure file generated by MAYGEN in step 2.
4. (Optional) Additional structure modifications
NOTE: These are performed in this example but may not be needed for curating other libraries.
- Pseudoatom replacement.
NOTE: Here, a pseudoatom is a unique atom used to represent a larger substructure shared by all generated structures, thus reducing MAYGEN's generation time. See Supplemental File 4 for an example of pseudoatom replacement.- Open an Anaconda prompt (see step 1.2.2.1) and navigate to the folder containing the Jupyter notebooks.
- Open the Jupyter notebook for pseudoatom replacement:
jupyter notebook [notebook file name] - In the designated cell at the start of the notebook, enter the full file path of the input .sdf file and the full file path of the desired .sdf output file as strings (within quotes).
- Restart the notebook kernel and run all the cells to get a .sdf file with the desired name in the specified output folder.
- Amino acid N- and C-termini capping
NOTE: This procedure is specific to alpha-amino acids, adding molecular caps to the N- and C-termini of alpha-amino acid backbones. See Supplemental File 5 for an example of amino acid capping.- Open an Anaconda prompt (see step 1.2.2.1) and navigate to the folder containing the Jupyter notebooks.
- Open the Jupyter notebook for amino acid capping:
jupyter notebook [notebook file name] - In the designated cell at the start of the notebook, enter the full file path of the input .sdf file and the full file path of the desired .sdf output file as strings (within quotes).
- Restart the notebook kernel and run all the cells to get a .sdf file with the desired name in the specified output folder.
5. Descriptor generation
- Prior to descriptor generation, place all .sdf files for which descriptors are to be calculated in a single folder.
NOTE: If not done already, give these files descriptive names for easy filtering after descriptor generation. - Open a command prompt, and navigate to the folder containing the PaDEL-Descriptor .jar file.
- Run PaDEL-Descriptor for the collected .sdf files using the following command:
java -jar PaDEL-Descriptor.jar -dir [directory of the .sdf files] -file [file path of a .csv file for results] -2d -retainorder -usefilenameasmolname
NOTE: The results file will have the molecule name in the first column and each descriptor in the subsequent columns. - Export these data to any spreadsheet software for further analysis.
Wyniki
| Library | Formula | Additional constraints | "Early" coded amino acids | Generation time (ms) | Structures | ||||
| Initial | Final | ||||||||
| 1 | Gly | C2H5NO2 | include Gly substructure | Gly | 192 | 84 | 1 | ||
| 2 | VAIL | PC0-3H3-9 | Val, Ala, Ile, Leu | 172 | 70 | 22 | |||
| 3 | DEST | PC0-3O1-2H3-5 | Asp, Glu, Ser, Thr | 481 | 1928 | 254 | |||
| 4 | Pro | C2-5NO2H7-11 | Include N-meGly or N-meAla substructure | Pro | 4035 | 79777 | 16 | ||
| 5 | VAIL_S | PSC0-2H3-7 | 122 | 65 | 31 | ||||
| 6 | DEST_S | PSC0-2O1-2H3 | 349 | 1075 | 79 | ||||
| 7 | Pro_S | C2-4SNO2H7-9 | Include N-meGly or N-meAla substructure | 3999 | 75734 | 10 | |||
Table 1: Compound libraries used in this example. Libraries built from formulae 1-4 (Gly, VAIL, DEST, and Pro) are based on previously published fuzzy formulae of the "early" coded amino acids21, while libraries built from formulae 5-7 (VAIL_S, DEST_S, and Pro_S) are based on variants of formulae 2-4 that imagine a divalent sulfur replacing one of the carbon atoms. Structure counts reflect the number of molecules generated by MAYGEN for each formula ("Initial") and the number of molecules remaining after filtering out those with unwanted substructures ("Final"). Abbreviations: VAIL = valine, alanine, isoleucine, leucine; DEST = aspartic acid, glutamic acid, serine, threonine; X_S = Divalent sulfur replaces one of the carbons in library X; N-meX = N-methylX.
The general methods above were applied to formulae based on the "early" coded amino acids, following the procedure of Meringer et al.21 Badlist structures were taken from this same source and converted to SMARTS strings to easily represent substructural patterns. Two badlist substructures were not used in this example: structure 018 (CH3-CH-N) matched near-isomers of proline that were not themselves unstable; structure 106 (R-C-C-OH, where R=alanine substructure attaching at the beta-carbon) matched glutamic acid, a coded amino acid. In addition to these chemical formulae, variants with divalent sulfur taking the place of a carbon atom and two hydrogen atoms were created. For performance reasons, several of these formulae use a trivalent phosphorus atom (e.g., a "pseudoatom") as a substitute for the beta-carbon of an alanine substructure. Table 1 lists the libraries generated in this example, the formulae used to generate them, and the number of compounds contained within. Library names are based on the coded amino acids from which they are derived: either using the 3-letter abbreviation (Gly = glycine, Pro = proline) or single-letter abbreviation (VAIL = Valine, Alanine, Isoleucine, Leucine; DEST = Aspartic acid, Glutamic acid, Serine, Threonine). The "_S" suffix indicates a sulfur was substituted for a carbon in the original library's formula (e.g., VAIL_S is built with the same fuzzy formula as VAIL, but with a divalent sulfur replacing one of the carbons).
After structure generation with MAYGEN, the resulting libraries were filtered of compounds containing at least one substructure contained in the badlist. Following this filtering, any phosphorus atoms were replaced with an alanine substructure. Next, "capped" versions of all structures were created, with an acetyl group added to the N-terminus and an N-methyl amide group added to the C-terminus. This was done to remove the effect on the hydrophobicity of the free amine and carboxylic acid groups in the alpha-amino acid backbone. PaDEL-Descriptor was used to calculate XLogP for all capped structures and calculated van der Waals volume (VABC) for all uncapped structures.
Figure 2 shows the chemical space of the filtered libraries, as defined by VABC and XLogP descriptors. Here, the range of possible logP values increases with molecular volume, even within libraries that lack explicitly hydrophilic sidechains (e.g., VAIL, Pro). Coded amino acids with hydrocarbon sidechains were more hydrophobic than most other amino acids of a comparable volume from their respective library. This also seems to be the case for Met and Cys compared to other members of the VAIL_S library with similar volumes. Coded amino acids with hydroxyl side chains (Ser and Thr) were among the smallest members of the DEST library, with Asp only slightly larger than Thr.
Figure 3 and Figure 4 show the impacts on volume and logP when a divalent sulfur replaces a carbon in an alpha-amino acid side chain. Sulfur substitution led to a slight increase in molecular volume in all libraries (Figure 3). The effect of sulfur substitution on logP is not as homogenous as for volume (Figure 4). The mean logP of the VAIL_S library is slightly lower than that of the VAIL library, but this effect is not seen in either of the other library pairs (DEST and DEST_S, Pro and Pro_S).
Figure 5 quantifies the effects on structure generation of a pseudoatom standing in for a common substructure; here, a trivalent P substituted for an alanine moiety during structure generation. Using a pseudoatom in structure generation greatly decreased the number of structures generated by ~3 orders of magnitude (Figure 5A) and the total time needed to generate those structures by 1-2 orders of magnitude (Figure 5B).

Figure 2: Chemical space of all filtered amino acid libraries. Black markers represent amino acids from libraries without sulfur; yellow markers represent amino acids from sulfur-enriched libraries. Circles: VAIL and VAIL_S; squares: DEST and DEST_S; triangles: Pro and Pro_S; stars: coded amino acids. Note that the two sulfur-containing coded amino acids (Met and Cys) are not considered "early" amino acids but are present in the VAIL_S library. Abbreviations: XLogP = partition coefficient; VAIL = valine, alanine, isoleucine, leucine; DEST = aspartic acid, glutamic acid, serine, threonine; X_S = Divalent sulfur replaces one of the carbons in library X. Please click here to view a larger version of this figure.

Figure 3: Mean van der Waals volumes (in Å3) of libraries with and without sulfur. Black bars represent the mean volumes of libraries without sulfur (VAIL, DEST, Pro), while yellow bars represent mean volumes of the sulfur-substituted versions of those libraries (VAIL_S, DEST_S, Pro_S). Error bars show standard deviation. Abbreviations: VAIL = valine, alanine, isoleucine, leucine; DEST = aspartic acid, glutamic acid, serine, threonine; X_S = Divalent sulfur replaces one of the carbons in library X. Please click here to view a larger version of this figure.
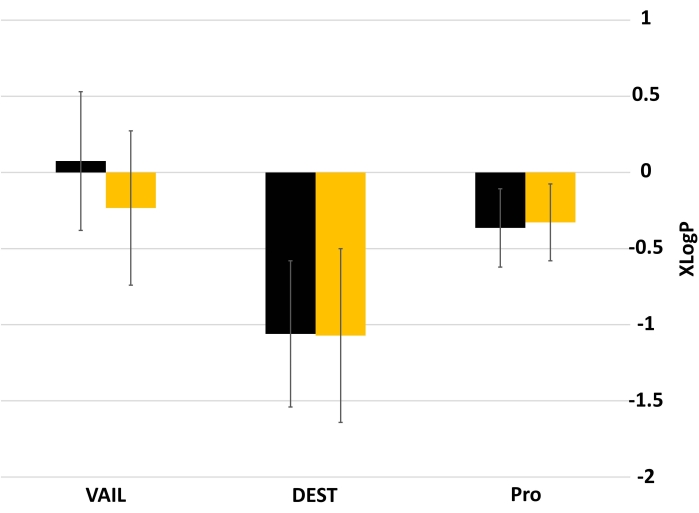
Figure 4: Mean XLogP values of libraries with and without sulfur. Black bars represent libraries without sulfur (VAIL, DEST, Pro), while yellow bars represent sulfur-substituted versions of those libraries (VAIL_S, DEST_S, Pro_S). Error bars show standard deviation. Abbreviations: XLogP = partition coefficient; VAIL = valine, alanine, isoleucine, leucine; DEST = aspartic acid, glutamic acid, serine, threonine; X_S = Divalent sulfur replaces one of the carbons in library X. Please click here to view a larger version of this figure.

Figure 5: Effects of a trivalent pseudoatom on MAYGEN structure generation. All tests were done on a PC with an Intel i7-7700HQ processor at 2.8 GHz, 16 GB of RAM, no saving structures to a file, and the -m option to use multithreading. Tests using a pseudoatom used the fuzzy formulae as described in Table 1. For tests without a pseudoatom, the fuzzy formulae used were the same as described in Table 1 with the following changes: P was replaced with N; carbon counts were increased by 3; hydrogen counts were increased by 7; oxygen counts were increased by 2. Black bars show libraries generated with a pseudoatom; gray bars show libraries generated without a pseudoatom. (A) Number of structures generated using the fuzzy formulae used to build the VAIL and DEST libraries with and without a trivalent phosphorus substituting for an alanine substructure. (B) Time (in ms) needed to build the VAIL and DEST libraries with and without a trivalent phosphorus substituting for an alanine substructure. Abbreviations: VAIL = valine, alanine, isoleucine, leucine; DEST = aspartic acid, glutamic acid, serine, threonine. Please click here to view a larger version of this figure.
Supplemental File 1: Substructure screening notebook. Please click here to download this File.
Supplemental File 2: Sample badlist. Please click here to download this File.
Supplemental File 3: Sample goodlist. Please click here to download this File.
Supplemental File 4: Pseudoatom replacement notebook. Please click here to download this File.
Supplemental File 5: Amino acid capping notebook. Please click here to download this File.
Dyskusje
One feature of the "early" amino acids is a lack of sulfur. The meta-analyses mentioned earlier generally consider the sulfur-containing coded amino acids (Cys and Met) to have been relatively late additions to the genetic code, conclusions supported by the lack of sulfur-containing amino acids in meteorites and spark tube experiments. However, organosulfur compounds are readily detected in comets and meteorites22, and reanalysis of spark tube experiments using H2S gas found amino acids and other organic compounds containing sulfur16. When considering an alternative amino acid alphabet, one enriched in sulfur is worth exploring.
In the above protocol, structure generation and substructure filtering are considered critical steps; depending on the composition of the finished structure library, a researcher may only need to perform those two steps. Instructions and software for additional actions (pseudoatom replacement and addition of substructures (in this case, amino acid capping)) are included for more relevant descriptor calculation (capping ensures that XLogP calculations are influenced by the sidechain and not the backbone amine or carboxyl groups) and faster structure generation via the use of a pseudoatom, which is discussed in more detail below. Additionally, descriptor calculation is done here as an easy way to visualize the diversity of the structures generated and compare the effects of sulfur enrichment in the finished libraries.
While PaDEL-Descriptor can calculate thousands of molecular properties, molecular volume (as calculated van der Waals volume) and partition coefficient (as XLogP) were used here for two distinct reasons. First, these two descriptors measure molecular properties (size and hydrophobicity, respectively) that are familiar to most chemists and biologists. Second, in the case of amino acids, these two properties are significant. For decades, amino acid size and hydrophobicity were known to influence the thermodynamics of protein folding23. These two properties help explain amino acid substitution frequencies that have been integral to understanding protein evolution24.
The above example shows that, in the two descriptors studied (molecular volume and hydrophobicity), substituting a divalent sulfur for a carbon and two hydrogens does not yield significant changes. The slight, nonsignificant increase in mean molecular volume from sulfur substitution (Figure 3) could be attributed to sulfur's larger covalent radius (~103 pm) compared to either sp3 (~75 pm) or sp2 (~73 pm) carbon25. Similarly, sulfur substitution has minimal effect on the mean XLogP (Figure 4). The largest effect was between the VAIL and VAIL_S libraries, likely due to a combination of the VAIL library being especially hydrophobic (the sidechains are only hydrocarbons) and sulfhydryl groups being much more acidic than the methyl groups they would replace. The minimal effect of sulfur substitution is apparent in Figure 2, where libraries with sulfur substitution occupy the same chemical space as analogous libraries without sulfur substitution.
The decrease in the number of structures (Figure 5A) and time needed to generate those structures (Figure 5B) when using a pseudoatom is unsurprising. Using a pseudoatom reduces the number of heavy atoms that need to be incorporated into a chemical graph, reducing the number of graph nodes and yielding exponential decreases in generation time and number of structures. Here, the choice of trivalent phosphorus as a pseudoatom stems from basic biochemistry (absent posttranslational addition of phosphate groups, no genetically coded amino acids contain phosphorus) and the valence of the atom that would replace it (a trivalent phosphorus can easily be replaced with a tetravalent carbon that is singly bonded to another atom or group of atoms). While the provided code for pseudoatom substitution is specific for replacing a trivalent phosphorus with an alanine substructure, users can customize the code to work with different pseudoatoms or replacement substructures, potentially using multiple pseudoatoms during initial structure generation followed by replacing each pseudoatom with a larger molecular substructure.
Structure generation methods similar to those employed by MAYGEN (and other methods such as neural networks) are already used in drug discovery to generate compound libraries for in silico screening; a recent review4 discusses these methods in more detail. As these methods are intended primarily for the creation of drug-like molecules, there are some limitations on their ability to generate molecules, such as using biological or pharmaceutical properties to limit the structures created (inverse QSPR/QSAR) or creating structures from a preset number of substructure building blocks. As astrobiology is focused more on the multitude of organic compounds that can form abiotically and less on any end products or their properties, MAYGEN's exhaustive structure generation is ideal for creating structure libraries to address astrobiological questions. The approach to substructure filtering described here (performed after structure generation via an external program) differs from the competitor program MOLGEN in that MOLGEN's substructure filtering occurs during structure generation. As MAYGEN is open-source, not only is it more accessible than MOLGEN due to MOLGEN's licensing cost, but individuals could implement new features such as substructure filtering during structure generation.
As written, the protocol described here is focused on generating and curating libraries of relatively small alpha-amino acids. To generate different libraries, users can give different molecular formulae to MAYGEN, change the substructure filtering by changing the maximum allowed ring size and bond valence, or edit the goodlist and badlist files to add or remove substructure patterns. Protocol modifications that involve changing how atoms and substructures are added or replaced (pseudoatom substitution and molecular capping) are feasible but will require more attention to valence restrictions to avoid RDKit errors about incorrect valences in modified structures.
The protocol detailed above is designed for small alpha-amino acids. However, the general format (comprehensive structure generation using pseudoatoms, followed by substructure filtering and molecular modifications) is highly flexible for compounds beyond small amino acids. Even in astrobiology, a similar recent procedure using MOLGEN was used to investigate constitutional isomers of nucleic acids26. In addition to the tools described above, MAYGEN can be paired with other open-source cheminformatics tools to make creating and analyzing novel chemical structures affordable and accessible to a broad array of research fields.
Ujawnienia
The authors have no conflicts of interest to disclose.
Podziękowania
MAY acknowledges funding by the Carl-Zeiss-Foundation. All figures were generated using Microsoft Excel.
Materiały
| Name | Company | Catalog Number | Comments |
| conda v. 4.10.3 | https://www.anaconda.com/products/individual | ||
| Java 17 | https://java.com/en/download/help/download_options.html | ||
| MAYGEN v. 1.8 | https://github.com/MehmetAzizYirik/MAYGEN/releases | ||
| PaDEL-Descriptor v. 2.21 | http://www.yapcwsoft.com/dd/padeldescriptor/ | ||
| python v. 3.7.11 | included in Anaconda environment | ||
| RDKit v. 2020.09.1.0 | https://www.rdkit.org/docs/Install.html, or installed via conda: https://anaconda.org/rdkit/rdkit | ||
| *These specific versions were used for this manuscript; user can obtain more recent versions if available. |
Odniesienia
- Ruddigkeit, L., van Deursen, R., Blum, L. C., Reymond, J. -. L. Enumeration of 166 billion organic small molecules in the chemical universe database GDB-17. Journal of Chemical Information and Modeling. 52 (11), 2864-2875 (2012).
- Buchanan, B. G., Feigenbaum, E. A., Webber, B. L., Nilsson, N. J. Dendral and Meta-Dendral: their applications dimension. Readings in Artificial Intelligence. , 313-322 (1981).
- Gugisch, R., Basak, S. C., Restrepo, G., Villaveces, J. L., et al. MOLGEN 5.0, A Molecular Structure Generator. Advances in Mathematical Chemistry and Applications. , 113-138 (2015).
- Yirik, M. A., Steinbeck, C. Chemical graph generators. PLOS Computational Biology. 17 (1), 1008504 (2021).
- Jaghoori, M. M., et al. PMG: multi-core metabolite identification. Electronic Notes in Theoretical Computer Science. 299, 53-60 (2013).
- Yirik, M. A., Sorokina, M., Steinbeck, C. MAYGEN: an open-source chemical structure generator for constitutional isomers based on the orderly generation principle. Journal of Cheminformatics. 13 (1), 48 (2021).
- Sims, C. C., Leech, J. Computational methods in the study of permutation groups. Computational Problems in Abstract Algebra. , 169-183 (1970).
- Mat, W. -. K., Xue, H., Wong, J. T. -. F. The genomics of LUCA. Frontiers in Bioscience. 13, 5605-5613 (2008).
- Fournier, G. P., Alm, E. J. Ancestral reconstruction of a pre-LUCA aminoacyl-tRNA synthetase ancestor supports the late addition of Trp to the genetic code. Journal of Molecular Evolution. 80 (3-4), 171-185 (2015).
- Higgs, P. G., Pudritz, R. E. A Thermodynamic basis for prebiotic amino acid synthesis and the nature of the first genetic code. Astrobiology. 9 (5), 483-490 (2009).
- Trifonov, E. N. Consensus temporal order of amino acids and evolution of the triplet code. Gene. 261 (1), 139-151 (2000).
- Cleaves, H. J. The origin of the biologically coded amino acids. Journal of Theoretical Biology. 263 (4), 490-498 (2010).
- Moosmann, B. Redox biochemistry of the genetic code. Trends in Biochemical Sciences. 46 (2), 83-86 (2021).
- Simkus, D. N., et al. Methodologies for analyzing soluble organic compounds in extraterrestrial samples: amino acids, amines, monocarboxylic acids, aldehydes, and ketones. Life. 9 (2), 47 (2019).
- Criado-Reyes, J., Bizzarri, B. M., García-Ruiz, J. M., Saladino, R., Di Mauro, E. The role of borosilicate glass in Miller-Urey experiment. Scientific Reports. 11 (1), 21009 (2021).
- Parker, E. T., et al. Primordial synthesis of amines and amino acids in a 1958 Miller H2S-rich spark discharge experiment. Proceedings of the National Academy of Sciences of the United States of America. 108 (14), 5526-5531 (2011).
- Bada, J. L. New insights into prebiotic chemistry from Stanley Miller's spark discharge experiments. Chemical Society Reviews. 42 (5), 2186-2196 (2013).
- Yap, C. W. PaDEL-descriptor: An open source software to calculate molecular descriptors and fingerprints. Journal of Computational Chemistry. 32 (7), 1466-1474 (2011).
- SMARTS - A language for describing molecular patterns. Daylight Chemical Information Systems, Inc Available from: https://www.daylight.com/html/doc/theory/theory.smarts.html (2019)
- Meringer, M., Cleaves, H. J., Freeland, S. J. Beyond terrestrial biology: charting the chemical universe of α-amino acid structures. Journal of Chemical Information and Modeling. 53 (11), 2851-2862 (2013).
- Zherebker, A., et al. Speciation of organosulfur compounds in carbonaceous chondrites. Scientific Reports. 11 (1), 7410 (2021).
- Tanford, C. The hydrophobic effect and the organization of living matter. Science. 200 (4345), 1012-1018 (1978).
- Grantham, R. Amino acid difference formula to help explain protein evolution. Science. 185 (4154), 862-864 (1974).
- Cordero, B., et al. Covalent radii revisited. Dalton Transactions. (21), 2832-2838 (2008).
- Cleaves, H. J., Butch, C., Burger, P. B., Goodwin, J., Meringer, M. One among millions: the chemical space of nucleic acid-like molecules. Journal of Chemical Information and Modeling. 59 (10), 4266-4277 (2019).
Przedruki i uprawnienia
Zapytaj o uprawnienia na użycie tekstu lub obrazów z tego artykułu JoVE
Zapytaj o uprawnieniaThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Wszelkie prawa zastrzeżone