Method Article
Oi-C: um método para estudar a arquitetura tridimensional de Genomas.
* Estes autores contribuíram igualmente
Neste Artigo
Erratum Notice
Resumo
O método de Hi-C permite a identificação imparciais, de todo o genoma de interações cromatina (1). Oi-C casais ligadura proximidade e seqüenciamento massivamente paralelo. Os dados resultantes podem ser usados para estudar a arquitetura genômica em múltiplas escalas: resultados iniciais identificadas características como territórios cromossomo, a segregação da cromatina abertas e fechadas, e estrutura da cromatina na escala megabase.
Resumo
O dobramento tridimensional de cromossomos do genoma e compartimenta e pode trazer distantes elementos funcionais, tais como promotores e enhancers, em estreita proximidade espacial 2-6. Decifrar a relação entre a organização e atividade do cromossomo do genoma ajudará na compreensão dos processos genômica, como transcrição e replicação. No entanto, pouco se sabe sobre como os cromossomos fold. Microscopia é incapaz de distinguir um grande número de loci simultaneamente ou em alta resolução. Até o momento, a detecção de interações cromossômicas usando captura de cromossomo conformação (3C) e suas adaptações posteriores necessária a escolha de um conjunto de loci alvo, fazendo estudos genômicos 70-10 impossível.
Nós desenvolvemos Hi-C, uma extensão do 3C que é capaz de identificar interações de longo alcance em uma imparciais, moda de todo o genoma. Em Hi-C, as células são fixadas com formaldeído, causando loci interagem para ser vinculadas entre si por meio de ligações covalentes DNA-proteína ligações cruzadas. Quando o DNA é posteriormente fragmentado, com uma enzima de restrição, estes loci permanecem ligados. Um resíduo biotinilado é incorporado como o 5 'saliências são preenchidos Em seguida, ligadura blunt-end é realizada sob condições que favorecem a diluir eventos ligadura entre os fragmentos cruzada DNA. Isso resulta em uma biblioteca do genoma de produtos ligadura, o que corresponde a pares de fragmentos que foram originalmente em estreita proximidade um do outro no núcleo. Cada produto ligadura é marcado com biotina no local da junção. A biblioteca é cortado, e as junções são puxados para baixo com contas estreptavidina. As junções purificado pode ser posteriormente analisados usando um seqüenciador de alto rendimento, resultando em um catálogo de fragmentos de interação.
A análise direta da matriz de contato resultante revela inúmeros recursos de organização genômica, tais como a presença de territórios cromossomo ea associação preferencial de pequenas gene-rico cromossomos. Análise de correlação pode ser aplicada à matriz de contato, demonstrando que o genoma humano é segregado em dois compartimentos: um compartimento menos densamente contendo cromatina aberta, acessível, e ativo, e um compartimento mais denso contendo fechada, inacessível, e inativos regiões de cromatina. Finalmente, a análise ensemble da matriz de contato, juntamente com derivações teóricas e simulações computacionais, revelou que na escala megabase Hi-C revela características consistentes com uma conformação glóbulo fractal.
Protocolo
Este método foi utilizado na pesquisa, publicada na Lieberman-Aiden et al. Ciência 326, 289-293 (2009) .
I. Crosslinking, digestão, Marcação de DNA Ends, e Blunt-end Ligadura
- Oi-C começa com reticulação de células, que é um traço comum entre todos os métodos de 3C-based. Para começar, crescer entre 2 x 10 7 e 2,5 x 10 7 células de mamíferos, seja aderente ou em suspensão, e crosslink as células. (Para detalhes sobre crosslinking de células, consulte: 11
- Lisar as células em 550 mL lysisbuffer (500 mL 10 mM Tris-HCl pH 8,0, 10 mM NaCl, 0,2% Igepal CA-630 e 50 mL inibidores da protease), utilizando um homogeneizador. Girar a cromatina menos 5.000 rpm e lavar o pellet duas vezes com 500 mL NEBuffer 1x 2.
- Ressuspender o cromatina em 1x NEBuffer 2, alíquota em 5 tubos numerados e adicione 1x NEBuffer 2 para um volume final de 362 mL. Adicionar 38 mL SDS 1%, misture cuidadosamente e incubar a 65 ° C por 10 minutos. Coloque os tubos de volta no gelo imediatamente após a incubação.
- Saciar a SDS, adicionando 44 mL Triton X-100 e misture com cuidado. Digerir a cromatina, adicionando 400 unidades de HindIII e incubar a 37 ° C durante a noite enquanto gira.
- Os próximos passos são Hi-C específicos e incluem a marcação do DNA termina com biotina e realizando blunt-end ligadura de fragmentos reticulado. Este passo vai permitir junções ligadura de ser purificados depois. Tubo 1 não devem ser submetidos a etapa Biotinilação e devem ser mantidos separados, em vez e servem como um controle de 3C para garantir que a digestão, e as condições de ligadura foram ótimas.
- Para preencher as saliências fragmento de restrição e marcar o DNA termina com biotina nos restantes 4 tubos, adicionar 1,5 mL 10 mM dATP, 1,5 mL 10 mM dGTP, 1,5 mL 10 mM dTTP, 37,5 mM 0,4 mL biotina-14-dCTP, e 10 mL Klenow 5U/μl para tubos de 2-5. Misture cuidadosamente e incubar por 45 minutos a 37 ° C.
- Coloque os tubos no gelo. Para inativar as enzimas, adicionar 86 mL SDS 10% para tubos de 1-5. Incubar os tubos a 65 ° C por exatamente 30 minutos e colocá-los no gelo imediatamente depois.
- A ligadura é realizada sob condições extremamente diluídas de forma a favorecer eventos ligadura entre os fragmentos reticulado. Trabalho sobre gelo, adicionar 7,61 ml mix ligadura [745 mL de 10% Triton X-100, 745 mL de buffer ligadura 10x (500 mM Tris-HCl pH 7,5, 100 mM MgCl 2, 100 mM DTT), 80 mL 10 mg / ml BSA , 80 mL 100 mM ATP e 5,96 ml de água] para cada uma das cinco numeradas de 15 tubos de ml. Transferência de cada mistura cromatina digerida para um tubo correspondente ml 15.
- Para ligadura 3C regular, adicionar 10 mL DNA ligase T4 1U/μl ao tubo 1. Para blunt-end ligadura Hi-C, adicionar 50 mL DNA ligase 1U/μl T4 para tubos de 2-5. Misture invertendo os tubos e incubar todos os tubos de 5 para 4 horas a 16 ° C.
- Ligações cruzadas são invertidos e proteína é degradada pela adição de 50 ul 10 mg / ml proteinase K por tubo e incubar os tubos durante a noite a 65 ° C. Adicionar um adicional de 50 ul 10 mg / ml proteinase K por tubo no dia seguinte e continuar a incubação a 65 ° C por mais 2 horas.
- Esfriar a mistura de reação à temperatura ambiente e transferi-los para cinco tubos de 50 ml cônico. Purificar o DNA nesses tubos através da realização de uma extração de fenol. Adicionar 10 ml de fenol pH 8,0 e vortex por 2 minutos. Girar os tubos por 10 minutos a 3.500 rpm e transferir cuidadosamente tanto da fase aquosa quanto possível para um tubo novo 50 ml.
- Repita a extração utilizando fenol pH 8,0: clorofórmio (1:1) e precipitar o DNA com etanol. (Para detalhes sobre a purificação do DNA, consulte: 11
- Após a centrifugação do etanol precipitado DNA, dissolver cada pellet de DNA em 450 mL 1x TE (10 mM Tris-HCl pH 8,0, 1 mM EDTA). Transfira a mistura de DNA para um tubo de centrifugação 1,7 ml.
- Outra rodada de purificação é realizada, fazendo 2 fenol: clorofórmio extrações. Adicionar 500 mL de fenol pH 8,0: clorofórmio (1:1) e vortex por 1 minuto. Centrifugue os tubos durante 5 minutos a 14.000 rpm e transferir a fase aquosa para um novo tubo. Após a segunda extração, precipitar o DNA pela adição de 0,1 x o volume de NaOAc, volume 2x de etanol 100% e incubar 30 minutos a -80 ° C.
- Depois de girar para baixo o DNA precipitado, lavar cada pellet de DNA com etanol 70% e ressuspender cada pellet de DNA em 25 mL 1x TE. Degradar qualquer RNA que podem estar presentes, adicionando 1 ml 1 mg / ml RNAse tubo A por e incubando os tubos durante 30 minutos a 37 ° C. Piscina o conteúdo Hi-C de tubos de 2-5, mantendo ainda um tubo separado como um controle de 3C.
- Agora é uma boa oportunidade para examinar a marcação Hi-C e eficiência ligadura. Esses controles são excelentes indicadores de saber se uma biblioteca de Hi-C vai ser bem sucedido.
- Para verificar a qualidade ea quantidade das bibliotecas, execute 2 l alíquotas e 6 mL de diluições 1:10 de ambos 3C e Hi-C bibliotecas em um gel de agarose 0,8%. (Veja Figura 2A)
- Oi-C marcação e Hi-C eficiência ligadura é verificada por um ensaio de PCR digerir. Bem sucedida de preenchimento e ligadura de um site HindIII (AAGCTT) cria um site para a enzima de restrição NheI (GCTAGC). Um produto ligadura especial formada por dois fragmentos de restrição por perto é amplificado por PCR (como em 3C 11 utilizando 0,2 mL de cada biblioteca como modelo. Os produtos de PCR são posteriormente digeridos com HindIII, NheI ou ambos. Depois de correr as amostras em um gel 2% , o número relativo de 3C e eventos Hi-C ligadura pode ser estimada pela quantificação da intensidade das bandas cortadas e sem cortes (Figura 2B).
- Alguns fragmentos não terá sido ligado: para evitar puxá-los para baixo mais tarde, remova biotina a partir destes termina sem ligadura usando a atividade exonuclease de DNA polimerase T4.
- Biotina-14-dCTP nas extremidades não-ligados DNA é removido com a atividade exonuclease de DNA polimerase T4. Misturar 5 mg de Hi-C biblioteca com 1 ml 10 mg / ml BSA, 10 mL NEBuffer 10x 2, 1 ml 10 mM dATP, dGTP 1 ml 10 mM e 5 Unidades de T4 DNA polimerase em um volume total de 100 l e incubar a mistura a 12 ° C por 2 horas. Se possível, múltiplas reações 5 mg são executadas.
- A reação é interrompida pela adição de 2 mL de EDTA 0,5 M pH 8.0.
- Para purificar o DNA, um fenol pH 8,0: clorofórmio extração (1:1) é feito seguido por precipitação de etanol.
- O sobrenadante é descartado e os pellets de DNA são ressuspenso e reunidos num volume total de 100 mL de água.
II. Shearing e Seleção Tamanho
- Para fazer o DNA biotinilado adequada para alto rendimento de sequenciamento, o DNA deve ser cortado em um tamanho de 300-500 pares de bases com um instrumento Covaris S2 (ciclo de 5, intensidade de 5, ciclos / estourar 200, o tempo de 60 segundos para 4 ciclos) .
- Para reparar o DNA termina cortado, adicionar 14 mL de buffer ligadura 10x, 14 mL 2,5 mM dNTP mix, 5 mL T4 DNA polimerase, 5 mL T4 polinucleotídeo quinase, uma DNA polimerase Klenow mL e 1 mL de água. Incubar por 30 minutos em temperatura ambiente.
- Após a incubação, use uma coluna MinElute Qiagen para purificar o DNA de acordo com recomendações do fabricante. Eluir o DNA por duas vezes com 15 mL 1x Tris-Low-EDTA (TLE: 10 mM Tris pH 8,0, 0,1 mM EDTA). Então, anexar um dATP para termina a 3 'do fim-reparado DNA pela adição de 5 mL NEBuffer2 10x, 10 ml 1 mM dATP, 2 mL de água e 3 mL Klenow (exo-). Incubar a reação por 30 minutos a 37 ° C.
- Para inativar o fragmento Klenow, incubar as reações durante 20 minutos a 65 ° C e, posteriormente, esfriar a reações no gelo. Usando um speedvac, reduzir os volumes de reação a 20 l.
- Em seguida, carregue o DNA em um gel de agarose 1,5% com TAE 1X e correr para 3,5 horas a 80-90V. Após a coloração do gel com SYBR green, visualizar o DNA em um DarkReader. Fragmentos de DNA especiais de consumo entre 300 e 500 pares de base e purificá-los com um kit de extração de gel Qiagen usando colunas 2-4, dependendo do peso do gel. Eluir o DNA com 50 mL TLE 1x.
- Combine o eluatos das colunas QIAquick e trazer o volume final de até 300 ml com 1x TLE. Finalmente, determinar a concentração de DNA com o ensaio Quant-iT usando o fluorímetro qubit e calcular a quantidade total de DNA.
III. Biotina Pull-down e emparelhados final de Seqüenciamento
- Nesta secção do protocolo, junções ligadura são purificados a partir do pool de DNA, permitindo a identificação eficiente de interagir fragmentos de cromatina por emparelhado fim-de seqüenciamento. Executar todas as etapas subseqüentes em tubos LoBind DNA.
- Prepare contas de biotina suspenso por lavagem 150 mL ressuspenso contas estreptavidina magnética duas vezes com 400 mL de buffer Tween (TB: 5 mM Tris-HCl pH 8,0, 0,5 mM EDTA, 1 M NaCl, Tween 0,05%).
Lavagens estas e futuras consistem em cinco passos:- Adicionar buffer para as contas
- Transfira a mistura para um novo tubo
- Girar a amostra por 3 minutos em temperatura ambiente
- Recuperar as contas usando um concentrador de partículas magnéticas
- Retire o sobrenadante
- Volte a suspender as contas em 300 mL 2x Não tampão Tween (2x NTB: 10 mM Tris-HCl pH 8,0, 1 mM EDTA, 2 M NaCl) e combinar com 300 DNA Hi-C mL. Permitir que a biotina marcada Hi-C de DNA para vincular as contas estreptavidina incubando a mistura em temperatura ambiente por 15 minutos com rotação.
- Recuperar o DNA ligado contas com a estreptavidina Concentr partículas magnéticasAtor, e remover o sobrenadante. Lave as contas em 400 mL 1x NTB (5 mM Tris-HCl pH 8,0, 0,5 mM EDTA, 1 M NaCl), seguido por 100 mL de buffer ligadura 1x. Volte a suspender as contas em 50 mL de buffer ligadura 1x e transferir a mistura para um novo tubo.
- Para preparar o DNA para o seqüenciamento End Illumina emparelhados, tomar a quantidade total de DNA utilizado como entrada para a biotina pull-down, que foi calculado anteriormente na etapa 2.6, e dividir por 20 para estimar a quantidade de Hi-C de DNA que tem foi puxado para baixo e está disponível para ligadura. Adicionar 6 picomoles da Illumina adaptadores End emparelhados por mg de Hi-C de DNA disponíveis para ligadura. Use 1.200 Unidades de DNA Ligase T4 para ligadura dos adaptadores ao DNA. Incubar por 2 horas em temperatura ambiente.
- Remover não ligados adaptadores End emparelhados com a recuperação de contas Hi-C DNA ligado e lavando as contas duas vezes com 400 mL TB 1x.
- Lave as contas com 200 mL NTB 1x, seguido de 200 mL e em seguida 50 mL NEBuffer 1x 2. Após a última lavagem, ressuspender as contas em 50 NEBuffer 1x 2 mL e transferir para um novo tubo.
- Para determinar o número de ciclos necessários para gerar produtos suficientes PCR para o seqüenciamento, criada quatro reações do teste de PCR com ciclos de 6, 9, 12 ou 15. (Para mais detalhes sobre a amplificação PCR, consulte: 12 Determine o número de ciclo ótimo, executando as reações de PCR em gel de poliacrilamida 5% e coloração com SYBR Green, garantindo a ausência de bandas espúrias ea presença de uma mancha entre 400 -. 600 pares de base, que é o comprimento dos produtos cortados após a ligadura para os adaptadores.
- Amplificar o resto das esferas Hi-C-biblioteca-bound estreptavidina em um PCR em larga escala com o número ideal de ciclos de PCR. Piscina dos produtos PCR dos poços separados e recuperar as contas. Manter 1% da escala grande de produtos PCR separada para executar em um gel e purificar o restante do produto da PCR com contas Ampure 1.8x volume de acordo com as recomendações do fabricante.
- Eluir o DNA com 50 mL de buffer TLE 1x e compare 1% do talão purificada Ampure produto da PCR para a alíquota de 1% do produto da PCR original em gel de poliacrilamida a 5%, garantindo a remoção do primers PCR.
- Recomendamos também a clonagem 1 ml da biblioteca Hi-C e determinar o produto de cerca de 100 clones usando seqüenciamento Sanger. Isto irá permitir que você avalie o número relativo de alignable Hi-C lê na mistura de PCR. Para os resultados típicos, ver Figura 3B.
- Seqüência biblioteca do Hi-C com seqüenciamento final Illumina emparelhados. Alinhar cada extremidade independentemente usando Maq (http://maq.sourceforge.net/) para identificar fragmentos de cromatina interagindo.
IV. Representante Hi-C Resultados
- Os seguintes resultados são esperados quando o protocolo de Hi-C é executada tecnicamente bem e podem ser considerados padrões de controle de qualidade.
- Medidas de controle de qualidade deve revelar que ambas as bibliotecas 3C e Hi-C run bandas como apertado em vez maior que 10 kb. Uma mancha DNA indica a eficiência ligadura pobres. Tipicamente, a eficiência ligadura é ligeiramente inferior em uma biblioteca de Hi-C, em comparação com um modelo 3C (ver Figura 2a).
- Oi-C marcação e eficiência ligadura pode ser estimado por digestão de um produto PCR gerados usando primers 3C. Junções 3C são cortadas por HindIII e não por NheI. O inverso é verdadeiro para Hi-C cruzamentos. Este PCR digerir mostra que 70% dos Hi-C amplicons são cortadas por NheI e não por HindIII, confirmando marcação eficiente de cruzamentos ligadura (Veja Figura 2B).
- Análise das leituras seqüenciadas deve mostrar que lê a partir de interações tanto intrachromosomal e interchromosomal, indicado pelas linhas azul e vermelha, alinhar significativamente mais perto de sítios de restrição HindIII, em comparação com geradas aleatoriamente lê, mostrado em verde (veja a Figura 3A).
- Em uma experiência de sucesso, 55% dos pares alignable ler representam interações interchromosomal. Quinze por cento representam as interações entre os fragmentos intrachromosomal menos de 20 kb de distância e 30% são intrachromosomal pares de ler que são mais de 20 kb de distância (veja a Figura 3B). Esta distribuição pode ser provado antes de high-throughput seqüenciamento, como uma forma de controle de qualidade; clonagem e sequenciamento Sanger de cerca de 100 clones é geralmente suficiente.
- As interações cromatina pode ser visualizado como um mapa de calor, onde o x-e y-eixos representam loci para genômica e cada pixel representa o número de interações observadas entre eles. Normalmente, os fragmentos de DNA que são muito próximos uns dos outros no genoma linear terá a tendência de interagir com freqüência com o outro. Isto é visto na heatmaps intrachromosomal como uma diagonal de destaque (Figura 4A).
- Os resultados a seguir mostram diferentes formas de análise dos dados a revelar vários níveis de organização do genoma. Locando a probabilit contatoy distância genômica versus (Ver Figura 5A) mostra que a probabilidade de contato diminui em função da distância genômica, eventualmente, atingir um platô. Em cada distância, as interações intrachromosomal, mostrado na linha sólida, são enriquecidos em relação às interações interchromosomal, representadas pelas linhas tracejadas. Esta implica diretamente a presença de territórios cromossomo.
- Cálculo do número observado / esperado de contatos interchromosomal entre todos os pares de cromossomos revela associação preferencial entre pares de cromossomos particular. Cromossomos gene-rico pequeno preferencialmente interagem uns com os outros, indicados pela cor vermelha brilhante (ver Figura 5B).
- Cromossomos individuais também podem ser examinadas. O heatmap-prima pode ser ajustada usando um heatmap esperado para dar conta da distância genômica entre pares de locos, resultando em um heatmap observados / esperados. Então, uma matriz de correlação podem ser produzidos, correlacionando as linhas e colunas da heatmaps observados / esperados. Usando análise de correlação, é demonstrado que o ser humano segrega genoma em dois compartimentos. Isto é ilustrado pelo padrão plaid-nos heatmaps correlação (Figura 4A-D). (Para mais detalhes sobre a análise dos dados Hi-C, consulte: 1.
- Usando Hi-C de dados, novas percepções foram obtidas em dobrar cromatina na escala megabase. O modelo clássico de polímero de condensação sugere que pacotes de cromatina em um glóbulo de equilíbrio. Probabilidade de contato plotagem em função da distância ilustra que as escalas de probabilidade de contato como uma lei de potência com a distância genômica cuja inclinação é de cerca de -1 (veja a Figura 6A). Isto não é consistente com o comportamento de um glóbulo de equilíbrio, mas não coincidem com o esperado para uma estrutura alternativa conhecida como um glóbulo fractal (Figura 6B).
- Aqui, duas estruturas globulares são mostrados. Coloração corresponde à distância de um ponto final, que vão do azul ao turquesa, verde, amarelo, laranja e vermelho (Veja os principais Figura 6C). Ao contrário dos glóbulos de equilíbrio, glóbulos fractal falta embaraços. Em um glóbulo fractal, loci que estão próximas ao longo do contorno tendem a ser próximos, em 3D, levando à presença de blocos monocromática (Veja a Figura 6C meio). Tais blocos não são encontrados no glóbulo de equilíbrio (Veja a parte inferior Figura 6C).
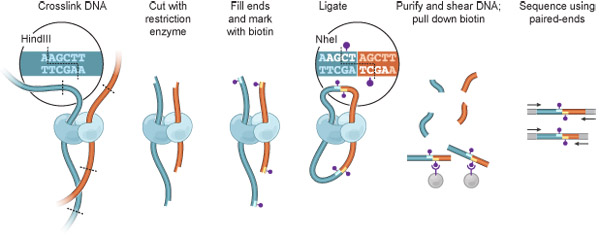
Figura 1. Panorama Hi-C. Células são ligadas com formaldeído, resultando em ligações covalentes entre os segmentos de cromatina espacialmente adjacentes (fragmentos de DNA: azul escuro, vermelho; Proteínas, que pode mediar tais interações, são mostrados na luz azul e ciano). Cromatina é digerido com uma enzima de restrição (aqui, HindIII; sítio de restrição: linha tracejada, ver caixa). As extremidades, resultando sticky são preenchidos com nucleotídeos, um dos quais é biotinilado (ponto roxo). Ligadura é realizada sob condições extremamente diluídas favorecendo eventos ligadura intramolecular; o site HindIII está perdido e um site NheI é criado (inset). DNA é purificado e cortado, e junções biotinilado são isolados usando pérolas de estreptavidina. Fragmentos de interação são identificados por par-end seqüenciamento.

Figura 2. Hi-C controles de qualidade da biblioteca. (A) quantidades cada vez maiores de um controle 3C e uma biblioteca de Hi-C foram resolvidos em gel de agarose 0,8%. Ambas as bibliotecas são executados como uma banda muito estreita com mais de 10 kb. Eficiência ligadura típico em uma biblioteca de Hi-C é ligeiramente inferior ao que é observado em um modelo 3C, e é indicado pela mancha nas pistas Hi-C. (B) PCR controle de digerir. A junção ligadura formada por dois fragmentos nas proximidades é amplificada usando condições padrão 3C PCR. Produtos de oi-C ligadura podem ser distinguidos daqueles produzidos em 3C convencionais por digestão do site ligadura. Oi-C junções são cortadas por NheI não, HindIII; o inverso é verdadeiro para junções de 3C. 70% de Hi-C amplicons foram cortados por NheI, confirmando eficiente marcação de junção ligadura. Duas repetições foram realizadas para garantir a quantificação confiável.

Figura 3. Hi-C ler controles de qualidade. (A) Lê a partir de fragmentos correspondentes a ambos os intrachromosomal (azul) e interchromosomal (vermelho) interações alinhar significativamente mais perto de sítios de restrição HindIII, em comparação com lê gerados aleatoriamente (verde). Tanto o intrachromosomal lê e lê interchromosomal curvas diminuem rapidamente como a distância do site aumenta HindIII até um platô é atingido a uma distância de ~ 500 pb. Isso corresponde ao tamanho do fragmento máxima utilizada para o seqüenciamento. (B) Normalmente, 55% dos pares alignable ler representam interações interchromosomal. Quinze por cento representam as interações entre os fragmentos intrachromosomal menos de 20 kbdistante e 30% são intrachromosomal pares ler que são mais de 20 kb de distância. Esta distribuição pode ser provado antes de high-throughput seqüenciamento, como uma forma de controle de qualidade; clonagem e sequenciamento Sanger de cerca de 100 clones é geralmente suficiente.

Figura 4. A análise de correlação demonstra que o núcleo é segregado em dois compartimentos. (A) Mapa de calor correspondente às interações intrachromosomal no cromossomo 14. Cada pixel representa todas as interações entre um locus de 1 Mb e outro lócus de 1 Mb; intensidade corresponde ao número total de leituras (intervalo: 0-200 leituras). Marcas aparecem a cada 10 Mb. A subestrutura heatmap exibe na forma de uma diagonal intensa e uma constelação de grandes blocos. (Cromossomo 14 é acrocêntrico;. Braço curto não é mostrado) Utilizando o conjunto de dados Hi-C para calcular a probabilidade média de contato por um par de loci a uma distância genômica dado, uma matriz expectativa é produzido (B) correspondente ao que seria observado se não houvesse estruturas de longo alcance. O quociente dessas duas matrizes é uma matriz observados / esperados (C) onde o esgotamento é mostrado em azul e em vermelho enriquecimento [intervalo: 0,2 (azul) a 5 (vermelho)]. O padrão de bloqueio se torna mais evidente. A matriz de correlação (D) ilustra a correlação [intervalo: -1 (azul) para 1 (vermelha)] entre os perfis de interação intrachromosomal de cada par de locos ao longo do cromossomo 14. O padrão xadrez marcante indica a presença de dois compartimentos dentro do cromossomo.

Figura 5. A presença e organização dos territórios cromossomo. (A) Probabilidade de contato diminui em função da distância genômica no cromossomo 1, acabou atingindo um platô em ~ 90MB (azul). O nível de contato interchromosomal (traços pretos) é diferente para diferentes pares de cromossomos; loci no cromossomo 1 são mais propensos a interagir com loci no cromossomo 10 (traços verde) e menos propensos a interagir com loci no cromossomo 21 (traços vermelhos). Interações Interchromosomal estão esgotados em relação às interações intrachromosomal. (B) observados / esperados número de contatos interchromosomal entre todos os pares de cromossomos. Vermelho indica enriquecimento e azul indica [intervalo: 0,5 (azul) a 2 (vermelho)] esgotamento. Pequeno, gene-rico cromossomos tendem a interagir mais com o outro.

Figura 6. A embalagem locais da cromatina é consistente com o comportamento de um glóbulo fractal. (A) a probabilidade de contato em função da distância genômica, em média, em todo o genoma (azul). A lei de escala proeminente poder é visto entre 500kb e 7Mb (região sombreada) com uma inclinação de -1,08 (ajuste mostrado em ciano). (B) Os resultados das simulações para a probabilidade de contato como uma função da distância para o equilíbrio (vermelho) e fractal (azul) glóbulos. A inclinação de um glóbulo fractal é quase -1 (cyan), confirmando nossa previsão romance teórica 1. A inclinação de um glóbulo de equilíbrio é -3 / 2, que corresponde às expectativas teórico prévio. A inclinação para o glóbulo fractal se assemelha a inclinação observada nos resultados Hi-C, enquanto que a inclinação para um glóbulo de equilíbrio não é visto nos dados Hi-C. (C) Superior: Um desdobrado cadeia polimérica, 4000 monômeros longo. Coloração corresponde à distância de um ponto final, que vão do azul ao turquesa, verde, amarelo, laranja e vermelho Médio:. Exemplo típico de um glóbulo fractal extraídos de nossa ensemble. Glóbulos Fractal falta embaraços. Loci que estão próximas ao longo do contorno tendem a ser próximos, em 3D, levando à presença de grandes blocos monocromáticas que são visíveis na superfície e na seção transversal inferior:. Um glóbulo de equilíbrio. A estrutura é altamente emaranhado; loci que estão próximas ao longo do contorno (cor similar) não precisam ser próximos, em 3D.
Discussão
Nós apresentamos um método de estudo da arquitetura 3-dimensional do genoma com o mapeamento interações cromatina em um imparciais, forma genome-wide. A etapa mais crítica experimental o que define esta tecnologia além de trabalhos anteriores - é a incorporação de nucleotídeos biotinilado na restrição acaba de fragmentos reticuladas antes blunt-end ligadura. Realizar esta etapa com sucesso permite profunda seqüência de todos os cruzamentos ligadura, e dá Hi-C seu alcance e poder.
O número de leituras irá determinar a resolução dos mapas de interação. Aqui, a 1 Mb mapa de interação para o genoma humano é apresentado por meio de ~ 30 milhões lê alignable. , A fim de aumentar a resolução "para todos os fins-'por um fator de n, o número de leituras deve ser aumentada por um fator de n 2.
A técnica de Hi-C pode ser facilmente combinada com outras técnicas, como a captura híbrida após geração biblioteca (para atingir partes específicas do genoma) e imunoprecipitação da cromatina após a ligadura (para analisar o ambiente de cromatina de regiões associadas com proteínas específicas).
Divulgações
Agradecimentos
Agradecemos a A. Kosmrlj para discussões e código; AP Aiden, XR Bao, M. Brenner, D. Galas, W. Gosper, A. Jaffer, A. Melnikov, A. Miele, G. Giannoukos, C. Nusbaum, AJM Walhout , L. Wood, e K. Zeldovich para discussões, e L. Gaffney e B. Wong para ajudar com a visualização.
Apoiado por uma Fannie e John Hertz Fundação de pós-graduação comunhão, um Defesa Nacional de Ciência e de pós-graduação de Engenharia comunhão, uma comunhão NSF de pós-graduação, o National Space Biomedical Research Institute, e não conceder. T32 HG002295 do National Human Genome Research Institute (NHGRI) (EL); i2b2 (Informática para a Integração da Biologia e da cabeceira), o NIH-suportados Centro de Computação Biomédica na Brigham and Women s Hospital (LAM), não conceder. HG003143 do NHGRI, e um Keck Fundação distinguiu prêmio jovem estudioso (JD). Dados da seqüência primas e mapeados Hi-C tenha sido depositado no banco de dados GEO ( www.ncbi.nlm.nih.gov/geo/ ), a adesão não. GSE18199. Visualizações adicionais estão disponíveis em http://hic.umassmed.edu .
Materiais
| Name | Company | Catalog Number | Comments |
| Protease inhibitors | Sigma-Aldrich | P8340-5ml | Step 1.2 |
| biotin-14-dCTP | Invitrogen | 19518-018 | Step 1.6 |
| Klenow | New England Biolabs | M0210 | Steps 1.6 and 2.2 |
| T4 DNA ligase | Invitrogen | 15224 | Step 1.9 |
| T4 DNA polymerase | New England Biolabs | M0203 | Steps 1.17 and 2.2 |
| 10x ligation buffer | New England Biolabs | B0202 | Steps 2.2 and 3.4 |
| T4 PNK | New England Biolabs | M0201 | Step 2.2 |
| Klenow (exo-) | New England Biolabs | M0212 | Step 2.3 |
| Dynabeads MyOne Streptavin C1 Beads | Invitrogen | 650.01 | Step 3.2 |
| T4 DNA ligase HC | Enzymatics | L603-HC-L | Step 3.5 |
| Phusion HF mastermix | New England Biolabs | F531 | Step 3.8 |
| Ampure beads | Beckman Coulter Inc. | A2915 | Step 3.9 |
Referências
- Lieberman-Aiden, E., Van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., Amit, I., Lajoie, B. R., Sabo, P. J., Dorschner, M. O., Sandstrom, R., Bernstein, B., Bender, M. A., Groudine, M., Gnirke, A., Stamatoyannopoulos, J., Mirny, L. A., Lander, E. S., Dekker, J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Kosak, S. T., Groudine, M. Form follows function: the genomic organization of cellular differentiation. Genes and Dev. 18, 1371-1384 (2004).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (2007).
- Dekker, J. Gene Regulation in the Third Dimension. Science. 319, 1793-1794 (2008).
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet. 2, 292-301 (2001).
- Sexton, T., Schober, H., Fraser, P., Gasser, S. M. Gene regulation through nuclear organization. Nat Struct and Mol Biol. 14, 1049-1055 (2007).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing Chromosome Conformation. Science. 295, 1306-1311 (2002).
- Zhao, Z., Tavoosidana, G., Sjölinder, M., Göndör, A., Mariano, P., Wang, S., Kanduri, C., Lezcano, M., Sandhu, K. S., Singh, U., Pant, V., Tiwari, V., Kurukuti, S., Ohlsson, R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1341-1347 (2006).
- Simonis, M., Klous, P., Splinter, E., Moshkin, Y., Willemsen, R., de Wit, E., van Steensel, B., de Laat, W. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1348-1354 (2006).
- Dostie, J., Richmond, T. A., Arnaout, R. A., Selzer, R. R., Lee, W. L., Honan, T. A., Rubio, E. D., Krumm, A., Lamb, J., Nusbaum, C., Green, R. D., Dekker, J. Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Res. 16, 1299-1309 (2006).
- Miele, A., Dekker, J. Mapping Cis- and Trans Chromatin Interaction Networks Using Chromosome Conformation Capture (3C). Methods Mol Biol. 464, 105-121 (2009).
- Maccallum, I., Przybylski, D., Gnerre, S., Burton, J., Shlyakhter, I., Gnirke, A., Malek, J., McKernan, K., Ranade, S., Shea, T. P., Williams, L., Young, S., Nusbaum, C., Jaffe, D. B. ALLPATHS 2: small genomes assembled accurately and with high continuity from short paired reads. Genome Biol . 10, R103-R103 (2009).
Erratum
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados