É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Determinação de metilação de DNA de genes marcados em Arabidopsis Endosperma
Neste Artigo
Resumo
Imprinting é um fenômeno em plantas e da reprodução de mamíferos. Metilação do DNA desempenha um papel importante nos mecanismos de imprinting. Isolando endosperma e determinação do estatuto de metilação de genes imprinted em Arabidopsis Pode ser difícil. Neste protocolo, descrevemos como isolar endosperma e determinar a metilação por seqüenciamento bissulfito.
Resumo
Arabidopsis thaliana é um organismo excelente modelo para estudar mecanismos epigenéticos. Uma das razões é o mutante nulo de perda de função de DNA metiltransferases é viável, proporcionando assim um sistema para estudar como a perda da metilação do DNA em um genoma afeta o crescimento e desenvolvimento. Imprinting refere-se a expressão diferencial dos alelos maternos e paternos e desempenha um papel importante no desenvolvimento da reprodução em ambos os mamíferos e plantas. Metilação do DNA é fundamental para determinar se os alelos maternos ou paternos de um gene é expresso impressa ou silenciados. Em plantas com flores, há um evento de fertilização dupla na reprodução: um espermatozóide fertiliza o óvulo para formar um embrião e esperma fusíveis segundo com a célula central para dar origem a endosperma. Endosperma é o tecido onde imprinting ocorre nas plantas. MEDEA, a SET domínio gene grupo Polycomb e FWA, um fator de transcrição que regulam a floração, são os dois primeiros genes mostrado para ser impressa no endosperma e sua expressão é controlada por metilação do DNA e desmetilação em plantas. , A fim de determinar o status de impressão de um gene e padrão de metilação no endosperma, precisamos ser capazes de isolar endosperma primeiro. Desde que a semente é pequena em Arabidopsis, ele continua sendo um desafio para isolar endosperma Arabidopsis e examinar a sua metilação. Neste protocolo de vídeo, relatamos como conduzir um cruzamento genético, para isolar o tecido de endosperma de sementes, e para determinar o estado de metilação de seqüenciamento bissulfito.
Protocolo
I. Genética Travessia
1. Castrar o pai do sexo feminino
A fim de distinguir os alelos maternos e paternos, usando polimorfismo de DNA seqüência, dois ecótipos diferentes, por exemplo, Columbia-0 (Col-0) e Ler, serão escolhidos como pais feminino e masculino. As plantas devem ser jovens e saudáveis. Pode-se castrar o pai do sexo feminino, usando um microscópio de dissecação, ampliando viseira, ou olho nu. Localize estágio-12 flores (Smyth et al., 1990) e remover qualquer flores ou siliques acima e abaixo deles pelo recorte da base do pedicelo com a tesoura. Esterilizar fórceps por imersão a base da ponta suavemente para um copo de etanol a 95%, o que irá remover quaisquer grãos de pólen sobre o forceps e matar o pólen também. Force suavemente para além dos botões florais utilizando fórceps, e remova cuidadosamente 4 sépalas, 4 pétalas e 6 estames, deixando o pistilo nua e intacta. Tente evitar danos ao carpelos durante este processo.
2. Escolher o doador de pólen e realizar a polinização
Os doadores de pólen são melhores anthesed flores abertas na fase 14 com as pétalas estendendo em um ângulo de 90 ° para o pistilo (Smyth et al., 1990), em que uma grande quantidade de pólen é derramamento. Pegue a flor na base e um pouco acima do pedicelo, que fará com que a flor a espalhar aberto. Poeira o estigma do pistilo preparado com a antera. Após a polinização, o estigma será coberto com o pólen amarelo, que pode ser facilmente observado sob um microscópio.
3. Rotulagem da cruz
Depois de polinizadores todos os pistilos emasculados em uma planta, nós rotulamos a cruz com uma etiqueta de jóias e informações dos pais do sexo feminino e masculino e data. Coloque uma estaca no solo próximo à planta, use uma corda para amarrar a haste da inflorescência para a fogueira, e cobrem os pistilos polinizados com um saco plástico.
II. Isolamento de tecidos endosperma em Arabidopsis
1. Preparando materiais
Costumamos receber materiais necessários pronto antes da colheita do endosperma e dos tecidos do embrião: um tanque de nitrogênio líquido, nitrogênio líquido, um microscópio de dissecação, dois novos pares de fina ponta pinças (5 INOX FST por Dumont biologia, na Suíça.), Lâminas de vidro de microscópio ( 3 "X 1" X 1.0 mm), um pH 5,7 solução de 0,3 M sorbitol e 5 mM MES.
2. Siliques coleta
Aos 7 - ou 8 dias de pós-polinização (DAP), as sementes estão prontas para serem colhidas em meados dos anos para a fase final da embriogênese torpedo e dissecados para endosperma e embrião. Às vezes, pode levar 90-10 DAP se as flores emasculadas são muito jovens.
3. Endosperma e embrião isolando
Desde sementes Arabidopsis são pequenos, nós usamos um microscópio de dissecação para isolar endosperma e embrião. Pôr silique 8 DAP-sob um microscópio, use uma pinça para segurar o pedicelo silique e usar a ponta de um outro par de fórceps para deslizar silique a céu aberto na margem onde fundir dois carpelos. Use uma pinça para pegar uma semente em soluções de pH 5,7 a 0,3 M sorbitol e 5 mM MES, faça um pequeno corte no final micrópila a deslizar para fora do embrião, aperte o fim uncut a empurrar para fora endosperma e endosperma separado do tegumento (Kinoshita et al., 2004). Coloque o embrião e endosperma em microtubos separados em nitrogênio líquido. Continue até que acumulamos embrião ou endosperma 10-15 siliques e depois guardar o tubo em um freezer -80 C °. Em alguns casos, endosperma não tem que ser separada da casca da semente, ou seja, pode-se isolar RNA total do endosperma e tegumento mistura para a análise de imprinting gene.
III. Seqüenciamento bissulfito
1. Reagentes necessários
Brometo de amônio cetiltrimetilamônio (CTAB) para a preparação do DNA genômico, enzimas de restrição, 3 M NaOH (recém-feitos), 6,42 M de uréia / 4 bissulfito de sódio M (2 metabissulfito de sódio M, Sigma-Aldrich, S9000, Na2S2O5, Peso Molecular: 190), 10 mM hidroquinona, um kit de purificação de DNA (Promega Assistente de DNA Sistema Clean-up, Cat. # A7280), TE buffer, 6,3 M NaOH (recém feitos), 10 M NH 4 OAc, 20 mcg / mL tRNA e etanol 100% .
2. Detalhes do protocolo de tratamento bissulfito
- Endosperma isolar DNA genômico ou embrião através de um procedimento CTAB (Rogers e Bendich, 1988).
- Digest 100 ng - 2 mg de DNA genômico em 20 mL a 100 mL com volume total enzimas de restrição que cortam fora da região a ser analisada. Para o promotor MEA, usamos XhoI, NdeI e PstI ou HindIII.
- Desnaturar as enzimas de restrição do DNA por fervura durante cinco minutos e depois apagar no gelo.
- Adicione 1 / 9 do volume (2,2 mL para 20 mL de DNA digerido) de 3 M NaOH e incubar a 37 ° C por 15 minutos.
- Transferir a solução para um tubo de PCR 250 mL.
- Dissolver 7,5 g de uréia em 10 mL de água destilada estéril; Lentamente, adicionar 7,6 g de metabissulfito de sódio durante 1-2 horas e aquecimento geralmente ajuda a dissolver; Ajustar o pH para 5 com recém feitos 10 M NaOH; Adicionar água destilada estéril para a final volume para 20 mL. Este é 6,24 M de uréia / 4 M de solução de bissulfito de sódio.
- Adicionar 6,24 M de uréia / 4 M de solução de bissulfito de sódio para uma concentração final de 5,36 M e 3,44 M, respectivamente (Paulin et al., 1998). Por exemplo, adicione 208 mL de 6,42 M de uréia / 4 M de solução de bissulfito de sódio para o mL 22,2 acima do DNA genômico desnaturado (Xiao et al., 2003).
- Adicionar 10 mM hidroquinona ao DNA em uma concentração final de 0,5 mM (12 mL para 20 mL a digestão).
- Realizar tratamento em uma máquina de bissulfito PCR: 30 ciclos de 55 ° C por 15 minutos e 95 ° C por 30 segundos.
- Dessalinizar o DNA bissulfito tratado utilizando o Assistente do Sistema DNA Clean-Up da Promega e acompanhar o protocolo (Jacobsen et al., 2000).
- Medir o volume exato de TE recuperado da coluna após dessalinização e adicionar 6,3 M NaOH a uma concentração final de 0,3 M. Incubar a 37 ° C por 15 minutos.
- Adicionar 10 M NH 4 OAc (pH 7,0) para uma concentração final de 3 M, 2 mL de 20 mcg / mL tRNA e 3 volumes de etanol 100% e, em seguida, misture. Centrifugar por 15 minutos a 14.000 rpm.
- Lave o pellet uma vez com etanol 70%, faça uma centrífuga curto, e remover o etanol extra.
- Pellet seco em um speedvac por 5-10 minutos e ressuspender em 25-100 mL tampão TE dependendo de partida quantidade de DNA. O tratado pelo bissulfito de sódio DNA está pronto para a análise PCR.
3. Amplificação por PCR
- Desde citosinas não metiladas são convertidas em uracila, é difícil para amplificar um fragmento grande usando o DNA tratado pelo bissulfito como um modelo. Assim, costumamos projetar primers para amplificar um produto não superior a 500 pb. Para a seqüência de 4 kb promotor MEA, nós projetamos muitos conjuntos de primers e amplificado 14 fragmentos sobrepostos para cobrir toda a região (Xiao et al., 2003).
- Para seqüenciar o top-fio, na concepção de uma cartilha para a frente, I) escolher um G (guanina) rica região a fim de ter uma maior temperatura de tratamento térmico, sem extras nucleotídeos na primers; II) mudança C (citosina) para Y ( pirimidina) em CG e contextos CNG e alterar o restante para C T (timina). Na concepção de um primer reverso, I), escolha uma região C-ricos; II) para mudar G R (purina) em CG e contextos CNG e alterar o G restantes para A (adenina).
- Para seqüenciar o fundo vertente, na concepção de uma cartilha para a frente, I), escolha uma região C-ricos; II) a mudança para R G em CG e contextos CNG e alterar o G restantes para A. Ao projetar um primer reverso, I) escolha uma região rica em G; II) mudança C para Y (pirimidina) em CG e contextos CNG e alterar o C restantes para T.
- Nós usamos geralmente 1-2 mL do sódio DNA tratado pelo bissulfito como um modelo para cada PCR (Xiao et al., 2003). O produto da PCR deve ser analisado por eletroforese em gel para confirmar o tamanho correto do fragmento, então a ser purificado com gel e clonados no vetor de clonagem TOPO TA pCR2.1 (Invitrogen), como uma inserção. A única colônia é captado e culta; DNA plasmidial extraídos e enviados para o seqüenciamento.
4. Análise da seqüência
O princípio do seqüenciamento bissulfito é que citosina unmethylated será convertido para uracil devido à desaminação hidrolítica pela alta concentração de bissulfito de sódio a pH5.0 que será amplificada como timina no produto de PCR, enquanto 5-metil citosina não será modificado por bissulfito de sódio e continuam a ser citosina após a amplificação (Clark et al, 1994;. Frommer et al, 1992).. Depois de obter o resultado de seqüenciamento, que compará-lo com o modelo vertente específica que é usado para a amplificação PCR. Se um resíduo citosina no modelo lê como uma timina no resultado seqüenciamento, isso indica que a citosina não está metilado. Se um resíduo citosina no modelo continua a ser uma citosina no seqüenciamento, isso significa que a citosina é metilado.
IV. Resultados representante
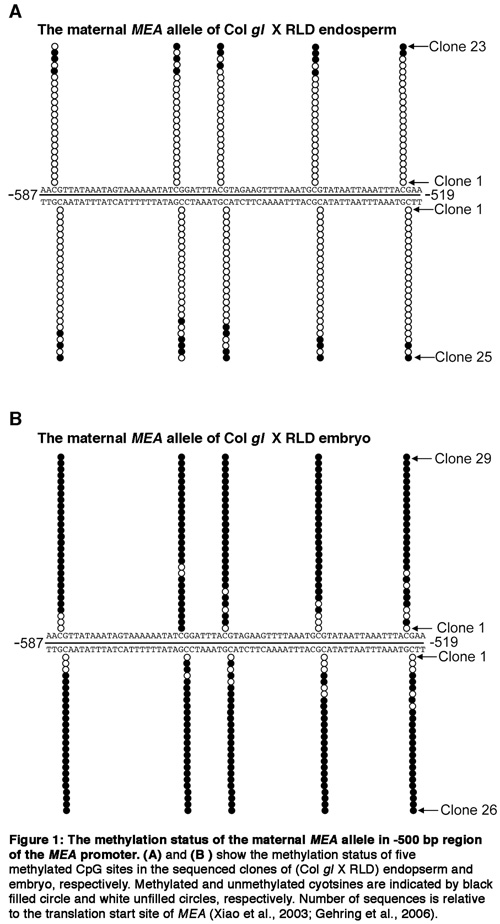 Figura 1.
Figura 1.
Discussão
É relativamente fácil separar embrião do endosperma e tegumento, mas é entediante para separar endosperma de tegumento, especialmente para sementes na fase inicial ou média torpedo da embriogênese. Desde tegumento contribui apenas com uma pequena quantidade de tecido, para alguns genes, por exemplo, MEA e FWA, não temos para separar endosperma de tegumento. Isso significa que podemos isolar RNAs de uma mistura de endosperma e tecidos tegumento, verificar a expressão dos alelos maternos e patern...
Divulgações
Agradecimentos
Os autores agradecem a Sra. Jennifer M. Lommel e Tara N. Rognan para a manutenção de plantas de Arabidopsis. Este trabalho foi financiado por fundos start-up de Saint Louis University e dos Institutos Nacionais de Saúde concede 1R15GM086846-01 e 3R15GM086846-01S1 para W. Xiao.
Materiais
Abastecimento
- Microscópio de dissecação
- Tesoura
- Pinça Ponta Fina
- Tag da jóia
- Estacas de plantas
- Barbante ou fios de arame
- 4 "X 2" X 8 "sacos de polietileno
- 3 "X 1" X 1,0 milímetros Lâminas de microscopia
- Nitrogênio líquido
- Recipientes de azoto líquido
- Bloco de calor
- PCR Tubes
- Termociclador
- Tubos de microcentrífuga
- Microcentrífuga
- Instalação de eletroforese em gel
- Arabidopsis thaliana Columbia-0 Plantas
- Arabidopsis thaliana Landsberg Plantas erecta
Reagentes
- Etanol 70%
- Etanol 95%
- O etanol 100%
- 0,3 M de sorbitol e 5 mM MES-pH 5,7
- Brometo de amônio cetiltrimetilamônio (CTAB)
- O etanol 100%
- Cholorform
- Enzimas de Restrição
- 3 M NaOH
- 6,3 M NaOH
- 6,24 M de uréia / 4 bissulfito de sódio M
- Destilada estéril H 2 O
- 10 mM hidroquinona
- Assistente de DNA Sistema Clean-Up (Promega)
- 10 M NH 4 OAc
- 20 mcg / mL tRNA
- Tampão TE
- O TOPO TA Cloning Kit (Invitrogen)
Referências
- Clark, S. J., Harrison, J., Paul, C. L., Frommer, M. High sensitivity mapping of methylated cytosines. Nucleic Acids Res. 22, 2990-2997 (1994).
- Cokus, S. J., Feng, S., Zhang, X., Chen, Z., Merriman, B., Haudenschild, C. D., Sriharsa Pradhan, S., Nelson, S. F., Pellegrini, M., Jacobsen, S. E. Shotgun bisulphite sequencing of the Arabidopsis genome reveals DNA methylation patterning. Nature. 452, 215-219 (2008).
- Frommer, M., McDonald, L. E., Millar, D. S., Collis, C. M., Watt, F., Grigg, G. W. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc Natl Acad Sci USA. 89, 1827-1831 (1992).
- Gehring, M., Huh, J. H., Hsieh, T. F., Penterman, J., Choi, Y., Harada, J. J., Goldberg, R. B., Fischer, R. L. D. E. M. E. T. E. R. DNA glycosylase establishes MEDEA polycomb gene self-imprinting by allele-specific demethylation. Cell. 124, 495-506 (2006).
- Henderson, I. R., Chan, S. R., Cao, X., Johnson, L., Jacobsen, S. E. Accurate sodium bisulfite sequencing in plants. Epigenetics. 5, 47-49 (2010).
- Hsieh, T. F., Ibarra, C. A., Silva, P., Zemach, A., Eshed-Williams, L., Fischer, R. L., Zilberman, D. Genome-wide demethylation of Arabidopsis endosperm. Science. 324, 1451-1454 (2009).
- Jacobsen, S. E., Sakai, H., Finnegan, E. J., Cao, X., Meyerowitz, E. M. Ectopic hypermethylation of flower-specific genes in Arabidopsis. Curr. Biol. 10, 179-186 (2000).
- Kinoshita, T., Miura, A., Choi, Y., Kinoshita, Y., Cao, X., Jacobsen, S. E., Fischer, R. L., Kakutani, T. One-way control of FWA imprinting in Arabidopsis endosperm by DNA methylation. Science. 303, 521-523 (2004).
- Lister, R., O'Malley, R. C., Tonti-Filippini, J., Gregory, B. D., Berry, C. C., Millar, A. H., Ecker, J. R. Highly integrated single-base resolution maps of the epigenome in Arabidopsis. Cell. 133, 523-536 (2008).
- Lister, R., Pelizzola, M., Dowen, R. H., Hawkins, R. D., Hon, G., Tonti-Filippini, J., Nery, J. R., Lee, L., Ye, Z., Ngo, Q. -. M. Human DNA methylomes at base resolution show widespread epigenomic differences. Nature. 462, 315-322 (2009).
- Paulin, R., Grigg, G. W., Davey, M. W., Piper, A. A. Urea improves efficiency of bisulfite-mediated sequencing of 5'- methylcytosine in genomic DNA. Nucl. Acids Res. 26, 5009-5010 (1998).
- Rogers, S. O., &, B. e. n. d. i. c. h., J, A. Extraction of DNA from plant tissues. Plant Molecular Biology Manual. A6, 1-10 (1988).
- Smyth, D. R., Bowman, J. L., Elliot, M., Meyerowitz, E. M. Early Flower Development in Arabídopsis. Plant Cell. 2, 755-767 (1990).
- Xiao, W., Gehring, M., Choi, Y., Margossian, L., Pu, H., Harada, J. J., Goldberg, R. B., Pennell, R. I., Fischer, R. L. Imprinting of the MEA Polycomb gene is controlled by antagonism between MET1 methyltransferase and DME glycosylase. Dev. Cell. 5, 891-901 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados