É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Produção de C. elegans Transgenes via Recombineering com o GalK Marcador selecionável
Neste Artigo
Resumo
A capacidade de produzir transgenes para Caenorhabditis elegans Usando DNA genômico realizado por fosmids é particularmente atraente como todos os elementos nativos reguladoras são retidos. Descrito é um procedimento simples e robusto para a produção de transgenes via recombineering com o GalK Marcador selecionável.
Resumo
A criação de animais transgênicos é amplamente utilizada em C. elegans pesquisa, incluindo o uso de proteínas de fusão GFP para estudar a regulação e padrão de expressão de genes de interesse ou de geração de conjunto de purificação de afinidade (TAP) com a tag versões de genes específicos para facilitar a sua purificação. Tipicamente transgenes são gerados pela colocação de um promotor a montante de um gene repórter GFP ou cDNA de interesse, e isso muitas vezes produz um padrão de expressão representativa. No entanto, elementos críticos da regulação de genes, como elementos de controle na região não traduzida 3 'ou promotores alternativos, podem ser perdidos por esta abordagem. Mais apenas uma variante única emenda pode ser geralmente estudado por este meio. Em contraste, o uso de DNA genômico verme transportado por clones DNA fosmid provavelmente inclui a maioria se não todos os elementos envolvidos na regulação de genes in vivo que permite a maior capacidade de capturar o padrão de expressão genuína e timing. Para facilitar a geração de transgenes usando DNA fosmid, descrevemos um E. coli baseado recombineering procedimento para inserir GFP, a TAP tag, ou outras seqüências de interesse em qualquer local do gene. O procedimento utiliza o gene galK como o marcador de seleção para as duas etapas de seleção positiva e negativa em recombineering o que resulta na obtenção da modificação desejada com alta eficiência. Além disso, plasmídeos contendo o gene galK ladeado por braços homologia com GFP comumente usados e TAP genes de fusão estão disponíveis que reduzem o custo de oligos em 50% ao gerar um GFP, ou proteína de fusão TAP. Estes plasmídeos use a origem de replicação R6K que impede a necessidade de purificação extensa produto da PCR. Finalmente, também demonstram uma técnica para integrar o marcador unc-119 para o backbone fosmid que permite que o fosmid a ser injetado diretamente ou bombardeados em vermes para gerar animais transgênicos. Este vídeo demonstra os procedimentos envolvidos na geração de um transgene através recombineering utilizando este método.
Protocolo
Visão global
Transgenes muitos usados na geração de transgênicos C. elegans consistem em seqüências de promotor e, talvez, um gene cDNA clonado em um dos vetores gerados pelo laboratório do Dr. Andy Fogo 1. Embora esses transgenes são frequentemente bem sucedidos no que diz respeito à elaboração de um gene repórter GFP ou expressar um cDNA em um padrão desejado, esses transgenes pode faltar os promotores alternativos, os elementos enhancer, e 3 'não traduzida região (UTR) elementos que desempenham papéis importantes no controle da expressão gênica in vivo 2. Por exemplo, tanto os genes daf-12 e fah-1 tem elementos importantes enhancer que estão fora do promotor proximal que foram perdidas na promotor só constrói 3,4,5. Ainda constrói transgene muitos usam o 3'UTR unc-54 que impede regulação pelos genes microRNA apropriado 6,7,8. Conseqüentemente, gerando transgenes com grandes segmentos de DNA genômico verme seria ideal para a captura de todos os promotores, as variantes de emenda, e 3 'UTR elementos de controle. Recentemente, um C. elegans biblioteca fosmid que consiste de ~ 40 kb regiões do DNA genômico e cobre quase todo o genoma foi construído. O uso do DNA genômico verme realizado por estes clones resultados DNA fosmid na maior capacidade de capturar o padrão de expressão genuína e tempo de genes específicos 2,8,9,10,11.
No entanto, trabalhar com grandes regiões de DNA genômico coloca desafios práticos, como as grandes dificuldades na utilização de técnicas de biologia molecular padrão 12. Para superar essas limitações, técnicas para modificar fosmids ou bacteriana cromossomos artificiais através de recombinação homóloga em E. coli foram desenvolvidos e são denominadas recombineering 12,13. Recombineering permite a inserção contínua de GFP, uma purificação de afinidade tandem (TAP), tag, ou outras seqüências de interesse em qualquer local do gene transportado pelo C. elegans fosmid clone 2,10,14. Recombinação homóloga ocorre entre um produto PCR ladeado por 50 regiões bp de homologia com o site de destino eo DNA alvo em E. especialmente modificado coli.
Descrevemos recentemente um processo de dois estágios para a modificação de C. fosmids elegans por recombineering que envolve a inserção do gene galK no local desejado e depois substituir esse gene com a sequência desejada 2. O gene galK serve como um marcador de seleção eficaz para ambas as etapas do processo, pois ele pode ser selecionado a favor e contra, através da utilização de meio de crescimento seletivo 15. Na primeira etapa da modificação fosmid, o gene é inserido galK via recombinação homóloga no local desejado, e os fosmids corretamente modificados, identificados pela seleção positiva para a capacidade de utilizar galactose como fonte de carbono 2,15. Na segunda etapa, o gene galK é substituído pela seqüência desejada, eo fosmids corretamente modificados são identificados através de seleção negativa contra o gene galK através da utilização do deoxygalactose galactose tóxicos derivados que mata as bactérias galK + 2,15. Uma vantagem do galK é a capacidade de um único gene a ser utilizado para as etapas de seleção positiva e negativa, em vez de outros marcadores que têm genes separados para cada etapa, e os resultados na obtenção da modificação desejada com alta eficiência 2,15.
Para facilitar a aplicação desta técnica para C. elegans pesquisa, fizemos várias alterações para os recursos disponíveis. Primeiro, as tags GFP e TAP são comumente usados para gerar transgenes worm, por isso construído em 50 regiões bp de homologia com cada uma dessas tags no pMOD4 galK-G e pMOD4 galK GT-plasmídeos, que servem como fonte do gene galK 2. Estas regiões permitir um único conjunto de oligos a ser utilizado para ambas as fases da modificação fosmid que poupa a necessidade de encomendar um segundo conjunto de oligos um pouco caro. Segundo, estes plasmídeos use a origem de replicação R6K que impede a necessidade de digerir o pai de purificação do produto plasmídeo ou PCR extensa como o pai plasmídeo não é capaz de se replicar em bactérias utilizadas para recombineering, e só podem se replicar em cepas especiais, tais como EC100 2 , 16 (Tabela 1 e Tabela 2). Finalmente, uma forma comum de geração de transgênicos C. elegans é através do uso de bombardeios biobalística seguido pela seleção de vermes transgênicos através do resgate da mutação unc-119 17. Para fazer o fosmids compatível com o bombardeio, que desenvolveu o pLoxP unc-119 plasmídeo que pode ser usado para integrar o marcador unc-119 para o backbone fosmid 2.
I. Oligo design
Recombineering com as seqüências desejado pode ser inserido em qualquer local dentro do gene. Sítios mais comuns são na extremidade 5 'ou 3' end dependendo domínios funcionais, splice variantes, ou modificações pós-translacionais, como a clivagem por proteases. A GFP pMOD4 plasmídeo criado por nosso laboratório pode ser usado para inserir uma tag FLAG-GFP em qualquer site como o plasmídeo inclui um códon iniciador e carece de um códon 3 'stop (Figura 1). Em contraste, a marca TAP tem versões específicas para 5 'e 3' fusões, devido à clivagem TEV usados durante a depuração 18,19.
- Planejar o local de inserção tag dentro do gene. Considere promotores alternativos, domínios funcionais, a alternativa de corte, e modificações pós-translacionais quando se considera o local de inserção. Diferentes locais de inserção poderia ser usada para marcar todas, uma ou algumas isoformas de um gene específico. Identificar 50 regiões pb a montante ea jusante do ponto de inserção.
- Projetar o oligos (Tabela 1). Ou escala nM 100 - oligos gel purificada ou oligos Ultramer de Tecnologias Integradas DNA pode ser usado para o procedimento.
Para executar recombineering galK, você precisa para projetar primers galK com 50 bp de homologia para uma área de acompanhamento do site desejado para ser modificado e 3 'final destes primers se ligam ao cassete galK, que está presente em ambos os pMOD4 galK-G e pMOD4 galK-GT (Figura 1). O oligo frente será 5'------- ------- 50 bp de homologia CCTGTTGACAATTAATCATCGGCA-3 'e uma reversa como 5'------- 50 bp de homologia na cadeia complementar - ------ TCAGCACTGTCCTGCTCCT-3 '.
Para executar recombineering galK com GFP, você precisa de design pMOD4 galK-G ou pMOD4 galK-GT primers com 50 bp de homologia para uma área de acompanhamento do site desejado a ser modificada (Figura 1). Certifique-se de manter a proteína de fusão, em frame. O ATG pode ser descartada, se desejar. Extremidade 3 'dos primers se ligam a regiões de homologia GFP acompanhamento a cassete galK. Nota: Os codons e sobrenome de GFP são sublinhados para demonstrar o quadro de leitura. O oligo frente será 5'------- ------- 50 bp de homologia ATG GATTACAAGGACGATGACGATAAGATGAG -3 '3' e inverter a um 5'------- 50 bp de homologia na complementares vertente ------- AGCTTGTGGGCTTTTGTATAG CAA-3 '
Para executar galK recombineering TAP C prazo, você precisa de design pMOD4 galK-GT primers com 50 bp de homologia para uma área de acompanhamento do site desejado a ser modificada (Figura 1). Certifique-se de manter a proteína de fusão, em frame. Extremidade 3 'dos primers se ligam a regiões de homologia TAP acompanhamento a cassete galK. Nota: Os códons primeiro e último da TAP estão sublinhados para demonstrar o quadro de leitura. O oligo frente será 5'------- ------ 50 bp de homologia ATG GAAAAGAGAAGATGGAAAAAG - -3 'e vice-versa um 5'------- 50 bp de homologia na cadeia complementar - ------ GGT -3 TGACTTCCCCGC '
Para executar galK recombineering TAP N-termo, você precisa de design pMOD4 galK-GT primers com 50 bp de homologia para uma área de acompanhamento do site desejado a ser modificada (Figura 1). Certifique-se de manter a proteína de fusão, em frame. Extremidade 3 'dos primers se ligam a regiões de homologia TAP acompanhamento a cassete galK. Nota: Os códons primeiro e último da TAP estão sublinhados para demonstrar o quadro de leitura. O oligo frente será 5'------- ------ 50 bp de homologia ATG GCAGGCCTTGCGC - -3 'e vice-versa um 5'------- 50 bp de homologia na cadeia complementar - ------ AAG -3 TGCCCCGGAGGATGAGATTTTCT ' - Gerar um conjunto de acompanhamento oligos para PCR em etapas posteriores, bem como para o seqüenciamento do fosmid. Estes são oligos PCR padrão que deve ligar ~ 100 pb a montante ea jusante do local de inserção.
II. Transferência para Fosmid SW016 Bactérias
O fosmids da C. elegans fosmid biblioteca são fornecidos na cepa EPI300 bacteriana (F-mcrA Δ (MRR-hsdRMS-mcrBC) φ80dlacZΔM15 ΔlacX74 recA1 endA1 araD139 Δ (ara, leu) 7697 galu galK λ-RPSL nupG trfA tona) (Biotecnologias Epicentro, Madison, WI ), que permite a expressão fosmid ser aumentado acima de uma única cópia por célula para melhorar o rendimento de DNA durante a depuração (Tabela 2). Para recombineering, o fosmid terá de ser transferido para a estirpe bacteriana SW106 (mcrA Δ (MRR-hsdRMS-mcrBC) ΔlacX74 Deor endA1 araD139 Δ (ara, leu) 7697 RPSL recA1 nupG φ80dlacZΔM15 [λc1857 (cro-bioA) <> Tet ] (cro-bioA) <> ARAC-PBAD Cre ΔgalK) estirpe (NCI-Frederick), que carrega a recombinação homóloga λred genes sob o controle de um λ sensível à temperatura e um repressor induzível arabinose cre recombinase (Tabela 2) 15.
- Ordem do clone fosmid de Gene de interesse (GOI) de GeneService (Cambridge, UK), utilizando Wormbase como um guia. Ao selecionar clones, nós escolhemos aqueles que têm o Governo indiano, no centro da seqüência. Aqueles que exclui genes vizinhos pode ser preferível, mas poderia ser difícil de encontrar.
- Cultura clonar o fosmid do Governo da Índia em LB contendo 12,5 mg / mL cloranfenicol a 37 ° C.
- Crescer uma cultura 1,5 mL da noite para o dia fosmid, e mini-prep DNA do fosmid usando o Epicentro fosmid prep kit (Biotecnologias Epicentro, Madison, WI). Nós seguimos o protocolo alternativo descrito nas instruções que envolve a adição da mistura Riboshredder em uma etapa anterior.
- Determinar a concentração de DNA fosmid com um espectrofotômetro.
- Prepare o electrocompetent SW106 células crescendo uma cultura de 5 ml da noite para o dia SW106 em um tubo de 14 mL pressão cap-com caldo LB com 12,5 cloranfenicol mcg / mL a 32 ° C.
- Inocular 1 mL em 100 mL de LB com cloranfenicol em um frasco de L 2. SW106 crescer bactérias para uma 600 OD 0,6-0,8. NÃO choque térmico.
- Pellet por centrifugação a 5000xg por 5 minutos, ressuspender o sedimento em vortex gentil, e adicionar 50 mL gelada glicerol 10%. Repita esta etapa de lavagem uma vez.
- Pellet SW106 o por centrifugação e aspirar todos, mas ~ 500 mL de cada sobrenadante
- Volte a suspender as pelotas em vortex gentil. 100 mL congelar alíquotas em nitrogênio líquido ou em gelo seco, e armazenar a -80 ° C para uso futuro.
- Transformar o DNA fosmid em electrocompetent SW106 células electroporating as bactérias com ~ 50 de ng DNA fosmid usando um Eppendorf 2510 a 1350 volts electroporator em 0,1 cubetas gap cm.
- Recuperar bactérias em 1 LB mL por 1 hora a 32 ° C.
- Alíquotas placa em placas de LB com cloranfenicol (12,5 mg / mL) e incubar a 32 ° C durante a noite.
- Verificar a presença do Governo da Índia por colônia PCR. Crescer uma cultura 5 mL durante a noite em LB com 12,5 mg / mL de cloranfenicol em 32 ° C. Adicionar 0,5 mL da cultura para uma reação de PCR com os oligos padrão de acompanhamento, e aumentar a inicial 95 ° C de incubação de cinco minutos para lisar as bactérias antes da PCR.
- Prepare um estoque de glicerol para armazenamento de longo prazo.
III. Inserção de Gene galK por Recombineering
Na primeira etapa da modificação fosmid, o gene galK é inserido no fosmid por recombinação homóloga, e os fosmids corretamente modificados são selecionados para pelo crescimento em meio mínimo contendo galactose como única fonte de carbono (Figura 2A). O SW106 bactérias crescem lentamente na mídia mínimo e 3-5 dias são necessários para ver colônias.
- Prepare MOPS placas mínima mídia contendo galactose 0,2%. MOPS media mínima está disponível a partir Teknova Inc. (Hollister, CA) (catálogo # M2106), mas não usar a glicose incluídos.
MOPS media mínimo com galactose 0,2% (1 L)
Autoclave 15 gramas de ágar em 870 mL de água
Esfriar a 55 ° C e adicionar:100 mL MOPS 10X media mínima 5 mL 0,2 mg / ml d-biotina (estéril filtrado) 4,5 mL 10 mg / ml L-leucina (1%, aquecido e, em seguida arrefecido e filtrado estéril) 10 mL Galactose 20% (autoclavado) 1 mL 12,5 mg / ml de cloranfenicol em EtOH 2,55 mL 20% NH 4 Cl 10 mL 0,132 M de fosfato de potássio dibásico - PCR amplificar o pMOD4 galK-G ou pMOD4 galK-GT cassetes utilizando os primers desenhado acima. Temos usado Phusion (New England Biolabs, Ipswich, MA) ou GoTaq (Promega, Madison, WI).
- Gel purificar a banda resultante. Quantificar o rendimento de gel ou espectrofotômetro Nanodrop. Este produto PCR está pronto para o passo 3.14.
- Inocular uma cultura overnight de SW106 células contendo o DNA fosmid em 5 ml LB com cloranfenicol (12,5 mg / mL). Crescer a 32 ° C.
- Definir tremendo banho de água a 42 ° C a warm-up com um frasco estéril 250 ml no suporte. Uso de um tremendo banho de água é crítica para a obtenção de alta eficiência.
- Adicionar 1 ml da cultura durante a noite para 100 ml de LB e cloranfenicol em um frasco L 2. Crescer a um OD 0,6-0,8. Isso normalmente leva 3-4 horas.
- Transferir 50 ml de células SW106 para o balão de 250 ml e de choque térmico a 42 ° C por exatamente 20 min. agitação em banho-maria a 100 rpm Deixe as bactérias remanescentes em 32 ° C como o controle uninduced.
- Arrefecer as bactérias induzido e uninduced no gelo por 10 minutos.
- Transferir as amostras para dois tubos de centrífuga estéreis e pellet em ~ 5000xg por 5 minutos.
- Deitar fora todo o sobrenadante e ressuspender o sedimento em 1 ml de glicerol gelada 10% em vortex suave (ie configuração 3-4).
- Quando ressuspenso, adicionar outro 49 ml gelada glicerol 10%, e as amostras de sedimento em ~ 5000xg por 5 minutos.
- Repita o passo 3.9, 3.10 e 3.11 novamente.
- Remova todas as sobrenadante invertendo os tubos, e ressuspender o sedimento em que o líquido restante (cerca de 500 mL cada). Alíquota em 100 amostras mL, congelar em gelo seco, e armazenar a -80 ° C. Estes são bons por semanas a meses. (Costumamos parar por aqui e realizar o eletroporação no dia seguinte).
- Electroporate o induzido e uninduced SW106 células com 150 ng de produto de PCR usando cubetas 0,1 cm de distância um conjunto electroporator Eppendorf 2510 a 1350 volts.
- Recuperar as bactérias em um LB mL em um tubo Falcon 14 ml. Incubar a 32 ° C por 4,5 horas.
- Pellet as bactérias em uma microcentrífuga a 13.200 rpm por 15 segundos. As bactérias são ressuspenso em M9 e, em seguida, lavados duas vezes remover qualquer meio rico (veja abaixo a receita).
- M9 médio (1 L)
- 6g Na 2 HPO 4
- 3g KH 2 PO 4
- 1g NH 4 Cl
- 0,5 g NaCl
- AUTOCLAVE
- Depois da segunda lavagem, o sobrenadante é removido eo pellet é ressuspenso em 1 mL M9 antes do plaqueamento de diluições de série em M9 (100 mL, 100 mL de uma diluição de 1:10 e 1:100 100 L) em MOPS media mínima.
- Incubar 3-5 dias a 32 ° C em uma incubadora. Nota: Seja paciente como verdadeiros positivos crescem lentamente.
- Streak algumas colônias em placas de agar MacConkey indicador (BD # 281810) suplementado com galactose 1% e cloranfenicol 12,5 mcg / mL. Todas as colônias que aparecem após o último passo deve ser + galK, mas para se livrar de qualquer galK - contaminantes, é importante para obter única, brilhante colônias rosa antes de prosseguir para a segunda etapa. O galK - colônias será branca / incolor e as bactérias + galK será brilhante vermelho / rosa devido a uma alteração do pH resultante da galactose fermentados após uma incubação overnight a 32 ° C.
- Escolha uma única colônia e inocular uma LB de 5 ml + cultura overnight cloranfenicol para o crescimento a 32 ° C.
- Confirmar a inserção do gene galK no local apropriado via PCR utilizando o acompanhamento oligos. Adicionar 0,5 mL da cultura para uma reação de PCR padrão e aumentar a inicial 95 ° C de incubação de cinco minutos para lisar as bactérias. O produto da PCR deve ser upshifted em tamanho, devido à presença do gene galK.
- Prepare um estoque de glicerol para armazenamento.
IV. Substituição de galK com seqüências Tag por Recombineering
Nesta fase, o gene galK é substituído pelo seqüências tag desejada eo fosmids corretamente modificados são selecionados por seleção contra o gene galK pela tóxicos galactose analógico deoxygalactose (DOG) (Figura 2B).
- Prepare MOPS placas mínima mídia contendo deoxygalactose 0,2% (DOG) e glicerol 0,2%. MOPS media mínima está disponível a partir Teknova Inc. (Hollister, CA) (catálogo # M2106), mas não usar a glicose incluídos.
MOPS media mínimo com DOG 0,2% e glicerol (1 L)
Autoclave 15 gramas de ágar em 860 mL de água
Esfriar a 55 ° C e adicionar:100 mL MOPS 10X media mínima 5 mL 0,2 mg / ml d-biotina (estéril filtrado) 4,5 mL 10 mg / ml L-leucina (1%, aquecido e, em seguida arrefecido e filtrado estéril) 10 mL Deoxygalactose 20% (estéril filtrado) 10 mL Glicerol 20% (autoclavado) 1 mL 12,5 mg / ml de cloranfenicol em EtOH 2,55 mL 20% NH 4 Cl 10 mL 0,132 M de fosfato de potássio dibásico - PCR amplificar os fragmentos de tag pMOD4 GFP, pBS1761 (N-termo TAP), ou pBS1479 (C prazo TAP), utilizando o oligos mesmo utilizado no primeiro turno ou usando mais curto ou GFP TAP oligos específicos (as seqüências internas no passo 1,2 ). Se você está fazendo construções múltiplas, é particularmente útil para usar o oligos mais curto como o produto de PCR mesmo pode ser usado para todas as construções.
- Gel purificar o produto de PCR e medir a concentração através de gel ou espectrofotometria.
- Gerar induzido e uninduced SW106 competente que o fosmid com o gene inserido galK seguintes passos 3,4-3,13 acima.
- Electroporate o induzido e uninduced SW106 células com ~ 100 ng de produto de PCR usando cubetas 0,1 cm de distância um conjunto electroporator Eppendorf 2510 a 1350 volts.
- Recuperar em um LB ml em um tubo de 14 ml pressão cap-and incubar em um shaker 32 ° C por 4,5 horas.
- Lavar e diluir as bactérias como nos passos 3.16 e 3.17. Bactérias na placa MOPS placas mínima meios contendo 0,2% 2-desoxi-galactose (DOG) e glicerol 0,2%.
- Incubar a 32 ° C por três dias.
- Quatro colônias são usados para fazer 5 mLculturas durante a noite em LB com 12,5 mcg / mL cloranfenicol. Estes são utilizados para a colônia PCR como descrito acima para confirmar que a cassete foi inserido. Nós usamos as GFP a curto / TAP oligos específicos e os oligos acompanhamento para demonstrar a inserir à direita e local certo. GFP é de aproximadamente 800 pb e TAP é de aproximadamente 550 pb ao passo que galK é de 1,4 kb.
- Prepare um estoque de glicerol.
V. A adição de unc-119 com a criação de Gene loxP Recombinação
Um meio comum de geração de animais transgênicos com o fosmids modificada é através do uso de bombardeio biobalística. Esta técnica utiliza partículas de DNA revestido de ouro para introduzir DNA fosmid em C. elegans. Animais transgênicos são normalmente identificados por meio de resgate do mutante unc-119 com um transgene unc-119. Nesta etapa, o gene unc-119 é adicionado para a espinha dorsal fosmid em cis com a criação de loxP recombinação com o pLoxP unc-119 plasmídeo (Figura 2C).
- Prepare competente SW106 bactérias carregando o fosmid modificado a partir do passo 4.9 usando as etapas 2,5-2,9. NÃO induzir a 42 ° C.
- Electroporate com 50 ng. pLoxP unc-119 a partir de um mini-prep usando cubetas gap 0,1 cm em um Eppendorf electroporator 2510 fixado em 1,350 volts.
- Recuperar bactérias em LB contendo arabinose de 0,1% para 1 hora a 32 ° C.
- Alíquotas placa em placas LB contendo 50μg/mL ampicilina e 12,5 mg / mL cloranfenicol. Incubar a 32 ° C durante a noite. que seleciona para a integração de pLoxP unc-119 no fosmid.
- Crescer uma cultura durante a noite em LB contendo 50μg/mL ampicilina e 12,5 mg / mL cloranfenicol. Use 0,5 mL para PCR verificar a presença do gene unc-119 com a unc-119 F (5'-CAAATCCGTGACCTCGACAC-3 ') e unc-119 R (5'-CACAGTTGTTTCTCGAATTTGG-3') oligos (Tabela 1).
- Faça um estoque de glicerol do fosmid final.
VI. Preparação Fosmid Large Scale
Para facilitar a obtenção da maior quantidade de DNA necessária para fosmid bombardeio, nesta etapa o fosmid é transferido para o EPI300 bactérias. Esta linhagem tem a capacidade de aumentar o número de cópias fosmid para aumentar a produtividade durante a preparação do DNA.
- Crescer uma cultura 5 mL noite para o dia das bactérias a partir do passo 5.5 em LB contendo ampicilina e cloranfenicol em 32 ° C. Use Epicentre fosmid prep kit para isolar o fosmid de 1,5 mL da cultura.
- Electroporate ~ 50 ng no EPI300 bactérias usando cubetas 0,1 cm de distância um conjunto electroporator Eppendorf 2510 a 1350 volts. O EPI300 bactérias pode ser comprado de Biotecnologias Epicentre (Madison, WI).
- Recuperar bactérias no LB por 1 hora a 37 ° C. Alíquotas placa em ágar LB contendo 50 mg / mL e 12,5 mcg / mL cloranfenicol.
- Crescer e induzir o EPI300 bactérias contendo o fosmid modificados usando as instruções incluídas. A 50 mL da cultura induzida dará> mcg 10 de purificados DNA fosmid. Purificar o fosmid com o fosmid Epicentre prep kit.
VII. Bombardeamento
- Use 10 mg. de DNA fosmid para bombardear a tensão verme DP38 como descrito (D. Hochbaum, A. Ferguson, e A. Fisher, Jove, no prelo).
VIII. Resultados representante
A modificação do fosmids via recombineering robusto e taxas de sucesso de> 90% na etapa de seleção negativa são rotineiramente observadas 2. Este protocolo também leva ~ 2 semanas para concluir o que torna a preparação de transgenes bastante rápida. O protocolo também foi tentada por outros laboratórios com sucesso 20.
| Oligo | Seqüência |
| C F prazo TAP | ATGGAAAAGAGAAGATGGAAAAAG |
| C-TAP R prazo | GGTTGACTTCCCCGC |
| BANDEIRA-GFP F | ATGGATTACAAGGACGATGACGATAAGATGAG |
| BANDEIRA-GFP R | CAAAGCTTGTGGGCTTTTGTATAG |
| N prazo F TAP | ATGGCAGGCCTTGCGC |
| N prazo TAP R | AAGTGCCCCGGAGGATGAGATTTTCT |
| galK F | CCTGTTGACAATTAATCATCGGCA |
| R galK | TCAGCACTGTCCTGCTCCT |
| unc-119 F | CAAATCCGTGACCTCGACAC |
| unc-119 R | CACAGTTGTTTCTCGAATTTGG |
Tabela 1. Oligonucleotídeos utilizados para PCR.
| Plasmídeos | Fonte | Disponível em |
| Fosmid clone | Geneservice Ltd. | Geneservice |
| pGalK | 15 | NCI-Frederick |
| pMOD4-RT-G | 2 | Addgene |
| pMOD4-galK-G | ||
| pMOD4-galK-GT | ||
| pLoxP-unc-119 | ||
| pMOD4-GFP | ||
| Bactérias | ||
| SW106 | 15 | NCI-Frederick |
| EPI300 | Biotecnologias Epicentre | Epicentro |
| EC100D pir-116 | ||
Tabela 2. Strain e disponibilidade vetor.
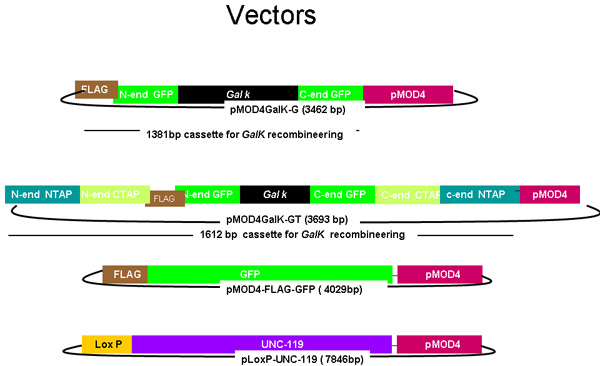
Figura 1.
Diagrama de pMOD4-galK-G, e pMOD4-galK-GT, pMOD4 GFP, pLoxP-unc-119
O plasmídeo pMOD4-galk-G consiste na cassete galK (preto) ladeado por 50 regiões de nucleotídeos idênticos aos 5 'e 3' termina de FLAG (Brown)-GFP (verde), enquanto PMOD 4 galK-GT consiste na galK cassete ladeado por ambas as regiões BANDEIRA GFP-homologia e 50 regiões de nucleotídeos idênticos aos 5 'e 3' extremidades N-terminal e C-terminal TAP (azul e laranja, respectivamente). pMOD4-FLAG-GFP consiste na cassete GFP completa com 5 'tag FLAG e pLoxP unc-119 consiste na unc-119 seqüência genômica (roxo) em um plasmídeo contendo um site loxP. Todos os plasmídeos utilizar o R6K baseado pMOD4 backbone (vermelho), que é incapaz de se replicar em SW106.
Figura 2.
Visão geral do processo galK recombineering
Figura 2A-2C valores distintos mostrando as etapas eo tempo envolvidos na recombineering utilizar a cassete galK. Estes são os mesmos valores que são fundidos na figura 2d, mas fornecidos separadamente para maior clareza e facilidade de leitura. A fosmid de interesse é o primeiro modificado em um procedimento em duas etapas que envolvem a inserção do cassete galK ladeado por 50 regiões bp de homologia com FLAG-GFP ou TAP (Figura 2A), seguido por substituição deste por cassete BANDEIRA GFP ou TAP ( Figura 2B). Mais tarde o marcador unc-119 para uso na geração de animais transgênicos é inserido no site LoxP no backbone fosmid (Figura 2C).
Figura 2D mostra uma figura mesclada de galK procedimento recombineering.
Esboço de galK recombineering procedimento como descrito acima no tempo, incluindo necessários para cada etapa da fusão Figura 2A-2C.
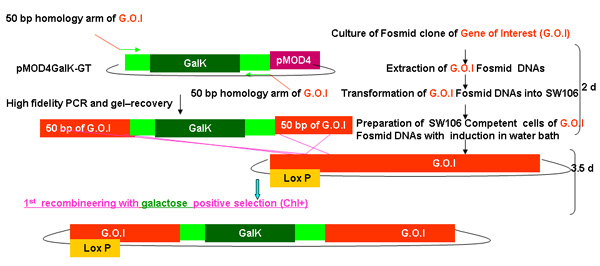
Figura 2a. A inserção galk em recombineering galk.
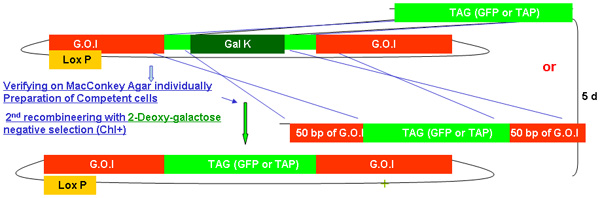
Figura 2b. O TAG de inserção (GFP / TAP) em recombineering galk.
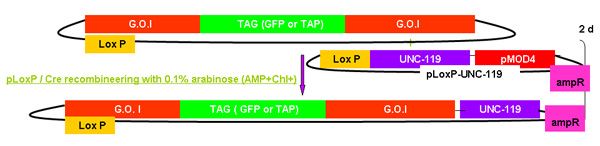
Figura 2c. A adição de unc-119.
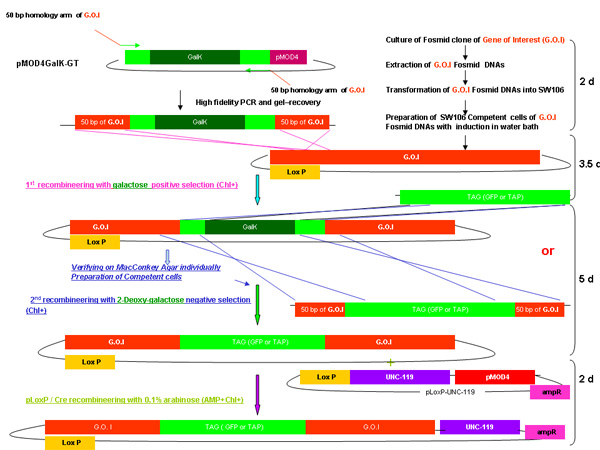
Figura 2d. A visão mesclada de recombineering galk.
Discussão
A geração de transgenes de fosmids oferece o benefício de manter todos os elementos promotor nativo, splice variantes, e três elementos 'UTR de regulamentação. Isso pode levar à construção de um transgene que é mais um reflexo do padrão de expressão nativa, ou a construção de um transgene funcional quando outras abordagens não 5. Os transgenes resultante pode carregar uma variedade de etiquetas de epítopo incluindo GFP ou um tag TAP.
A construção de transgene...
Divulgações
Agradecimentos
Os autores gostariam de agradecer a Lindsey Nash para ajudar a desenvolver a técnica. Este trabalho foi financiado pelo NIH conceder AG028977 a ALF, uma bolsa de projeto piloto da Universidade de Pittsburgh OAIC (AG024827), e fundos de sementes da Universidade de Pittsburgh.
Materiais
| Name | Company | Catalog Number | Comments |
| FosmidMAX kit | Epicentre Biotechnologies | FMAX046 | |
| GoTaq | Promega Corp. | M7122 | |
| MOPS Media | TEKnova, Inc. | M2120 | |
| 0.132 M Potassium phosphate solution | TEKnova, Inc. | M2102 | |
| D-galactose | Sigma-Aldrich | G0750 | |
| 2-deoxygalactose | Sigma-Aldrich | D4407 | |
| Biotin | Sigma-Aldrich | B4639 | |
| Leucine | Sigma-Aldrich | L8000 | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Phusion DNA polymerase | New England Biolabs | F-530S | |
| MacConkey agar base | BD Biosciences | 281810 | |
| Arabinose | Sigma-Aldrich | A3131 | |
| Chloramphenicol | Sigma-Aldrich | C1919 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S5136 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Sodium chloride | Sigma-Aldrich | S5886 | |
| Glycerol | Sigma-Aldrich | G2025 | |
| Bacto Agar | BD Biosciences | 214010 |
Referências
- Mello, C., Fire, A. DNA transformation. Methods Cell Biol. 48, 451-482 (1995).
- Zhang, Y., Nash, L., Fisher, A. L. A simplified, robust, and streamlined procedure for the production of C. elegans transgenes via recombineering. BMC Dev Biol. 8, 119-119 (2008).
- Antebi, A., Yeh, W. H., Tait, D., Hedgecock, E. M., Riddle, D. L. daf-12 encodes a nuclear receptor that regulates the dauer diapause and developmental age in C. elegans. Genes and Development. 14, 1512-1527 (2000).
- Snow, M. I., Larsen, P. L. Structure and expression of daf-12: a nuclear hormone receptor with three isoforms that are involved in development and aging in Caenorhabditis elegans. Biochim. Biophys. Acta. 1494, 104-116 (2000).
- Fisher, A. L., Page, K. E., Lithgow, G. J., Nash, L. The Caenorhabditis elegans K10C2.4 Gene Encodes a Member of the Fumarylacetoacetate Hydrolase Family. A CAENORHABDITIS ELEGANS MODEL OF TYPE I TYROSINEMIA. J Biol.Chem. 283, 9127-9135 (2008).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75, 855-862 (1993).
- Lehrbach, N. J. LIN-28 and the poly(U) polymerase PUP-2 regulate let-7 microRNA processing in Caenorhabditis elegans. Nat Struct Mol Biol. 16, 1016-1020 (2009).
- Tursun, B., Cochella, L., Carrera, I., Hobert, O. A toolkit and robust pipeline for the generation of fosmid-based reporter genes in C. elegans. PLoS One. 4, e4625-e4625 (2009).
- Bamps, S., Hope, I. A. Large-scale gene expression pattern analysis, in situ, in Caenorhabditis elegans. Brief. Funct. Genomic. Proteomic. , (2008).
- Dolphin, C. T., Hope, I. A. Caenorhabditis elegans reporter fusion genes generated by seamless modification of large genomic DNA clones. Nucleic Acids Res. 34, e72-e72 (2006).
- Sarov, M. A recombineering pipeline for functional genomics applied to Caenorhabditis elegans. Nat. Methods. 3, 839-844 (2006).
- Yang, X. W., Model, P., Heintz, N. Homologous recombination based modification in Escherichia coli and germline transmission in transgenic mice of a bacterial artificial chromosome. Nat Biotechnol. 15, 859-865 (1997).
- Court, D. L., Sawitzke, J. A., Thomason, L. C. Genetic engineering using homologous recombination. Annu.Rev.Genet. 36, 361-388 (2002).
- Westenberg, M., Bamps, S., Soedling, H., Hope, I. A., Dolphin, C. T. Escherichia coli MW005: lambda Red-mediated recombineering and copy-number induction of oriV-equipped constructs in a single host. BMC Biotechnol. 10, 27-27 (2010).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36-e36 (2005).
- Penfold, R. J., Pemberton, J. M. An improved suicide vector for construction of chromosomal insertion mutations in bacteria. Gene. 118, 145-146 (1992).
- Praitis, V., Casey, E., Collar, D., Austin, J. Creation of low-copy integrated transgenic lines in Caenorhabditis elegans. Genetics. 157, 1217-1226 (2001).
- Puig, O. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat.Biotechnol. 17, 1030-1032 (1999).
- Achilleos, A., Wehman, A. M., Nance, J. PAR-3 mediates the initial clustering and apical localization of junction and polarity proteins during C. elegans intestinal epithelial cell polarization. Development. 137, 1833-1842 (2010).
- Maduro, M., Pilgrim, D. Identification and cloning of unc-119, a gene expressed in the Caenorhabditis elegans nervous system. Genetics. 141, 977-988 (1995).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados