É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Uma ferramenta analítica-box para avaliação bioquímica, estrutural e Transcriptoma abrangente de Biofilmes Oral Mediada por estreptococos do grupo mutans
Neste Artigo
Resumo
Biofilmes formados nas superfícies dos dentes são altamente complexos e expostos a constante inata e exógenos desafios ambientais, que modulam sua arquitetura, fisiologia e transcriptoma. Nós desenvolvemos uma caixa de ferramentas para examinar a composição, organização estrutural e expressão gênica de biofilmes orais, que pode ser adaptado para outras áreas de pesquisa biofilme.
Resumo
Biofilmes são altamente dinâmicas, comunidades organizadas e estruturadas de células microbianas enredados em uma matriz extracelular de densidade variável e composição 1, 2. Em geral, os biofilmes se desenvolvem a partir de fixação microbiana inicial em uma superfície seguido pela formação de grupos de células (ou microcolônias) e um maior desenvolvimento e estabilização do microcolônias, que ocorrem em uma matriz complexa extracelular. A maioria dos exopolissacarídeos biofilme matrizes porto (EPS), e biofilmes dentários não são excepção, especialmente aqueles associados à doença cárie, que são principalmente mediadas por estreptococos mutans 3. O EPS são sintetizados por microrganismos (S. mutans, um dos principais contribuintes), por meio de enzimas extracelulares, tais como glucosyltransferases usando principalmente sacarose como substrato 3.
Estudos de biofilmes formados nas superfícies dos dentes são particularmente desafiadora devido à sua exposição constante aos desafios ambientais associados a complexos dieta-host-microbial interações que ocorrem na cavidade oral. Melhor compreensão da dinâmica de mudança da organização estrutural e composição da matriz, fisiologia e transcriptoma / profile proteoma de biofilme células em resposta a essas interações complexas seria avançar o conhecimento atual de como biofilmes orais modular a patogenicidade. Por isso, temos desenvolvido uma caixa de ferramentas analíticas para facilitar a análise do biofilme em níveis estruturais, bioquímicos e moleculares, combinando técnicas comumente disponíveis e romance com custom-made software para análise de dados. Padrão de análise (ensaios colorimétricos, RT-qPCR e microarrays) e novas técnicas de fluorescência (para a rotulagem simultânea de bactérias e EPS) foram integrados com software específico para análise de dados para resolver a complexa natureza da pesquisa biofilme oral.
A caixa de ferramentas é composto por 4 etapas distintas, mas interligadas (Figura 1): 1) Os bioensaios, 2) entrada de dados brutos, 3) Processamento de Dados, e 4) Análise de Dados. Nós usamos o nosso modelo in vitro de biofilme e específicas condições experimentais para demonstrar a utilidade ea flexibilidade da ferramenta-box. O modelo de biofilme é simples, reprodutíveis e várias réplicas de um único experimento pode ser feito simultaneamente 4, 5. Além disso, permite a avaliação temporal, inclusão de diversas espécies microbianas 5 e avaliação dos efeitos de diferentes condições experimentais (por exemplo, tratamentos 6; comparação de mutantes knockout vs cepa parental 5; carboidratos disponibilidade 7). Aqui, descrevemos dois componentes específicos da caixa de ferramentas, incluindo (i) novo software para microarray de mineração de dados / organização (MDV) e análise de imagens de fluorescência (DUOSTAT), e (ii) in situ EPS rotulagem. Nós também fornecemos um caso experimental mostrando como a caixa de ferramentas pode ajudar com biofilmes análise, os dados da organização, integração e interpretação.
Protocolo
1. PASSO 1 - bioensaios
O método de biofilme usa discos de hidroxiapatita (HA) como substituto do dente (Clarkson Cromatografia Products, Inc., South Williamsport, PA, EUA; superfície = 2,7 ± 0,2 centímetros 2) revestido com saliva (imitando a presença de película adquirida), colocado na posição vertical, 4, 5, 8.
- Ensaios bioquímicos.
- Os biofilmes são (i) homogeneizadas por sonicação 9 ou (ii) mantidos intactos para os ensaios bioquímicos 8. A suspensão homogeneizada biofilmes pode ser utilizado para determinação da biomassa (peso seco), proteína total, inorgânico e extracelular e intracelular polissacarídeos composição / conteúdo (ver detalhes em Lemos et al. Protocolos para estudar a fisiologia de biofilmes Oral 8). Os biofilmes intacta pode ser analisado para as suas respostas fisiológicas utilizando padrão pH glicolítico-gota, o ácido matá-permeabilidade de prótons e ensaios F-ATPase de atividade (ver detalhes em Lemos et al. Protocolos para estudar a fisiologia de biofilmes Oral 8).
- As análises de transcriptoma.
- Padrão de cDNA microarray e RT-qPCR protocolos (http://pfgrc.jcvi.org/index.php/microarray/protocols.html por exemplo) pode ser usado em combinação para a determinação do transcriptoma-resposta de patógenos específicos (por exemplo, S. mutans) dentro de biofilmes a vários desafios ambientais e terapêuticos em vários estágios de desenvolvimento 6, 7, 10, 11. Na caixa de ferramenta, que incluiu dois métodos que são críticos para a análise do transcriptoma de biofilmes dentários: 1) RNA de qualidade (por causa da natureza heterogênea das populações biofilme e presença de EPS-matriz), e 2) mineração de dados e interpretação (devido a grande conjunto de dados gerados por microarray).
- RNA isolamento. Para abordar a questão da qualidade do RNA extraído de biofilmes, nós usamos um protocolo otimizado desenvolvido especificamente para RNA isolamento e purificação de células bacterianas enredados em EPS matriz rica em 12 (http://dx.doi.org/10.1016/j . ab.2007.03.021). Este protocolo fornece RNA Número Integrity (RIN) superior a 8.5, que é considerado ideal para a análise de transcriptoma usando RT-qPCR tanto e Microarrays (para exemplos, ver 6, 11).
- Padrão de cDNA microarray e RT-qPCR protocolos (http://pfgrc.jcvi.org/index.php/microarray/protocols.html por exemplo) pode ser usado em combinação para a determinação do transcriptoma-resposta de patógenos específicos (por exemplo, S. mutans) dentro de biofilmes a vários desafios ambientais e terapêuticos em vários estágios de desenvolvimento 6, 7, 10, 11. Na caixa de ferramenta, que incluiu dois métodos que são críticos para a análise do transcriptoma de biofilmes dentários: 1) RNA de qualidade (por causa da natureza heterogênea das populações biofilme e presença de EPS-matriz), e 2) mineração de dados e interpretação (devido a grande conjunto de dados gerados por microarray).
- Fluorescência Imaging. Organização estrutural de biofilmes.
- A maioria dos protocolos de imagens de fluorescência confocal disponíveis na literatura centra-se na rotulagem microbiana. A análise da EPS como um importante componente do biofilme tem sido amplamente negligenciada na investigação biofilme oral envolvendo imagens de fluorescência, com poucas exceções 5, 13, 14. Nós desenvolvemos uma técnica de rotulagem romance e software específico para visualização reprodutível e quantificação de células bacterianas EPS e simultaneamente dentro de biofilmes intacta. Amira 5 (Visage Imaging GmbH, Berlin, Alemanha) é usado para a reconstrução 3D do biofilme, enquanto COMSTAT (disponível em http://www.imageanalysis.dk) e DUOSTAT (http://www.imageanalysis.dk) fornecer o análise quantitativa.
- Imagem da organização estrutural do EPS-bactérias em biofilmes. A Fluor Alexa 647-dextran marcado conjugado é usado para visualizar o EPS-matriz. O dextran marcado fluorescente serve como um primer para estreptocócica glucosyltransferases-GTFS (particularmente GtfB e GtfC), e podem ser simultaneamente incorporada durante a síntese da matriz exopolissacarídeo ao longo do desenvolvimento do biofilme 11.
EPS rotulagem:- Revestimento de discos de hidroxiapatita com a saliva esterilizada por filtração, por 1h, na sequência da "Preparação biofilme" protocolo 8.
- Pipetar 2,8 mL de meio de cultura em cada poço de 24 placas bem, e levar a uma sala escura.
- Adicione 1 mM de dextran conjugados Alexa Fluor em meio de cultura, misturar corante em meio de cultura por pipetagem cima e para baixo (cerca de 10 vezes).
- Dip-lavar a saliva revestido HA (SHA) discos (após 1 h de incubação) duas vezes em tampão AB estéril e colocá-los no meio contendo dextrano conjugados Alexa Fluor.
- Cubra o prato com papel-alumínio e incubar a 37 ° C, 5% CO 2. A duração do tempo de incubação depende de cada projeto experimental. O dextran fluorescente etiquetado não mancha as células bacterianas em concentrações utilizadas neste comodizer 11. Outros EPS coloração técnicas, como com calcofluor 14, pode ser usado em combinação.
- Rotulagem bactérias:
- Bactérias em biofilmes componentes são rotulados no final da formação de biofilmes com ácido nucléico mancha fluorescente padrão (por exemplo, SYTO 9); outras técnicas de fluorescência (por exemplo, espécies específicas fluorescente etiquetado anticorpos 15 ou células que expressam GFP 16) pode ser naturalmente usado para detectar uma ou mais espécies de bactérias no biofilme. A Figura 2 mostra rotulagem simultânea de EPS (vermelho) e bactérias / microcolônias (verde) em uma imagem 3D de um 24 h de idade S. mutans do biofilme formado na superfície de um disco SHA.
- Aquisição de imagens: Cada biofilme é digitalizado em 5-10 posições selecionados aleatoriamente, e z série são gerados por seccionamento óptico em cada uma dessas posições por varredura a laser microscopia confocal utilizando protocolos padrão. Nós usamos uma Olympus FV 1000 de dois fótons microscópio (Olympus, Tóquio, Japão) equipado com x10 (Carl Zeiss, abertura numérica 0.45; trabalhando milímetros de distância 3-4) ou x25 (Olympus LPlan N; NA 1,05; WD mm 2) de água objetivos de imersão. O comprimento de onda de excitação é 810 nm e emissão de comprimento de onda de filtro para SYTO 9 (bactérias) é 495/540 OlyMPFC1 filtro, enquanto o filtro de Alexa Fluor 647 (EPS) é HQ655/40M-2P filtro. Salvar e armazenar imagens brutas.
- Imagem da organização estrutural do EPS-bactérias em biofilmes. A Fluor Alexa 647-dextran marcado conjugado é usado para visualizar o EPS-matriz. O dextran marcado fluorescente serve como um primer para estreptocócica glucosyltransferases-GTFS (particularmente GtfB e GtfC), e podem ser simultaneamente incorporada durante a síntese da matriz exopolissacarídeo ao longo do desenvolvimento do biofilme 11.
- A maioria dos protocolos de imagens de fluorescência confocal disponíveis na literatura centra-se na rotulagem microbiana. A análise da EPS como um importante componente do biofilme tem sido amplamente negligenciada na investigação biofilme oral envolvendo imagens de fluorescência, com poucas exceções 5, 13, 14. Nós desenvolvemos uma técnica de rotulagem romance e software específico para visualização reprodutível e quantificação de células bacterianas EPS e simultaneamente dentro de biofilmes intacta. Amira 5 (Visage Imaging GmbH, Berlin, Alemanha) é usado para a reconstrução 3D do biofilme, enquanto COMSTAT (disponível em http://www.imageanalysis.dk) e DUOSTAT (http://www.imageanalysis.dk) fornecer o análise quantitativa.
2. PASSO 2 - entrada de dados brutos
Dados brutos de entrada de ensaios bioquímicos e RT-qPCR diretamente no arquivo de dados brutos (RDF-MS Excel). Para os dados de microarrays, a carga de um único canal de imagens de slides digitalizados em JCVI Spotfinder (http://pfgrc.jcvi.org/index.php/bioinformatics.html) ou software similar. Criar uma grade local de acordo com as especificações JCVI e depois ajustar manualmente para caber todos os pontos dentro da grade. Medir os valores de intensidade de cada local e salvar em ". MeV" arquivos e armazenados em "dados brutos Microarray".
3. PASSO 3 - PROCESSAMENTO DE DADOS
Organize os dados brutos (bioquímicos e RT-qPCR) na RDF para análise estatística. Transferi-los para "Arquivo de Dados Processados" (DPF - MS Excel). Para microarray e fluorescência de análise de imagens, software específico (atualmente disponíveis e custom-made) são utilizados para processar os dados.
- Dados de microarray:
- Apresentar os dados em bruto gerado na etapa 2 (armazenados em "dados brutos Microarrays") aos dados passo a normalização utilizando software específico. Normalizar os dados usando o JCVI microarray de análise de dados software MIDAS (http://www.tm4.org/midas.html). Use LOWESS e regularização desvio padrão com as configurações padrão, seguido de análise em slide-replicar. Armazenar os arquivos contendo os dados normalizados. Nesta fase (arquivos de dados brutos e arquivos de normalizados os dados prontos), os dados de microarray depósito em um banco de dados de acesso público (por exemplo, NCBI Expressão Gênica Omnibus (GEO) banco de dados em http://www.ncbi.nlm.nih.gov/geo), e registrar o número de adesão para referência futura.
- Dados de fluorescência de imagem
- Biofilme quantificação estrutura.
- Os dados quantitativos correspondentes a cada componente estrutural (EPS e bactérias) de biofilmes é calculado pela COMSTAT eo DUOSTAT recém-desenvolvido, que foram escritos como script em MATLAB 5.1. As imagens de fluorescência-primas são carregadas no software e analisados como segue:
- Use COMSTAT para calcular a biomassa, a espessura da camada de distribuição e outros parâmetros que quantificar e caracterizar a estrutura tridimensional de biofilmes (consulte o manual em http://www.imageanalysis.dk).
- Use DUOSTAT para calcular a co-localização de dois componentes do biofilme, tais como EPS e bactérias (ou espécies microbianas diferentes e / ou componentes extracelulares):
- Configurar o caminho de DUOSTAT em MATLAB 5.1, e pastas imagem aberta, que incluem as imagens dos dois componentes do biofilme.
- Escolha os dois canais que serão correlacionados e analisados. As duas pilhas de imagens também devem ter tamanhos de pixels idênticos em todas as três dimensões (x, y, z), eo mesmo número de imagens em cada pilha.
- Criar limites para cada canal de acordo com o homemual de COMSTAT. Siga as instruções na interface do software de operação (ver artigo de vídeo; manual de DUOSTAT em http://www.imageanalysis.dk). Introduza os dados obtidos em DPF. Veja o exemplo na Figura 4.4.
- Reconstrução tridimensional biofilmes.
- A arquitetura tridimensional dos biofilmes é visualizada utilizando Amira. Importar as imagens de fluorescência armazenados em "Folder Imagens Raw" em software, e usar o processamento voltex e iso-superfície para criar 3-D renderings de cada um dos componentes do biofilme (ver Amira manual em http://www.amira.com / documentação / manuais-e-solte-notes.html). Veja o exemplo na Figura 4.4.
- Os dados quantitativos correspondentes a cada componente estrutural (EPS e bactérias) de biofilmes é calculado pela COMSTAT eo DUOSTAT recém-desenvolvido, que foram escritos como script em MATLAB 5.1. As imagens de fluorescência-primas são carregadas no software e analisados como segue:
- Biofilme quantificação estrutura.
4. PASSO 4 - ANÁLISE DE DADOS
Os dados quantitativos de bioquímica, RT-qPCR e COMSTAT-DUOSTAT ensaios na DPF estão prontos para análise estatística. Após a análise estatística é realizada, gráficos e / ou tabelas podem ser construídos (ver "Resultados Representante" secção).
1. Microarray organização de dados usando o software Microarray Dados Visualizer (MDV).
Devido à complexidade e à saída de grandes conjuntos de dados ao usar microarrays e várias condições experimentais, foi elaborado um software de mineração de dados e organização de dados chamado Microarray Visualizer (MDV) 7 (disponível em http://www.oralgen.lanl.gov/) .
Depois de realizar a análise estatística utilizando-ArrayTools BRB (http://linus.nci.nih.gov/BRB-ArrayTools.html) com um valor P de corte de 0,001 para predição de classes e para a comparação de classes, os dados gerados podem ser submetidos a MDV, como segue:
- Usando o Excel, converter os dados em arquivos BRB MDV texto delimitado por tabulações. Remova todas as cartas que vêm com o número de Tag Gene Locus, por exemplo: SMU.1423c, apagar a letra "c".
- Abra MDV. Vá ao menu Arquivo e selecione "Select Source Annotation". Escolha "Arquivo Flat". Quando apresentado com a caixa de diálogo, use os botões Procurar para escolher os arquivos que contêm anotação para o nome do gene, Gene Ontology (GO), número caminho, e os dados de classificação funcional.
- Vá ao menu Arquivo e importe cada um dos arquivos do item 1. Cada um dos arquivos representa as condições experimentais para análise. Se existem várias condições para a análise, por exemplo, dois tratamentos distintos (tratamento 1 e 2) e controle (veículo) a partir do ponto 4.
- Neste caso, as comparações possíveis são: (A) um tratamento de controle versus (por genes diferencialmente expressos devido ao tratamento 1), (B) tratamento 2 vs controle, e (C) tratamento 1 tratamento vs 2. Dependendo da hipótese de trabalho, conjunto diferente de genes podem ser selecionados.
- Selecione apenas genes diferencialmente expressos na comparação. (Genes únicos afetados pelo tratamento 1 somente) Neste caso, use a função Subtract no menu funções de conjunto. Subtrair os genes de B comparação de A, e salvar o resultado. Então, subtrair C a partir dos dados resultantes. As mesmas ações pode ser feito para obter apenas os genes na B (genes únicos afetados pelo tratamento 2 apenas).
- Para selecionar somente os genes detectados em ambos A e B, use a função União a partir do menu Funções Set para combinar o resultado de comparações A e B, e salvar o resultado. Em seguida, subtrair C a partir dos dados de resultado.
- Essas ações podem ser visualizados usando diagrama de Venn, apresentado na MDV, que é mais intuitiva, e facilita a hipótese-driven organização de dados. O diagrama de Venn saída de dados será exibida na tela (veja a exibição da tela na Figura 3 e artigo de vídeo).
- Para salvar os dados em arquivos de texto delimitado por tabulações, vá ao menu Arquivo e escolha Exportar.
- Exportar os arquivos de texto delimitado por tabulações. Abri-los usando o Excel, e organizar a saída de dados a partir de MDV em tabelas e / ou secretas para uma apresentação gráfica (ver Figuras 4.2, 4.3 e artigo de vídeo).
5. RESULTADOS REPRESENTANTE
Aqui nós fornecemos um exemplo de como a caixa de ferramentas analíticas integra a várias análises de um estudo de biofilme com múltiplas variáveis e condições experimentais.
Caso experimental:
Dinâmica de Streptococcus mutans transcriptoma em resposta ao amido e de sacarose durante o desenvolvimento do biofilme 7.
Background:
As interações de amido na dieta e sacarose com amilase salivar e anfitrião glucosyltransferases estreptocócica poderia melhorar a formação e virulência de S. mutans no biofilme por incrementosasing exopolissacarídeos síntese, o metabolismo do açúcar e acidogenicidade 11. Essa interação patógeno-hospedeiro dieta do complexo podem modular a formação de biofilmes patogênicos relacionados à doença cárie dentária. Realizamos uma análise abrangente bioquímicos e transcriptomic (incluindo perfil genômico inteiro) para entender melhor como S. mutans responde ao amido e sacarose em estágios distintos de desenvolvimento do biofilme na presença de amilase 7.
A caixa de ferramentas de análise foi usado para nos ajudar a integrar os ensaios bioquímicos e moleculares de biofilmes formados sob diferentes condições experimentais e os pontos de tempo. A saída de dados em geral utilizando a caixa de ferramentas de análise é apresentado de forma sequencial na Figura 4 (4,1-4,4). Vale ressaltar que o foco principal aqui é demonstrar a utilidade da caixa de ferramentas, em vez de interpretação de dados e discussão.
- Combinando os ensaios bioquímicos e RT-qPCR para selecionar as condições experimentais para análise microarrays mais. Inicialmente, testamos seis sacarose distintas e 5 de amido e combinações diferentes de sacarose para selecionar as concentrações ideais dos carboidratos para o desenvolvimento de biofilmes por S. mutans utilizando o nosso modelo in vitro. A capacidade de analisar, simultaneamente, a saída de dados a partir do bioensaios nos guiou na escolha de três concentrações específicas (em destaque na Figura 4.1) com base (elevada) quantidade de exopolissacarídeos e insolúveis (avançado) expressão de gtfB (responsáveis pela síntese de glucano insolúvel) em biofilmes .
- Microarray análise. Selecionado (três) concentrações de carboidratos na dieta foram usados para formar biofilmes, que foram removidos em tempo específico de pontos refletindo as várias fases do processo de desenvolvimento de biofilmes. Os biofilmes (divididos em 3 grupos experimentais e 4 horários pontos) foram submetidos à análise de transcriptoma, combinando perfil genômico inteiro (microarrays de cDNA), com matriz BRB-Ferramentas e MDV.
O RNA foi extraído e purificado utilizando protocolo específico biofilme-12, e então submetidos a análise de microarray através da 4 etapa do processo da Analytical caixa de ferramentas. Figura 4.2 ilustra como o MDV ajudou a selecionar os genes de interesse de acordo com nossa hipótese de trabalho que a combinação de sacarose e amido desencadeia resposta transcricional específicos associados com a virulência maior (potencial cariogênico) de S. mutans no biofilme. Os dados brutos gerados pelo BRB-ArrayTools (Figura 4.2a) foi processada pelo MDV usando o diagrama de Venn (Figura 4.2b). Neste estudo, o grupo de genes relacionados com a nossa hipótese são as diferencialmente expressos em comparação A (0,5% de sacarose + 1% de amido versus 1% de sacarose) e B (0,5% de sacarose + amido 1% vs 0,5% de sacarose), mas não os genes em comparação C (0,5% de sacarose versus 1% de sacarose) (Figura 4.2ae 4.2b). Conforme mostrado na Figura 4.2c, o MDV reduziu muito o número total de genes a ser analisados e, ao mesmo tempo filtrada-out dos genes não diretamente relacionado com a influência da sacarose e amido em combinação. A MDV saída de dados também foi convertido em um display gráfico mostrando os dados organizados por classe funcional de cada um dos genes diferencialmente expressos (cima e para baixo-regulado), no amido + sacarose-biofilmes (vs. sacarose-grown biofilmes) em cada dos 4 tempos (Figura 4.3). O software MDV é user-friendly e facilitou a mineração / organização dos dados grandes e complexos conjuntos de experimentos nosso microarray. - . Biofilme imagem Concomitantemente, a organização estrutural do biofilme foi analisado utilizando-COMSTAT DUOSTAT-Amira incluídos na caixa de ferramentas (veja um exemplo da reconstrução em 3D e análise quantitativa do amido + sacarose-grown biofilme na Figura 4.4; ver também artigo vídeo). A morfologia, distribuição e relacionamento estrutural de EPS e microcolônias pode ser visualizada usando renderização 3D de superfície por Amira. Close-up vista para área selecionada pode ser gerado, o que ilustra específicas de bactérias EPS relações estruturais em microescala (Figura 4.4). Além disso, o mesmo conjunto de imagens confocal pode ser processado por COMSTAT DUOSTAT para biomassa / microcolony medições, distribuição espacial e co-localização de bactérias e células EPS simultaneamente. Por exemplo, a distribuição vertical de EPS e bactérias da superfície do disco a fase fluida foi calculado a partir de cada secção óptica da imagens tridimensionais biofilme confocal usando COMSTAT-DUOSTAT (gráfico da Figura 4.4;. Veja o artigo de vídeo). Os dados mostram maior proporção de EPS (linha vermelha) do que as bactérias (linha verde) em toda a profundidade de biofilmes. Além disso, a maioria das células bacterianas estão associadas com EPS (linha azul), especialmente nas camadas média e externa do biofilme. Esta observação indica que os biofilmes cultivados em amido e sacarose são especialmente ricos em exopolímeros, que são emalhamento (em estreito contato com a maioria) das células bacterianas; organização estrutural, a aumentar a estabilidade e coesão do biofilme 13. Os renderings tridimensional e medições quantitativas de imagens confocal fornecer informações adicionais sobre a estrutura do biofilme, que complementa os dados bioquímicos e expressão de genes (aumento GtfB tipo síntese-glucano insolúvel por S. mutans em amido + sacarose-grown biofilmes) (para ver exemplos adicionais 5, 6, 11, 13).
A Analítica Ferramenta Caixa-nos ajudaram a obter, organizar e integrar os dados de bioensaios diferentes, o que proporcionou uma análise abrangente de como S. mutans podem responder à complexidade das mudanças ambientais como resultado de uma dieta-hospedeiro interações encontrado na cavidade oral (ver detalhes em Klein et al, 2009 11;.. Klein et al, 2010 7).

Figura 1. Fluxograma da Analytical caixa de ferramentas para análise de Biofilme.

Figura 2. Imagens de fluorescência confocal de EPS e bactérias em biofilmes. Visualização simultânea de EPS (vermelho) e bactérias / microcolônias (verde) na prestação tridimensional do biofilme formado sobre Streptococcus mutans SHA superfície do disco.

Figura 3. Microarray Data Mining e Organização de Dados Usando Microarray Visualizer (MDV) Software. Uso de Venn característica diagrama para selecionar a genes de interesse, e seguido pela adição de nome de gene e anotação classe funcional.

Figura 4.1. Avaliação da formação de biofilme por S. mutans utilizando ensaios bioquímicos (A) e RT-qPCR (B). Os dados do INS (exopolissacarídeos insolúvel) correlaciona bem com o padrão de expressão gtfB, e com a biomassa do biofilme. Sacarose a 1% foi a concentração máxima para a formação de INS, expressão gtfB e acúmulo de biofilme na superfície SHA sacarose enquanto 0,5% foi a concentração mínima necessária para o desenvolvimento de biofilme melhor usar nosso modelo in vitro. S. mutans células cultivadas na presença de sacarose 0,5% + 1% de amido rendeu a maior biomassa, e apresentou mais de biofilmes INS outros, que se correlacionou com maior expressão gtfB (B2). Estas concentrações de carboidratos foram selecionados para análise do transcriptoma mais.
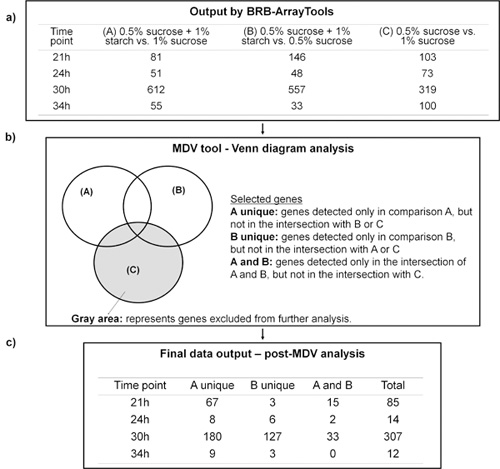
Figura 4.2. Microarray análise de dados usando BRB-Array Tools em conjunto com software MDV. a) Representar o número de genes detectados como diferencialmente expressos em cada comparação (A, B ou C) e tempo avaliados usando BRB-Array Tools. b) Dados Microarray Visualizer (MDV), utilizando o diagrama de Venn para selecionar genes de interesse. c) Os genes selecionados de acordo com a análise MDV.

Figura 4.3. S. mutans genes diferencialmente expressos em amido + sacarose-biofilmes (vs. sacarose-biofilme) em vários pontos temporais organizado pela classe funcional. Gene anotações são baseados em informações fornecidas pelo Los Alamos National Laboratory (www.oralgen.lanl.gov) ou por literatura publicada disponível no mesmo site.

Figura 4.4. Tridimensional e renderização COMSTAT DUOSTAT análise de amido + sacarose biofilme.
Access restricted. Please log in or start a trial to view this content.
Discussão
Nesta apresentação, demonstramos dois componentes críticos do Analytical caixa de ferramentas (EPS / bactérias imagem e microarray de mineração de dados / processamento), a versatilidade e utilidade das várias análises integradas no sistema. Claramente, a caixa de ferramentas facilitou a análise abrangente (comparativo) e simultânea dos vários aspectos da bioquímica biofilmes, arquitetura e expressão gênica em resposta às diferentes condições experimentais, utilizando um modelo in vitro.
Access restricted. Please log in or start a trial to view this content.
Divulgações
Agradecimentos
Os autores gostariam de agradecer ao Dr. Gary Xie e Herbert Lee para o desenvolvimento do MDV. Agradecemos também os drs. Simone Duarte, Ramiro Murata, Jae-Gyu Jeon, Jacqueline Abranches, e Ms. Stacy Gregoire por sua contribuição técnica e científica para os componentes analíticos da caixa de ferramentas. Este estudo foi apoiado em parte pela USPHS Research conceder DE018023 do Instituto Nacional de Pesquisa Dental e Craniofacial.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Syto 9 | Invitrogen | S34854 | |
| Syto 60 | Invitrogen | S11342 | |
| Dextran conjugated alexa 647 | Invitrogen | D22914 | |
| Olympus FV1000 two-photon laser scanning microscope | Olympus Corporation |
Referências
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- Leme, P. aes, Koo, A. F., Bellato, H., Bedi, C. M., G, J. A. C. ury The role of sucrose in cariogenic dental biofilm formation--new insight. J Dent Res. 85, 878-887 (2006).
- Koo, H., Schobel, B. D., Scott-Annem, K., Watson, G., Bowen, W. H., Cury, J. A., Rosalen, P. L., Park, Y. K. Apigenin and tt-farnesol with fluoride effects on S. mutans biofilms and dental caries. J Dent Res. 84, 1016-1020 (2005).
- Koo, H., Xiao, J., Klein, M. I., Jeon, J. G. Exopolysaccharides produced by Streptococcus mutans glucosyltransferases modulate the establishment of microcolonies within multispecies biofilms. J Bacteriol. 192, 3024-3032 (2010).
- Jeon, J. G., Klein, M. I., Xiao, J., Gregoire, S., Rosalen, P. L., Koo, H. Influences of naturally occurring agents in combination with fluoride on gene expression and structural organization of Streptococcus mutans in biofilms. BMC Microbiol. 9, 228-228 (2009).
- Klein, M. I., DeBaz, L., Agidi, S., Lee, H., Xie, G., Lin, A. H. M., Hamaker, B. R., Lemos, J. A., Koo, H. Dynamics of Streptococcus mutans transcriptome in response to starch and sucrose during biofilm development. PLoS ONE. , 0013478-0013478 (2010).
- Lemos, J. A., Abranches, J., Koo, H., Marquis, R. E., Burne, R. A. Protocols to Study the Physiology of Oral Biofilms. Methods Mol Biol. 666, 87-102 (2010).
- Koo, H., Hayacibara, M. F., Schobel, B. D., Cury, J. A., Rosalen, P. L., Park, Y. K., Vacca-Smith, A. M., Bowen, W. H. Inhibition of Streptococcus mutans biofilm accumulation and polysaccharide production by apigenin and tt-farnesol. J Antimicrob Chemother. 52, 782-789 (2003).
- Koo, H., Seils, J., Abranches, J., Burne, R. A., Bowen, W. H., Quivey, R. G. Influence of apigenin on gtf gene expression in Streptococcus mutans UA159. Antimicrob. Agents Chemother. 50, 542-546 (2006).
- Klein, M. I., Duarte, S., Xiao, J., Mitra, S., Foster, T. H., Koo, H. Structural and molecular basis of the role of starch and sucrose in Streptococcus mutans biofilm development. Appl Environ Microbiol. 75, 837-841 (2009).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Anal Biochem. 365, 208-214 (2007).
- Xiao, J., Koo, H. Structural organization and dynamics of exopolysaccharide matrix and microcolonies formation by Streptococcus mutans in biofilms. J Appl Microbiol. 108, 2103-2113 (2010).
- Thurnheer, T., Gmür, R., Shapiro, S., Guggenheim, B. Mass transport of macromolecules within an in vitro model of supragingival plaque. Appl Environ Microbiol. 69, 1702-1709 (2003).
- Chalmers, N. I., Palmer, R. J. J. r, Du-Thumm, L., Sullivan, R., Shi, W., Kolenbrander, P. E. Use of quantum dot luminescent probes to achieve single-cell resolution of human oral bacteria in biofilms. Appl Environ Microbiol. 73, 630-636 (2007).
- Deng, D. M., Hoogenkamp, M. A., Ten Cate, J. M. >, Crielaard, W. Novel metabolic activity indicator in Streptococcus mutans biofilms. J Microbiol Methods. 77, 67-71 (2009).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados