É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Única partícula Reconstrução Microscopia Eletrônica do Complexo exossomo Usando o método Tilt Aleatório Conical
Neste Artigo
Resumo
Este artigo descreve um método padrão para obter uma reconstrução tridimensional (3D) de macromoléculas biológicas por microscopia eletrônica de coloração negativa (EM). Neste protocolo, vamos explicar como obter a estrutura 3D do complexo exossomo Saccharomyces cerevisiae em média resolução usando o método de reconstrução cônica aleatória de inclinação (RCT).
Resumo
Microscopia eletrônica única partícula (EM) de reconstrução recentemente se tornou uma ferramenta popular para obter a estrutura tridimensional (3D) de complexos macromoleculares grandes. Comparado a cristalografia de raios X, ele tem algumas vantagens exclusivas. Primeiro, única partícula EM reconstrução não precisa cristalizar a amostra de proteína, que é o gargalo em cristalografia de raios X, especialmente para grandes complexos macromoleculares. Em segundo lugar, ele não precisa de grandes quantidades de amostras de proteínas. Em comparação com miligramas de proteínas necessárias para a cristalização, única partícula de reconstrução EM só precisa de vários micro-litros de solução de proteína em concentrações nano-molar, utilizando o método de coloração negativa EM. No entanto, apesar de uma assembleias macromolecular poucos com simetria de alta, única partícula EM é limitada a uma resolução relativamente baixa (inferior a 1 nm Resolução) para muitos espécimes especialmente aqueles sem simetria. Esta técnica também é limitada pelo tamanho das moléculas em estudo, ou seja, 100 kDa para os espécimes negativamente coradas e 300 kDa para congelados hidratado espécimes em geral.
Para uma nova amostra de estrutura desconhecida, geralmente usamos uma solução de metais pesados para incorporar as moléculas através da coloração negativa. A amostra é então examinado em um microscópio eletrônico de transmissão de tomar duas dimensões (2D) micrografias das moléculas. Idealmente, as moléculas de proteína têm uma estrutura homogênea 3D, mas apresentam diferentes orientações na micrografias. Essas micrografias são digitalizadas e processadas em computadores como "partículas single". Usando dois-dimensional de alinhamento e técnicas de classificação, as moléculas homogêneas no mesmos pontos de vista são agrupados em classes. Suas médias melhorar o sinal de formas 2D da molécula. Depois vamos atribuir as partículas com a orientação adequada relativa (ângulos de Euler), seremos capazes de reconstruir as imagens das partículas 2D em um volume 3D virtual.
Na única partícula de reconstrução 3D, é um passo essencial para atribuir corretamente a orientação adequada de cada partícula. Existem vários métodos para atribuir o ponto de vista de cada partícula, incluindo a reconstituição angular 1 e aleatória cônica tilt método (RCT) 2. Neste protocolo, descrevemos nossa prática no sentido de obter a reconstrução 3D do complexo levedura exossomo usando coloração negativa EM e RCT. Deve-se notar que o nosso protocolo de microscopia eletrônica e processamento de imagem segue o princípio básico da RCT, mas não é a única maneira de executar o método. Primeiro, descrevem como incorporar a amostra de proteína em uma camada de uranilo Formate com uma espessura comparável ao tamanho da proteína, utilizando uma grade de carbono holey coberto com uma camada de película de carbono contínua fina. Em seguida, a amostra é inserida em um microscópio eletrônico de transmissão de recolher untilted (0 graus) e inclinada (55 graus) pares de micrografias, que será usado posteriormente para o processamento e obtenção de um modelo inicial em 3D do exossomo levedura. Para este fim, realizamos RCT e refinar o modelo inicial em 3D usando o método de projeção combinando refinamento 3.
Protocolo
1. Princípio do Método Tilt Aleatório Conical
- O princípio do método de inclinação aleatórios requer cônica tendo um par de micrografias da mesma região da amostra no interior do microscópio eletrônico. Uma foto é tirada da amostra em uma posição untilted (Figura 1A) ea outra imagem é tirada da amostra inclinada em um ângulo entre 50 a 70 graus (no nosso caso, usamos 55 graus). (Figura 1B)
- Usando o computador, o par micrografia digitalizado é colocado lado a lado e imagens do mesmo partículas são selecionados. (Figura 1C)
- Em coordenadas tridimensionais, as imagens de partículas untilted e seus parceiros inclinado são correlacionados entre si pela direção do eixo de inclinação eo ângulo de inclinação. (Figura 1D)
- O alinhamento das imagens de partículas untilted traz as imagens de partículas inclinado aos seus correspondentes locais azimutal. (Figura 1E)
- Usando várias imagens de partículas inclinado preenchendo o espaço azimutal, a estrutura tridimensional da molécula pode ser reconstruído usando um algoritmo de retro-projeção. (Figura 1F)
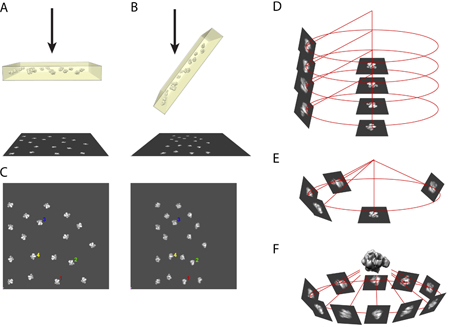
Figura 1. Uma ilustração do princípio de RCT reconstrução.
2. Prepare as Grades de Holey carbono revestido com carbono Fino
Justificativa: Nós utilizamos método de coloração negativa para fixar a amostra de proteínas para a reconstrução de inclinação aleatórios cônico. A fim de preservar as macromoléculas sem muito achatamento durante a secagem, tentamos incorporar as moléculas de proteína em uma mancha profunda, com uma espessura sobre a dimensão de proteínas 4. Em geral, o carbono contínua é usada na tomada de amostras negativamente manchado. Esse tipo de carbono, no entanto, é difícil controlar a espessura da mancha em torno das partículas de proteínas. Nós, portanto, o uso home-made grades de carbono holey coberto com uma fina camada de filme de carbono (~ 5 nm de espessura) para fazer espécimes negativamente manchado. Pequenos poços formados pelos furos permitem manter a solução de proteína ea solução mancha no grid por isso é muito mais fácil de incorporar proteína em uma espessura ideal mancha. Além disso, a fina camada de carbono sobre o furo reduz o ruído de fundo muito.
- Preparar a solução Formvar 0,5%. Na capela, adicionar 0,45 g de resina polivinil formal e 90 mL de clorofórmio num copo de vidro de 100 mL. Use papel alumínio para cobrir o copo, e usar uma barra de agitação pequenos para ajudar na dissolução de resina formais sobre um agitador magnético. Demora cerca de 15 minutos para dissolver a resina.
- Durante a dissolução do Formvar, vidro de microscopia limpa slides em metanol e seque com Kimwipes.
- Após a resina Formvar é completamente dissolvido em clorofórmio, adicionar 1 mL de glicerol 50% da superfície da solução. Ajustar o volume do glicerol adicionado afeta a densidade dos buracos no carbono holey. Mergulhe a ponta de um ultrasonicator na solução em cerca de 1 centímetro de profundidade e usar a potência máxima sonicate 1 min para fazer emulsão de gotas de glicerol em solução Formvar. A solução torna-se leitoso após esta etapa. Mais sonicação causas tamanho menor de buracos no carbono holey.
- Imediatamente após a sonicação, mergulhe a lâminas de vidro limpas verticalmente no emulsão para 1 segundo, tirá-los, e blot fundo dos slides usando filtro de papel para formar uma película plástica fina sobre a superfície das lâminas. Após a evaporação choloroform, verificar a densidade e tamanho dos furos no filme sob um microscópio de luz. Ajustar a condição de preparação de acordo com as necessidades. Com a condição descrita aqui, nós geralmente obter furos com diâmetro de 3 a 4 micro-metros e 10 ~ 20 buracos em cada quadrado de uma grade de malha 400.
- Após a lâminas de vidro são secos, corte a ponta do filme plástico sobre a superfície da lâmina. Float o filme off na superfície da água destilada. A película fina na superfície da água pode ser observado em um ângulo de olhar contra a reflexão da luz. Grades lugar 400 mesh de cobre sobre o filme, um por um, com as grades 'superfície lisa voltada para baixo.
- Pegue o filme plástico com grades nele com um pedaço de papel. Vire o papel e deixe secar numa placa de Petri. Mergulhe o papel em metanol para remover o glicerol residual nos buracos e deixe o papel secar ao ar.
- Brasão das grades com uma camada de carbono com a espessura de 20 nanômetros ~ em um evaporador de carbono. A espessura pode ser determinada pela cor cinza do carbono.
- Mergulhe as grades com carbono em clorofórmio por meia hora para remover o Formvar. Após as grades são secas, obtivemos home-made grades de carbono holey.
- Evaporar fina camada de carbono com cerca de 5 nanômetros de espessura na superfície de mica recém clivada.
- Cuidadosamente coloque as grades de carbono holey debaixo de água destilada. Float o carbono fina da superfície mica sobre a superfície da água e depositá-lo sobre o carbono holeygrades lentamente. Seca as grades de uma capela.
3. Coloração negativa do Complexo exossomo
Justificativa: Há muito poucas soluções de heavy metal mancha que pode ser usado para EM coloração negativa, incluindo acetato de uranila, formate uranila, ácido fosfotúngstico, molibdato de amônio e outros. Solução mancha diferentes apresentam propriedades únicas. Por exemplo, acetato de uranila fornece alto contraste da partícula, mas pode falhar complexos de proteínas que não gostam de ambiente ácido. Para essas amostras, ácido phosphotungestic em pH neutro pode ser uma boa solução mancha. Nós escolhemos Formate uranilo saturada (UF), devido à sua solução de granularidade fina e alta capacidade de penetração em moléculas.
- Ferva água destilada por 1 min. Esfriá-la lentamente à temperatura ambiente. Este passo é remover o oxigênio dissolvido da água.
- Faça fresco 2% uranilo Formate solução (UF). Misture 1 ml de água e 20 mg UF em um tubo de 1,5 mL. Vortex por 10 min.
- Ajustar o valor de pH para 5,0 adicionando 2 microlitro de 10 M de hidróxido de potássio. Misturar imediatamente. A solução deve ser de cor mais amarelada. O pH da solução não deve ser muito alta, caso contrário, a mancha precipitados.
- Colocar o tubo na vortexer para outra 10 min.
- Girar a solução em uma centrífuga de mesa na velocidade máxima por 10 min.
- Filtrar a solução através de uma membrana de PVDF 0,2 micrômetro. Esta é a solução UF fresco. Cobrir o tubo de solução em um pedaço de papel alumínio para evitar a luz. A solução tem que ser usado no mesmo dia.
- Descarga luminescente um thin-carbono-over-holey grade de carbono usando um aparelho de descarga luminescente por 30 segundos a 25 mA.
- Coloque um pedaço de parafilme limpa no banco. Coloque três gotas de solução de 50 UF microlitro mancha em cima do parafilme.
- Diluir o complexo exossomo a uma concentração de 50 ~ 100 nm usando o tampão de diluição (25 mM Tris-HCl pH 7,5, 100 mM NaCl, 2 mM DTT). Colocar 4 microlitro da proteína diluído no brilho descarregada grid. Deixe a amostra ficar na grade por um minuto. (Nota:. Essa concentração final de moléculas geralmente dá uma densidade ideal de bem-partículas dispersas no grid negativamente coradas Para formate uranilo ou solução de uranilo acecate mancha, o fosfato ou alta concentração de sal (mais de 0,5 M) em geral é não é bom para obter resultados de coloração bem. Nossa experiência sugere que Hepes ou Pipes trabalhar bem com as soluções de uranila mancha.)
- Use um pedaço de papel de filtro para apagar a solução residual da borda da rede e virar a grade imediatamente no topo das gotículas mancha e lave a grelha por cerca de 10 segundos em cada gota.
- Após a última lavagem, deixe a mancha permanecer no grid por mais 1 min e depois apagar a mancha de distância de um pedaço de papel de filtro. Manter uma fina camada de solução de manchas na superfície da grade, a fim de obter um bom resultado profunda mancha. Deixe a grade de secar rapidamente em uma capela.
4. Microscopia Eletrônica do Complexo exossomo
Justificativa: Qualquer microscópio eletrônico de transmissão com um palco de inclinação pode ser usado para coletar pares de inclinação da amostra para RCT reconstrução. Em teoria, quanto maior o ângulo do espécime pode ser inclinado para coletar dados, melhor. Na prática, devido ao desenho do porta-amostra ea geometria da grelha, o ângulo máximo operável é limitado 50-70 graus. Neste protocolo, apenas descrevem o nosso procedimento usando um FEI Tecnai-12 microscópio eletrônico. Para os outros modelos de microscópios, as operações precisam ser ajustados de acordo com a exigência do projeto e à propriedade do instrumento.
- Coloque a grade amostra no porta amostra e em seguida, colocar o titular em um microscópio eletrônico FEI Tecnai -12. O microscópio é operado a 120 kV. Usamos Gatan Ultrascan4000 câmera CCD para tirar fotos. Certifique-se que o 'Flip em torno do eixo vertical "na caixa de diálogo' câmera config 'da interface Micrografia Digital está desmarcada para assegurar a determinação da lateralidade correta. (Nota:. Isso é importante, especialmente se o leitor se baseia em procedimentos SPIDER RCT reconstrução de obter um modelo 3D)
- Confira a grade da amostra em baixa ampliação para encontrar as melhores praças manchado. Esse tipo de praças deve ter uma dúzia de buracos com dimensão de cerca de 1 ~ 2 micrômetro e escura mancha áreas neles. (Figura 2)

Figura 2. A micrografia baixa ampliação de uma grade mostrando quadrados com manchas bons e ruins. - Ativar o modo dose baixa de interface do usuário FEI e alinhar a pesquisa, o foco ea exposição posição no modo de baixa dosagem. Usamos ampliações de 150.000 para foco, exposição e 52.000 para 1,5 metros de comprimento da câmara em difração para pesquisa. O tempo de exposição é ajustado para 1 segundo. Definir o foco posição 2 micrômetro de distância da pos exposiçãoition ao longo do eixo de inclinação.
- Encontrar os buracos com coloração bem em modo de busca, e salvar os locais. Os buracos têm, geralmente, boa gradiente mancha escura neles observados sob o modo de busca. Tire uma foto CCD da praça. Inclinação da amostra a 55 graus, tirar outra foto. Compare as duas imagens, identificar os buracos emparelhado nas duas micrografias. (Figura 3)
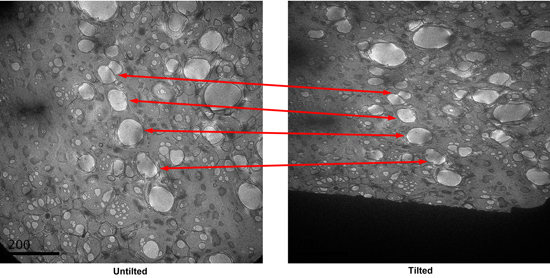
Figura 3. Um par de inclinação de micrografias de uma praça no modo de busca. Correspondentes furos emparelhados são indicados. - Incline o estágio de volta para 0 grau. Usando o kit de baixa dosagem para tirar fotos de cada buraco identificadas no modo de busca em grandes ampliações. O defocus utilizado é de cerca de -0,7 micrômetro.
- Depois de todos os buracos foram tomadas imagens de, incline o palco a 55 graus. Tome micrografias da amostra inclinada com a mesma ampliação como os untilted com uma desfocagem de cerca de -1,2 micrômetro. Identificar os pares correspondentes inclinado de micrografias com base nos padrões da micrografias baixa ampliação. O padrão irregular do home-made grades holey de carbono ajuda a correlação. Examine pares inclinação de micrografias e remover as micrografias com manchas ruins como rasas áreas manchadas (partículas parecem ter um halo em torno deles sob a condição inclinado). (Figura 4)
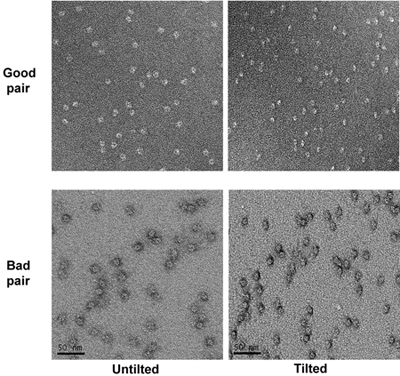
Figura 4. Dois pares de inclinação de micrografias da amostra em grandes ampliações. As micrografias bons e maus são marcados.
5. Processamento de Imagem dos Dados
Justificativa: Existem diferentes opções e pacotes de software para realizar a reconstrução RCT no computador. O mais geralmente usado é SPIDER 5. Um protocolo básico para executar RCT em SPIDER podem ser encontradas na página http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Um protocolo detalhado para executar RCT em SPIDER é descrito no artigo de Shaikh et al. 6 Em nosso protocolo, usamos uma combinação de IMAGIC-5 7 e SPIDER na versão em vídeo do protocolo. Nós também fornecemos um procedimento alternativo para o único uso SPIDER na versão texto do protocolo.
- Configuração dos programas. Usamos o proc2d na 8 REMA pacote para mudar o formato de imagem a partir de imagem digital para Gatan SPIDER formato de imagem. IMAGIC-5 é usado para fazer o alinhamento 2D. SPIDER é usado para fazer a reconstrução 3D e requinte.
Sub-seção 1: Escolher pares de inclinação das partículas.
- Converter o *. dm3 Gatan imagem digital para SPIDER formato de imagem usando o comando proc2d no REMA. Os pares inclinado e untilted são nomeados no padrão como *** *** t.spi e u.spi, respectivamente.
- Escolha os pares de partículas usando o programa 'WEB' distribuídos em SPIDER pacote do programa. Siga as instruções na Coordenadas são salvos em automaticly dcu ***. ***. spi e dct spi para partículas untilted e inclinado, respectivamente. Outro documento dcb ***. spi contém as informações ângulo de inclinação entre as partículas inclinado e untilted (Nota:. Os três ângulos durante a montagem na WEB entre os pares de inclinação não garantem a lateralidade correta da reconstrução final porque theta não tem o sinal (valor positivo) e phi e gama contar com os valores iniciais definidas para eles. Examinando a direção do gradiente defocus na micrografia inclinado iria ajudar a definir os valores corretos inicial do phi e gama antes da colocação, a fim de obter uma correta lateralidade da figura de reconstrução. 5 ilustra a convenção correta.)
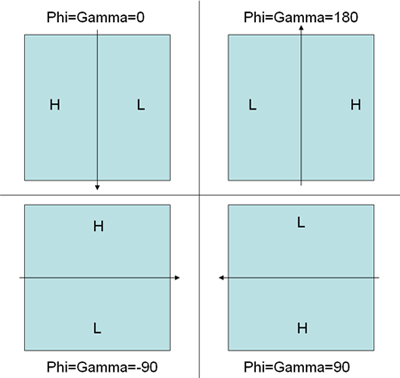
Figura 5. Uma ilustração para explicar a convenção de determinação do ângulo de inclinação de partículas WEB par-picking modo de ajuste determinado pelo gradiente de desfocagem. H representa alta defocus área, enquanto L representa baixa defocus área. A seta representa eixo de inclinação. O correspondente correta ângulos iniciais para PHI e GAMMA são indicados para cada esquema. - Box para fora todas as partículas escolheu usar uma versão modificada do script SPIDER como mostrado na página http://www.wadsworth.org/spider_doc/spider/docs/partpick.html . Script poupa as pilhas de partículas untilted e inclinado como u.spi e t.spi. O número de micrografia onde as partículas são de deve ser salvo em particle_list.spi. (Nota: Isto é muito importante para gerar os arquivos ângulo correto para eulerRCT).
Sub-seção 2: Two-dimensional de alinhamento e classificação das imagens de partículas untilted.
- Converter as partículas untilted em IMAGIC-5 formato usando o programa em2em em Imagic-5 pacote. Alinhar e classificar as partículas em classes homogêneas iterativamente usando o IMAGIC-5 programas (Anexo A). Usando o comando msa-nomes-in-class em IMAGIC-5 para gerar classes de tabela de pesquisa de partículas, que salvar como imagic_classes.lis. (Nota:. A classificação e alinhamento é aumentar o número de partículas com a mesma forma, bem como reduzir a variação em uma classe de Variância mapa de cada classe pode fornecer informações sobre a qualidade da classe.)
- Gerar um arquivo de plotagem (ali_50.plt na demonstração em vídeo) para translação e rotação valores de alinhamento de cada partícula usando o comando cabeçalho em IMAGIC-5.
- Converter as classes tabela look-up no documento SPIDER arquivos base_file ***. spi utilizando um script perl lis2spi.pl distribuídos em http://cryoem.berkeley.edu .
- Converter os valores translação e rotação de alinhamento de cada partícula a partir do arquivo gerado enredo para a tradução e os valores de rotação da etapa em 5,6 SPIDER ali_50.spi arquivo de documento usando um roteiro plt2spi.pl distribuídos em http://cryoem.berkeley.edu .
Usamos IMAGIC-5 para o alinhamento em 2D e classificação, porque dá o melhor desempenho neste trabalho em nossas mãos. Estratégia alternativa no SPIDER para o alinhamento de duas dimensões e classificação pode ser encontrada em http://www.wadsworth.org/spider_doc/spider/docs/align.html . Temos também utilizado SPIDER para realizar a análise 2D sobre as partículas untilted do exossomo. Segue-se um procedimento simples.
Alternativa 5.5) Use o alinhamento de referência livre, conforme descrito na alinhar as imagens. Dois scripts simples podem ser encontrados Salvar a rotação e mudança de todas as partículas em um angular_file.spi arquivo de documento.
Alternativa 5.6) Classifique as partículas alinhadas em grupos com a mesma visão, conforme descrito no Temos usado o método K-means para a classificação. Gerar base_file ***. spi com base na classificação.
Sub-seção 3: A reconstrução tridimensional usando as imagens das partículas inclinada.
- Filtro passa-banda, máscara e centro as partículas inclinada assim como para as partículas em untilted IMAGIC-5. Gerar um novo conjunto de dados para partículas intitulado. (Nota: Este passo é opcional, pode ser feito em SPIDER..)
- Criar arquivos de documentos anglular das classes look-up table documentos ali_50.spi, o dcb arquivos ***. spi gerado pelo web 5.3 e no passo a partícula partile_list.spi arquivo de lista a partir do passo 5,4 usando o script SPIDER como no Apêndice B.
- Use scripts de reconstrução do SPIDER a fazer a reconstrução de cada classe, conforme descrito no http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Cada classe de partículas contribui para um volume de reconstrução 3D. Os modelos 3D podem ser examinados em UCSF-Chimera 9. A projeção bidimensional do modelo 3D no ângulo de euler (0,0,0) pode ser comparado com a média de classe correspondente 2D das partículas untilted para verificar a qualidade da reconstrução. Encontrar volumes similares, alinhar e fundi-los para gerar volumes iniciais após o procedimento http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html .
Sub-seção 4: Refinamento da reconstrução em 3D usando imagens de partículas untilted.
- Do refinamento projeção do volume de correspondência fundiu inicial contra todas as partículas untilted para obter uma resolução maior volume sem cone desaparecidos e artefato achatamento usando scripts SPIDER, conforme descrito no http://www.wadsworth.org/spider_doc/spider/docs/techs/recon/refine.html .
6. Resultados representativos:
Usando o método RCT, obtivemos cerca de 50 reconstruções do exossomo de um total de 5.000 pares de inclinação (Figura 6). Dos 50 modelos 3D, podemos ver diferentes orientações da sessão complexos na rede, principalmente com duas projeções ortogonais. Um artefato achatamento também é detectável em muitos dos volumes na direção perpendicular à superfície de carbono. Foi realizado o alinhamento e fusão dos volumes 3D para gerar dois volumes iniciais com as vistas ortogonais. Usando a 5.000 imagens de partículas untilted, obtivemos a reconstrução 3D de última mesmo o exossomo em cerca de 18 Angstrom resolução de ambos os modelos iniciais (Figura 7). A estrutura revelou a arquitetura do exossomo fermento e fornecido informações sobre o caminho de recrutamento substrato RNA 10.

Figura 6. 50 modelos 3D do complexo exossomo por RCT reconstrução.
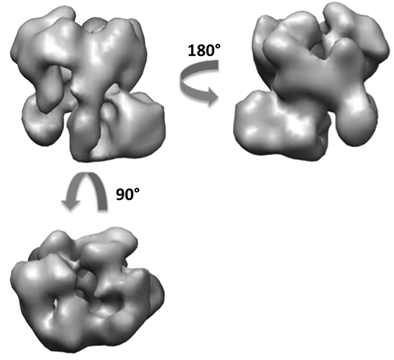
Figura 7. Reconstrução 3D do complexo exossomo após a refinamento.
Apêndice:
Apêndice A. O arquivo de script para alinhamento 2D e classificação em IMAGIC-5.
File: auto_align_i.sh
Clique aqui para arquivo
Apêndice B. O arquivo de script para gerar arquivo angular para a reconstrução 3D em SPIDER.
File: generate_angular_file.spi
Clique aqui para arquivo
Discussão
Neste artigo apresentamos um protocolo detalhado de preparação de amostras e reconstrução tridimensional do complexo exossomo meio de microscopia eletrônica negativa coloração. Usando este método, obtivemos a reconstrução em 3D usando o método aleatório inclinação cônica sem qualquer conhecimento prévio da estrutura. Método aleatório inclinação cônica não exige necessariamente uma amostra homogênea, mas o seguinte projeção passo de refinamento de correspondência seria necessário uma amostra ho...
Divulgações
Agradecimentos
Os autores gostariam de agradecer aos membros de Nogales laboratório na Universidade da Califórnia-Berkeley em ajudar a estabelecer os protocolos iniciais e os membros do Wang laboratório na Universidade de Yale em sua ajuda para estabelecer os protocolos completa. Reconhecemos também as equipes em crio-EM facilidade e alto desempenho Centro de Computação na Universidade de Yale School of Medicine, pelo seu apoio. HW é um Smith Família premiado.
Materiais
| Name | Company | Catalog Number | Comments |
| Polyvinyl Formal Resin | Electron Microscopy Sciences | 63450-15-7 | |
| Uranyl Formate | Electron Microscopy Sciences | 22451 | |
| Superfrost Microscope Slides | Thermo Fisher Scientific, Inc. | 4951F-001 | |
| 400 mesh grid regular | SPI Supplies | 3040C | |
| Carbon coater Auto 306 | Edwards Lifesciences | ||
| Tecnai-12 Electron Microscope | FEI | ||
| Glow Discharger | BAL-TEC | Sputter Coater SCD 005 |
Referências
- van Heel, M. Angular reconstitution: a posteriori assignment of projection directions for 3D reconstruction. Ultramicroscopy. 21, 111-123 (1987).
- Radermacher, M. Three-dimensional reconstruction of single particles from random and nonrandom tilt series. J Electron Microsc Tech. 9, 359-394 (1988).
- Penczek, P. A., Grassucci, R. A., Frank, J. The ribosome at improved resolution: new techniques for merging and orientation refinement in 3D cryo-electron microscopy of biological particles. Ultramicroscopy. 53, 251-270 (1994).
- Ohi, M., Li, Y., Cheng, Y., Walz, T. Negative staining and image classification - powerful tools in modern electron microscopy. Biol Proced Online. 6, 23-34 (2004).
- Frank, J., Radermacher, M., Penczek, P., Zhu, J., Li, Y., Ladjadj, M., Leith, A. SPIDER and WEB: processing and visualization of images in 3D electron microscopy and related fields. J Struct Biol. 116, 190-199 (1996).
- Shaikh, T. R., Gao, H., Baxter, W. T., Asturias, F. J., Boisset, N., Leith, A., Frank, J. SPIDER image processing for single particle reconstruction of biological macromolecules from electron micrographs. Nat Protoc. 3, 1941-1974 (2008).
- Heel, M. v. a. n., Harauz, G., Orlova, E. V., Schmidt, R., Schatz, M. A new generation of the IMAGIC image processing system. J Struct Biol. 116, 17-24 (1996).
- Ludtke, S. J., Baldwin, P. R., Chiu, W. EMAN: semiautomated software for high-resolution single-particle reconstructions. J Struct Biol. 128, 82-97 (1999).
- Pettersen, E. F., Goddard, T. D., Huang, C. C., Couch, G. S., Greenblatt, D. M., Meng, E. C., Ferrin, T. E. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25, 1605-1612 (2004).
- Wang, H. W., Wang, J., Ding, F., Callahan, K., Bratkowski, M. A., Buttler, J. S., Nogales, E., Ke, A. Architecture of the yeast Rrp44 exosome complex suggests routes of RNA recruitment for 3' end processing. Proc Natl Acad Sci USA. 104, 16844-16849 (2007).
- Scheres, S. H., Nunez-Ramirez, R., Sorzano, C. O., Carazo, J. M., Marabini, R. Image processing for electron microscopy single-particle analysis using Xmipp. Nat Protoc. 3, 977-990 (2008).
- Yoshioka, C., Pulokas, J., Fellmann, D., Potter, C. S., Milligan, R. A., Carragher, B. Automation of random conical tilt and orthogonal tilt data collection using feature-based correlation. J Struct Biol. 159, 335-346 (2007).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados