Method Article
Armadilha cromossomo associados para Identificar Interações de longo alcance DNA
Neste Artigo
Resumo
O associado armadilha cromossomo ensaio (ACT) é um novo método imparcial para identificar as interações de longo alcance DNA. A caracterização das interações DNA de longo alcance nos permitirá determinar a relação da arquitetura nuclear para a expressão do gene em ambos os fisiologia normal e em estados doente.
Resumo
Informação genética codificada pelo DNA é organizado em uma estrutura complexa e altamente regulamentado cromatina. Cada cromossomo ocupa um território específico, que pode mudar de acordo com o estágio de desenvolvimento ou do ciclo celular. Expressão gênica pode ocorrer em fábricas especializadas em segmentos transcricional da cromatina podem loop out cromossomo a partir de territórios diversos, levando a co-localização de segmentos de DNA que podem existir em cromossomos diferentes ou distantes no mesmo cromossomo. A Associated cromossomo Armadilha ensaio (ACT) fornece uma metodologia eficaz para identificar essas associações de longo alcance DNA de uma maneira imparcial, estendendo e modificando o cromossomo técnica de captura de conformação. O ensaio ACT torna possível para nós para investigar os mecanismos de regulação da transcrição em trans, e podem ajudar a explicar a relação da arquitetura nuclear para a expressão do gene na fisiologia normal e durante estados de doença.
Protocolo
1. Fixação em formol de interações de longo alcance cromatina
- Cultura linha celular humana HL-60 em RPMI1640 média com 15% FBS e 1 x penicilina / estreptomicina a confluência de 80-90% em uma incubadora fornecido com 5% de CO 2 a 37 ° C.
- Coletar as células em um tubo de 50 ml Nunc, centrifugar a 1.200 rpm por 15 minutos e remova o meio por aspiração
- Adicionar 5 ml de meio de cultura para voltar a suspender as pelotas de células, e contar as células usando um hemocitômetro. Demorar cerca de 1 x 10 7 células para um volume de 40 ml com FBS RPMI1640 / 10%, em seguida, adicionar 1,7 ml de formaldeído 37% para corrigir a cromatina diluído.
- Incubar à temperatura ambiente por 10 minutos, agitando suavemente, e depois apagar com 2,4 ml de glicina 2M. Centrifugar por 15 minutos a 1.200 rpm a 4 ° C, e remover o sobrenadante. Lave o pellet uma vez com 40 ml de gelo frio PBS, em seguida, girar o pellet e remover o PBS.
2. Lise celular para isolar núcleos
- Ressuspender as células em 40 ml de gelo tampão de lise gelada (10 mM Tris-HCl, pH8.0, 10 mM NaCl, 0,2% NP-40) com inibidores da protease recém adicionado (1:500 diluição) e 0,1 mM PMSF. Incubar em uma sala fria, com rotação de 90 minutos, centrifugar a 2.500 rpm por 15 minutos, e remover o sobrenadante.
3. Digestão com enzima de restrição Bgl II
- Ressuspender os núcleos em 0,5 ml de tampão NEB 1 x 3, e adicionar 15 μlof SDS 10%. Incubar a 37 ° C por 1 hora com agitação, em seguida, adicione 45 mL de 20% Triton X-100 para seqüestrar o SDS. Incubar a 37 ° C por 1 hora com agitação.
- Use uma alíquota de 1 x 10 6 núcleos (cerca de 15 mg, 1 / 10 das células original) para a digestão enzima de restrição. Retire 55 ml de solução núcleos da Etapa 3.1 e completar a 500 mL com 433 mL de um tampão NEB x 3 e 12 mL de Bgl II (50U/ul). Incubar a 37 ° C durante a noite.
4. Ligadura de segmentos de DNA interagindo
- Inativar a enzima de restrição, adicionando 95 mL de SDS 10%, e desnaturados por aquecimento a 65 ° C por 20 minutos em banho-maria.
- Adicionar 7 ml de um tampão ligadura x (30 mM Tris-HCl, pH 8,0, 10 mM MgCl 2, 10 mM DTT, 1 mM ATP) e 360 de mL Triton 20% X-100 e incubar a 37 ° C por 1 hora .
- Abaixe a temperatura para 16 ° C e adicionar 50 ul de 400 U / mL DNA ligase T4. Incubar a amostra a 16 ° C por 4 horas e, em seguida, em temperatura ambiente por 30 minutos.
5. Purificação de DNA
- Adicionar 300 mg de proteinase K, e incubar a 65 ° C durante a noite.
- Adicionar 5 mg de RNase A e incubar a 37 ° C por 30 minutos
- Purificar DNA por extração de fenol / clorofórmio, eo DNA precipitado em isopropanol. Dissolver o DNA em 150 mL de água destilada estéril.
6. Digestão com Msp I e ligadura com linkers oligonucleotídeo
- Incubar 2μg de DNA purificado com 5 unidades de Msp I, a 37 ° C por 4-6 horas. Inativar Msp I a 65 ° C por 10 minutos, em seguida, precipitar o DNA em etanol com 1 ml de glicogênio 5 mg / ml. Dissolver o sedimento de DNA em 50 ul de água destilada estéril.
- Mix 50 ul de DNA Msp I-tratados com 2 l de um oligonucleotídeo 20 L M linker (5'-gctgaccctgaattcgcacgtgcctgtcgttagcggacacagggcgattcac-3 '), 1 ml de um 20 mM oligonucleotídeo S (5'-cggtgaatc-3'), 1 ml de água destilada estéril e 6 mL de tampão ligase 10 x T4 DNA. Cubra a mistura com cera líquida.
- Oligonucleotides desnaturar a 50 ° C por 1 minuto e deixe arrefecer gradualmente para 10 ° C em um gradiente de 0,5 ° C / minuto em um termociclador.
- Adicionar 1 ml de 400 U / mL DNA ligase T4 e incubar a 15 ° C durante a noite. Purificar o DNA linker-ligado com um kit de purificação QIAquick PCR, e eluir em 50 l de água destilada estéril.
7. PCR amplificação e análise de seqüência de
- Escolha um primer para a região específica do DNA que você deseja examinar para interações de longo alcance (isto é, a 'isca'). Neste exemplo, usamos ABL-1.
- Tomar 1 ml do DNA purificado para realizar a primeira rodada PCR utilizando 1 ml de 20 mM de primer -1 ABL específica # 4656 (5'-gttcaagcgattctcctgcctcga-3 '), 1 ml de 20 mM de primer específico linker # 2963 (5'- gctgaccctgaattcgcacgtgcct-3 '), 3μl de 3 x Klen Taq DNA polimerase I cocktail e 3 l de água destilada estéril. Dois mL de 32 P-dCTP é adicionado 100 ul de 3 x DNA polimerase Taq I Klen cocktail antes da amplificação PCR. O ciclo térmico de um hot start PCR é de 72 ° C por 2 min, 95 ° Cpara 2 minutos, 18 ciclos de 95 ° C por 20 segundos, 65 ° C por 40 segundos e 72 ° C por 1 minuto; extensão da final é realizada a 72 ° Cpor 5 minutos.
- Purificar os primeiros produtos rodada PCR usando um kit de purificação QIAquick PCR e DNA eluída em 30 mL de água destilada estéril.
- Tomar 1 ml de 100 x diluída produto PCR primeiro a realizar uma segunda rodada de PCR usando primers nested adicionando 1 ml de 20 mM ABL-1 primer específico # 4626 (5'-ggagaatttttatctgcctctgtga-3 ') (Figura 1a), 1 ml de 20 mM de primer específico linker # 2961 (5'-gtcgttagcggacacagggcgattc-3 '), 3 mL de 3 x Klen Taq DNA polimerase I cocktail e 3 l de água destilada estéril. A programação de ciclos térmicos é de 25 ciclos de 95 ° C por 20 segundos, 67 ° C por 40 segundos e 72 ° C por 1 minuto, seguido por extensão a 72 ° C por 5 minutos.
- Visualize os produtos de PCR, executando a 5% gel uréia-PAGE e digitalização a tela exposta em um PhosphoImager (Figura 1b). Cada banda PCR pode ser reciclado a partir do gel, dissolvendo as tiras de gel em um tubo Eppendorf contendo 60 mL de água destilada estéril e incubar a 95 ° C por 5 minutos. Centrifugar brevemente a 10.000 rpm por 10 segundos para recolher todas as amostras. Retire 1 ml para usar como molde de DNA para realizar PCR com o par de iniciadores 2961/4626 usando as mesmas condições, como descrito acima. Análise da seqüência pode ser realizada depois da purificação usando um kit de purificação QIAquick PCR.
- Seqüências de DNA são analisadas utilizando uma ferramenta online para determinar a sua localização cromossômica no site: http://genome.ucsc.edu (Figura 1c). Clique em 'BLAT' para introduzir uma nova janela que permite colar uma seqüência de DNA, uma nova janela aparece após clicar em 'Enviar', e mostra o 'Resultados da Pesquisa BLAT. Clique em "browser" da batida com 100% de identidade para entrar numa janela ao lado mostra a localização da seqüência de DNA.
8. Resultados representante
1. ACT ensaio usando ABL-1 região como isca para determinar suas interações DNA de longo alcance
Como ilustrado na Figura 1a, dois sites Bgl II e um site que eu BAMH foram escolhidos para o ensaio ACT. Na segunda rodada de PCR, primer set 4626/2961 foi usado para amplificar ABL-M1, 4630/2961 foi usado para ABL-M2, e 4636/2961 foi usado para ABL-M3. Um padrão típico mostra um gel de várias bandas (Figura 1b). Cada fragmento de um ensaio ACT consiste em dois segmentos de DNA combinado: um segmento é derivada da região do DNA isca, enquanto o segundo segmento vem do parceiro associado, que se juntou ao segmento região isca pela primeira seqüência de reconhecimento enzima de restrição. A seqüência de reconhecimento segunda enzima irá aparecer no final da seqüência de parceiro associado (Figura 1c). O fragmento clonado ABL-M1 contém DNA da região de ABL1 localizado no cromossomo 9q32.4 de +133,592,306-133,592,399, e os parceiros associados está localizado no cromossomo 3p13 de +71,869,882-71,870,107. As identidades dos parceiros associados são descobertos por jateamento suas seqüências usando o browser genoma UCSC (GRCh7/hg19 lançado em fevereiro de 2009). PROK2 foi identificado como o parceiro associado no locus chr3p13.
Da mesma forma, a ABL-M2 parceiro associado foi localizada chr5q21.1, enquanto clone ABL-M3 foi identificado como uma associação intra-cromossômicas perto do lugar-1 ABL.
2. Determinar menos prevalente interações de longo alcance usando ensaio ACT
Outros ensaios com base em 3C relataram muitos mais parceiros do que a interação que temos mostrado na Figura 1 e na IGF2 / H19 lugar 12. A metodologia que temos delineado irá selecionar as mais prevalentes interações de longo alcance. No entanto, ao aumentar o número de ciclos de PCR, é possível identificar adicional, associações menos freqüentes, bem como (Figura 2).
3. Diferenças nas interações de longo alcance em células de câncer quando comparados com os tecidos normais.
O ensaio ACT também pode ser usado para identificar diferenças na arquitetura nuclear e interações de longo alcance entre células normais e células cancerosas (Figura 3). Estas diferenças podem refletir mudanças arquitetura nuclear que ocorrem durante a transformação celular, e, portanto, este ensaio pode vir a ser aplicáveis para fins de diagnóstico. Os padrões de gel semelhantes que ocorrem em ambos os normais e os tecidos de câncer indicou que o ensaio ACT é confiável e repetível. Enquanto o ensaio ACT pode identificar parceiros de longo alcance DNA, análises adicionais, tais como FISH e ensaios ChIP, são obrigados a verificar a presença das associações identificadas entre loci distantes. Estudos genéticos, fisiológicos e bioquímicos podem então ser realizados para elucidar as implicações biológicas dessas associações DNA de longo alcance.
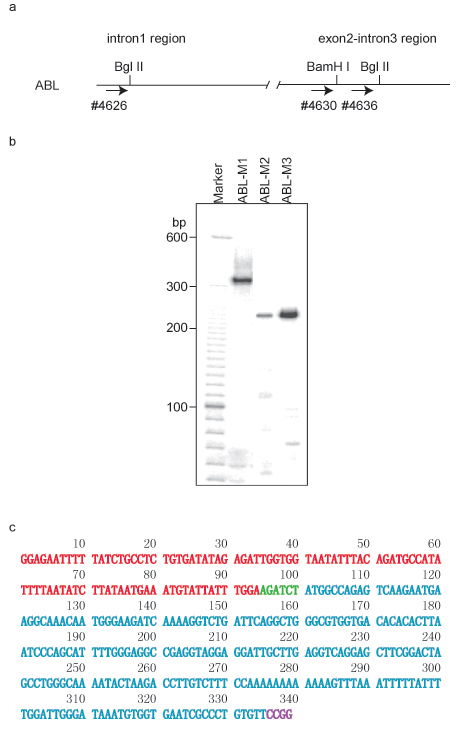
Figura 1 usando ensaio ACT ABL-1 DNA região como isca em células HL-60. a. Struct DNAure na ABL-1 região. A enzima de restrição usada pela primeira vez no ACT ensaio foi Bgl II. Os primers para a segunda rodada de PCR também estão marcados por setas e números primário correspondente. b. Gel padrão do ensaio ACT em 5% de uréia-PAGE. O par de iniciadores 4626/2961 foi usado para amplificar clone ABL-M1 na PCR nested, 4630/2961 foi usada para clonar ABL-M2, e 4636/2961 foi usada para clonar ABL-M3. c. Seqüência de DNA do clone ABL-M1. Fragmento de DNA a partir de ABL-1 DNA isca é marcado em vermelho, eo parceiro de DNA associado é rotulado em ciano. Bgl II site (AGACTC) foi marcado em verde, e Msp site que eu (CCGG) é marcado em roxo.

Figura 2 ciclos de PCR afetar os resultados do ensaio ACT. Usando a região de controle imprinting (ICR) no locus Igf2/H19 como isca DNA em células de fibroblastos de rato, os programas de ciclismo diferentes foram aplicadas no primeiro e segundo turnos da PCR no ensaio ACT. 18-20 ciclos no primeiro turno da PCR não amplificar o sinal suficiente para ser visualizado. Vinte e cinco ciclos nos resultados da segunda PCR em bandas claras, enquanto aumenta o número de ciclos para a segunda rodada de PCR induziu um padrão de manchas, além de bandas mais.

Figura 3 detecção de diferenças na arquitetura nuclear entre o tecido normal e tecido do cólon câncer de cólon em ensaio ACT. A. Estrutura do DNA da região ICR em IGF2/H19 locus e gene IGF2. Locais DPN II para o ensaio ACT são rotulados. Primers utilizados na PCR segunda rodada estão marcados por setas e números em cores diferentes que correspondem a cada pista no painel b da figura. b. Gel padrão do ensaio ACT em 5% de uréia-PAGE. Tecido do cólon normal, MAD03-1423 e tecido de câncer de cólon MAD04-149 foi obtida a partir da Rede Cooperativa de Tecidos Humanos (CHTN) Divisão Ocidental. Depois de cada tecido do cólon foi homogeneizado, ensaio ACT foi realizada seguindo os procedimentos aqui descritos. Faixa 1 representa os resultados da PCR usando primers # # 2961and 4161 (5'-tctgcgccatcagggcagtgagac-3 ') marcado em rosa em um painel; lane 2 representa os resultados da PCR usando primers # 2961 e # 4163 (5'-gccgcgcggccacttccgattcc-3 ') marcado em laranja no painel a; faixa 3 representa os resultados da PCR usando primers # 2961 e # 5145 (5'-gccatgcaggtaggatttgagctg-3') marcada em azul no painel a; lane 4 representa os resultados da PCR usando primers # 2961 e # 5151 (5'-gtctcaaataggggccagctagcttgg-3 ') marcado em verde no painel de a. Bandas originais que só aparecem no tecido do cólon normal são marcados por setas amarelas, e as bandas que apareceu somente no tecido de câncer de cólon são rotulados de setas vermelhas. ICR, região de controle imprinting; DMR, região metilado diferentes.
Discussão
Dekker et al. Desenvolveu o cromossomo conformação capturar abordagem (3C) para detectar a freqüência de interação entre dois loci genômicos 1 e 3C tem sido amplamente utilizado para investigar associações cromossômicas intra-e inter-cromossômicas entre duas regiões do DNA em células de mamíferos conhecidos dois -9. Embora recém-desenvolvida metodologia de Hi-C pode ser aplicado para o estudo do genoma de DNA associação, ensaio ACT ainda é uma técnica eficaz para o estudo do locus específico de interação DNA 10-11. Nós modificamos essa abordagem para identificar regiões do DNA desconhecidos que estão associados a uma região do DNA conhecido no rato cultivadas e células humanas (Figura 1). Chamamos este método na armadilha cromossomo associado ensaio (ACT), uma vez que nos forneceu um método confiável e reprodutível para identificar novos parceiros DNA desconhecidos que se associam a uma região conhecida de DNA alvo 12. Um ensaio bem sucedido 3C com controles adequados é feita antes de executar os aspectos novos do ensaio ACT 13. A fim tofind tantas regiões do DNA associado quanto possível, é necessário o uso de várias combinações de enzimas de restrição primeiro e segundo. É especialmente importante o uso de enzimas de restrição que são insensíveis a metilação CpG para realizar a etapa ligadura primeiro 3C. A ligação às proteínas e metilação do DNA também podem influenciar a eficiência restrição de digestão enzimática e pode levar à insuficiência de ligadura das regiões DNA associado para digestões certas enzimas de restrição. A ocorrência de ligadura intra ou inter-cromossômicas depende de proteínas DNA-cross-linking e apropriado mapas físicos de ambos das regiões DNA associado. Assim, alguns experimentos preliminares são essenciais para estabelecer uma possível concentração de formaldeído eficaz e tempo de tratamento no ensaio ACT. A gama de concentrações finais de formaldeído (from1.5% para 2%) têm sido utilizados em diversos laboratórios durante a parte 3C do ensaio 7,9. Alternativamente, os oligonucleotídeos utilizados como linkers pode ser projetado para combinar com o final coesa cortado pela enzima de restrição segundo. Embora descobrimos que 18-20 ciclos na primeira rodada de PCR e 20-25 ciclos no segundo turno da PCR poderia fornecer bandas claras, é necessário estabelecer as melhores condições de PCR para cada experimento (Figura 2). Intra-molécula annealing entre seqüência adaptador 5'-end e 3'-end complementares de uma cadeia de DNA pode ocorrer em PCR, inibe específicas do adaptador primário de recozimento com a molécula de DNA, e resultou em eficiência de amplificação muito mais baixo nos primeiros ciclos diversos. Após o DNA alvo foi ampliado para os ciclos, o montante pode ser muito maior do que estas reações inespecíficas, e pode facilitar a competição de primer annealing para as moléculas de DNA alvo. É também por isso nós podemos ver a amplificação de fundo, e porque o primeiro produto da PCR rodada tem que ser diluído para diminuir amplification.It fundo é importante para remover excesso de primers a partir da reação de PCR, a fim de diminuir a fundo na segunda rodada de PCR. Como em todos os experimentos baseados em PCR, é essencial para projetar primers que não estão localizados em regiões de repetição de seqüência, que constituem a maioria do DNA humano e do rato. Embora recém-desenvolvida metodologia de Hi-C pode ser aplicado para o estudo do genoma de DNA associação, ensaio ACT ainda é uma técnica eficaz para o estudo do locus específico de interação DNA.
Divulgações
Agradecimentos
Nós graças Adelle Murell e Wolf Reik muito para compartilhar seu protocolo 3C. Este trabalho foi financiado pelo Departamento de Defesa eo Serviço de Pesquisa do Departamento de Assuntos de Veteranos.
Materiais
| Name | Company | Catalog Number | Comments |
| RPMI1640 medium | Invitrogen | 22400-105 | |
| acrylamide | Invitrogen | 15512-023 | |
| ATP solution, 10mM | Invitrogen | AM8110G | |
| fetal bovine serum | Invitrogen | 16000-044 | |
| penicillin-streptomycin | Invitrogen | 15140-122 | |
| 1M Tris pH8.0 | Invitrogen | AM9856 | |
| RNase A | Invitrogen | 12091-039 | |
| SDS | Invitrogen | 15525-017 | |
| urea | Invitrogen | 15505-035 | |
| BamH I | New England Biolabs | R0136T | |
| Bgl II | New England Biolabs | R0144M | |
| Dpn II | New England Biolabs | R0543T | |
| Msp I | New England Biolabs | R0106S | |
| dNTPs | New England Biolabs | N0447L | |
| proteinase K | New England Biolabs | P8102S | |
| T4 DNA ligase | New England Biolabs | M0202T | |
| 37% formaldehyde | Sigma-Aldrich | F8775 | |
| Bis-acrylamide | Sigma-Aldrich | 146072 | |
| dithiothreitol | Sigma-Aldrich | 43815 | |
| glycine | Sigma-Aldrich | 50046 | |
| PMSF | Sigma-Aldrich | 93482 | |
| proteinase inhibitor | Sigma-Aldrich | S8830 | |
| Nonidet P-40 | Roche Group | 11754599001 | |
| KlenTaq1 | Ab peptides | 1001 | |
| dCTP alpha P32 | PerkinElmer, Inc. | BLU513H250UC | |
| PTC-100 Thermal Cycler | MJ Research | mjptc100 | |
| Power Supply | Bio-Rad | 164-5056 | |
| OmniPAGE Maxi | Aurogene Life Science | VS20D | |
| Typhoon 9400 | GE Healthcare | 63-0055-78 |
Referências
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295, 1306-1311 (2002).
- Apostolou, E., Thanos, D. Virus Infection Induces NF-kappaB-dependent interchromosomal associations mediating monoallelic IFN-beta gene expression. Cell. 134, 85-96 (2008).
- Duan, Z. A three-dimensional model of the yeast genome. Nature. 465, 363-367 (2010).
- Lomvardas, S. Interchromosomal interactions and olfactory receptor choice. Cell. 126, 403-413 (2006).
- Murrell, A., Heeson, S., Reik, W. Interaction between differentially methylated regions partitions the imprinted genes Igf2 and H19 into parent-specific chromatin loops. Nat Genet. 36, 889-893 (2004).
- Schoenfelder, S. Preferential associations between co-regulated genes reveal a transcriptional interactome in erythroid cells. Nat Genet. 42, 53-61 (2010).
- Spilianakis, C. G., Lalioti, M. D., Town, T., Lee, G. R., Flavell, R. A. Interchromosomal associations between alternatively expressed loci. Nature. 435, 637-645 (2005).
- Vakoc, C. R. Proximity among distant regulatory elements at the beta-globin locus requires GATA-1 and FOG-1. Mol Cell. 17, 453-462 (2005).
- Xu, N., Tsai, C. L., Lee, J. T. Transient homologous chromosome pairing marks the onset of X inactivation. Science. 311, 1149-1152 (2006).
- Lieberman-Aiden, E. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Berkum, N. L. v. a. n. Hi-C: a method to study the three-dimensional architecture of genomes. J Vis Exp. , (2010).
- Ling, J. Q. CTCF mediates interchromosomal colocalization between Igf2/H19 and Wsb1/Nf1. Science. 312, 269-272 (2006).
- Dekker, J. The three 'C's of chromosome conformation capture: controls, controls, controls. Nat Methods. 3, 17-21 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados