É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Ensaio para imunoprecipitação da cromatina Genes Tissue-específicos usando embriões em início de carreira do rato
Neste Artigo
Resumo
Nós demonstramos uma imunoprecipitação da cromatina (CHIP) para identificar as interações fator em tecido-específicos genes durante ou após o início da tecido-específicos de expressão gênica em camundongos tecido embrionário. Este protocolo deve ser amplamente aplicáveis para o estudo do tecido-específicos ativação do gene como ocorre durante o desenvolvimento embrionário normal.
Resumo
Cromatina imunoprecipitação (chip) é uma ferramenta poderosa para identificar proteínas: interações cromatina que ocorrem no contexto de células vivas 1-3. Esta técnica tem sido amplamente explorada em células de cultura de tecidos, e, em menor grau, no tecido primário. A aplicação do chip para o tecido embrionário de roedores, sobretudo em momentos iniciais de desenvolvimento, é complicado pela quantidade limitada de tecido e da heterogeneidade das células e tecidos no embrião. Aqui apresentamos um método para realizar chip usando um dia dissociada embrionárias 8.5 (E8.5) do embrião. Cromatina tosquiada de um embrião E8.5 única pode ser dividido em até cinco alíquotas, que permite que o investigador material suficiente para controles e para a investigação de proteínas específicas: interações cromatina.
Temos utilizado esta técnica para começar a documentar proteína: interações cromatina durante a especificação de tecido programas específicos de expressão gênica. A heterogeneidade de tipos de células em um embrião, necessariamente, restringe a aplicação desta técnica porque o resultado é a detecção de proteínas: interações cromatina sem distinguir se as interações ocorrem em todos, um subconjunto de, ou um único tipo de células (s). No entanto, o exame de tecido específico genes durante ou após o aparecimento de tecido-específicos de expressão gênica é viável por dois motivos. Primeiro, imunoprecipitação de fatores tecido específico necessariamente isola cromatina do tipo de célula onde o fator é expresso. Segundo, imunoprecipitação de coativadores e histonas contendo modificações pós-translacionais que estão associados com a ativação do gene só deve ser encontrada em genes e seqüências regulatórias no tipo de células onde o gene está sendo ou foi ativado. A técnica deve ser aplicado ao estudo da maioria dos tecidos específicos eventos de ativação do gene.
No exemplo descrito abaixo, utilizamos E8.5 e E9.5 embriões de camundongos para analisar fator determinante em um esqueleto promotor do gene do músculo específico. Somitos, que são os tecidos precursor a partir do qual os músculos esqueléticos do tronco e membros formarão, estão presentes em E8.5-9.5 4,5. Miogenina é um fator de regulação necessários para a diferenciação do músculo esquelético 6-9. Os dados demonstram que miogenina está associado ao seu próprio promotor em E8.5 e E9.5 embriões. Porque miogenina é expresso apenas em somitos nesta fase de desenvolvimento 6,10, os dados indicam que as interações com miogenina seu promotor própria já ocorreu em células do músculo esquelético precursor em E8.5 embriões.
Protocolo
1. Isolamento de embriões
Nota: Todas as operações envolvendo ratos deve ser realizada de acordo com o cuidado de animais e políticas adequadas de uso e protocolos
- Verificar a presença de um plugue de acasalamento em camundongos fêmeas da manhã depois do acasalamento e as fêmeas separadas dos machos acasalaram parafuso, colocando-os em uma gaiola diferente. Meio-dia do dia que a ficha de acasalamento se observa é considerado o dia embrionárias 0,5 (E0.5) do desenvolvimento.
- Em E8.5, (ou o estágio desejado, se for diferente), o sacrifício do mouse usando um protocolo aprovado.
- Molhar a barriga do animal sacrificado com etanol 70% e abrir a cavidade abdominal. Os locais de implantação, indicando a presença de embriões em desenvolvimento são vistos como uma "contas em um colar" arranjo ao longo do comprimento de cada corno uterino (Figura 1A). Com uma tesoura, retire os dois cornos uterinos, corte cada local de implantação para separar individualmente (Figura 1B) e coloque-os em um prato de 100 milímetros de petri contendo temperatura ambiente (RT), médio dissecção (DMEM, 10% de soro fetal bovino, 20 mM HEPES pH 7,4 , 60 mg / ml de penicilina, estreptomicina 2 mM).
- Utilizando uma pinça, remova cada embrião e as membranas que envolvem desde o tecido uterino (Figura 1C) e colocá-los em meio dissecção fresco.
- Isolar embriões individuais sob um microscópio de dissecação com a pinça. Neste estágio de desenvolvimento, os embriões são rodeados por parietal saco vitelino, que em contato com o tecido deciduum, o saco vitelino visceral eo âmnio. O âmnio é uma membrana transparente que está em contato direto com o embrião. O saco vitelino visceral está localizado entre o âmnio eo saco vitelino parietal e é facilmente distinguido em E8.5-E9.5 embriões pela presença de vasos sanguíneos que nutrem importantes do embrião. Retire cada embrião de sua membranas que envolvem e transferência individualmente em um novo prato contendo media dissecção para remover o excesso de sangue. O estágio de desenvolvimento pode variar de embrião para embrião e entre ninhadas; representante E8.5 embriões e um representante do embrião E9.5 são mostrados na Figura 1D.
- Transferência de cada embrião para um tubo eppendorf 1,5 ml contendo 200 mL de mídia dissecção (descrito na etapa 4 acima).
2. Homogeneização da Embryo isolados
- Adicionar 20 unidades de colagenase tipo II em um volume de 200 ul (diluído em fosfato 1X Dubbecco de Buffered Saline; DPBS) a cada tubo eppendorf.
- Agite suavemente amostras em um shaker 37 ° C a 100 rpm por 20 min.
- Ressuspender bem pipetando para perturbar aglomerados de células.
- Aplicar a suspensão de células na parte superior do filtro de células estéreis (40 malhagem mm) colocado sobre um tubo eppendorf 1,5 ml.
- Aplicar imediatamente 600 mL de temperatura ambiente 1X DPBS em cima do filtro de celular para completar a separação. Descartar o filtro.
- Centrifugar as amostras a 4 ° C a 4000 xg por 5 min.
- Desprezar o sobrenadante.
- Ressuspender o sedimento em 1 ml de DPBS RT 1X.
- Centrifugar as amostras a 4000 xg por 1 min em temperatura ambiente.
- Desprezar o sobrenadante.
- Ressuspender o sedimento em 200 mL de mídia dissecção RT.
- Contar as células embrionárias. Há um embrião 3-5 x 10 6 cells/E8.5 média.
3. Cross-link Chromatin
- Adicionar 5,6 mL de formaldeído 37% para as 200 amostras mL para uma concentração final de formaldeído a 1%.
- Incubar as amostras por 10 min em temperatura ambiente.
- Centrifugar as amostras a 4 ° C a 4000 xg por 3 min.
- Desprezar o sobrenadante.
- Ressuspender as células com frio 1X DPBS contendo 80 mL / cocktail inibidor da protease ml (PIC).
- Centrifugar as amostras a 4 ° C a 4000 xg por 3 min.
- Desprezar o sobrenadante. Nesta etapa, as amostras podem ser congeladas a -80 ° C. Os congelados, amostras reticuladas são estáveis por vários meses.
4. Sonicação
- Ressuspender o pellet celular em 100 ul de tampão SDS RT lise (50 mM Tris-HCl (pH 8,1), EDTA 10 mM e SDS 1% com PIC recém adicionado ao volume de 1 ml PIC/800 mL total). Quando ressuspenso, adicionar um adicional de 100 ul de tampão de lise RT SDS para promover a ressuspensão. Misture bem por pipetagem e inversão.
- Incubar a suspensão de células no gelo por 10 min.
- Sonicate células lisadas de DNA de cisalhamento para entre 200 e 500 pares de bases usando um Bioruptor Diagenode ™ UCD200 (5 min x 8 vezes: definição de amplitude de 30 seg on/30 sec fora do poder (320 watts) de altura). As amostras devem ser cercado por uma pasta de gelo. Não permita que as amostras para aquecer acima de 4 ° C ou congelar. Existem muitos tipos de sonicators disponíveis. Sonicação condições devem ser otimizados para cada sonicador resultar em ruptura do DNA cruzada para um comprimento de aproximadamente 500 pb.
- Centrifugar amostras sonicado a 4 ° C a 14000 xg por 10 min.
- TRANSFERÊNCIA o sobrenadante para um tubo eppendorf frescos pré-resfriado em gelo.
- Dividir a amostra sonicado em um máximo de cinco alíquotas. Temos empiricamente determinou que um embrião E8.5 pode ser dividido em cinco alíquotas. Tamanhos de amostra menores ou maiores podem ser dimensionados de acordo. Nesta etapa, a amostra pode ser armazenada a -80 ° C até à sua utilização.
- Reserve uma alíquota para uso como entrada e armazenar a -20 ° C até a etapa 6, quando o cross-links serão revertidas. Diluir as alíquotas outras 10 vezes em tampão de diluição ChIP (16,7 mM Tris-HCl (pH 8,1), 1,2 mM EDTA, 1,1% Triton X-100, 167 mM NaCl e SDS 0,01%), contendo PIC recém-added at 1 μl/800 mL de buffer.
5. Pré-compensação e imunoprecipitação
- Pré-claro sobrenadante diluído em 75 mL de esperma de salmão-DNA / proteína A ou G-Agarose suspensão de 50% a 4 ° C, com rotação, por 1 hora. Uso de qualquer proteína A ou G grânulos de proteína deve ser determinada pelo anticorpo a ser utilizado para o chip. Por exemplo, a proteína A contas são recomendados para anticorpos de coelho, enquanto esferas de proteína G são recomendados para anticorpos IgG de cabra ou um rato anticorpos. Para informações mais detalhadas, veja as recomendações feitas pelo fabricante do grânulo.
- Centrífuga a 4 ° C a 2500 xg por 1 min.
- Transferir o sobrenadante para um novo tubo eppendorf pré-resfriada.
- Adicionar anticorpo (4 mg / amostra) à alíquota de pré-limpo e incubar overnight a 4 ° C com rotação.
6. Lavar o Complexo de proteína-cromatina-bead
- Adicionar 60 mL de esperma de salmão DNA / proteína A ou G agarose suspensão em cada frasco e incubar a 4 ° C com rotação de 1 hr. Use o mesmo tipo talão utilizado para pré-limpeza no passo 3 acima.
- Centrífuga a 4 ° C a 1000 xg por 1 min.
- Remova cuidadosamente o sobrenadante e descartar. Manter o complexo DNA-proteína-grânulo no gelo.
- Ressuspender e lavar o pellet, conforme indicado abaixo com buffers de lavagem 1, 2, 3 e 4. Lavar buffers 03/01 devem ser refrigerados a 4 ° C e lavagens com esses buffers deve ser realizada a 4 ° C. Tampão de lavagem 4 deve estar em temperatura ambiente e lavagens com esse buffer deve ser realizada à temperatura ambiente. Cada lavagem é de 5 minutos com rotação à temperatura adequada. Após cada lavagem centrífuga, a 4 ° C (temperatura ambiente por 4 lavagens tampão de lavagem) a 1000 xg por 1 min, em seguida, aspirar cuidadosamente ou remover o sobrenadante.
Tampão 1: tampão de lavagem Low sal (20 mM Tris-HCl (pH 8,1), 2 mM EDTA, 1% Triton X-100,
150 mM NaCl e SDS 0,1%), 1 ml x 2 lavagens.
Buffer de 2: tampão de lavagem de alta sal (20 mM Tris-HCl (pH 8,1), 2 mM EDTA, 1% Triton X-100,
500 mM NaCl e SDS 0,1%), 1 ml x 2 lavagens.
Tampão 3: LiCl tampão de lavagem de sal (10 mM Tris-HCl (pH 8,1), 1 mM EDTA, 1% IGEPAL-CA630,
0,25 M LiCl e ácido deoxicólico 1% (sal sódico)), 1 x 1 ml de lavagem.
Tampão 4: tampão TE (10 mM Tris-HCl (pH 8,1), 1 mM EDTA), 1 ml x 2 lavagens.
7. Eluição do Complexo Chromatin-anticorpo
- Ressuspender o complexa proteína-cromatina-bead lavados em 250 L de tampão de eluição recém-preparada (1% SDS, 0,1 M NaHCO 3). Vortex vigorosamente a amostra por 5 segundos e, em seguida incubar a RT por 15 min com rotação.
- Centrifugar a 2.500 xg por 1 min em temperatura ambiente e transferir o eluato em um tubo eppendorf novo.
- Repita as etapas 1 e 2 e combinar os eluatos no tubo eppendorf mesmo. Os eluatos deve ser mantido em temperatura ambiente. Após a conclusão desta etapa, os eluatos podem ser armazenadas a -20 ° C até à sua utilização.
8. Reverso Cross-link e recuperar DNA
- Adicionar 20 l 5 M NaCl por 500 mL eluato (40 mL / ml para o controle de entrada).
- Aqueça o eluatos a 65 ° C em banho-maria por 4 horas durante a noite.
- 10 ml de cada amostra (incluindo o controle de entrada) podem ser executados em um gel de agarose 2% ao lado de marcadores de tamanho medindo 0,1-1 kbp para verificar o tamanho do fragmento de DNA. O DNA vai aparecer como uma mancha, e não uma banda afiada. O restante das amostras podem ser deixados a 65 ° C, enquanto o gel está sendo executado.
- Recuperar o DNA de cada amostra usando um QIAquick Gel Extraction Kit (Qiagen). Adicionar 600 mL tampão QG (fornecido no kit) para otimizar o DNA de adsorção com a membrana de sílica em uma coluna QIAquick e 200 mL de isopropanol em uma amostra mL 500. Note-se que tampão QG contém um indicador de pH permitindo a determinação fácil do pH da amostra. Se a cor da amostra passa de amarelo para roxo (pH> 7,5) após a mistura, adicione 10 ml de 3 NaAcetate M (pH 5,2) para a amostra e misturar. Isso reduz o pH da mistura para maximizar o DNA de ligação para as contas na coluna.
- Antes de carregar a coluna, verifique a mistura para determinar se qualquer precipitado SDS está presente. Se SDS precipitação ocorreu, a amostra deve ser aquecida em um42 ° C banho-maria até que o precipitado desaparece.
- Carga 700 mL da mistura na coluna QIAquick (coluna roxo no kit) e centrifugar a 14.000 xg na RT para 1 min. Descartar o fluxo através da coluna antes de continuar e repetir este passo com o restante da amostra.
- Adicionar 500 mL tampão QG para lavar a coluna. Centrifugar a 14.000 xg na RT por 2 min. Descartar o fluxo através da coluna antes de prosseguir.
- Lavar a coluna com 700 mL de tampão de lavagem PE (fornecido no kit) para que o etanol foi adicionado conforme as instruções do fabricante. Centrifugar a 14.000 xg na RT para 1 min. Descartar o fluxo através da coluna antes de prosseguir.
- Repita a etapa de centrifugação para remover completamente o restante do tampão PE na coluna. Descartar o fluxo através do tubo e coleção.
- Eluir o DNA da coluna em um tubo eppendorf fresco, adicionando 60 mL tampão EB (tampão de eluição fornecido no kit).
- Centrifugar a temperatura ambiente em 14.000 xg por 1 min. O DNA eluído podem ser armazenadas a -20 ° C até a detecção. A 260 leituras do material recuperado normalmente indicam concentrações de 90-120 ng / ul, para uma recuperação total de ~ 6-7 ug por alíquota. No entanto, a proporção de 260/280 Um desses exemplos é tipicamente> 1,8, sugerindo que a RNA está presente na amostra. Assim, a quantidade exata de DNA recuperado pode ser menor.
9. Análise de DNA recuperado
- SDS pode interferir com a actividade da PCR Taq polimerase. SDS deve ser completamente removido antes de realizar PCR. Incubar a DNA em gelo para precipitar qualquer SDS residual e centrifugar a 4 ° C em 14.000 xg por 1 min. Transfira o sobrenadante para um novo passo tube.This eppendorf é fortemente recomendado.
- Os eluatos DNA podem ser detectadas quer por PCR convencional ou pela quantitativa PCR em tempo real (Q-PCR) com primers específicos para cada seqüência a ser analisado. ML de eluato 50-10 DNA total pode ser utilizado para um PCR ou Q-PCR reação. PCR com o controle de entrada pode ser realizada com uma diluição de 50-10 vezes do DNA em comparação com a quantidade de outras amostras. PCR e Q-PCR condições podem variar, no entanto, a quantidade limitada de matéria-prima exige ciclos adicionais PCR. Para a detecção de PCR convencional, recomendamos que comece com 40 ciclos e ajustar conforme necessário.
10. Resultados representante
Nós temos usado esse protocolo para realizar ChIP de ambos E8.5 E9.5 e embriões (Figura 2). Os resultados demonstram que miogenina está presente no promotor miogenina em E8.5 e E9.5 embriões. DNAs ChIP purificados foram analisados por PCR convencional (Figura 2A) e pelo quantitativo real-time PCR (Figura 2B). Em contraste, não houve indicação de miogenina ligação ao promotor miogenina no saco vitelino, onde miogenina não é expresso. O interferon-γ (IFNγ) promotor, que contém seqüências correspondentes do site miogenina vinculativo, foi usado como um controle de seqüência negativa. Como esperado, miogenina não estava preso ao promotor IFNγ em qualquer uma das amostras de tecido testado. Em E8.5 e 9,5 embriões, miogenina é especificamente expresso na somitos (Figura 3; 6,10), que são as precursoras de músculo esquelético. Assim, os resultados indicam que a miogenina é vinculado ao promotor miogenina na somitos E8.5 e E9.5.
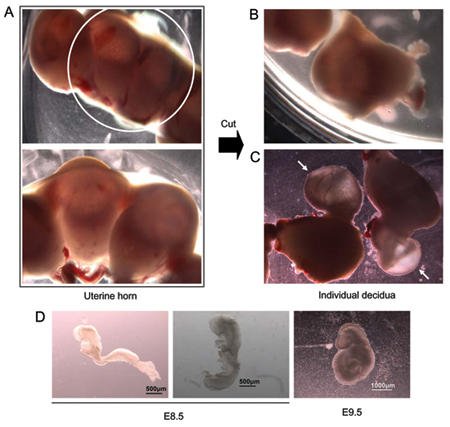
Figura 1. E8.5 dissecção embrião. (A) chifre Isolated uterina (painel superior; maior ampliação mostrado no painel inferior). (B) uterina chifre cortado com tesoura para separar locais de implantação individuais contendo embriões individuais. (C) Embriões projetando-se de tecido uterino durante a dissecção. Setas marcam os embriões ainda cobertos por extra-embrionária do tecido. (D) Dois E8.5 embriões de mesma ninhada. O embrião do lado esquerdo não começou o processo de transformação. O embrião do lado direito está passando por torneamento. Extrema-direita - embrião E9.5 representante.
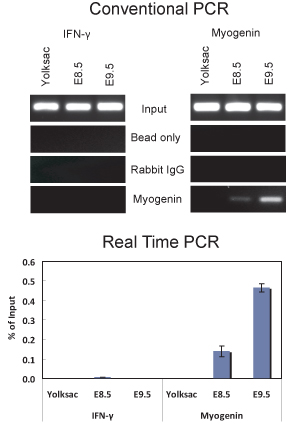
Figura 2. ChIP ensaio demonstrando miogenina ligação ao promotor miogenina em E8.5 e E9.5 embriões. (Top) análise de PCR convencional de 5 ul de DNA purificada a partir de experimentos chip usando um anticorpo miogenina ou não-específicas IgG foi realizada com primers que amplificam uma porção do promotor miogenina de -79 a 69 em relação ao local de início da transcrição que contém um sítio de ligação localizado no miogenina -12 ou primers que amplificam uma porção do promotor IFNγ que contém uma seqüência de combinação do site miogenina ligação localizada ~ 1075 pb a montante do local de início da transcrição. As seqüências de primers utilizadas foram IFNγ 5'-GCT GAC TCA GGC AGA CCC CGA-3 'e 5'-GGA TGG TGA GGC AGG AGG CC-3'. (Inferior) quantitativa em tempo real a análise PCR das mesmas amostras usadas em (A). Oos dados são plotados como% de entrada + / - desvio padrão.
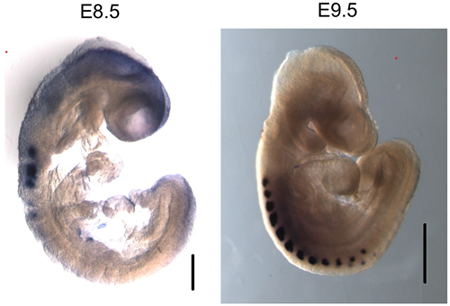
Figura 3. Miogenina é especificamente expresso na somitos de E8.5 E9.5 e embriões. Montar toda a hibridização in situ de miogenina mostra a expressão do mRNA específico no somitos. Bar tamanho em E8.5 imagem - 200 mM. Bar tamanho em E9.5 imagem - 500 mm.
Access restricted. Please log in or start a trial to view this content.
Discussão
No protocolo ChIP descrito, mostramos que a miogenina regulador miogênico está associada com o promotor miogenina no tecido músculo esquelético precursor presente em E8.5 E9.5 única e embriões. Estudos anteriores extensamente caracterizada miogenina ligação a caixa de E contendo seqüências, começando com a inicial em experimentos in vitro, utilizando gel shift in vitro traduzido ou bactérias produzidas DNA miogenina e radiolabeled codificação a parte relevante de seqüências-alv...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Agradecimentos
Este trabalho foi financiado pelo NIH R01 GM56244 a ANI, que inclui fundos concedidos através da Lei de Recuperação e Reinvestimento de 2009, e pelo NIH R01 GM87130 para JARP
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| ChIP Assay Kit | Upstate, Millipore | 17-295 | |
| Collagenase Type II | Invitrogen | 17101015 | Dilution by 1 x PBS |
| Dulbecco’s modified eagle medium (DMEM) | GIBCO, by Life Technologies | 12100-061 | High glucose content |
| Dulbecco’s phosphate buffered saline 1X (DPBS) | GIBCO, by Life Technologies | 14190-144 | Calcium chloride free, Magnesium chloride free |
| Fetal bovine serum (FBS) | Mediatech, Inc. | 35-010-CV | |
| Gel extraction kit | QIAquick | 28704 | 50 reaction kit |
| Penicillin/streptomycin stock solution | GIBCO, by Life Technologies | 5000 μg/ml concentration | |
| Protease Inhibitor Cocktail | Sigma-Aldrich | P8340 | |
| Salmon sperm DNA /Protein A agarose | EMD Millipore | 16-157 | |
| myogenin antibody | Santa Cruz Biotechnology, Inc. | sc-576 | |
| Normal rabbit IgG | EMD Millipore | 12-370 | |
| Platinum PCR Supermix | Invitrogen | 11306-016 | |
| GoTaq Q-PCR master mix | Promega Corp. | A6001 |
Referências
- Minard, M. E., Jain, A. K., Barton, M. C. Analysis of epigenetic alterations to chromatin during development. Genesis. 47, 559-572 (2009).
- Kuo, M. H., Allis, C. D. In vivo cross-linking and immunoprecipitation for studying dynamic Protein:DNA associations in a chromatin environment. Methods. 19, 425-433 (1999).
- Johnson, K. D., Bresnick, E. H. Dissecting long-range transcriptional mechanisms by chromatin immunoprecipitation. Methods. 26, 27-36 (2002).
- Yusuf, F., Brand-Saberi, B. The eventful somite: patterning, fate determination and cell division in the somite. Anat Embryol (Berl). 211, Suppl 1. 21-30 (2006).
- Buckingham, M., Bajard, L., Chang, T., Daubas, P., Hadchouel, J., Meilhac, S., Montarras, D., Rocancourt, D., Relaix, F. The formation of skeletal muscle: from somite to limb. J Anat. 202, 59-68 (2003).
- Wright, W. E., Sassoon, D. A., Lin, V. K. Myogenin, a factor regulating myogenesis, has a domain homologous to MyoD. Cell. 56, 607-617 (1989).
- Edmondson, D. G., Olson, E. N. A gene with homology to the myc similarity region of MyoD1 is expressed during myogenesis and is sufficient to activate the muscle differentiation program. Genes Dev. 3, 628-640 (1989).
- Nabeshima, Y., Hanaoka, K., Hayasaka, M., Esumi, E., Li, S., Nonaka, I. Myogenin gene disruption results in perinatal lethality because of severe muscle defect. Nature. 364, 532-535 (1993).
- Hasty, P., Bradley, A., Morris, J. H., Edmondson, D. G., Venuti, J. M., Olson, E. N., Klein, W. H. Muscle deficiency and neonatal death in mice with a targeted mutation in the myogenin gene. Nature. 364, 501-506 (1993).
- Sassoon, D., Lyons, G., Wright, W. E., Lin, V., Lassar, A., Weintraub, H., Buckingham, M. Expression of two myogenic regulatory factors myogenin and MyoD1 during mouse embryogenesis. Nature. 341, 303-307 (1989).
- Brennan, T. J., Olson, E. N. Myogenin resides in the nucleus and acquires high affinity for a conserved enhancer element on heterodimerization. Genes Dev. 4, 582-595 (1990).
- Rosenthal, N., Berglund, E. B., Wentworth, B. M., Donoghue, M., Winter, B., Bober, E., Braun, T., Arnold, H. H. A highly conserved enhancer downstream of the human MLC1/3 locus is a target for multiple myogenic determination factors. Nucleic Acids Res. 18, 6239-6246 (1990).
- Braun, T., Gearing, K., Wright, W. E., Arnold, H. H. Baculovirus-expressed myogenic determination factors require E12 complex formation for binding to the myosin-light-chain enhancer. Eur J Biochem. 198, 187-193 (1991).
- Chakraborty, T., Brennan, T., Olson, E. Differential trans-activation of a muscle-specific enhancer by myogenic helix-loop-helix proteins is separable from DNA binding. J Biol Chem. 266, 2878-2882 (1991).
- French, B. A., Chow, K. L., Olson, E. N., Schwartz, R. J. Heterodimers of myogenic helix-loop-helix regulatory factors and E12 bind a complex element governing myogenic induction of the avian cardiac alpha-actin promoter. Mol Cell Biol. 11, 2439-2450 (1991).
- Brennan, T. J., Chakraborty, T., Olson, E. N. Mutagenesis of the myogenin basic region identifies an ancient protein motif critical for activation of myogenesis. Proc Natl Acad Sci U S A. 88, 5675-5679 (1991).
- Lassar, A. B., Davis, R. L., Wright, W. E., Kadesch, T., Murre, C., Voronova, A., Baltimore, D., Weintraub, H. Functional activity of myogenic HLH proteins requires hetero-oligomerization with E12/E47-like proteins in vivo. Cell. 66, 305-315 (1991).
- Chakraborty, T., Brennan, T. J., Li, L., Edmondson, D., Olson, E. N. Inefficient homooligomerization contributes to the dependence of myogenin on E2A products for efficient DNA binding. Mol Cell Biol. 11, 3633-3641 (1991).
- Cserjesi, P., Olson, E. N. Myogenin induces the myocyte-specific enhancer binding factor MEF-2 independently of other muscle-specific gene products. Mol Cell Biol. 11, 4854-4862 (1991).
- Braun, T., Arnold, H. H. The four human muscle regulatory helix-loop-helix proteins Myf3-Myf6 exhibit similar hetero-dimerization and DNA binding properties. Nucleic Acids Res. 19, 5645-5651 (1991).
- Serna, dela, L, I., Ohkawa, Y., Berkes, C. A., Bergstrom, D. A., Dacwag, C. S., Tapscott, S. J., Imbalzano, A. N. MyoD targets chromatin remodeling complexes to the myogenin locus prior to forming a stable DNA-bound complex. Mol Cell Biol. 25, 3997-4009 (2005).
- Blais, A., Tsikitis, M., Acosta-Alvear, D., Sharan, R., Kluger, Y., Dynlacht, B. D. An initial blueprint for myogenic differentiation. Genes Dev. 19, 553-569 (2005).
- Cao, Y., Kumar, R. M., Penn, B. H., Berkes, C. A., Kooperberg, C., Boyer, L. A., Young, R. A., Tapscott, S. J. Global and gene-specific analyses show distinct roles for Myod and Myog at a common set of promoters. EMBO J. 25, 502-511 (2006).
- Ohkawa, Y., Yoshimura, S., Higashi, C., Marfella, C. G., Dacwag, C. S., Tachibana, T., Imbalzano, A. N. Myogenin and the SWI/SNF ATPase Brg1 maintain myogenic gene expression at different stages of skeletal myogenesis. J Biol Chem. 282, 6564-6570 (2007).
- Davie, J. K., Cho, J. H., Meadows, E., Flynn, J. M., Knapp, J. R., Klein, W. H. Target gene selectivity of the myogenic basic helix-loop-helix transcription factor myogenin in embryonic muscle. Dev Biol. 311, 650-664 (2007).
- Metivier, R., Penot, G., Hubner, M. R., Reid, G., Brand, H., Kos, M., Gannon, F. Estrogen receptor-alpha directs ordered, cyclical, and combinatorial recruitment of cofactors on a natural target promoter. Cell. 115, 751-763 (2003).
- Ausubel, F. M., Brent, R., Kingston, R. E., Moore, D. D., Seidman, J. G., Smith, J. A., Struhl, K. Current Protocols in Molecular Biology. , John Wiley & Sons, Inc. (2010).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados