É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
ampliPHOX Detecção colorimétrica em um Microarray de DNA para Influenza
Neste Artigo
Resumo
ampliPHOX tecnologia de detecção colorimétrica é apresentado como uma alternativa barata para detecção de fluorescência para microarrays. Com base na fotopolimerização, ampliPHOX produz spots polímero sólido visível a olho nu em apenas alguns minutos. Resultados são então fotografada e automaticamente interpretados com um pacote de software simples, mas poderosa.
Resumo
DNA microarrays têm emergido como uma poderosa ferramenta para a detecção de patógenos. 1-5 Por exemplo, muitos exemplos da capacidade de tipo e subtipo do vírus influenza tem sido demonstrado. 6-11 A identificação e subtipos de gripe em microarrays de DNA tem aplicações tanto em público saúde e da clínica para a detecção precoce, intervenção rápida, e minimizar o impacto de uma pandemia de influenza. Fluorescência tradicional é atualmente o método de detecção mais comumente utilizados microarray. No entanto, como tecnologia de microarray progride para uso clínico, uma substituindo a instrumentação caros com tecnologia de detecção de baixo custo que apresentem características de desempenho semelhante ao de fluorescência fará ensaios de microarray mais atrativo e rentável.
A tecnologia de detecção colorimétrica ampliPHOX é destinado a aplicações de pesquisa, e tem um limite de detecção dentro de uma ordem de magnitude da fluorescência tradicionais 11, com uma vantagem principal é um valor aproximado de dez vezes menor custo em comparação com o instrumento scanners microarray confocal de fluorescência necessários para microarray detecção. Outra vantagem é o tamanho compacto do instrumento que permite a portabilidade e flexibilidade, ao contrário dos tradicionais instrumentos de fluorescência. Porque a tecnologia de polimerização não é tão linear como inerentemente detecção de fluorescência, no entanto, é mais adequado para aplicações de menor densidade microarray em que um sim / não respondeu para a presença de uma determinada seqüência é desejada, como, por matrizes de detecção de patógenos. Atualmente a densidade local máxima compatível com a detecção ampliPHOX é de aproximadamente 1.800 pontos / matriz. Devido às limitações de densidade local, microarrays maior densidade não são adequados para a detecção ampliPHOX.
Aqui, apresentamos ampliPHOX tecnologia de detecção colorimétrica como um método de amplificação de sinal em um microarray de baixa densidade desenvolvido para a detecção e caracterização dos vírus influenza (FluChip). Embora este protocolo utiliza o FluChip (um microarray de DNA) como uma aplicação específica do ampliPHOX detecção, qualquer microarray incorporando-alvo biotinilado pode ser rotulada e detectado de forma similar. O projeto microarray e Biotinilação do alvo a ser capturada são de responsabilidade do usuário. Uma vez que o alvo biotinilado foi capturado na matriz, detecção ampliPHOX pode ser realizada por marcação a primeira matriz com um conjugado de estreptavidina-label (ampliTAG). Após a exposição à luz usando o instrumento ampliPHOX Leitor de polimerização, de uma solução de monômero (ampliPHY) ocorre somente em regiões contendo ampliTAG marcado alvos. O polímero formado pode ser posteriormente coradas com uma solução não tóxica para melhorar o contraste visual, seguido de imagem e análise usando um pacote de software simples (ampliVIEW). O ensaio FluChip inteira de un-extraído de amostra para resultado pode ser realizado em cerca de 6 horas, e as etapas de detecção de ampliPHOX descrito acima pode ser concluído em cerca de 30 min.
Protocolo
1. Ampliação da amostra utilizando RT-PCR
- Extrato de RNA viral a partir de material clínico ou de um isolado viral utilizando o Kit Qiagen MinElute rotação de vírus em conjunto com a plataforma de extração de ácidos nucleicos QIAcube automatizado. Extrações são realizadas em 200 mL de amostra com um volume de eluição final de 60 mL. Extratos armazenar a -70 ° C ou menor para uso posterior.
- Em um modelo de área de livre, prepare o RT-PCR Master Mix no gelo acordo com o protocolo do fabricante. Para incorporar biotina durante a RT-PCR, use uma mistura dNTP biotinilado no lugar do fabricante fornecido mix dNTP. O custo de usar uma mistura dNTP biotinilado é menos de $ 1 USD ensaio /. Métodos alternativos de incorporação biotina, como a utilização de um primer biotinilado também pode ser usado. Adicionar cartilha misturas em concentrações finais de 1,0 mM para a Gripe A, B 1,0 M para Flu, e 0,14 mM para o controle interno. O Flu Um conjunto de primers amplifica o segmento de gene de matriz (1032 nt produto) eo conjunto de primers Flu B amplifica um segmento de gene não-estruturais (811 produto nt). Cada conjunto contém uma cartilha FluChip cartilha com um 5 'grupo fosforil para facilitar a geração de DNA de fita única via digestão enzimática com exonuclease lambda seguintes PCR. O produto resultante de fita simples requer tempos de hibridização muito menor do que o equivalente dupla fita e reduz significativamente o tempo de ensaio geral. Uma mistura pré-preparada de estes primers, bem como o modelo de RNA de controles internos estão disponíveis a partir InDevR para os pesquisadores interessados em uma base limitada.
- Brevemente vortex e distribuir 18 mL da mistura de mestre em tubos de paredes finas PCR.
- Transferência de tubos no gelo a uma área de trabalho adequado para além do modelo, e adicionar 2 mL para cada modelo de tubo de reação.
- Tubos de transferência de PCR para um termociclador e executar o seguinte perfil térmico: transcrição reversa a 50 ° C por 30 min, a inativação da enzima / ativação de 95 ° C por 15 min, 40 ciclos de PCR de 95 ° C por 30 s, 55 ° C por 30s e 72 ° C por 1 min, e uma extensão final a 72 ° C por 10 min.
2. Hibridação de RT-PCR produtos para microarrays de baixa densidade
- A fim de gerar DNA de fita simples para a hibridização FluChip, prepare a mistura de digestão enzimática através da combinação de 1,0 mL da enzima exonuclease lambda, 2.2 l do tampão de reação de acompanhamento, e 0,8 mL de água livre de nuclease. Estes montantes são para uma única amostra, mas pode ser simplesmente escalado para o número total de amostras a serem digeridas. Remover amostras do termociclador e adicionar 4 mL da mistura preparada para cada reação de digerir a vertente fosforilada do produto da PCR. Retorno de amostras para o termociclador e programar o termociclador a 37 graus Celsius por 15 minutos seguido de 95 graus Celsius por 10 minutos para completar a digestão enzimática e fragmentação passos calor.
- Os microarrays influenza personalizados usados são impressas em lâminas de vidro aldeído funcionalizado pela Applied Microarrays Inc. (Tempe, AZ). 5'-amino capturar seqüências terminadas são combinados com um buffer otimizado spotting e impresso em uma concentração final de 20 mM (exceto para a sequência do controlo positivo, que além disso tem uma modificação 3'-biotina e é avistado em uma concentração final de 500 nM) . Um contato não-spotting método é usado, com um diâmetro do ponto ideal de 300 mm, e centro para lançar centro de 700 mM.
- Remover microarrays da caixa de armazenamento e aplicar poços de uso único de hibridização em todo o microarrays, removendo a folha de protecção e pressionando firmemente em torno do perímetro bem.
- Colocar as lâminas em uma lata de lavagem contendo 110 ml de água purificada para a 5 min de lavagem pré-hibridização utilizando um agitador orbital de 60-90 rpm. Secar a matriz, tocando suavemente um limpador de tecido até a borda do poço e permitindo que a água a ser mau de distância.
- Combine 22 mL de tampão de hibridação 2x com cada um dos produtos fragmentados ssDNA e Pipete 40 das soluções de hibridização nos poços microarray.
- Permitir slides de hibridizar em câmara húmida fechada por 60 minutos.
- Remove os slides da câmara de umidade, brevemente lavar matrizes com Wash Buffer D em uma garrafa lavar antes de colocar no rack de slides. Normalmente o volume de lavagem de Wash Buffer D é de 2 ml por matriz. Rack de lugar slide contendo slides no bin lavar contendo 110 ml Tampão bin A. Coloque em agitador orbital de 60-90 rpm durante 1 minuto.
- Remover porta-lâminas de tampão de lavagem A, brevemente enxaguar com Wash Buffer D, rack de transferência de slide para uma caixa contendo B tampão de lavagem, e agitar a 60-90 rpm por 5 minutos.
- Seque suavemente a matriz em cada slide, slides e colocar secas em câmara de umidade, em preparação para as etapas de detecção de ampliPHOX colorimétrico.
3. ampliPHOX: rotulagem dos produtos cruzados e fichas de calibração com ampliTAG
- Combinar 10 ml de ampliTAG, 20 l de tampão ampliTAG 2x, e 10 ml de água purificada para cada matriz para ser processado. Os volumes de reagente podem ser simplesmente dimensionados para o número total de amostras. Certifique-se de preparar a mistura rotulagem suficientes para explicar todas as matrizes de amostra e matrizes de calibração necessário. O número de chips de aferimento depende se você estiver executando uma calibração completa (para um instrumento novo ou novo lote de reagentes), ou apenas uma verificação de instrumento. Para uma calibração completa, 3 chips de calibração devem ser rotulados, e para uma verificação de reagentes, apenas um chip único de calibração devem ser rotulados. Por favor, note que antes de as matrizes de calibração são rotulados, eles devem passar pela etapa de lavagem pré-hibridação que foi descrito anteriormente para as matrizes de influenza.
- Transferir 40 mL da mistura de etiqueta para cada matriz e permitir a rotulagem de reação para avançar em uma câmara de umidade fechado por 5 minutos.
- Lave imediatamente matrizes com Wash Buffer D em uma garrafa de lavar antes de colocar slides em um rack de slide. Transferência de rack na bin contendo 110 ml de Tampão C, e agitar a 60-90 rpm por 5 minutos.
- Usando um bin segunda lavagem com água purificada, execute três vezes consecutivas quedas de água breve para remover os resíduos de sal. Seca as matrizes por tocar suavemente um limpador de tecido para a beira dos poços.
- Os microarrays estão devidamente rotulados com ampliTAG, eo restante do procedimento de detecção ampliPHOX pode ser realizada. Fotoativação e de imagem de matrizes com a etiqueta deve ser concluída no prazo de 24 horas, e arrays adicionais podem ser armazenados em uma caixa de lâminas escuras até o uso.
4. ampliPHOX: calibração, amplificação de sinal e imagem
- Por sua vez ampliPHOX leitor sobre software e garantir ampliVIEW está pronto para fotoativação.
- Determinar o tempo de fotoativação ideal o uso de chips de calibração. Além da breve introdução que se segue, este procedimento também é descrito em detalhe no Manual de Operação ampliPHOX. Os chips de calibração contém uma série de diluições de uma seqüência de controle biotinilado que são utilizados para otimizar a sensibilidade do ensaio, com o objetivo de maximizar o número de linhas do chip de calibração que produzem um sinal positivo, conforme determinado pelo software.
- Remover ampliPHY de 4 ° C, permitem a temperatura ambiente, e vortex brevemente para misturar. Pipete 3 de ampliPHY enhancer no frasco ampliPHY e vortex cuidadosamente por 10 segundos.
- Uniformemente transferir 40 ml de solução ampliPHY para o bem microarray contendo o ampliTAG marcado matriz, garantindo que nenhum bolhas estão presentes. Fechar o frasco ampliPHY entre cada aplicação. Inserir o slide microarray na baía fotoativação do Leitor ampliPHOX.
- Para a matriz primeira calibração, usar o tempo de fotoativação padrão no 'Time' caixa no painel esquerdo do software, e clique no verde 'Start' para iniciar a fotoativação. Depois de concluído, retire a matriz e enxágüe com água purificada para remover ampliPHY excesso. Formação de polímeros claras sobre alguns dos pontos deverão ser agora visíveis.
- Permitir que as manchas de polímero para secar por 2 minutos, em seguida, distribua duas gotas de ampliRED para a matriz e permitir que a coloração para prosseguir por 2 minutos.
- Em seguida, enxágüe rapidamente microarray com água purificada e seque com um lenço de papel.
- Inserir microarray na baía de imagens do Leitor ampliPHOX, e clique no botão 'nova imagem de captura "na guia Imagem. Uma vez digitalizado, ajustar a recortar e salvar a imagem.
- Na guia de análise, selecione a máscara Chip de calibração e no botão "Colocação Auto 'para iniciar a análise. O software vai automaticamente produzir um 'Record Resumo' mostrando os resultados quantificados. Com base nesses resultados, o tempo de fotoativação é ajustado por 10 segundos para a matriz de calibração segundo, eo procedimento é repetido.
- Uma vez que o tempo ideal fotoativação foi determinada, as matrizes de amostra podem ser processados usando a amplificação de sinal mesmo e protocolo de imagem usado para as matrizes de calibração.
- Depois que a imagem é capturada para as matrizes de amostra, uma máscara para o seu layout de matriz específico como o layout variedade de gripe descrito aqui pode ser created.The ampliVIEW software é capaz de realizar análise de imagem automática e gerando resultados influenza subtipagem para cada amostra.
5. Resultados representativos:
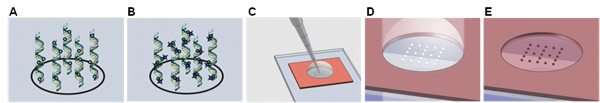
Figura 1. Ilustração esquemática do método de detecção ampliPHOX colorimétrico. (A) DNA alvo biotinilado é hibridizada com cada ponto na matriz, e (B) marcada com ampliTAG. (C) ampliPHY solução é então adicionado, e (D) exposto à luz para formar manchas de polímero visível. (E) As manchas de polímero formado são posteriormente marcadas com um corante não tóxico para melhorar o contraste.
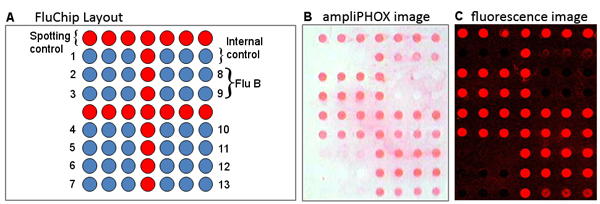
Figura 2. (A) layout da Influenza microarray de baixa densidade. Seqüências de 1-7 e 10-13 influenza A meta, e as seqüências de 8, 9 alvo influenza B. (B) ampliPHOX e (C) as imagens de fluorescência de um romance de 2009 H1N1 ("gripe suína") Espécime mostrando o mesmo padrão de detecção por ambos os métodos.
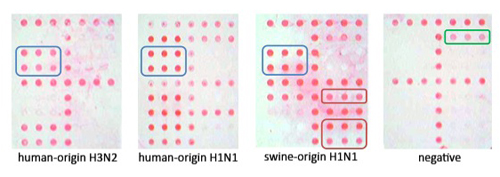
Figura 3. Da esquerda para a direita, imagens representativas ampliPHOX para influenza A H3N2, de origem humana H1N1, 2009 romance H1N1 (suína de origem), e um espécime negativo. Todos os três subtipos mostram padrões visualmente distintas na matriz. Aviso no negativo que apenas o controle interno é visto MS2, indicando a amplificação RT-PCR não foi inibida.
Discussão
A tecnologia de detecção colorimétrica ampliPHOX aqui apresentada é uma alternativa rápida e barata para detecção de uma única cor de fluorescência para aplicações de menor densidade de microarray. Mostrado esquematicamente na Figura 1, o princípio de detecção é baseado no uso de uma etiqueta fotoiniciador (1B). Na presença de uma solução de monômero contendo (1C), a exposição à luz faz com que o fotoiniciador (ampliTAG) para desencadear uma reação de polimerização apenas em re...
Divulgações
Agradecimentos
InDevR reconhece NIH / NIAID U01AI070276 e R43AI077112 para financiamento deste trabalho.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagente / equipamentos | Fabricante | Catalogo | Comentários |
|---|---|---|---|
| Kit Qiagen MinElute rotação Virus | Qiagen | 57704 | eluição 60 mL única |
| QIAcube | Qiagen | 9001292 | opcional |
| ABI 9800 Rápido Termociclador | Applied Biosystems | 4441166 | |
| Qiagen OneStep RT-PCR kit | Qiagen | 210210 | kit dNTPs não utilizados |
| Tampão Spotting 2x | InDevR Inc. | MI-5007 | |
| Mix dNTP biotinilado | InDevR Inc. | MI-5009 | |
| Exonuclease Lambda | Biotecnologias Epicentre | LE032K | 2500 U, 10U/μl |
| FluChip mistura de iniciadores | InDevR | N / A | ainda não disponível para venda |
| Orbital Shaker | Madell Tecnologia | ZD-9556-A | |
| Lavar Bins | InDevR Inc. | MI-4002 | |
| Lavar Racks | InDevR Inc. | MI-4003 | |
| Tampão de hibridação 2x | InDevR Inc. | MI-5004 | |
| Fichas de calibração | InDevR Inc. | AP-5006 | |
| Lavar Buffers AD | InDevR Inc. | MI-5005 | |
| ampliRED | InDevR Inc. | AP-5004 | |
| ampliTAG | InDevR Inc. | AP-5001 | |
| 2x ampliTAG buffer | InDevR Inc. | AP-5002 | |
| ampliPHY, ampliPHY enhancer | InDevR Inc. | AP-5003 |
Referências
- Kumar, R. M. The Widely Used Diagnostics "DNA-Microarray"-A Review. Amer J Inf Dis. 5, 207-218 (2009).
- Miller, M. B., Tang, Y. W. Basic Concepts of Microarrays and Potential Applications in Clinical Microbiology. Clin Microbiol Rev. 22, 611-633 (2009).
- Mikhailovich, V., Gryadunov, D., Kolchinsky, A., Makarov, A. A., Zasedatelev, A. DNA microarrays in the clinic: infectious diseases. BioEssays. 30, 673-682 (2008).
- Call, D. R. Challenges and opportunities for pathogen detection using DNA microarrays. Crit Rev Microbiol. 31, 91-99 (2005).
- Raoult, D., Fournier, P. E., Drancourt, M. What does the future hold for clinical microbiology. Nat Rev Microbiol. 2, 151-159 (2004).
- Dawson, E. D., Rowlen, K. L., Wang, Q., Tao, Y. J. MChip: A Single Gene Diagnostic for Influenza A. Influenza: Molecular Virology. , (2010).
- Gall, A., Hoffman, B., Harder, T., Grund, C., Ehricht, R., Beer, M. Rapid hemagglutinin subtyping and pathotyping of avian influenza viruses by a DNA microarray. J Virol Meth. 160, 200-205 (2009).
- Townsend, M. B., Dawson, E. D., Mehlmann, M., Smagala, J. A., Dankbar, D. M., Moore, C. L., Smith, C. B., Cox, N. J. FluChip: Experimental evaluation of a diagnostic influenza microarray. J Clin Microbiol. 44, 2863-2871 (2006).
- Wang, Z., Daum, L. T., Vora, G. J., Metzgar, D., Walter, E. A., Canas, L. C., Malanosky, A. P., Lin, B., Stenger, D. A. Identifying influenza viruses with resequencing arrays. Emerg Inf Dis. 12, 638-646 (2006).
- Kessler, N., Ferraris, O., Palmer, K., Marsh, W., Steel, A. Use of the DNA Flow-Thru Chip, a three-dimensional biochip, for typing and subtyping of influenza viruses. J Clin Microbiol. 42, 2173-2185 (2004).
- Kuck, L. R., Taylor, A. W. Photopolymerization as an innovative detection technique for low-density microarrays. Biotechniques. 45, 179-186 (2008).
- Avens, H. J., Bowman, C. N. Development of fluorescent polymerization-based signal amplification for sensitive and non-enzymatic biodetection in antibody arrays. Acta Biomat. 6, 83-89 (2010).
- Sikes, H. D., Jenison, R., Bowman, C. N. Antigen detection using polymerization-based amplification. Lab on a Chip. 9, 653-656 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados