É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Fotobranqueamento Ensaios (FRAP & FLIP) a Medida Dynamics Protein Chromatin em Viver Células-Tronco Embrionárias
Neste Artigo
Resumo
Descrevemos os métodos fotobranqueamento inclusive Recuperação de Fluorescência Após Fotodegradação (FRAP) e perda de fluorescência em Fotodegradação (FLIP) para monitorar a dinâmica de proteínas da cromatina no tronco embrionárias (ES) células. Proteínas da cromatina dinâmica, que é considerado um dos meios para estudar a plasticidade da cromatina, é reforçada em células pluripotentes.
Resumo
Recuperação de fluorescência Após Fotodegradação (FRAP) e perda de fluorescência em Fotodegradação (FLIP) permitem o estudo da dinâmica de proteínas em células vivas, com resolução espacial e temporal bem. Aqui nós descrevemos como executar FRAP e ensaios FLIP de proteínas da cromatina, incluindo H1 e HP1, em tronco embrionárias de camundongos (ES) células. Em um experimento FRAP, as células são transfectadas, seja transitoriamente ou de maneira estável, com uma proteína de interesse em fusão com a proteína verde fluorescente (GFP) e suas derivadas (YFP, PCP, Cherry, etc.) No transfectadas, as células fluorescentes, um feixe de laser intenso focado alvejantes uma região relativamente pequena de interesse (ROI). O comprimento de onda do laser é selecionado de acordo com a proteína fluorescente usada para a fusão. A luz do laser de forma irreversível o sinal alvejantes fluorescentes de moléculas no ROI e, imediatamente após o branqueamento, a recuperação do sinal fluorescente na área branqueada - mediada pela substituição das moléculas branqueada com as moléculas crus - é monitorado usando imagens de lapso de tempo. Gerada curvas de recuperação de fluorescência fornecer informações sobre a mobilidade da proteína. Se as moléculas fluorescentes são imóveis, nenhuma recuperação de fluorescência será observado. Em uma abordagem complementar, perda de fluorescência em Fotodegradação (FLIP), o feixe de laser alvejantes mesmo lugar repetidamente e da intensidade do sinal é medido em outras partes da célula fluorescentes. Experimentos FLIP, portanto, medida de decaimento do sinal, em vez de recuperação de fluorescência e são úteis para determinar a mobilidade de proteínas assim como a proteína vaivém entre compartimentos celulares. Vinculação transitória é uma propriedade comum da cromatina proteínas associadas. Embora a maior fração de cada proteína cromatina é obrigado a cromatina em um dado momento no estado estacionário, a ligação é transitória e proteínas mais cromatina têm uma alta rotatividade de cromatina, com um tempo de residência na ordem de segundos. Estas propriedades são cruciais para a geração de elevada plasticidade no genoma de expressão 1. Experimentos fotobranqueamento são, portanto, particularmente útil para determinar plasticidade cromatina usando GFP fusão versões da cromatina proteínas estruturais, especialmente em células ES, onde a troca dinâmica de proteínas da cromatina (incluindo heterocromatina proteína 1 (HP1), histona H1 linker e histonas core) é maior do que em células diferenciadas 2,3.
Protocolo
1. Chapeamento as células ES
T = 0 hrs
Plating MEF
- Brasão da imagem live 8 bem-μ-Slides (ibidi; Munique, Alemanha) com gelatina ou em copos chambered tampa (Lab-Tek, Rochester, NY) ou em pratos de vidro de fundo de cultura (Mattek; Ashland, MA). Deixe por 50-30 min e aspire a gelatina de distância livre.
- Semente 22.000 MEFs / poço em 250 mL de volume total de DMEM [suplementado com 10% soro fetal bovino (FBS)]. Permitem que as células a crescer em uma cultura de tecidos incubadora (37 ° C, 5% de CO2).
T = 6 horas
ES revestimento celular
- Aspirar DMEM.
- Semente de cada MEF revestido bem com 15.000 células R1 / poço em 250 mL de volume total de células ES media [suplementado com 10% ESC-grade de soro fetal bovino (FBS), piruvato de sódio 1 mM, 0,1 mM aminoácidos não essenciais, 0,1 mM β- mercaptoetanol, e 1000 fator de leucemia U / ml inibitória (LIF)], para obter 30% a 50% confluência no dia seguinte.
2. Transfecting as células ES
T = 24 hrs
Transfecção transiente
- Substituir a mídia de células ES com 250 mL / poço de mídia celular fresco ES.
- Num tubo de ensaio 1,5 ml estéril, adicionar 100 ml de soro de mídia livre [Opti-MEM (Gibco)], em seguida, adicione 10 mL de trânsito LT1 reagentes de transfecção (Mirus) diretamente para a mídia livre de soro. Mix pipetando suave e incubar em temperatura ambiente por 50-20 min.
- Adicione 1,5 g de DNA de fusão GFP plasmídeo (H1E, H1o ou HP1) ao diluída trânsito LT1 reagente. Mix pipetando suave e incubar à temperatura ambiente por 15-30 min.
- Adicionar 13,5 mL / poço da mistura de transfecção. Agitar o 8-bem-μ Slides para assegurar uma dispersão. Depois de 24 horas substituir a mídia antiga cela ES com 250 mL dos meios de comunicação celular fresco ES.
3. Realizar FRAP e FLIP
T = 48-72 hrs
- O experimento pode ser realizado em qualquer microscópio confocal de varredura a laser (CLSM) mas desde que em um experimento FRAP / FLIP normal, muitas imagens consecutivas são adquiridos, é recomendado o uso de um microscópio confocal girando do disco, que permite velocidade de aquisição e garante que não amostra indesejada branqueamento ocorrerá após o evento inicial intencional de branqueamento. Aqui, nós recomendamos o uso do sistema de disco giratório Revolução confocal (www.Andor.com), com a cabeça do disco Yokogawa CSU-X spinning. Este sistema tem a capacidade dupla para photobleach usando um módulo Frappa especializada, com um sistema de ponto de leitura, e rapidamente mudar a luz de volta para coletar imagens usando o disco girando. Os três proteínas fluorescentes mais comum usado para fotobranqueamento experimentos são GFP, YFP e Cherry. Se GFP ou YFP são usados, a ~ 488 nm do laser é necessária. Para Cherry, usam um laser ~ 560 nm. Em todos os casos, recomendamos o uso de lasers de estado sólido. Ter um estágio automatizado é útil mas não é obrigatório. Uma vez que as células vivas são gravadas, é imprescindível a utilização de uma câmara ambiental (usamos um de LIS, Suíça), o oxigênio controlar, umidade, CO 2 e temperatura. FRAP é realizada utilizando máxima intensidade do laser, enquanto imagem é feito com a potência do laser mínimo exigido (geralmente na área de 10%, quando o nível de fluorescência é adequada).
Observe as células com luz fluorescente do comprimento de onda apropriado e selecione uma célula expressando GFP usando uma lente de imersão 60X de petróleo. Assegurar a distribuição subcelular correta. Ocasionalmente, quando os níveis de expressão são muito altos, a localização da proteína pode 'derrame' de outros compartimentos, como o nucléolo. Tais células não deve ser selecionado. - Agora definir um protocolo de imagem: coletar 3-5 frames antes do fotobranqueamento, então photobleach em eucromatina ou heterocromatina (visto como condensado GFP focos) e coletar 90-120 frames após a photobleach, com intervalos 250-1000 ms: H1E-GFP, 1000 ms, H1o e HP1-GFP, 250 ms (intervalo de tempo muda de acordo com a dinâmica das proteínas, onde as proteínas altamente dinâmicos requerem tempo mais curto intervalo). Normalmente usamos intensidade do laser 8-10% para fotobranqueamento com um pulso de laser de μseconds 20-40 (1-2 iterações), mas esses números podem mudar de acordo com a proteína analisada e os níveis de expressão. Quando fotobranqueamento é apropriado, você deve observar um "buraco negro" em sua fluorescência da GFP. O buraco negro está gradualmente re-cheia de recuperação após fluorescência. Embora o disco giratório pode obter até cerca de 60 imagens por segundo (com boa intensidade de fluorescência e quando o zoom em uma única célula), não recomendamos utilizar o sistema a velocidades tão altas devido à baixa qualidade de imagem e maior potencial de fototoxicidade.
- Para um experimento FLIP, estabeleceu um protocolo de imagem diferente: coletar 3-5 frames antes do branqueamento, em seguida, iniciar repetida de branqueamento no mesmo local durante a coleta de imagens. Para cada sec H1E-GFP, lixívia a cada 5 segundos, para H1o-GFP, bleach 2, e para HP1-GFP lixívia cada 1 seg. Bleach umd coletar imagens repetidamente durante todo o experimento.
- Para uma ou outra técnica, repita o processo em 20-30 células. Para fins estatísticos, repetir a experiência de três vezes ou mais, de preferência em dias diferentes. Em populações homogêneas e configurações corretas, o desvio padrão é geralmente baixa (<5%).
Para ambos os FRAP e FLIP, o tamanho ea forma da região branqueada influencia a dinâmica de recuperação e deve permanecer constante dentro de um experimento. Além disso, quando duas células são comparados, protocolos idênticos devem ser utilizados e as células devem ser analisados em seqüência, no mesmo dia em que o poder do laser e outras condições podem variar e pode afetar o resultado do experimento.
4. FRAP e Análise de Dados FLIP
- Em todos os quadros FRAP coletados, medir a intensidade de fluorescência no ROI (ROI b = área branqueada), a área de fundo (ROI bg), ea área nuclear não-branqueada (ROI nb) como uma função do tempo antes e após o clareamento. Quando a região é insignificante branqueada todo o núcleo pode ser selecionado para fins de normalização.
- Para cada ponto do tempo, normalizar os dados de acordo com a fórmula: (ROI b - ROI bg) / (ROI nb - ROI bg) / (b pbROI - pbROI bg) / (pbROI nb - pbROI bg), pb denota pré-branqueada. Para a pré-bleach imagens deverá obter um valor de aproximadamente 1. A primeira imagem após a lixívia irá indicar a profundidade Bleach. Subtrair o valor de 1 para o valor real Bleach Profundidade. Repita a operação para cada célula e média 20-30 células de cada experimento.
- Em todos os quadros FLIP coletados, medir a intensidade de fluorescência na área não-branqueada nuclear, ea área de fundo (ROI nb = área não-branqueada, ROI bg de fundo =). Cálculo dos dados FLIP é semelhante a uma curva de FRAP, apenas o ROI analisados (ROI nb) deve ser diferente do que a região real branqueada, que não é utilizado para o cálculo: (ROI nb - ROI bg) / (pbROI nb - pbROI bg) . Também é possível usar uma célula vizinha (ROI n = célula vizinha) para fins de normalização: (ROI nb - ROI bg) / (ROI n - ROI bg) / (pbROI nb - pbROI bg) / (pbROI n - pbROI bg ).
Após coleta de dados, é possível ajustar os dados experimentais para simulação de computador. Isto permite calcular, com uma boa proximidade, a fração móvel, a fração de imóvel e da metade do valor máximo. Nós não vamos discutir os aspectos matemáticos e estatísticos de análises FRAP aqui e remeter o leitor para outras publicações excelente 4-9. A profundidade de água sanitária refere-se à distância (no eixo y) entre o pré-lixívia de sinal (100%) e a primeira imagem depois de lixívia, a fração móvel refere-se à distância (no eixo y) entre a profundidade e lixívia o sinal recuperado quando a cinética atinge um platô, ea fração de imóvel refere-se à distância (no eixo y) entre o sinal recuperado e os pré-lixívia de sinal (100%) (ver Figuras 1B e 2B). Além desta análise, não são bons modelos matemáticos para ajustar os dados. Para um único expoente, a equação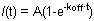
onde t é o tempo, A é a fração móvel, 1-A é a fração imóvel e off k é a constante de dissociação, pode ser usado para ajustar os dados, e uma estimativa direta da taxa fora de ligação (k off) pode ser obtidos, bem como para o parâmetro A, que pode ser usado para calcular a taxa de associação.
5. Resultados representativos:
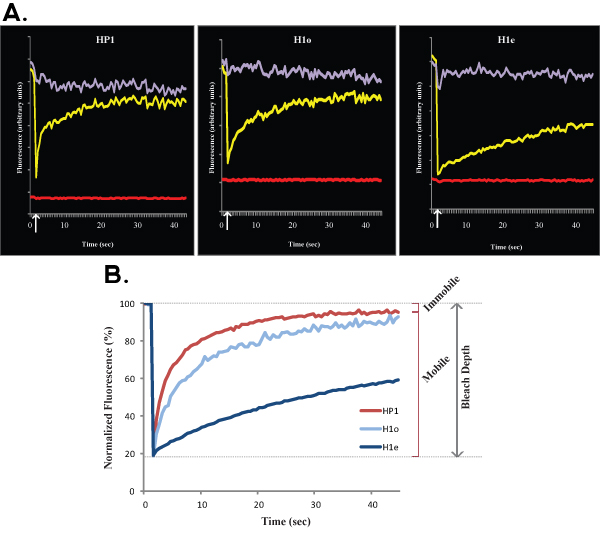
Figura 1. A e B mostram curvas representativas FRAP de HP1 (esquerda), H1o (meio) e H1E (direita) em células ES R1. Figura de simplicidade e clareza 1A mostra os dados brutos de uma única célula, antes de qualquer normalização e cálculo. A curva amarela corresponde à região branqueada, a curva de púrpura corresponde à área não-branqueada nuclear (quando a região branqueada é insignificante todo o núcleo pode ser selecionado para fins de normalização), ea linha vermelha corresponde à fluorescência de fundo, que é mínima neste caso. Seta vertical representa o tempo de lixívia. Dados normalizados e em média é mostrado na Figura 1B. Observe a recuperação mais lenta da H1 (azul) em comparação com HP1 (vermelho). Também a variante H1E (azul escuro) é mais lenta do que a variante H1o (azul claro). Frações móvel e imóvel e Profundidade Bleach são indicados para HP1.
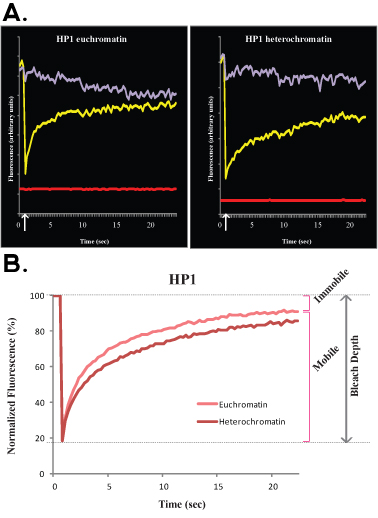
Figura 2. A e B mostram curvas representativas FRAP comparando eucromatina (esquerda) com heterocromatina (direita) de HP1 em células ES R1. Da mesma forma a Figura 1, Figura 2A mostra os dados brutos de uma única célula, a curva amarela corresponde à região branqueada, curva roxa corresponde à área não-branqueada nuclear, ea linha vermelha corresponde à fluorescência de fundo. Seta vertical representa o tempo de lixívia. Dados normalizados e em média é mostrado na Figura 2B. Observe a recuperação mais lenta da heterocromatina (vermelho escuro) em comparação com eucromatina (luz vermelha). Frações móvel e imóvel e Profundidade Bleach são indicados para eucromatina.
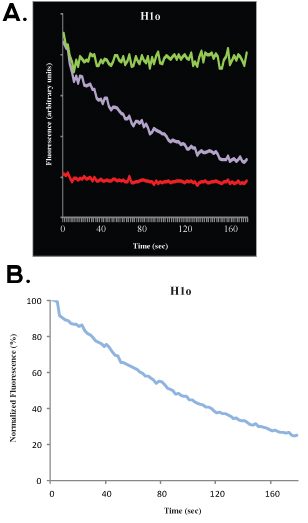
Figura 3.. Um experimento típico FLIP de células ES H1o R1 é mostrado na Figura 3A (cru, não-normalizada de dados) e B (dados normalizados e média). Neste experimento, a curva de púrpura corresponde à área não-branqueada nuclear, a linha verde corresponde a um núcleo da célula vizinha ea linha vermelha corresponde à fluorescência de fundo.
Discussão
Ao contrário das técnicas mais disponíveis, que envolvem cromatina purificada a partir de populações de células ou células fixas, experimentos FRAP seguir as mudanças na dinâmica de proteínas da cromatina em células vivas. Encontramos dinâmica de proteínas da cromatina para ser um bom indicador para a plasticidade da cromatina. No entanto, porque requer a fusão do gene de interesse com GFP, a adição da tag fluorescentes podem interferir com a função da proteína. Assim, antes de prosseguir com FRAP, a ...
Divulgações
Agradecimentos
Agradecemos a membros do laboratório Meshorer, especialmente Shai Melcer, Adi Alajem, Edupuganti Raghu Ram, Badi Sri Sailaja, Anna Mattout e Alva Biran, por comentários críticos e para a resolução de problemas experimentos fotobranqueamento em uma base diária. EM é um H. José e Belle R. Braun Professor de Ciências da Vida e é apoiado pela Fundação Ciência Israel (ISF 943/09), o Ministério da Saúde, Israel (6007) da União Europeia (IRG-206872 e 238176), Israel Cancer Research Foundation, o Internal Applicative Grants de Medicina da Universidade Hebraica e do Instituto de Psicobiologia Israel.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
|---|---|---|---|
| DMEM | Sigma | D5671 | |
| Gelatina | Merck | 1,04078 | |
| Opti-MEM | Gibco | 31985 | |
| Trânsito LT1 | Mirus | MIR2300 | |
| 8-bem-μ Slides | ibidi | 80826 |
Referências
- Phair, R. D. Global nature of dynamic protein-chromatin interactions in vivo: three-dimensional genome scanning and dynamic interaction networks of chromatin proteins. Mol Cell Biol. 24, 6393-6402 (2004).
- Meshorer, E., Girard, L. . Imaging chromatin in embyonic stem cells in StemBook. , (2008).
- Meshorer, E. Hyperdynamic plasticity of chromatin proteins in pluripotent embryonic stem cells. Dev Cell. 10, 105-116 (2006).
- Bancaud, A., Huet, S., Rabut, G., Ellenberg, J. . Fluorescence perturbation techniques to study mobility and molecular dynamics of proteins in live cells: FRAP, photoactivation, photo conversion, and FLIP. , (2009).
- Dundr, M., Misteli, T. Measuring dynamics of nuclear proteins by photobleaching. Curr Protoc Cell Biol. Chapter 13, Unit 13-Unit 13 (2003).
- Ellenberg, J. Nuclear membrane dynamics and reassembly in living cells: targeting of an inner nuclear membrane protein in interphase and mitosis. J Cell Biol. 138, 1193-1206 (1997).
- Lenser, T., Weisshart, K., Ulbricht, T., Klement, K., Hemmerich, P. Fluorescence fluctuation microscopy to reveal 3D architecture and function in the cell nucleus. Methods Cell Biol. 98, 2-33 (2010).
- Mueller, F., Mazza, D., Stasevich, T. J., McNally, J. G. FRAP and kinetic modeling in the analysis of nuclear protein dynamics: what do we really know. Curr Opin Cell Biol. 22, 403-411 (2010).
- Phair, R. D., Misteli, T. Kinetic modelling approaches to in vivo imaging. Nat Rev Mol Cell Biol. 2, 898-907 (2001).
- Poser, I. BAC TransgeneOmics: a high-throughput method for exploration of protein function in mammals. Nat Methods. 5, 409-415 (2008).
- Sigal, A. Generation of a fluorescently labeled endogenous protein library in living human cells. Nat Protoc. 2, 1515-1527 (2007).
- Cohen, A. A. Dynamic proteomics of individual cancer cells in response to a drug. Science. 322, 1511-1516 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados