É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Ensaio in vitro da adesão bacteriana em mamíferos Células Epiteliais
Neste Artigo
Resumo
Este protocolo é um ensaio de adesão bacteriana simples que consiste na contagem do número de unidades formadoras de colônia de bactérias que são aderidos em células em cultura. O ensaio é robusto, independente da adesina estudado, e inúmeras variações são usadas na maioria dos laboratórios que trabalham com patogênese bacteriana.
Resumo
Para causar infecções, as bactérias precisam colonizar seu hospedeiro. Patógenos bacterianos expressar várias moléculas ou estruturas capazes de promover a ligação às células hospedeiras 1. Estas adesinas confiar em interações com os receptores da célula hospedeira superfície ou proteínas solúveis agindo como uma ponte entre bactérias e hospedeiro. Adesão é um primeiro passo crítico antes da invasão e / ou secreção de toxinas, por isso, é um evento-chave a ser estudado na patogênese bacteriana. Além disso, as bactérias aderiram muitas vezes induzem exquisitely aperfeiçoá-lo respostas celulares, os estudos que deram origem ao campo da 2 'celular microbiologia. Ensaios robusta para adesão bacteriana em células hospedeiras e sua invasão, portanto, têm um papel fundamental em estudos de patogenia de bactérias e têm sido muito utilizados em muitos laboratórios pioneira 3,4. Estes ensaios estão agora praticado pela maioria dos laboratórios que trabalham com patogênese bacteriana.
Aqui, descrevemos um teste de aderência normal ilustrando a contribuição de uma adesina específica. Usamos a cepa de Escherichia coli 2787 5, uma cepa patogênica humana expressando o autotransporter Adhesin Envolvidos na adesão difusa (AIDA). Como controle, utilizamos uma estirpe mutante sem o gene Aida, 2787Δ aida (F. e M. Berthiaume Mourez, não publicado), e uma cepa de laboratório comercial de E. coli, C600 (New England Biolabs). As bactérias são deixados para aderir às células do comumente usado HEp-2 linha celular humana epiteliais. Este ensaio tem sido menos extensivamente descrito anteriormente 6.
Protocolo
1. Preliminar: cepas bacterianas e células epiteliais.
Manipulações de células e bactérias são realizados de forma asséptica, debaixo de uma capela de fluxo laminar.
- Recém isolar a E. cepas de Escherichia 2787, 2787Δ Aida, e C600 de stocks glicerol em lisogenia Caldo (LB) ágar (triptona 1%, cloreto de sódio 0,5%, extrato de levedura 0,5%, ágar 1,5%) e crescer a 37 ° C. Para minimizar a variabilidade no ensaio, é aconselhável usar sempre cepas recém-banhado e para manter as cepas a 4 ° C em placas de Petri seladas apenas para um máximo de um par de semanas.
- Cultura de células HEp-2 (ATCC CCL-23) em alta de glicose Modified Dulbecco Eagle Medium (DMEM) suplementado com 10% de soro bovino inactivado pelo calor (inativação de calor é realizada a 56 ° C por 30 min). Durante a cultura de rotina também adicionar 10 U / ml de penicilina e estreptomicina 10 mg / ml.
- Crescer células Hep-2, a 37 ° C em uma célula incubadora com uma atmosfera contendo 5% CO 2. Use procedimentos padrão de cultura celular para manter as células, que são cultivadas em 75 cm 2 frascos e cada vez que eles atinjam subcultivadas confluência. Usamos nossas células cultivadas até atingirem 30-40 passagens e depois descartá-las.
- Prepare um ensaio quando as células HEp-2 em um frasco de 75 cm 2 são quase atingindo confluência e, portanto, estão prontos para um ensaio de adesão.
2. DIA 1: Preparação do inóculo e as células epiteliais
- Lavar a células HEp-2 no frasco uma vez com fosfato de Dulbecco quente de solução salina tamponada (DPBS: 8 g / L NaCl, 0,2 g / L KCl, 0,2 g / L KH 2 PO 4, 0,21 g / L Na 2 HPO 4: 7H 2 O)
- As células são incubadas com tripsina 0,05% - EDTA por 5 min antes de adicionar meio morno fresco completo. Depois de meio fresco é adicionado, as células são ressuspendidas pipetando cima e para baixo.
- Centrifugar a suspensão celular a 2.000 rpm por 5 min e ressuspender o pelotas no DPBS e centrifugar novamente.
- Ressuspender as células em meio fresco suplementado com 10% de soro, mas não contendo antibióticos, a uma concentração de 2x10 5 células / ml.
- Mililitro uma semente de suspensão de células em três conjuntos de poços duplicado (um para cada cepa) no centro de uma placa de 24 poços e incubar a placa durante a noite em uma cela cultura incubadora.
Nota: É importante que o soro ser cuidadosamente decomplemented e que os antibióticos são omitidos após esta fase. Não fazer isso pode matar a bactéria infectante. - Inocular uma colônia isolada de cada estirpe bacteriana (2787, 2787Δ Aida e C600) em 5 ml de caldo LB (triptona 1%, cloreto de sódio 0,5%, extrato de levedura 0,5%) e crescer durante a noite a 37 ° C com agitação vigorosa (180 rpm ).
3. DIA 2: Infecção das células
- Inspecionar as células HEp-2 em microscópio invertido para verificar que eles são pelo menos 90% confluentes e não contaminados.
- Lavou as células com DPBS quente e, a cada poço, adicione 1 ml de meio fresco suplementado com 10% de soro, mas não contendo antibióticos. Também adicionar meio fresco a três poços sem células. Isso será usado para determinar o número total de bactérias no inóculo para cada cepa.
- Medir a densidade óptica a 600 nm (OD 0,600 nm) das culturas bacterianas. Adicionar uma alíquota de cada cultura bacteriana a um conjunto de poços duplicados contendo células Hep-2 e para um bem, não contendo células. Geralmente, nós usamos um volume de cultura overnight correspondente a 10 6 unidades formadoras de colônia (UFC). Isso representa uma multiplicidade de infecção (MOI) de 5:1 (bactérias: células). Embora nós usar diretamente nossas culturas durante a noite, às vezes, é aconselhável centrífuga as bactérias e ressuspender-los em DPBS para evitar efeitos deletérios de moléculas secretadas presentes nas culturas durante a noite (como citotoxinas, por exemplo)
Nota: O MOI pode variar entre 100:1 e 01:10. MOI maior variabilidade e maior rendimento de fundo e as bactérias tendem a se ater ao plástico da placa. Menor MOI também produz alta variabilidade. Uma vez que um MOI é escolhido, é imperativo para ser coerente e manter esta MOI. - Incubar as células infectadas na cultura de células incubadora durante 3 horas a 37 ° C com 5% de CO 2.
Nota: Também é possível centrífuga brevemente a placa em baixa velocidade (por exemplo, 1.000 xg por 1-2 min), a fim de trazer todas as bactérias diretamente em contato com as células. Isto tem a vantagem adicional de sincronizar a infecção, e permitindo menor tempo de incubação (tão pouco quanto 15-30 min). - Remova a mídia a partir de células infectadas e lavar as células 3 vezes com DPBS quente. Nesta etapa, as bactérias aderentes geralmente pode ser visto com um microscópio normal e rotineiramente realizar esta verificação.
- Para lisar as células e retirar o bac aderiutérios, adicione 100 ml de 1% Triton X-100 a cada poço contendo as células. Detergentes podem ser utilizados outros (como saponina por exemplo).
- Incubar as células por 10 min em temperatura ambiente e em seguida, adicione 900 mL de meio LB.
- Suavemente homogeneizar as suspensões por repetidas up-and-down pipetagem. Os poços sem células contendo o inóculo são igualmente suavemente homogeneizado.
Nota: Algumas bactérias podem ser muito sensíveis ao detergente, nesse caso, destacamos as células epiteliais através da incubação com tripsina 0,05% - EDTA por 20 min a 37 ° C. Células juntamente com as bactérias aderiram também pode ser raspado dos poços com uma pipeta. - Prepare série de 10 diluições das suspensões de bactérias aderidas e inóculo utilizando caldo LB e 100 mL placa de 3 diluições (geralmente as diluições 1:1.000, 1:10.000 e 1:100.000) em ágar LB e incubar durante a 37 ° C .
4. DIA 3: ufc contagem e apresentação dos dados.
- Contagem de colônias nas placas e calcular o número de ufc de bactérias aderidas e do inóculo pela média de cada série de diluições. Placas somente com entre 10-300 colônias devem ser contadas.
5. Resultados representativos:
A seguinte tabela mostra os resultados típicos da ufc contagem de bactérias aderidas e inóculo de 3 experimentos realizados em dias diferentes:

Os resultados podem ser reportados diretamente como aderiu ufc, como mostrado na Figura 1A. Uma vez que os tamanhos de inóculo pode variar entre linhagens por causa das diferenças nas taxas de crescimento ou erros de pipetagem, muitas vezes é aconselhável para relatar os resultados em percentagem de bactérias aderidas. A porcentagem de bactérias aderidas pode ser calculado dividindo o número de ufc de bactérias aderidas pelo número de ufc do inóculo, como mostrado na Figura 1B. Ao realizar uma medida repetidas ANOVA e Bonferroni pós-testes para comparar todas as colunas da Figura 1B, podemos ver que existem diferenças significativas (p <0,05) entre 2787 e 2787Δ Aida, bem como entre 2787 e C600, mas não houve diferença significativa entre 2787Δ Aida e C600.
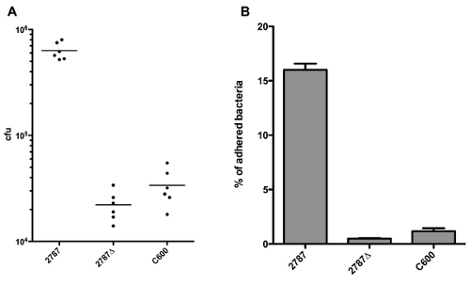
Figura 1. Representação dos resultados, ufc de bactérias aderidas (A) ou a porcentagem de bactérias aderidas (B)
O percentual de adesão observada é freqüentemente muito dependente do set-up experimental (e, em menor medida, no experimentador). Especialmente importantes são os MOI e do número de lavagens, de modo que o percentual não deve ser interpretada literalmente.
As bactérias no inóculo às vezes pode crescer muito mais rapidamente do que as bactérias na presença de células, desviando o tamanho do inóculo, em comparação com o número real total de bactérias em poços com células. Para aliviar este problema, recomenda-se: (i) usar os poços com células para determinar o inóculo: células HEp-2 são semeadas em um poço adicional e infectados. No final do ensaio, em vez de descartar o sobrenadante e lavar roupa, 100 ul de 10% Triton X-100 é adicionado para o bem. Lisar as células e os lisado contendo bactérias aderidas e não aderidas é suavemente homogeneizada por repetidas up-and-down pipetagem;. Ou (ii) recolher os sobrenadantes de células infectadas no final do ensaio, bem como os sobrenadantes dos as lavagens DPBS e determinar o número de ufc nos sobrenadantes reunidos. O resultado será o número de ufc de bactérias não aderiu. A porcentagem de bactérias aderidas pode então ser calculada dividindo o número de ufc de bactérias aderidas pela adição do número de ufc de bactérias aderidas e não aderidas.
Para ilustrar a robustez do ensaio, podemos comparar este protocolo com o ensaio realizado com S4074, uma cepa patogênica adesivo suína de Actinobacillus pleuropneumoniae 7, incubados com as células de uma linhagem de células recém-criada traqueal leitão, nptr 8. Além das diferenças nos meios de comunicação necessários para o crescimento das bactérias e as células, a única diferença é que as bactérias usadas para a infecção são de uma cultura em crescimento exponencial, e não de uma cultura em fase estacionária durante a noite. Com A. pleuropneumoniae é também muito importante respeitar um MOI de 10:1 e um máximo de 3 horas depois da infecção, caso contrário, a secreção de toxinas irá causar a morte celular e viés nos resultados. Além disso, esta cepa de A. pleuropneumoniae pode facilmente aderir ao plástico por isso é importante o uso de células confluentes e verifique visualmente que as células não são sloughing off.
Discussão
Este protocolo descreve um ensaio de adesão padrão de bactérias que podem ser modificados para estudar a invasão (por exemplo, utilizando gentamicina ensaio de proteção 3). A contagem de unidades formadoras de colônia permite quantificar, em comparação com abordagens contando com técnicas de visualização padrão sob um microscópio, como coloração Giemsa. Este último só dá uma visão qualitativa de adesão, mas muitas vezes é um complemento útil, pois pode diferenciar vários padrões de ad...
Divulgações
Agradecimentos
Trabalho nos laboratórios dos autores é suportado por concessões do Ciências Naturais e Pesquisa de Engenharia Council of Canada, os Institutos de Pesquisa em Saúde do Canadá, eo Canadá Research programa presidentes. JL é apoiado por uma bolsa de estudos do Groupe d'Étude des Protéines Membranaires (GEPROM) através do financiamento do Fonds de Recherche en Santé du Québec.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagente | Companhia | Número de catálogo | |
|---|---|---|---|
| DMEM alta glicose | GIBCO-Invitrogen | 12430-054 | |
| DDPBS | GIBCO-Invitrogen | 14190-144 | |
| Serum Crescimento bovina | Hyclone | SH3054 | |
| Penicilina / estreptomicina | GIBCO-Invitrogen | 15140-148 | |
| 24 placas bem | Corning | 3337 | |
| Triton X-100 | Fisher Scientific | BP151-100 |
Referências
- Kline, K. A. Bacterial adhesins in host-microbe interactions. Cell Host Microbe. 5, 580-580 (2009).
- Cossart, P., Boquet, P., Normark, S., Rappuoli, R. Cellular microbiology emerging. Science. 271, 315-315 (1996).
- Elsinghorst, E. A. Measurement of invasion by gentamicin resistance. Methods Enzymol. 236, 405-405 (1994).
- Pizarro-Cerda, J., Lecuit, M., Cossart, P., Sansonetti, P., Zychlinsky, A. . Molecular Cellular Microbiology. , 161-161 (2002).
- Srivastava, A., Isberg, R. R., Sansonetti, P., Zychlinsky, A. . Molecular Cellular Microbiology. , 179-179 (2002).
- Benz, I., Schmidt, M. A. Cloning and expression of an adhesin (AIDA-I) involved in diffuse adherence of enteropathogenic Escherichia coli. Infect Immun. 57, 1506-1506 (1989).
- Charbonneau, M. E., Berthiaume, F., Mourez, M. Proteolytic processing is not essential for multiple functions of the Escherichia coli autotransporter adhesin involved in diffuse adherence (AIDA-I). J Bacteriol. 188, 8504-8504 (2006).
- Jacques, M., Paradis, S. E. Adhesin-receptor interactions in Pasteurellaceae. FEMS Microbiol Rev. 22, 45-45 (1998).
- Auger, E. Host-pathogen interactions of Actinobacillus pleuropneumoniae with porcine lung and tracheal epithelial cells. Infect Immun. 77, 1426-1426 (2009).
- Jacques, M. Role of lipo-oligosaccharides and lipopolysaccharides in bacterial adherence. Trends Microbiol. 4, 408-408 (1996).
- Nataro, J. P., Kaper, J. B. Diarrheagenic Escherichia coli. Clin Microbiol Rev. 11, 142-142 (1998).
- Jallat, C., Darfeuille-Michaud, A., Rich, C., Joly, B. Survey of clinical isolates of diarrhoeogenic Escherichia coli: diffusely adhering E. coli strains with multiple adhesive factors. Res Microbiol. 145, 621-621 (1994).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados