Method Article
Competitiva Screens Genomic de Bibliotecas Yeast Barcoded
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Nós desenvolvemos abrangente, imparcial genome-wide telas para entender gene de drogas e interações gene-ambiente. Métodos para a triagem dessas coleções mutantes são apresentados.
Resumo
Em virtude dos avanços em tecnologias de próxima geração de sequenciamento, temos acesso a novas seqüências do genoma quase que diariamente. O ritmo destes avanços está se acelerando, prometendo uma maior profundidade e amplitude. À luz desses avanços extraordinários, a necessidade de rápido, métodos paralelos para definir a função do gene torna-se cada vez mais importante. Coleções de mutantes do genoma exclusão em leveduras e E. coli têm servido como burros de carga para a caracterização funcional da função dos genes, mas esta abordagem não é escalável, atual gene-apagamento abordagens exigem cada um dos milhares de genes que compõem o genoma a ser excluído e verificada. Somente após esse trabalho é completo pode buscamos high-throughput fenotipagem. Durante a última década, nosso laboratório tem refinado uma carteira de competitivo, miniaturizado, do genoma de alto rendimento ensaios que podem ser executadas em paralelo. Esta paralelização é possível por causa da inclusão de 'tags' DNA, ou "código de barras," em cada mutante, com o código de barras que serve como um proxy para a mutação e pode-se medir a abundância de código de barras para avaliar aptidão mutante. Neste estudo, procuramos preencher a lacuna entre seqüência de DNA e código de barras coleções mutante. Para conseguir isso, apresentamos uma abordagem combinada transposon interrupção código de barras, que abre os ensaios de código de barras paralelas para recém seqüenciados, mas mal caracterizada micróbios. Para ilustrar esta abordagem, apresentamos uma nova Candida albicans coleção rompimento com código de barras e descrever a forma como o microarray-based e próxima geração de plataformas baseadas em seqüenciamento pode ser usado para coletar 10.000 - 1.000.000 gene-gene e gene-drogas interações em um único experimento.
Protocolo
1. Informação de fundo
Existem várias maneiras para gerar mutantes que carregam etiquetas de código de barras. O padrão ouro atual é o fermento KnockOut coleção (YKO) criado por um consórcio de laboratórios e concluída em 2002 1. Desde o YKO original foi introduzida, coleções outra levedura foram gerados, em contextos diferentes cepas, utilizando sobre-expressão construções, e em outros micróbios como E. coli 2. Em paralelo, o esforço para criar bibliotecas de código de barras shRNA está avançando rapidamente, e de fato, muitos dos princípios de design para estas coleções de mamíferos foram adotadas a partir de levedura. Para demonstrar como transposons código de barras pode ser uma estratégia rápida e amplamente aplicáveis para a criação de coleções sistemáticas mutante, nos concentramos em uma coleção que recentemente criado no patógeno humano de fungos, Candida albicans. Nosso trabalho sobre Candida foi baseado no sucesso das telas de código de barras em S. cerevisiae, e é usado aqui como um organismo exemplo. O protocolo de amostra, com pequenas modificações pode ser usado para analisar qualquer organismo que podem ser cultivadas em cultura de suspensão. Porque poucos organismos têm as taxas exigidas alta de transformação e recombinação mitótica eficiente necessário para criar mutantes de deleção perfeito, portanto, nós desenvolvemos um protocolo que utiliza transposon mutagênese in vitro para induz mutações uma biblioteca de DNA genômico, e depois transformado esses fragmentos genômicos com código de barras em Candida albicans 3 , 4. Inspirado pelo sucesso da coleção YKO original e seu papel em descobertas fundamentais sobre a natureza do gene redes 5-8, genome-wide haploinsuficiência 9, alvo da droga eo mecanismo de ação 10,11, e da essencialidade de todos os genes no genoma 12 prevemos alargar esta abordagem a outros micróbios será extremamente frutífera.
O protocolo a seguir pressupõe que a coleção desejada mutante foi criada (por exemplo, YKO ou Candida albicans coleta de perturbação) e está disponível como cepas individualmente arquivados. Para uma descrição detalhada da construção estirpe ver 1,13,14.
2. Combine mutantes individuais em um único pool
- Permitir uma semana para gerar alíquotas de células agrupadas (pode ser armazenada por tempo indeterminado a -80 ° C).
- Descongelar os estoques glicerol congelados para as cepas de interesse completamente, mas não deixe que as células permanecem descongelado para> 2hrs.
- Esterilizar uma ferramenta pin de 96 poços, mergulhe a ferramenta pin em água para remover as células restantes, seguido por dois mergulhos em banhos de etanol a 70% (por exemplo, tampas de caixa de pipeta ponta), chama a ferramenta pin e fresco por 1 minuto. Tome cuidado para chama a ferramenta pin de distância dos banhos de etanol. O nível dos banhos de etanol deve ultrapassar o nível do banho de água para garantir que todos carry-over células são flamed e removido. Substituir a água a cada 4-6 pinnings.
- Insira a ferramenta pin estéril de 96 poços em um redemoinho de 96 poços da placa descongelado, delicadamente e transferir células a uma bandeja Omni Nunc contendo ágar-YPD incluindo o antibiótico adequado. Colônias crescem até atingirem o tamanho máximo a 30 ° C (2-3d). Para conservar placas, achamos mais útil para consolidar quatro placas de 96 bem em um único Omni-bandeja com ~ 384 cepas.
- Depois de colônias cresceram, note qualquer falta ou estirpes de crescimento lento e repin estes em 2X ~ a massa celular como o resto das cepas.
- Trabalhando em um ambiente de microbiologia (com chama e material de laboratório estéril, placa de inundação com a mídia ml 10/05, de molho por 5 min e colônias ressuspender com um espalhador de celular. Despeje o líquido mais células em um tubo de centrífuga 50 ml, e adicionar o glicerol para DMSO 15% ou a 7% (vol / vol).
- Medir o OD 600 da piscina e ajuste (por diluição ou centrifugação) para uma concentração final de 50 OD 600 ml / com a mídia contendo glicerol 15% ou 7% DMSO.
- Alíquota em 40 volumes mL em tubos de strip PCR e congelar a -80 ° C.
3. Crescimento piscina Experimental
Este procedimento é descrito na Figura 1.
- Descongelar alíquotas piscina (em tubos de PCR) no gelo. Se não estiver usando a robótica, pule para o passo 5.
- Imediatamente diluído (delicadamente!) a piscina em mídia com drogas ou condição de escolha, inoculando em um OD 600 de 0,0625 em um volume total de 700 mL em um prato de 48 também. Incluir pelo menos um controle apropriado solvente sobre o prato. Para os experimentos que se estendem para além de 5 gerações de crescimento (ou seja, mais do que um bem), preencher poços adjacentes com a mídia ou a condição de escolha, mas NO células.
- Selar com um selo de placa de plástico, se a condição requer um crescimento aeróbico (por exemplo, as fontes não-fermentável de carbono), use uma agulha de calibre 21 para furar os buracos na vedação de membrana para o lado de cada poço.
- Crescer em um espectrofotômetro, balançando a 30 ° C com uma determinação experimentalned tremendo esquema (por exemplo, agitar 14 min na configuração mais alta (ou temperatura controlada shaker), leia poços, retomar a agitação). Parte da suspensão de células podem ser colhidas pelo robô e salvos em uma placa fria no deck robótica em pontos de geração de user-defined tempo, normalmente 5, 10, 15 e 20 gerações de crescimento. (Http://med.stanford.edu/sgtc/technology/access.html, para contactos C. Nislow ou Giaever G.).
- Para o crescimento celular manual, inocular uma cultura de 50 ml em uma OD de partida 600 de 0.002 em um frasco de 250 ml da cultura. Agite a 30 ° C a 250 rpm até as células chegar a um final de 600 OD de 2,0 (por Saccharomyces ou Candida) para ~ 10 gerações de crescimento. Gerações adicionais de crescimento pode ser obtido pela diluição de células em um OD600 volta de 2,0 a 0,02 num frasco de doce.
- Colheita, pelo menos, OD ~ 2 600 unidades de células para cada amostra / hora ponto em tubos de microcentrífuga Safe-Lock.
Nota: Sempre coletar uma amostra de células de partida (ou seja, um "ponto no tempo T0") para avaliar a representação tensão inicial em qualquer piscina recém-criado pela adição de 1-2 OD 600 de piscina diretamente do freezer alíquotas para um tubo de 1,5 ml e processamento como descrito abaixo.
4. Extração do DNA genômico, PCR e hibridização microarray ou seqüenciamento
- Purificar o DNA genômico de ~ 2 OD 600 de células com a Research Zymo Yeastar kit de acordo com as instruções do fabricante (Protocolo I se estiver usando DNA de levedura), ou outro método adequado específicos para o organismo de interesse (extração de fenol / clorofórmio padrão seguido por precipitação álcool funciona bem para diversos micróbios). Se usar o kit Yeastar, eluir o DNA com 300 mL de 0,1 x em vez do TE mL 60 de 1X TE especificado no protocolo. DNA genômico pode ser armazenada por tempo indeterminado a -80 ° C.
- Configurar duas reações de PCR para cada amostra, um para o uptags e um para o downtags, com as condições de reação da seguinte forma: 33 mL DDH 2 O, 6 mL tampão PCR 10X sem MgCl 2, 3 mL 50 mM MgCl 2, 1,2 mL 10 mM dNTPs, 1,2 mL 50 mM Up ou mistura de iniciador Down, 0,6 mL 5 U / mL de Taq polimerase, 0,1 mg ~ DNA genômico em 15 mL. Volume total é de 60 mL. Thermocycle sob as seguintes condições: 94 ° C min 3, 30 ciclos de 94 ° C 30s, 30s 55 ° C, 72 ° C 30s; então 72 ° C 3min, e mantenha a 4 ° C. Confira os produtos resultantes da PCR em um gel, um produto 60 bp para ambos os PCRs é esperado para amplicons usado para hibridação e 130bp para amplicons para o seqüenciamento do código de barras). Os produtos de PCR podem ser armazenados a -80 ° C por tempo indeterminado.
- Pré-aquecer a temperatura do forno de hibridação a 42 ° C e configurar um banho de água fervente e balde de gelo contendo uma pasta fluida de gelo-água.
- Pré-molhar o arrays por lentamente, com 120 mL 1X tampão de hibridação.
- Incubar no tampão de hibridação a 42 ° C e 20 rpm por 10 minutos.
- Prepare 90 mL de mistura de hibridização por amostra, mais um extra como um buffer, da seguinte forma: 75 mL 2X tampão de hibridação, 0,5 mL de oligonucleotídeos controle B213 (0,2 fm / mL), 12 mL oligonucleotides mista (12:05 / mL), 3 mL solução 50X Denhardt) em lock-top tubos de 0,5 ml.
- Adicionar 30 mL uptag PCR e 30 mL downtag PCR para 120 mL de hibridização mix para um volume total de 150 mL. Ferva por 2 minutos e colocar em água gelada por pelo menos 2 minutos. Brevemente girar os tubos antes do uso.
- Remova o buffer de pré-hibridização dos arrays e adicionar 90 mL de hibridização / mix PCR. Para evitar a evaporação, cubra as juntas matriz com uma situação difícil. Hibridizar durante 16 horas a 42 ° C, 20 rpm.
- Recém preparar 600 mix rotulagem mL biotina por amostra, mais um extra, como segue: 180 mL 20X SSPE, 12 mL 50X Denhardt, 6 mL 1% Tween 20 (vol / vol), 1 ml 1 mg / ml estreptavidina-ficoeritrina, 401 mL DDH 2 O. Armazenar todas as amostras estreptavidina-PE no escuro. Alíquota de 600 mL em tubos de 2 ml. Remover Tough-Spots de chips.
- Remover lentamente mix de hibridização dos arrays com uma pipeta e preencha microarrays com 120 mL Wash A. Primeiro a Affymetrix estação fluídica.
- Lavar os arrays usando uma Affymetrix fluídica estação de acordo com as instruções do fabricante, usando "Gene-Flex_Sv3_450" protocolo com as seguintes modificações: 1 etapa extra com Wash A (1 ciclo, 2 misturas) antes de manchas, lavagem B temperatura 42 ° C em vez de 40 ° C, mancha a 42 ° C em vez de 25 ° C. Também é possível realizar a lavagem de hibridização post, a coloração biotina, a lavagem e coloração pós manualmente (veja p. 396, em referência 15). Seguintes operações de fluidos, execute a estação fluídica "SHUTDOWN_450" protocolo.
- Após a lavagem, garantir que não haja bolhas de ar estão presentes. Se necessário adicionar 90 mL de lavagem A e pipeta lentamente até que as bolhas desapareçam. Se houver quaisquer marcas ou smudges na superfície da matriz, limpar a janela de vidro com isopropanol e um tecido que não solte fiapos. Aplicar novas Tough-Spots sobre as juntas / septos e colocar as matrizes no scanner.
- Digitalizar em um scanner GeneArray Affymetrix em um comprimento de onda de emissão de 560 nm.
5. Análise de matriz (ver Figura 2 para um exemplo obtido utilizando a Candida albicans coleta de interrupção)
- Mascaramento Outlier: Porque o Affymetrix série tag4 contém 5 repetições de cada complemento de código de barras dispersos aleatoriamente qualquer sonda de código de barras que se desvia da replica pode ser mascarado e descartados. Para fazer isso, para cada característica da matriz que parece ser um outlier baseado em seu sinal em comparação com os outros 4 repetições deste recurso, o nosso primeiro software examina a 5 características que cercam o outlier suspeito, produzindo uma matriz de 25 características com o suspeito característica no centro. Se> 13 / 25 sondas na região diferem de cada um dos seus individuais significa duplicar as suas aparadas (a média dos três meio repetições, excluindo a maior ea menor repetições) em mais de 10%, esta sonda é então descartado da análise. Outliers porque tais são na maioria das vezes o resultado de inconsistências hibridização pós-lavagem, nós expandir ou "pad" da região contendo sondas suspeito. Sondas tais Pad, incluindo todas as sondas dentro de um raio de 5 sonda, conforme definido por ((x 1-x 2) 2 + (y 1-y 2) 2) ½ <6, onde x 1, x 2, y 1, e y 2 são as coordenadas x e y para as duas características. Por fim, descartar recursos para que o desvio padrão (incluso no arquivo. Cel para arrays Affymetrix) de pixels característica / média pixels recurso. Depois de remover outliers, valores médios da intensidade para todas as restantes repetições.
- A remoção de tags inutilizável: Tags com baixa intensidade valores dará pobres resultados de qualidade e deve ser removido. Um corte de exclusão para essas sondas de baixa intensidade pode ser calculada da seguinte forma:
- Para qualquer par tratamento de controle de vetores, calcular log 2 ((i c-b g) / (i t-b g)) para cada tag, onde i c é a intensidade de controle, t i é a intensidade do tratamento, e b g é a intensidade média das sondas tag não atribuído.
- Emparelhar o uptag e rácios downtag pela tensão e para cada par de tags, tome a intensidade mínima para as duas marcas em duas amostras. Classificar os pares por esta razão intensidade mínima.
- Use um tamanho de janela de correr de 50 pares na relação de classificados, calcular a correlação de uptag e downtag pares relação dentro da janela. Também calcular a média das intensidades mínimas calculadas no passo anterior.
- Deslize a janela por 25 pares, e repita o passo anterior até que todos os pares foram cruzados.
- Trama a intensidade média mínima em relação a correlação uptag-downtag para todas as janelas.
- Finalmente, escolha de um limiar de intensidade, geralmente usamos o valor de intensidade, onde a primeira correlação atinge 80% do seu nível máximo. Bandeira e remover quaisquer etiquetas análise mais aprofundada abaixo deste limite.
- Correção de saturação: Como cada recurso no microarray de código de barras podem ficar saturadas, o sinal da matriz tag4 não é linearmente relacionada com a concentração tag. Para corrigir essa saturação seguir o protocolo descrito na referência 16.
- Normalização matriz: Para cada matriz, normalizar a uptags e downtags separadamente. Para quantil normalizar, classificar os valores obtidos a partir de cada matriz para uptags e downtags em ordem crescente de intensidade. Para normalizar significa para cada conjunto de uptags e downtags, divida pela média. A normalização média de todas as matrizes é um segundo passo seguinte transformando os dados brutos para a média de cada array.
- Cálculo pontuações sensibilidade para controle do tratamento comparações: Para usar log 2 índices como uma métrica de sensibilidade: Para cada cepa, calcular log 2 ((μ c-b g) / (μ t-b g)), onde μ c é a intensidade média para as amostras controle, μ t é a intensidade média para as amostras, tratamento e b g é a intensidade média das sondas não atribuído. Cepas com uma positiva relação de log 2 são sensíveis ao tratamento, e aqueles que são resistentes têm negativas log 2 rácios.
6. Avaliação da aptidão de cepas de leveduras com código de barras por seqüenciamento
- Isolar o ADN das piscinas exclusão como descrito para microarrays.
- Amplificar a cada 20-mer uptag código de barras com primers composto composto de seqüências dos primers de código de barras comum e as seqüências necessárias para hibridação para a célula de fluxo Illumina (Ver Tabela de primers Illumina e Figura 3 para um diagrama do amplicon). Estesprimers podem ser usados sem purificação adicional dessalinizada. PCR é realizada em 100μL, usando Platinum Invitrogen PCR Supermix (Cat. No. 11306-016) com as seguintes condições: 95 ° C / 3 min, 25 ciclos de 94 ° C/30 segundos, 55 ° C/30 segundos, 68 ° C/30 seg, seguido de 68 ° C/10 min.
- Purificar o produto de PCR (~ 130bp) com Qiagen MinElute 96 UF PCR Purification Kit (Cat. No. 28051).
- Após purificação PCR, quantificar DNA com o Quant-iT Invitrogen dsDNA BR Assay Kit (Cat. Q32853). Não confie em 260/280 leituras!
- Normalizar a concentração de DNA para 10μg/ml e piscina volumes iguais de DNA normalizado.
- DNA agrupados separada em um gel de poliacrilamida 12% TBE por 3-4 horas dependendo da tensão utilizada.
- Mancha géis com brometo de etídio (Sybr Verde deve funcionar também) por 30 minutos.
- Localize a banda de juros sobre uma mesa de luz UV de ondas longas (usando proteção para o rosto adequado), cortá-la para fora e extraiu o DNA usando o método de esmagamento e absorver 17 seguido de precipitação de etanol.
- Confirmar que o DNA de tamanho apropriado (130bp) foi isolado e que primers foram removidos usando o Agilent Bioanalyzer Alta Sensibilidade DNA kit (Cat. 5067-4626).
- Seqüenciamento da amostra:
- Illumina plataforma GAIIx:
- Gerar clusters em um célula de fluxo único Leia utilizando o Kit de Geração CBOT e Single-Leia Cluster (Cat. GD-300-1001). Para Ler 1, UP e DOWN tag modificados primers de seqüenciamento são agrupados em uma concentração de ações 100um e adicionado a um strip-tubo (0.6μL de cada primer seqüenciamento 100um em 120 mL HT1). SR_Amp_Block_StripTubeHyb_v7.0 receita é usada para gerar grupos R1.
- Seqüenciamento do Genoma Analyzer IIx. Após 18 ciclos de seqüenciamento, o módulo emparelhados-end é utilizado para retirar a fita sintetizada primeiro e rehybridize a célula de fluxo, usando o R1 Illumina (abaixo). Clusters são regenerados e seqüenciado para 5 ciclos para capturar a marca de índice.
- Illumina plataforma GAIIx:
- A seqüência tag Index é usado para seqüências bin em caixas experimental.
- Dentro de cada bin experimental, as sequências de código de barras de levedura são computados para dar um número total de contagens para cada código de barras.
- Contagens são normalizados quantil para cada experimento tem a mesma contagem de distribuição. Por analogia com código de barras experiências microarray fitness, índices de fitness defeito para cada cepa são calculados e expressos como a razão log 2 (controle / tratamento). Pontuações positivas defeito de fitness significar uma diminuição na abundância tensão durante o tratamento de drogas e sugerem que a versão do tipo selvagem do gene suprimido em que a tensão é necessária para a resistência a essa droga ou inibidor.
Nota: Considerando Bar seq-como uma alternativa à hibridização matriz. Com os custos de high-throughput seqüenciamento decrescente, usando high-throughput seqüenciamento como uma leitura da abundância tag está se tornando viável e em muitos casos, é mais rentável 18. Desta forma, amplificado produto da PCR é medido diretamente como "conta" ao invés de intensidade de sinal como hibridizados para uma matriz. Isso elimina falsos negativos e positivos que surgem a partir tag contaminação cruzada, a saturação, ou questões decorrentes da intensidade de sinal muito alta ou muito baixa. Além disso, vários experimentos podem ser combinados antes do seqüenciamento pela adição de uma base de DNA 08/04 índice de 19. Porque os códigos de barras de levedura são 20 bp, um único passo 2 leitura de 26-28 bases capta tanto índice multiplex e código de barras único, permitindo extrema 100 + multiplexação. No momento da redação deste texto, Bar-seq oferece uma vantagem de custo sobre código de barras microarrays, e, além disso, Bar-seq é inerentemente flexíveis, que, como aumenta o número de leituras / executado, o nível de multiplexação pode aumentar para diminuir os custos adicionais . Vários "mid-capacidade" sequenciadores de todos os fabricantes de plataformas principais democratizar ainda mais Bar-seq, com seqüenciamento probabilidade de se tornar a leitura de escolha.
Este protocolo também foi validado no HiSeq2000 Illumina.
Uma excelente demonstração do uso de Bar-seq para tratar de uma questão biológica fundamental em Saccharomyces cerevisiae controlar o crescimento é apresentado em um recente estudo de Gresham et al. 20 que descrevem o design experimental e várias importantes orientações de interpretação.
7. Validação dos dados agrupados de triagem
Os resultados a partir de qualquer tela funcional genômica deve ser verificado utilizando as cepas individuais em cultura isolada. Porque cada experiência será diferente em termos do número de cepas sensíveis, selecionando o número de cepas de candidato para confirmar é algo arbitrário. Como um guia, classificando as cepas mais sensíveis pela relação log 2 ou z-score e testar a 25-50% superior dos candidatos (que normalmente se traduz em 03/02 padrão deviations a partir da média de todas as estirpes na piscina) é um bom equilíbrio entre custos e benefícios. Confirmações individuais podem ser realizados em qualquer frasco, mas realizamos esses testes há 5 gerações de crescimento em 96 placas de poço, utilizando um inóculo inicial de 0,06 OD 600 em 100 l de mídia em um espectrofotômetro agitando, fazendo medições a cada 15 minutos (Figura 4) .
8. Resultados representante
Uma vez que uma tela de todo o genoma está completa, e as matrizes foram normalizadas eo comportamento de cada cepa em comparação com um tratamento controle (por exemplo, comparando intensidades microarray ou seqüenciamento contagens / tensão) os dados são mais facilmente manipulados em um arquivo excel com genes classificado pela log2 índices de controle / experiência. Desta forma, maior a proporção log2 negativo, o mais sensível tensão que em particular é a condição de teste. Esses arquivos excel podem ser plotados em uma variedade de pacotes de software gráfico. Encontramos mais simples que a parcela dos rácios log2 no eixo Y eo gene ou nomes ORF no eixo X. No exemplo mostrado na Figura 2a, como traçar um tratamento de clotrimazol (um conhecido agente antifúngico) é mostrado. Toda a tensão que são significativamente sensíveis ao tratamento com uma relação de 2 log2 são destacadas em vermelho, e nós normalmente verificar muitas dessas estirpes em ensaios de crescimento individual de cada mutante na presença da mesma concentração de drogas. Neste exemplo, 4 cepas são destaque, NCP1, ERG2 e 2 alelos independentes de ERG11, o alvo proteína conhecida de clotrimazol. Cada um destes quatro genes está diretamente envolvido na biossíntese de ergosterol, o fermento equivalentes de colesterol. Por exemplo, NCP1 codifica uma NADP redutase-citocromo P450 que está envolvido na biossíntese de ergosterol e que está associado e coordenadamente regulado com Erg11. Este exemplo destaca o fato de que o alvo da droga conhecida (Erg11) é identificado nesta tela imparciais, assim como vários outros componentes-chave da via-alvo. Finalmente, vários dos genes destacado em vermelho representam genes que podem estar envolvidos na biossíntese de ergosterol ou em diferentes processos biológicos. Como mencionado acima, cada cepa detectada como em uma tela sensível agrupados devem ser verificados quanto à sua sensibilidade no ensaio de crescimento individual. No exemplo mostrado na Figura 2b, quatro cepas estão confirmados para ser sensíveis ao clotrimazol com base em seu crescimento diminuiu em relação à estirpe selvagem, BWP17. Estas curvas de crescimento individuais destacar uma característica importante deste gene pool de drogas telas, que é a classificação absoluta de uma cepa especial não refletem, necessariamente, o seu nível exacto de sensibilidade. Além disso, a Figura 2b mostra também o valor de ter múltiplos alelos para cada gene, neste caso, os dois mutantes erg11 perturbação têm ligeiramente diferentes sensibilidades. Correlacionando a natureza dessas rupturas com o grau de sensibilidade pode fornecer informações adicionais sobre o mecanismo de ação de drogas.
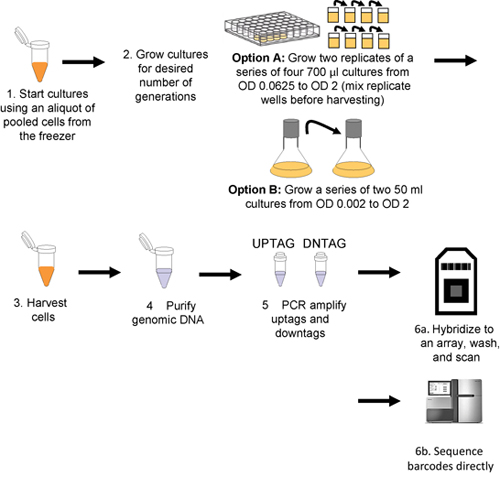
Figura 1. Workflow para ensaio de crescimento agrupados e detecção de código de barras. Culturas são inoculadas com alíquotas de células descongeladas pooled (passo 1), e então cultivadas para o número desejado de gerações (passo 2) ou roboticamente (Opção A) ou manualmente (Opção B). As células são colhidas por centrifugação (passo 3) e DNA genômico é, então, isolado a partir de células colhidas (passo 4), e uptags downtags são independentemente amplificado (passo 5) e hibridizados para uma matriz (passo 6a ou seqüenciados diretamente passo 6b).
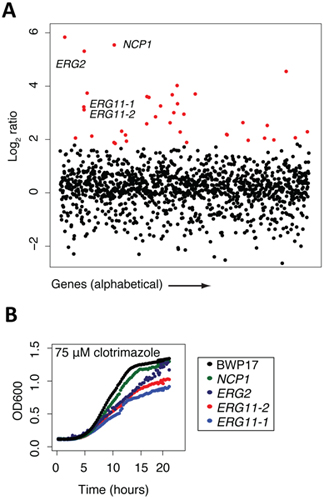
Figura 2. Os dados da amostra coletada em certos pontos do protocolo. (A) Os dados da amostra a partir dos resultados de triagem (adaptado de 13). A piscina de mutantes tag foi cultivado por 20 gerações, na presença de clotrimazol e DMSO (controle). Log 2 ratio (intensidade controle de intensidade / tratamento) foi calculado e plotado como uma função do gene. Cepas altamente sensível (vermelho), incluído o alvo conhecido de clotrimazol, ERG11p. Note-se que este ensaio freqüentemente descobre outros mutantes sensíveis, além de alvo real do composto. Geralmente, esses mutantes são aqueles que agem de forma sintética com o alvo, aquelas que fazem parte de uma resposta ao estresse / tratamento geral, ou são falsos positivos que não conseguem confirmar. (B) Exemplo de dados de confirmação (adaptado de 13). Resultados dos ensaios de crescimento combinada pode ser validado pelo crescente a tensão na cultura individual e comparada com o crescimento do tipo selvagem (preto).
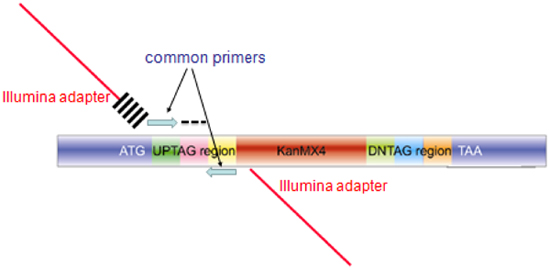
Figura 3. Estrutura do amplicon produzido a partir de ensaios de código de barras agrupados para hibridização microarray ou sequenciamento Barcode. O amplicon produzido para cada mutante in a coleção contém homologia com o genoma para a integração (regiões azuis rotulados ATG e TAA), códigos de barras único (identificado como AG e indicado por um traço preto). Para a hibridização microarray, os primers azul comuns são usados para amplificar uma sonda para a hibridização 60bp microarray. Para o seqüenciamento do código de barras, primers estendida são usados na reação de PCR, composto de seqüências de codificação o adaptador Illumina (barra vermelha), e índice de 6bases cross-escotilhas) eo primer azul comum para o primer a montante, eo primer mesmo composto (menos o índice de base 6) para o primer segundo.
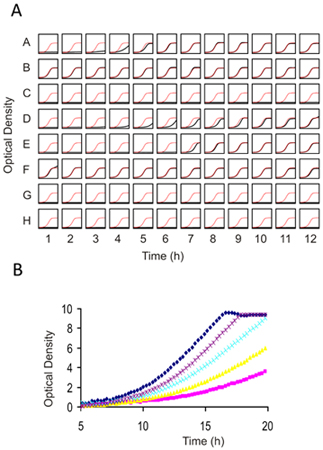
Figura 4. Ensaios de crescimento individual para 1) prescreening compostos contra fungos do tipo selvagem para determinar uma dose apropriada para o genoma de despistagem e 2) Confirmando os resultados de telas genoma de largura. (A) A 96 placa de fundo bem plano é preenchido com 100 ml de suspensão de células em uma OD de 0,062. Cada poço pode conter a mesma estirpe (para a dose-determinação) ou combinações diferentes tensão e de drogas (para ensaios de confirmação). 2 l de composto (normalmente dissolvido em DMSO) é adicionado e as células são cultivadas com constante agitação por 16-20 horas a 30 ° C. A concentração final de DMSO não deve exceder 2%. Neste exemplo, em cada poço da placa da curva de crescimento é plotado em preto contra um gráfico da curva de crescimento de controle em vermelho. (B) imagem com resolução mais alta de prescreens várias obtido com uma droga exemplo sobrepostos em cima da outra. Nesta série de titulação, uma IC 15/10 é obtido com a dose de roxo e seria adequado para o perfil de eliminação (HIP HOP e). Devido à não-linearidade de maior densidade óptica, Tecan (ou qualquer leitor de placas similar) ODs deve ser calibrado utilizando os obtidos com um 1 milímetro "tradicional" cuvete caminho de comprimento.
Discussão
Aqui, esboçamos um protocolo que, com a alteração modesta, pode ser facilmente adaptado a uma ampla gama de colecções existentes de código de barras coleções mutantes de microorganismos diferentes para criar coleções tag mutantes. Ressaltamos que, embora tenhamos relatado um protocolo sobre a mutagênese transposon marcadas para a levedura patogênica C. albicans, um protocolo muito semelhante poderia ser adaptado a uma grande variedade de fungos unicelulares. Modificada, este protocolo funciona bem em bactérias 13 e, atualmente, coleções para um número adicional de genomas de fungos e bactérias estão em construção. Atualmente, este ensaio fornece a única abrangente, a tela do genoma imparcial para interações gene-pequena molécula. Um aspecto particularmente atraente do ensaio é que nenhum conhecimento prévio do gene ou molécula pequena é necessária. Apesar do alcance e do poder destes ensaios, a sua transferência para outros laboratórios tem sido dificultada pelo pouco investimento de capital inicial e as ferramentas de informática para a análise dos resultados. Com a adoção de uma leitura próxima geração seqüência combinada com ferramentas robustas para análise, esperamos a sua aprovação a aumentar.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Agradecemos a Ron Davis, Adam Deutschbauer, eo laboratório HOP toda HIP da Universidade de Toronto para discussões e aconselhamento. CN é suportado por concessões do National Human Genome Research Institute (Grant Número HG000205), RO1 HG003317, CIHR MOP-84305, e Canadian Cancer Society (# 020380). JO foi apoiado pelo Programa de Formação de Stanford Genome (Grant Número T32 HG00044 do National Human Genome Research Institute) e os Institutos Nacionais de Saúde (Grant Número GH000205 P01). GG é suportado pelo RO1 NHGRI HG003317 e da Sociedade Canadense do Câncer, Grant # 020380, TD e do Centro de Seqüenciamento Donnelly é suportado, em parte por doações da Fundação Canadense para Inovação, para as Dras. Brenda e Jack Andrews Greenblatt. AMS é apoiado por uma Universidade de Toronto Fellowship Open.
Materiais
| Name | Company | Catalog Number | Comments |
| Antibióticos | Número de fornecedores e catálogo | ||
| Carbenicilina | Sigma, parte # C1613 | ||
| Canamicina | Sigma, parte # K1876 | ||
| espectinomicina | Sigma, parte # S0692 | ||
| Cloranfenicol | Sigma, parte # C0378 | ||
| DNA Clean-up e kits de concentração | |||
| QIAprep rotação Miniprep Kit | Qiagen, parte # 27106 | ||
| Kit Maxi HiSpeed Plasmid | Qiagen, parte # 12663 | ||
| QIAquick Kit de Purificação PCR | Qiagen, parte # 28106 | ||
| QIAquick Gel Extraction Kit | Qiagen, parte # 28704 | ||
| PCR e reagentes eletroforese | |||
| Taq DNA polimerase (Mg-free) do buffer | New England Biolabs, parte # M0320L | ||
| Mix Solução desoxinucleotídeo | New England Biolabs, parte # N0447L | ||
| 25 mM MgCl 2 | Sigma, parte # 63036 | ||
| Agarose, corante e ácido nucléico mancha adequado para eletroforese em gel | Vários | ||
| Tampão TAE 10X | Sigma, parte # T8280 | ||
| 1 Kb DNA Ladder Mais | Invitrogen, parte # 10787026 | ||
| YPD caldo | |||
| 10 g de extrato de levedura | Sigma, parte # Y1625 | ||
| 20 g de peptona Bacto | BD Biosciences, parte # 211677 | ||
| 20 g de dextrose | Sigma, parte # D9434 | ||
| Material de laboratório | |||
| Pipetas multicanal (1000, 200 e 20 mL) | Vários | ||
| Descartáveis reservatórios pipetagem | Vários | ||
| 15 e 50 mL tubos de centrífuga | Vários | ||
| 96 - e 384-poço profundo Placas Bem | Axygen Scientific, parte # P-2ML-SQ-CS & P-384240SQCS | ||
| De 96 poços e 384 placas de bem-PCR e filme de vedação | Vários | ||
| Rolo de chapa para a selagem de poços múltiplos placas | Sigma, parte # R1275 | ||
| 30 ° C e 37 ° C agitando incubadoras para o crescimento de bactérias e leveduras em placas e em tubos | Vários | ||
| In vitro transposon mutagênese | |||
| EZ-Tn5 transposase | Biotecnologias Epicentro, parte # TNP92110 | ||
| Alto rendimento de transformação | |||
| Seqprep 96 HT Kit Prep Plasmid | Borda Biosystems, parte # 84359 | ||
| polietileno glicol, peso molecular 3350 | Vários | ||
| acetato de lítio | Sigma, parte # 517992 | ||
| 6-bem pratos, estéril | Corning, parte # 3335 | ||
| 50 mg / mL uridina | Sigma, parte # U3750 | ||
| 100X solução tampão Tris-EDTA | Sigma, parte # T9285 | ||
| 1X TE/0.1M LiOAc | Vários | ||
| salmão testículo DNA | Sigma, parte # 1626 | ||
| Crescimento das coleções de código de barras | |||
| 48 placas bem, se crescer culturas em placas | Greiner, parte # M9437 | ||
| Colantes placa | ABgene, parte # AB-0580 | ||
| 200 frascos de cultura mL | Vários | ||
| Espectrofotômetro capaz de absorbância | Vários | ||
| Temperatura controlada shaker para balões de 250 ml ou espectrofotômetro agitação | Vários | ||
| Seguro-Lock tubo de microcentrífuga, 2 mL | Eppendorf, parte # 0030 120.094 | ||
| Equipamentos de hibridização | |||
| Forno de hibridização 640 | Affymetrix, parte # 800138 | ||
| GeneChip Fluidic Station 450 | Affymetrix, parte # 00-0079 | ||
| GeneArray Scanner 3000 | Affymetrix, parte # 00-0212 | ||
| Banho de água fervente com suporte flutuante | Vários | ||
| Consumíveis de hibridização | |||
| Genflex Tag Matriz 16K v2 | AffymEtrix, parte # 511331 | ||
| Solução Denhardt, a concentrar-50X | Sigma, parte # D2532 | ||
| Estreptavidina, R-ficoeritrina conjugada (SAPE) | Invitrogen, parte # S866 | ||
| Seguro-Lock tubo de microcentrífuga de 0,5 mL | Eppendorf, parte # 0030 123.301 | ||
| Teeny Tough-Spots | Diversified Biotech, parte # LTTM-1000 | ||
| 0,5 M EDTA | BioRad, parte # 161-0729 | ||
| 10% Tween | Sigma, parte # T2700 | ||
| MES monohidrato do ácido livre | Sigma, parte # M5287 | ||
| MES sal de sódio | Sigma, parte # M5057 | ||
| 5 M NaCl | Sigma, parte # 71386 | ||
| 20X SSPE | Sigma, parte # S2015 | ||
| Água de grau de biologia molecular | Sigma, parte # W4502 | ||
| Primers hibridação | Diversos fornecedores (dessalinização standard) | ||
| Uptag | "GATGTCCACGAGGTCTCT 3 '5 | ||
| Buptagkanmx4 | 5 'biotina-GTCGACCTGCAGCGTACG 3' | ||
| Dntag | 5 'CGGTGTCGGTCTCGTAG 3' | ||
| Bdntagkanmx4 | 5 'biotina-GAAAACGAGCTCGAATTCATCG 3' | ||
| B213 | 5 'biotina-CTGAACGGTAGCATCTTGAC 3' | ||
| Uptagkanmx | 5 'GTCGACCTGCAGCGTACG 3' | ||
| Dntagkanmx | 5'GAAAACGAGCTCGAATTCATCG 3 ' | ||
| Uptagcomp | 5'AGAGACCTCGTGGACATC 3 ' | ||
| Dntagcomp | 5'CTACGAGACCGACACCG 3 ' | ||
| Upkancomp | 5'CGTACGCTGCAGGTCGAC 3 ' | ||
| Dnkancomp | 5'CGATGAATTCGAGCTCGTTTTC 3 ' | ||
| Seqüenciamento Primers: Plataforma Illumina | Diversos fornecedores | ||
| UpTag Forward (100um) | 5 'CAA GGC GAC GAA GCA ATA CGA GCT CTT ATC CCG T GAT GTC GTC GAG CAC TCT 3' | ||
| UpTag Reverse (100um) | 5 'GAT AAT ACG GCG ACC ACC GAC ACT CTT TCC CTA CAC GAC GCT CTT ATC CCG T NNNNN GTC GAC CTG CAG CGT ACG 3' | ||
| DownTag Forward (100um) | 5 'CAA GGC GAC GAA GCA ATA CGA GCT CTT ATC CCG T GAA GAG AAC CTC TTC GAA ATC G 3' | ||
| DownTag Reverse (100um) | 5 'GAT AAT ACG GCG ACC ACC GAC ACT CTT TCC CTA CAC GAC GCT CTT ATC CCG T NNNNN CGG TGT CGG TCT CGT AG 3 ` | ||
| Leia um UP-tag cartilha seq (100um) | 5 'GAC GTC CTG CAG CGT ACG 3' | ||
| Leia um BAIXO tag-primário seq (100um) | 5 'CGG TGT CGG TCT CGT AG 3' | ||
| Ler 2 cartilha Seqüenciamento Index (padrão primário Illumina R1) (100um) | 5 'ACT AC CTT TCC CTA CAC GAC GCT CTT ATC CCG T 3' | ||
| Reagentes adicionais Seqüenciamento / Equipamentos | |||
| Qiagen MinElute 96 UF Kit de purificação de PCR | Qiagen, parte # 29051 | ||
| Bomba de vácuo | Qualquer fornecedor | ||
| Macherey-Nagel Manifold Vacuum | Macherey-Nagel, parte # 740 681 | ||
| Invitrogen Quant-iT dsDNA BR Assay Kit | Invitrogen, parte # Q32853 | ||
| Ensaio Invitrogen tubos Qubit | Invitrogen, parte # Q32856 | ||
| 40 acrilamida% mais 1% N, N'-metileno-bis-acrilamida, 37.5:1 | Bio Rad, parte # 161-0148 | ||
| Tris Base de Dados | Sigma, parte # T1503-1KG | ||
| Ácido bórico | Sigma, parte # B6768-500G | ||
| EDTA 0,5 M, pH 8,0 | Teknova, parte # E0306 | ||
| Persulfato de amônio | Sigma, parte # A3678-25G | ||
| Temed | Bioshop, parte # TEM001.25 | ||
| Solução de brometo de etídio | Bioshop, parte # ETB444.10 | ||
| Acetato de amônio 0,5 M | Teknova, parte # A2000 | ||
| Tetra acetato de magnésio 10mM | Sigma, parte # M0631-100G | ||
| 1mM EDTA, pH 8,0 | Ver EDTA 0.5M, pH 8.0 | ||
| Etanol | Vários | ||
| Acetato de sódio, pH 5,2 | Teknova, parte # S0297 | ||
| Vácuo velocidade | Vários | ||
| Leia Kit Single-Generation Cluster | Illumina, parte # GD-300-1001 | ||
| 36c Seqüenciamento Kit v4 | Illumina, parte # FC-104-4002 | ||
Receita TBE 10X
| Quantidades | Reagentes |
| 108 gramas | Tris Base de Dados |
| 55 gramas | Ácido bórico |
| 40mL | EDTA 0,5 M (pH 8,0) |
| Adicionar dH 2 O para 1L marca | |
12% da receita em gel de poliacrilamida
| Volumes | Reagentes |
| 5,8 ml | 40 acrilamida% mais 1% N, N'-metileno-bis-acrilamida, 37.5:1 |
| 12 ml | dH 2 O |
| 2 ml | TBE 10X |
| 140 mL | Persulfato de amônio 10% |
| Volume total: 20ml |
Uptag mistura de iniciadores:
Ressuspender Uptag e Buptagkanmx4 cada um em DDH 2 O a 100 mM, em seguida, misture em uma proporção de 1:1 para uma concentração final de 50 mM cada um. Armazenar a -20 ° C.
Downtag mistura de iniciadores:
Ressuspender Dntag e Bdntagkanmx4 cada um em DDH 2 O a 100 mM, em seguida, misture em uma proporção de 1:1 para uma concentração final de 50 mM cada um. Armazenar a -20 ° C.
Oligonucleotides misto:
Ressuspender cada um dos seguintes oito oligos (standard já dessalgado) em DDH 2 O a 100 mM:
Uptag, Dntag, Uptagkanmx, Dntagkanmx, Uptagcomp, Dntagcomp, Upkancomp, Dnkancomp.
Misturar um volume igual de oito oligonucleotídeos para uma concentração final de 12,5 mM cada um.
12X de ações MES:
Para 10 ml, dissolver 0,7 g MES monohidrato do ácido livre e 1,93 g de sal de sódio MES em 8 ml de água de grau de biologia molecular. Depois de misturar bem, verificar o pH e se necessário ajustar a um pH 6,5-6,7. Adicionar água a um volume total de 10 ml. Filtro de esterilizar e armazenar a 4 ° C protegido da luz (por exemplo, envolva o tubo em papel alumínio). Substituir se a solução torna-se visivelmente amarelo ou após 6 meses.
2X tampão de hibridação:
Para 50 ml, misturar 8,3 ml de 12X MES estoque (de 2.9.14), 17,7 ml de 5 M NaCl, 4,0 ml de 0,5 M EDTA, 0,1 ml de 10% Tween 20 (vol / vol), e 19,9 ml filtrados DDH 2 O. Filtro de esterilizar.
Lavar A: Misturar 300 ml 20X SSPE, 1 mL de Tween 10% (vol / vol), 699 ml DDH 2 O. Filtro de esterilizar.
Lavar B: Misture 150 ml 20X SSPE, 1 mL de Tween 10% (vol / vol), 849 ml DDH 2 O. Filtro de esterilizar.
Seqüenciamento primers de código de barras
Em seqüências UP tag-primário a 5 'da cauda (negrito) são seqüências de Illumina adaptador específico incorporado o primer F e R. A seqüência variável (itálico) representa a marca de indexação 5-mer usado em multiplexação / index de leitura. A cauda do 3 '(sublinhado) representa o primer comum de acompanhamento do código de barras uptag e é necessário para ampliar os códigos de barras de levedura.
Em Down tag-primário seqüências 5 'cauda é idêntica à 5' cauda de UP-tag primers (seqüência específica Illumina), no entanto, a 3 'cauda (sublinhado) é substituído pelo primers comuns que são usados para amplificar o DOWN- tag barcodes.
Referências
- Giaever, G. Functional profiling of the Saccharomyces cerevisiae genome. Nature. 418, 387-391 (2002).
- Baba, T. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular systems biology. 2, 2006.0008-2006.0008 (2006).
- Claus, H., Frosch, M., Vogel, U. Identification of a hotspot for transformation of Neisseria meningitidis by shuttle mutagenesis using signature-tagged transposons. Mol Gen Genet. 259, 363-371 (1998).
- Hava, D. L., Camilli, A. Large-scale identification of serotype 4 Streptococcus pneumoniae virulence factors. Molecular microbiology. 45, 1389-1406 (2002).
- Costanzo, M. The Genetic Landscape of a Cell. Science. 327, 425-431 (2010).
- Tong, A. H. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Pan, X. A robust toolkit for functional profiling of the yeast genome. Molecular cell. 16, 487-496 (2004).
- Schuldiner, M. Exploration of the Function and Organization of the Yeast Early Secretory Pathway through an Epistatic Miniarray Profile. Cell. 123, 507-519 (2005).
- Deutschbauer, A. M. Mechanisms of haploinsufficiency revealed by genome-wide profiling in yeast. Genetics. 169, 1915-1925 (2005).
- Giaever, G. Chemogenomic profiling: identifying the functional interactions of small molecules in yeast. Proceedings of the National Academy of Sciences of the United States of America. 101, 793-798 (2004).
- Lum, P. Y. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116, 121-137 (2004).
- Hillenmeyer, M. E. The chemical genomic portrait of yeast: uncovering a phenotype for all genes. Science. 320, 362-365 (2008).
- Oh, J. A universal TagModule collection for parallel genetic analysis of microorganisms. Nucleic acids research. 38, e146-e146 (2010).
- Oh, J. Gene annotation and drug target discovery in Candida albicans with a tagged transposon mutant collection. PLoS pathogens. 6, (2010).
- Nislow, C., Giaever, G., Stark, I., Stansfields, M. J. R. Chapter 387. Yeast Gene Analysis. , 387-414 (2007).
- Pierce, S. E., Davis, R. W., Nislow, C., Giaever, G. Genome-wide analysis of barcoded Saccharomyces cerevisiae gene-deletion mutants in pooled cultures. Nature protocols. 2, 2958-2974 (2007).
- Sambrook, J., Russell, D. W. . Molecular cloning : a laboratory manual. , (2001).
- Smith, A. M. Quantitative phenotyping via deep barcode sequencing. Genome Res. , (2009).
- Hamady, M., Walker, J. J., Harris, J. K., Gold, N. J., Knight, R. Error-correcting barcoded primers for pyrosequencing hundreds of samples in multiplex. Nature. 5, 235-237 (2008).
- Gresham, D. System-Level Analysis of Genes and Functions Affecting Survival During Nutrient Starvation in Saccharomyces cerevisiae. Genetics. 187, 299-317 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados