É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Extração de DNA de alto peso molecular de Esteiras Microbianas
Neste Artigo
Resumo
Nós fornecemos um protocolo melhorado para a extração de DNA de alto peso molecular de tapetes microbianos hipersalinos. Células microbianas são separados da matriz mat antes da extração e purificação DNA. Isso aumenta a concentração, qualidade e tamanho do DNA. O protocolo pode ser usado para outras amostras refratário.
Resumo
Análise bem sucedida e precisas e interpretação de dados metagenomic é dependente da extração eficiente de alta qualidade, de alto peso molecular DNA comunidade (HMW). No entanto, as amostras de mat ambientais muitas vezes apresentam dificuldades para a obtenção de grandes concentrações de DNA de alta qualidade HMW,. Tapetes microbianos hipersalinos contêm grandes quantidades de substâncias poliméricas extracelulares (EPS) 1 e sais que podem inibir a aplicações a jusante do DNA extraído. Métodos diretos e duras são muitas vezes utilizados na extração de DNA a partir de amostras refratário. Estes métodos são normalmente utilizados, pois o EPS em esteiras, uma matriz adesiva, liga-se 2,3 DNA durante a lise direta. Como resultado de métodos mais duros de extração, o DNA torna-se fragmentado em tamanhos pequenos 4,5,6.
O DNA, assim, torna-se inadequado para os grandes inserir clonagem do vetor. A fim de contornar essas limitações, apresentamos uma metodologia melhorada para extrair DNA HMW de boa qualidade e quantidade de tapetes microbianos hipersalinos. Nós empregamos um método indireto envolvendo a separação de células microbianas a partir da matriz mat fundo através de mistura e centrifugação diferencial. A combinação de procedimentos mecânicos e químicos foi usado para extrair e purificar o DNA das células microbianas extraído. Nosso protocolo produz cerca de 2 mg de HMW DNA (35-50 kb) por grama de amostra de esteira, com um A 260 / 280 relação de 1,6. Além disso, a amplificação do 16S rRNA genes 7 sugere que o protocolo é capaz de minimizar ou eliminar os efeitos inibitórios de contaminantes. Nossos resultados fornecem uma metodologia apropriada para a extração de DNA a partir de HMW tapetes microbianos para estudos funcionais metagenomic e pode ser aplicável a outras amostras ambientais a partir do qual a extração de DNA é um desafio.
Protocolo
1. Extração de células microbianas:
- Homogeneizar tapetes microbianos com um pilão de moer estéril, misturando bem. Coloque todos os cerca de 30 g (peso úmido) de material mat homogeneizadas em recipiente estéril de liquidificador Waring, adicionar cerca de 100 mL de NaCl 1 M (ou uma concentração específica para amostra em uso), e mistura três vezes em velocidade média por 1 min com resfriamento intermitente em um freezer -20 ° C por 1 min. Transfira a pasta em um frasco de centrífuga de 250 mL e completar o volume vazio restante com 1 M NaCl. Prepare a solução de NaCl em água autoclavada DI e filtro de esterilizá-lo.
- Além disso desalojar as células microbianas a partir da matriz agitando (150 rpm) usando um vortexer (Vortex-Genie R 2) em temperatura ambiente por 30 min.
- Centrifugar a baixa velocidade (500x g) por 15 min a 4 ° C. Gentilmente transferir o sobrenadante para um balão com perturbação mínima para o sedimento.
- Usando o sedimento peletizada, repita os passos de mistura e 1.2 e 1.3 quatro vezes mais com a adição de NaCl fresco para cada pellet de sedimentos no final do passo 1.3. O sobrenadante de cada extração de NaCl é transferido para um balão fresco.
- Combine os sobrenadantes dos 5 celulares extrações e centrifugar em alíquotas de 200 mL em alta velocidade (25.000 xg) por 15 min a 4 ° C. Desprezar o sobrenadante, ressuspender cada pellet celular em 10 mL de hexametafosfato de sódio a 2%, combine pellets e fazer aumentar o volume para 200 mL com hexametafosfato de sódio a 2%. Lave as células por agitação (150 rpm) em temperatura ambiente por 30 min. No final desta etapa você deve ter apenas um tubo de células para prosseguir para a próxima etapa.
- Centrífuga em alta velocidade (25.000 xg) por 15 min a 4 ° C. Desprezar o sobrenadante, ressuspender pellet celular em 200 mL de TE (50 mM EDTA), e lavar as células por agitação (150 rpm) em temperatura ambiente por 10 min.
- Centrífuga em alta velocidade (25.000 xg) por 15 min a 4 ° C, desprezar o sobrenadante, ressuspender em 15 mL de TE (10 mM EDTA), e armazenar de 200 mL alíquotas a -80 ° C até ser necessário para DNA extrações.
2. Extração de DNA e Purificação:
- Adicionar 200 mL de NaCl 5 M, 200 mL SDS 10%, e de 100 L 14,3 M β-mercaptoetanol para a alíquota de 200 mL de células microbianas. Misture delicadamente por inversão 4-6 vezes.
- Mistura o assunto para 3 rounds de congelamento e descongelamento por submersão em nitrogênio líquido por 2 minutos seguido de descongelamento em banho-maria a 65 ° C por 5 min. Estender finais descongelamento a 10 min.
- Adicionar 200 mL de acetato de potássio 5 M (pH 5,5) e colocar no gelo por 10 min. Centrifugar a 10.000 xg por 10 min a 4 ° C e transferir o sobrenadante para um novo tubo usando uma ponteira de grande furo. Adicionar 3 mL de RNase A e incubar a 37 ° C por 1h.
- Adicionar igual volume de clorofórmio, agitar brevemente por inversão, e centrifugar a 15.000 xg por 10 min em temperatura ambiente.
- Transferir cuidadosamente a camada superior do sobrenadante para um novo tubo usando uma ponteira de grande furo. Evite pipetagem a camada de interface branco, localizado entre as camadas superior e inferior. Repita o procedimento de extração clorofórmio.
- Adicionar um volume igual de refrigerados (a -20 ° C) isopropanol para o sobrenadante e incubar no gelo por 30 min para o DNA precipitado.
- Centrifugar à máxima velocidade (20.800 xg) por 10 min a 4 ° C para pellet de DNA.
- Desprezar o sobrenadante e lavar DNA pellet com 1 mL de refrigerados (a -20 ° C) etanol 70%.
- Centrifugar à máxima velocidade (20.800 xg) por 10 min a 4 ° C, desprezar o sobrenadante, repita lavar etanol 70%, e de ar seco pellet de DNA por 10 min. Repita lavar etanol 70%.
- Ressuspender o DNA em 25 mL TE (10 mM EDTA), quente a 65 ° C por 5 min, se combinam em um tubo se tubos múltiplos têm sido utilizados, e make-up volume de 500 mL com TE.
- Adicionar 500 mL gelada (ou a 4 ° C) 20% de polietileno glicol (PEG) (preparado em 1,2 M NaCl). Misture delicadamente por inversão e incubar no gelo durante 10 min, e centrifugar a velocidade máxima por 10 min a 4 ° C para pellet de DNA. Remover e descartar o sobrenadante e lavar o DNA peletizadas com 1 mL de etanol 70% gelado, ar seco por 10 min, e ressuspender em 30-50 mL TE ou água de grau molecular. Facilitar a ressuspensão de DNA por aquecimento a 65 ° C por 5 min e DNA loja a -80 ° C.
3. Purity DNA, Concentração e Determinação Tamanho:
- Determinar a qualidade do DNA através da medição da 260/280 A e A 260/230 relações usando um espectrofotômetro 1000 Nanodrop ou quaisquer outros instrumentos apropriados.
- Determinar a concentração de DNA usando Quant-iT dsDNA kit de Ensaio de acordo com as instruções do fabricante.
- Determinar o tamanho do DNA usando pulso eletroforese em gel de campo. Prepare 1% agarose gel com 0,5 X TBE e usar os seguintes parâmetros na Mapper CHEF XA System; tempo de troca inicial de 0,35 s, um tempo de troca final de 7,67 s, e 120 ° incluído ângulo de um gradiente de 6,0 V / cm em um r linearamplificação fator. Mancha gel com 1 x SYBR Green I para 30 min. Carga entre 700-1000 ng DNA total em gel. Ver fig. 3 para uma forma condensada do protocolo completo.
Resultados representativos:
Extração de células:
Extratos microbianos seqüencial celular (sobrenadante) mostraram que a turbidez dos extratos diminui à medida que aumenta o número de extrações. Isto sugere uma redução no número de células após cada extração de células sucessivas. Importante notar aqui é que, como cada etapa de extração de células adicionais proporciona a oportunidade para a introdução de contaminantes, o número de células extrações devem ser minimizados para que o final de pellet celular é representante da comunidade microbiana total. Outras fontes de contaminação foram controlados através da adopção de técnicas de laboratório adequado estéril. Por exemplo, as soluções foram preparadas em água DI autoclavado e esterilizado por filtração. Recipientes foram esterilizados com álcool, autoclavados e tratados sob luz UV e reticulador ultravioleta.
Concentração de DNA e determinação de qualidade:
O protocolo rendeu cerca de 2 mg de HMW DNA (35-50 kb) (Fig. 4) por grama de amostra tapete com um A 260 / 280 relação de 1,6, e um A proporção de 260/230 0,7 (Tabela 1). Embora a razão A 260 / 230 parece ser baixa, sem inibição de aplicação de base molecular a jusante, como a amplificação do 16S rRNA genes foi observado em um estudo separado 7. É importante notar que o DNA de hipersalinos esteiras tem duas principais fontes de contaminação; EPS e sais. Portanto, é possível que traços destes contaminantes podem estar influenciando a A 260/230 relações, apesar dos esforços tremendos para reduzir seus efeitos sobre as aplicações de fluxo baixo.
DNA determinação do tamanho:
Pulso de eletroforese em gel de campo emprega um fluxo pulsante atual com interruptores intermitentes direcional, resultando em um esfregaço de DNA, como mostrado na figura 3. Nosso protocolo rendeu um DNA HMW de aproximadamente 35-50 kb. Enquanto alguns tamanho DNA pode ser menor que 30 kb, é importante ter parte do esfregaço acima de 35 kb dado que a clonagem fosmid requer ~ 40 kb de DNA e insere fragmentos maiores de DNA proporcionar maior acesso às vias biossintéticas intacta. Em nossos estudos, o esfregaço DNA acima de 35 kb foi retirado e purificada de grande vetor de inserção clonagem e outras aplicações molecular.

Figura 1. Microbiana hipersalinos local de amostragem mat (Lagoa Grande) localizado na Eleuthera, nas Bahamas.

Figura 2. A seção transversal do tapete microbiano hipersalinos utilizados neste estudo. O tapete microbiano foi obtido a partir de uma lagoa hipersalina localizado na Eleuthera, nas Bahamas.
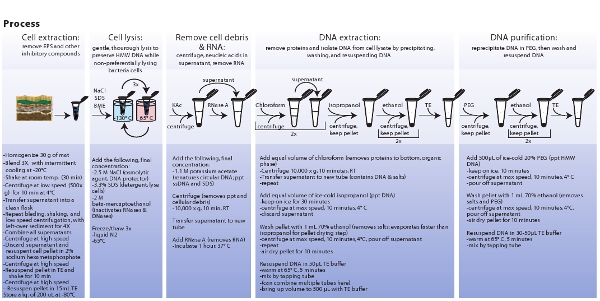
Figura 3. Representação esquemática dos processos envolvidos na extração de células microbianas, lise celular, extração e purificação do DNA metagenomic. Clique aqui para ver uma imagem maior.

Figura 4. Caracterização do peso molecular do DNA extraído usando pulso eletroforese em gel de campo. Pista M é um marcador HMW, e faixas 1 e 2 são réplicas de DNA metagenomic extraído do tapete hipersalinos Eleuthera usando o protocolo acima.
| Método de extração | Concentração ng / g | A 260/280 | A 260/230 | |
| PEG | Rep 1 | 1806,1 | 1,64 | 0,76 |
| Rep 2 | 2010,8 | 1,61 | 0,75 |
Tabela 1. A medição da concentração e qualidade do DNA extraído de mat hipersalinos microbiana.
Access restricted. Please log in or start a trial to view this content.
Discussão
Dado que a remoção total de células a partir de amostras mat complexa e altamente diversificada microbiana não é prático, a principal preocupação é o quão bem as células extraídas representar a comunidade microbiana total mat. Em um estudo anterior, PCR-DGGE análise dos genes 16S rRNA microbiana mostrou que as cinco etapas de remoção de células utilizadas neste protocolo de extratos de células que são representativas da comunidade em geral tapete microbiano 7. O número real de extração de...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Agradecimentos
Este trabalho foi financiado pela National Science Foundation Programa Ambiental Genomics (Grant No. EF-0723707).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
|---|---|---|---|
| β-mercaptoetanol | Sigma-Aldrich | M3148 | |
| Polietileno glicol 8000 | Promega | V3011 | 20% em 1,2 M NaCl |
| Acetato de potássio | Fisher Scientific | Fisher Scientific | |
| Quant-iT Assay dsDNA kit | Invitrogen | Q33130 | |
| RNase | Epicentro | MRNA092 | |
| Cloreto de sódio | BDH Chemicals | BDH8014 | Conc apropriado. |
| Dodecilsulfato de sódio | Fisher Scientific | 03-500-509 | 10% em água |
| hexametafosfato de sódio | Merck Química | SX0583-3 | 2% da água |
| TBE | Fisher Scientific | BP1333-1 | |
| CHEF Mapper XA Sistema | Bio-Rad Laboratories | 170-3670 | |
| NanoDrop 1000 espectrofotômetro | Thermo Scientific | ND-1000 | |
| Vortexer | Scientific Industries Inc. | ||
| Agente de crosslinking ultravioleta | UVP | ||
| Waring Blender | Waring laboratório | LB10S |
Referências
- Decho, A. W. Microbial biofilms in intertidal systems: an overview. Cont. Shelf Res. 20, 1257-1273 (2000).
- Dupraz, C., Visscher, P. T. Microbial lithification in marine stromatolites and hypersaline mats. Trends Microbiol. 13, 429-438 (2005).
- Steffan, R. J., Goksoyr, J., Boj, A. K., Atlas, R. M. Recovery of DNA from soils and sediments. Appl. Environ. Microbiol. 54, 2908-2915 (1988).
- Lee, Y. K., Kim, H. W., Liu, C. L., Lee, H. K. A simple method for DNA extraction from marine bacteria that produce extracellular materials. J. Microbiol. Methods. 52, 245-250 (2003).
- Roose-Amsaleg, C. L., Garnier-Sillam, E., Harry, M. Extraction and Purification of Microbial DNA from Soil and Sediment Samples. Appl. Soil Ecol. 18, 47-60 (2001).
- de Lipthay, J. R., Enzinger, C., Johnsen, K., Aamand, J., Sørensen, S. J. Impact of DNA extraction method on bacterial community composition measured by denaturing gradient gel electrophoresis. Soil Biol. Biochem. 36, 1607-1614 (2004).
- Bey, B. S., Fichot, E. B., Dayama, G., Decho, A. W., Norman, R. S. Extraction of High Molecular Weight DNA from Microbial Mats. Biotechniques. 49, 631-640 (2010).
- Kakirde, S. K., Parsley, L. C., Liles, M. R. Size does matter: Application-driven approaches for soil metagenomics. Soil Biology & Biochemistry. 42, 1911-1923 (2010).
- Rodon, M. R., August, P. R., Bettermann, A. D., Brady, S. F., Grossman, T. H., Liles, M. R., Loiacono, K. A., Lynch, B. A., MacNeil, I. A., Minor, C., Tiong, C. L., Gilman, M., Osburne, M. S., Clardy, J., Handelsman, J., Goodman, R. M. Cloning the Soil Metagenomic: A Strategy for Accessing the Genetic and Functional Diversity of Uncultured Microorganisms. Appl. Environ. Microbiol. 66, 2541-2547 (2000).
- Beja, O., Aravind, L., Koonin, E. V., Suzuki, M. T., Hadd, A., Nguyen, L. P., Jovanovich, S. B., Gates, C. M., Feldman, R. A., Spudich, J. L., Spudich, E. N., DeLong, E. F. Bacterial Rhodopsin: Evidence for a New Type of Phototrophy in the Sea. Science. 289, 1902-1906 (2000).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados