É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Ensaios biofísicos para investigar as propriedades mecânicas do núcleo da célula Interphase: Aplicação de substrato Strain e Manipulação de microagulhas
Neste Artigo
Resumo
Nós apresentamos dois independentes, microscópio de ferramentas baseadas em medir as deformações induzidas nucleares e citoesqueleto em single, vivendo células aderentes em resposta à aplicação de tensão global ou localizada. Estas técnicas são utilizadas para determinar a rigidez nuclear (ou seja, deformabilidade) e para transmissão de força sonda intracelular entre o núcleo e citoesqueleto.
Resumo
Na maioria das células eucarióticas, o núcleo é a maior organela e é geralmente de 2 a 10 vezes mais duro do que o citoesqueleto redor e, conseqüentemente, as propriedades físicas do núcleo contribuir significativamente para o comportamento global biomecânica de células em condições fisiológicas e patológicas. Por exemplo, na migração de neutrófilos e invadindo as células cancerosas, rigidez nuclear pode representar um grande obstáculo durante o extravasamento ou passagem através de espaços estreitos dentro dos tecidos. 1 Por outro lado, o núcleo de células no tecido mecanicamente ativos, tais como o músculo requer suporte estrutural suficiente para suportar o estresse mecânico repetitivo. Importante, o núcleo é totalmente integrado à arquitetura celular, que está fisicamente ligado ao citoesqueleto circundante, que é um requisito essencial para o movimento intracelular e posicionamento do núcleo, por exemplo, em células polarizadas, núcleos sináptica nas junções neuromusculares, ou em células de migração. 2 Não é de surpreender que mutações em proteínas do envelope nuclear, como lamins e nesprins, que desempenham um papel crítico na determinação da rigidez nuclear e nucleo-citoesqueleto acoplamento, tem sido mostrado recentemente para resultar em uma série de doenças humanas, incluindo Emery- Dreifuss distrofia muscular, cinturas pélvica e escapular distrofia muscular e miocardiopatia dilatada. 3 Para investigar a função de diversas proteínas biofísicos envelope nuclear eo efeito de mutações específicas, desenvolvemos métodos experimentais para estudar as propriedades físicas do núcleo em um único, células vivas submetido a global ou localizada perturbação mecânica. Medição induzidos deformações nucleares em resposta ao pedido precisamente aplicado substrato tensão gera informações importantes sobre a deformabilidade do núcleo e permite a comparação quantitativa entre mutações diferentes ou linhas de células específicas para deficientes proteínas do envelope nuclear. Aplicação esforço localizado citoesqueleto com microagulhas é utilizado para complementar este ensaio e pode fornecer informações adicionais sobre a transmissão de força entre o núcleo intracelular e citoesqueleto. Estudar a mecânica nuclear em células vivas intactas preserva a arquitetura normal intracelular e evita artefatos potenciais que podem surgir quando se trabalha com núcleos isolados. Além disso, a aplicação tensão substrato apresenta um bom modelo para o estresse fisiológico experimentado por células em músculos ou outros tecidos (por exemplo, células musculares lisas vasculares expostos a tensão navio). Por último, enquanto que essas ferramentas têm sido desenvolvidas principalmente para estudar mecânica nuclear, eles também podem ser aplicados para investigar a função das proteínas do citoesqueleto e sinalização Mecanotransdução.
Protocolo
1. Aplicação tensão substrato
A medição da tensão nuclear normalizado inclui a preparação de pratos com tensão transparente, membranas de silicone elástico como a superfície de cultura de células, cultivo de células para os pratos, e aquisição de imagens das células antes, durante e depois (uniaxial ou biaxial) a aplicação de tensão.
Preparação de pratos silicone membrana e adesão de células
- Cada prato consiste em um esforço feito por prato fundo de plástico com um diâmetro de 3 "e um plástico-ring para manter uma membrana de silicone, que serve como substrato de cultura de células. Para a preparação dos pratos tensão, braçadeira de 4" x 4 "pedaço de membrana de silicone entre o O-ring eo prato. cuidadosamente cortar a membrana excesso, enxágüe com água deionizada, e autoclave os pratos tensão.
- Marca um ponto de referência no centro inferior da membrana (do lado de fora) antes do revestimento das membranas de silicone com moléculas da matriz extracelular (por exemplo, fibronectina). Este marco ajudará a identificar as mesmas células durante os experimentos tensão. (Opcional: Para aplicação tensão uniaxial, duas tiras paralelas de fita adesiva são aplicados sobre o ponto de referência para restringir a deformação da membrana em uma dimensão.)
- Para fornecer adesão celular ideal, revestimento das membranas de silicone com 3 fibronectina mcg / ml diluído em 10 ml PBS ou qualquer adequado proteínas da matriz extracelular. Cubra o prato de tensão com um prato invertido 10 centímetros de poliestireno, e incubar os pratos durante a noite a 4 ° C.
- No dia seguinte, lavar as membranas uma vez com tampão fosfato (PBS) para remover o excesso de proteínas. Preencha prato com 10ml de meio de crescimento (Modified Dulbecco Eagles Médio (glicose alta) suplementado com 10% de soro fetal bovino e 1% de penicilina / estreptomicina) e reserve.
- Fibroblastos de rato embrionárias são tripsinizados com tripsina 0,05% e semeadas em meio de crescimento em torno de 30% sobre a confluência prato revestido de silicone de membrana e incubar por 24-48 horas sob condições de cultura normal.
Tensão experimentos substrato
- Set-up do microscópio para experimentos. Os experimentos são realizados em um microscópio invertido com uma câmera digital adequada para microscopia de fluorescência, contraste de fase ou DIC, utilizando objetiva de 60x e software apropriado aquisição de imagem (por exemplo, ou IPLab Metamorph). Um microscópio vertical não é adequado para esta aplicação. O dispositivo de tensão consiste de uma placa de base que se encaixa no palco microscópio e detém um cilindro central, cilíndrico, que serve para aplicar pressão para a seção central da membrana de silicone, uma placa móvel que mantém o prato tensão e que pode deslizar para cima e para baixo em quatro pinos de orientação, bem como uma chapa de peso de 5 lb de aplicar uma carga.
- Para visualizar núcleos, incubar as células no prato de tensão com 1 mg / ml de Hoechst 33342 por 15 min a 37 ° C. Aspirar o meio off e substituir com 15 ml de meio de crescimento de fenol vermelho-livre (Modificado de fenol vermelho-livre Dulbecco Médio Eagles (glicose alta) com 25 mM Hepes, suplementado com 10% de soro fetal bovino e 1% de penicilina estreptomicina /). Parafuso do prato tensão na placa titular prato. Cuidadosamente aplique graxa (graxa de vácuo Braycote 804) ao perímetro da parte inferior da membrana de silicone para garantir deslizamento da membrana ao longo da placa central. Certifique-se manter a parte central da membrana transparente.
- Coloque a placa de base para o palco microscópio. Placa de montagem titular prato cuidadosamente sobre a placa de base. Assegurar que na posição inicial de repouso, a membrana de silicone do prato tensão vagamente repousa sobre o cilindro central.
- Primeiro, o foco na parte inferior da membrana de silicone e encontrar o ponto de referência central preto. O ponto servirá como ponto de partida para todas as aquisições de imagem e ajuda a localizar as mesmas células durante e após o estiramento. Nós usamos uma custom-written programa de imagem automatizado para armazenar as posições das células e para realocar essas células durante o experimento, mas isso também pode ser alcançado manualmente.
- A partir do ponto, ajustar o foco para visualizar as células e no topo da membrana de silicone. Localize bem distribuídos células com núcleos centralmente localizados e adquirir um contraste de fase e uma imagem da fluorescência da Hoechst nuclear mancha. A imagem de contraste de fase deve incidir sobre o contorno da célula e da membrana de silicone, enquanto as imagens de fluorescência deve se concentrar no plano central do núcleo.
- Depois de adquirir imagens de 5 a 15 células, voltar para o ponto central. Lentamente aplicar o peso para o prato de tensão, resultando em aplicação tensão uniforme no centro do prato. A tensão máxima aplicada substrato é limitado por espaçadores de nylon colocado nos pinos de alinhamento vertical (pinos de orientação).
- Concentre-se no fundo da membrana de silicone e localizar o ponto de referência novamente. A partir dadot, relocar as mesmas células e novamente adquirir um contraste de fase e uma imagem de fluorescência das células e núcleos sob pressão total, tentando aproximar-se dos planos focais das imagens iniciais. Este processo não deve exceder 10 min para evitar remodelação ativa e adaptação da célula ao substrato tensas.
- Depois de todas as imagens correspondentes foram adquiridos, mova o estágio do microscópio de volta para o ponto de partida. Remova cuidadosamente o peso do prato titular prato e permitir que a membrana de silicone para relaxar. Se necessário, empurre o prato pressão até que esteja na posição inicial. Então adquirir imagens de contraste de fase e fluorescência das células pós-deformação, como descrito acima para as imagens tensão.
Análise
- Imagens de células e núcleos fluorescente etiquetado antes, durante e depois da aplicação de deformação são analisados para calcular a tensão normalizada nuclear. Em nosso laboratório, usamos uma custom-written script MATLAB para a análise, mas várias opções alternativas estão disponíveis. A análise é realizada em três etapas.
- Primeiro, para calcular a tensão aplicada substrato, as posições de 3 a 6 pontos de controle localizados na membrana são manualmente compensada entre as correspondentes pré-full-e pós-deformação imagens. O programa MATLAB, em seguida, calcula a tensão de membrana aplicada ao comparar as posições dos pontos de controle de correspondência entre as imagens pré-tensão e full-tensão e também a tensão residual entre a tensão pré-e as imagens pós-deformação. Ao mesmo tempo, os pontos de controle são usados para registrar os pares de imagem, que vai ajudar a detectar células danificadas ou retirar (ver Figura 1).
- Em uma segunda etapa, os núcleos são selecionados manualmente usando um programa MATLAB que calcula separados tensão nuclear para cada núcleo individual por correspondência ou o tamanho nuclear ou marcadores intranuclear entre correspondentes pré-full-e pós-deformação imagens fluorescentes. Dar conta de pequenas variações na tensão de membrana aplicada entre diferentes experimentos, expressamos resultados como Strain Nuclear normalizado, definido como a relação de tensão nuclear induzida a tensão de membrana aplicada que é calculado para cada núcleo. Os scripts MATLAB estão disponíveis no laboratório Lammerding mediante solicitação.
- Por fim, cada núcleo é validado, excluindo medidas a partir de células que desligar ou ficar danificado durante a aplicação tensão (Figura 1).
2. Microagulhas ensaio de manipulação
Preparação de pratos, as células aderentes, e microagulhas
- Incubar 35 milímetros de vidro de fundo pratos de cultura de células com baixa concentração de fibronectina (0.5ug/ml) em solução salina de Hank Sal Buffered (HBSS) ou qualquer adequado proteínas da matriz extracelular por 2 horas a 37 ° C. Lavar pratos com HBSS duas vezes e adicionar 2 ml de meio de crescimento para o prato antes de prosseguir para a próxima etapa.
- Fibroblastos de rato embrionárias são tripsinizados com tripsina 0,05% e semeadas em meio de crescimento 2 ml semeado em 7,5 x 10 4 células / ml para a fibronectina revestido pratos fundo de vidro. Células lugar de volta na incubadora durante a noite. Células lugar de volta na incubadora durante a noite. Deve-se otimizar o número de células para obter único, aderente, não confluente de células de outros tipos celulares.
- Puxe a microagulhas, feito de borosilicato capilares, a ponta diâmetros de aproximadamente 1 a 3 mm com uma pipeta extrator comercial (por exemplo, Sutter Instrument Company).
Microagulhas experimento de manipulação
- No dia seguinte, incubar as células com MitoTracker mitocondrial mancha (600 mM; Invitrogen) e Hoechst 33342 nuclear mancha (1 mg / mL) adicionado ao meio de crescimento durante 30 minutos em 37 ° C incubadora.
- Lave as células uma vez em HBSS durante 5 minutos em temperatura ambiente e em seguida, adicione fenol vermelho meio de crescimento livre para as células para geração de imagens.
- Adquirir uma imagem da única célula, sem a microneedle inserido no citoesqueleto em contraste de fase, uma imagem fluorescente de 33.342 a Hoechst imagem mancha e uma fluorescente do mitocondrial mancha, com um objetivo 60x (0,70 NA, Plano Achromat) a uma invertida microscópio com uma câmera dispositivo digital charge-coupled.
- Usando um micromanipulador (por exemplo, InjectMan NI 2, Eppendorf), insira cuidadosamente a microagulhas para o citoplasma de uma célula uma distância fixa (normalmente de 5 mm) de distância da periferia nuclear e tomar uma imagem de contraste de fase, uma imagem fluorescente da Hoechst 33342 mancha e uma imagem fluorescente da mancha mitocondrial. Para esta aplicação, que ajuda a controlar o micromanipulador através de um computador, por exemplo, Windows Hyperterminal, para alcançar os procedimentos de micromanipulação consistente.
- Mover o microagulhas, uma distância específica (normalmente 10 ou 20 mm) em direção à periferia celular em 1 mícron / seg, ao mesmo tempo coletando imagens de fluorescência e de contraste de fase a cada 10segundos. Como o microneedle está se movendo a uma distância específica em direção à periferia celular. Com os parâmetros escolhidos, como microagulhas está se movendo a uma distância específica para a periferia celular, esta corresponderá a 03/02 frames durante o processo de manipulação.
- Finalmente adquirir imagens adicionais após a microagulhas é removido do citoesqueleto.
Análise
- Mapas de deslocamento são calculados usando um custom-script MATLAB escrito com base no rastreamento características fluorescente etiquetado do núcleo e citoplasma. (O script MATLAB está disponível no laboratório Lammerding a pedido). O programa utiliza um algoritmo de correlação cruzada normalizada entre as regiões imagem pequena (cerca de 10 M x 10 M em tamanho e espaçamento 5 mm de intervalo) em quadros de imagem subseqüentes. Para cada centro de região, o deslocamento na x-e y-indicações são computados como a mudança entre a posição original ea posição recém-identificadas e apresentadas como um vetor de deslocamento e também armazenado como valores numéricos. A partir dos mapas de deslocamento, os deslocamentos médios dentro das regiões pré-definidas podem ser calculadas. Note-se que os deslocamentos do citoesqueleto são baseados no canal de fluorescência para os marcadores do citoesqueleto (por exemplo, MitoTracker mitocondrial mancha), enquanto os deslocamentos nuclear são computados a partir do canal de fluorescência correspondente à Hoechst 33342 sinal. Para a nossa aplicação, nós rotineiramente examinar as seguintes regiões: (i) deformação do citoesqueleto no local de aplicação de tensão, ou seja, o local de inserção de microagulhas, (ii) a tensão nuclear em uma região dentro do núcleo em direção ao local de aplicação tensão, (iii) nuclear tensão em uma região nuclear de distância do local de aplicação, e (iv) a tensão do citoesqueleto em uma região citoplasmática através do núcleo. Além disso, também se pode medir diretamente o alongamento nuclear de contraste de fase ou 33342 Hoechst sequências de imagens de fluorescência. Neste caso, aplicada a tensão nuclear é calculado dividindo o alongamento nuclear (DL = L - L 0) pelo comprimento inicial, L 0, onde L é o comprimento final do núcleo no final da aplicação tensão e L 0 é o comprimento inicial do núcleo. Para as células com um acoplamento nucleo-citoesqueleto intacto, o núcleo irá alongar em direção ao local de aplicação de tensão. Em contraste, nas células em que nucleo-citoesqueleto acoplamento é interrompido, ou seja, as forças são transmitidas de forma menos eficiente entre o citoesqueleto eo núcleo, o núcleo está prevista para alongar muito menos na direção do local de aplicação de tensão. Assim, diminuiu deformações nuclear em resposta à aplicação de tensão do citoesqueleto implicam um desacoplamento (parcial) entre o núcleo e citoesqueleto.
3. Resultados representativos:
Aplicação tensão substrato
Adquirimos as imagens antes, durante e após a aplicação tensão para fibroblastos de rato embrionárias a partir de lamin heterozigotos e homozigotos A / C-deficiente (LMNA + / - e LMNA - / -), e do tipo selvagem (LMNA + / +) camundongos e posteriormente calculado a tensão normalizada nuclear para cada célula. Após análise, os núcleos são validados e células que se tornam danificados ou retrair durante a aplicação de deformação são excluídos da análise. Figura 1A mostra núcleos de três células que são válidos, enquanto que Figura 1B mostra células que devem ser excluídos da análise. Normalizado dados de deformação nuclear são obtidos a partir de pelo menos três experimentos independentes (cada um contendo medidas de núcleos ~ 5-10) e comparadas com outras células ou grupos de tratamento pela análise estatística. Aumentou a tensão normalizada nuclear indica reduzida rigidez nuclear, como visto em células com expressão reduzida de proteínas do envelope nuclear lamin A / C (Figura 2).
Microagulhas ensaio de manipulação
Para o ensaio de manipulação de microagulhas, que imaged deslocamentos nuclear e do citoesqueleto durante a aplicação do citoesqueleto esforço localizado. Células que se tornam danificados ou destacados são excluídos da análise. Para a análise, medimos a magnitude dos movimentos nucleares e citoesqueleto em direção ao local de aplicação de força em um único, células aderentes. Por exemplo, na Figura 3, faixa mitocondrial (marcador para o citoesqueleto) deslocamentos antes e depois da tensão do citoesqueleto e, em seguida, traçar os deslocamentos como vetores. Cada vetor representa o deslocamento calculado como a mudança entre a posição original ea posição recentemente identificados. Regiões com baixa intensidade da imagem ou textura insuficiente (por exemplo, as regiões fora da célula) são excluídos da análise. Os deslocamentos do citoesqueleto e nuclear são então quantificados em áreas selecionadas a distâncias cada vez maior a partir do local de aplicação tensão (Figura 4, as áreas correspondentes à cor boxes em inset). Em fibroblastos embrionárias de camundongos com acoplamento nucleo-citoesqueleto intacto, as forças são transmitidas através de todas as células, resultando em deformações induzidas nucleares e citoesqueleto, que lentamente se dissipam longe do local de aplicação tensão (Figura 4). Em contraste, fibroblastos com acoplamento perturbado nucleo-citoesqueleto (ou organização do citoesqueleto alterado) exibem deslocamentos localizada perto do local de aplicação, como mostrado na Figura 4 e só pouco deformações induzidas mais longe. Aplicação tensão comparável citoesqueleto no local de inserção de microagulhas (caixa laranja) é observado para ambos os fibroblastos controle (mCherry sozinho) e fibroblastos, com uma ruptura de acoplamento nucleo-citoesqueleto (DN KASH). No entanto, induzida deslocamentos nucleares e citoesqueleto (azul, amarelo, vermelho e caixas) em outras regiões foram significativamente menores nos fibroblastos com interrompidas acoplamento nucleo-citoesqueleto (DN KASH) do que em células controle (mCherry sozinho) (Figura 4). Assim, a diminuição dos deslocamentos do citoesqueleto e nucleares longe do local de aplicação de tensão, indica que a transmissão de força entre o citoesqueleto eo núcleo foi perturbado.
Importante, temos também que as mitocôndrias são validados marcador do citoesqueleto adequado, através da realização de manipulação de microagulhas em fibroblastos de rato embrionárias transfectadas com GFP-actina ou mCherry e GFP-vimentina e fluorescente etiquetado com Mitotracker Verde ou Vermelho. Mapas de deslocamento do citoesqueleto foram calculadas de forma independente do sinal fluorescente da mitocôndria e do citoesqueleto de actina ou vimentina. O deslocamento absoluto médio foi calculado para quatro regiões distintas do citoesqueleto, a distâncias cada vez maior longe do local de aplicação de tensão. Os valores de inclinação e R-quadrado foram calculados a partir da regressão linear entre as medidas obtidas a partir de mitocôndrias e de actina ou vimentina, respectivamente. Para actina, a inclinação foi de 0,99 eo valor de R 2 foi 0,986; para vimentina, a inclinação foi de 1,04 eo valor de R2 foi 0,971, confirmando que os deslocamentos mitocondrial servir como indicadores confiáveis para as deformações do citoesqueleto.
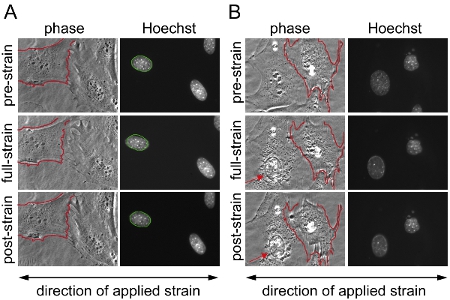
Figura 1. Aplicação substrato pressão sobre fibroblastos de rato embrionárias (MEFs). Fibroblastos de rato embrionárias distribuídos por duas áreas distintas na membrana de silício foram fotografadas com contraste de fase e microscopia de fluorescência antes, durante e após a aplicação de 20% de deformação uniaxial. (A) Exemplo de uma experiência bem sucedida com núcleos válida a partir de células que sobreviveram à aplicação tensão sem qualquer dano ou desapego e (B) exemplo de células que retrair / parcialmente destacada durante a aplicação tensão; resultados das células representadas em (B) são excluídas da análise. Em (B), a célula do lado esquerdo mostra sinais de danos e colapso do citoesqueleto nuclear (seta), enquanto a célula do lado direito destaca parcialmente e retrai durante a aplicação de tensão. Esta pode ser uma indicação de aplicação tensão excessiva. Para uma melhor comparação, em (A) e (B) a fronteira de uma das membranas celulares unstretched é delineada em vermelho e sobreposta à mesma célula durante e após a aplicação de tensão. Em (A) a fronteira do núcleo unstretched é descrito em verde e sobreposto ao mesmo núcleo durante e após a aplicação de tensão.
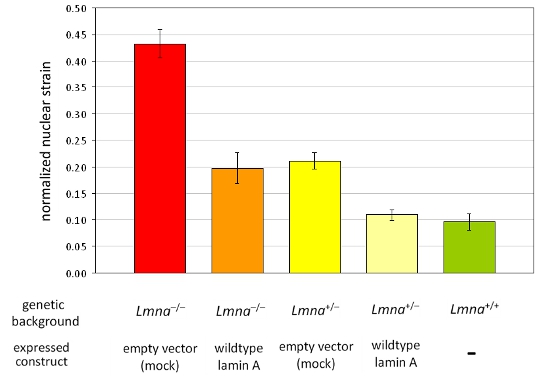
Figura 2. Análise de tensão nuclear normalizada em um painel de linhas celulares diferentes MEF MEFs do LMNA -. / - E LMNA + / - os antecedentes genéticos ectopicamente expressar tanto um vetor vazio ou tipo selvagem lamin A foram analisadas. Em comparação com MEFs de tipo selvagem littermates (LMNA + / +), perda de lamin A / C resultados de expressão em diminuição da rigidez nuclear que pode ser totalmente restaurado pela reintrodução do tipo selvagem lamin A. Notavelmente, reduzido a rigidez nuclear é refletida pela aumento dos valores da tensão nuclear normalizado. As barras de erro representam os erros padrão.

Figura 3. Ensaio de manipulação de microagulhas para medir a força de transmissão intracelular. Contraste de fase (A, B) e de fluorescência (C, D) imagens de um fibroblasto rotulados com mancha nuclear (azul) e MitoTracker mitocondrial mancha (verde). A microneedle foi inserido no citoesqueleto, a uma distância definida a partir do núcleo (A e C) e, posteriormente, mudou-se para a periferia da célula (B, D). Deslocamentos do citoesqueleto e nuclear foram quantificados através do rastreamento núcleo fluorescente etiquetado e mitocôndrias usando um custom-written algoritmo de correlação cruzada. (E) mapa de deslocamento do citoesqueleto final (verde) calculado a partir de deformações série de imagens de fluorescência, comprimento seta é ampliada em 2x para melhor visibilidade. Barra de escala, 10 mM.
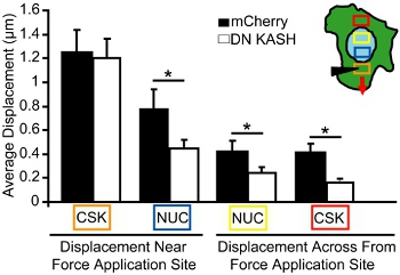
Figura 4. Análise de transmissão de força durante a manipulação intracelular microagulhas. Induzida deslocamentos do citoesqueleto e nuclear durante a manipulação de microagulhas, medido nas áreas correspondentes às caixas coloridas (inset em A). A caixa de laranja é o local de aplicação de tensão. Apesar da aplicação tensão semelhante no citoesqueleto (caixa laranja), induzida deslocamentos nucleares e citoesqueleto (azul, amarelo, vermelho e caixas) foram significativamente menores nos fibroblastos embrionárias de camundongos que com uma ruptura de acoplamento nucleo-citoesqueleto (DN KASH) comparado ao grupo controle ( mCherry sozinho) células.
Discussão
Ensaio de tensão substrato
Aplicação cepa tem sido usada com sucesso por nós e outros grupos para estudar as deformações induzidas nuclear em células submetidas a estresse mecânico e investigar a contribuição das proteínas específicas envelope nuclear à rigidez nuclear. 08/04 A vantagem desta técnica é que ela sondas propriedades mecânicas núcleos de viver em seu ambiente normal celular e do citoesqueleto e que a aplicação tensão substrato assemelha-se a aplicaç?...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi financiado pelo National Institutes of Health (R01 e R01 HL082792 NS059348) e do Hospital Brigham and Women Award do Grupo de Liderança Cardiovascular.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
| Fibronectina | Millipore | FC010 | |
| MitoTracker Red FM e FM Verde | Invitrogen | M22425 e M-7514 | |
| Hoechst 33342 | Invitrogen | H3570 | |
| Saline Hank Sal Buffered | Invitrogen | 14185 | |
| Livre de fenol, DMEM | Invitrogen | 21063 | |
| Soro fetal bovino | Aleken Biologicals | FBSS500 | |
| Penicilina / estreptomicina | Sigma | P0781-100ML | |
| Vidro borosilicato com filamento | Instrumento Sutter | BF100-78-10 | |
| Gloss / folhas silicone Gloss não-reforçada, 0.005 " | Especialidade Manufacturing Inc. | ||
| Phosphate Buffered Saline Dulbecco | Invitrogen | 14200 | |
| 35 milímetros de vidro de fundo placas de cultura (FluoroDish) | Mundial Precision Instruments, INC | FD35-100 | |
| Braycote graxa de vácuo 804 | Suprimentos Spi | 05133A-AB |
Referências
- Friedl, P., Wolf, K., Lammerding, J. Nuclear mechanics during cell migration. Curr Opin Cell Biol. 23, 55-64 (2011).
- Mejat, A., Misteli, T. LINC complexes in health and disease. Nucleus. 1, 40-52 (2010).
- Worman, H. J., Fong, L. G., Muchir, A., Young, S. G. Laminopathies and the long strange trip from basic cell biology to therapy. J Clin Invest. 119, 1825-1836 (2009).
- Caille, N., Tardy, Y., Meister, J. J. Assessment of strain field in endothelial cells subjected to uniaxial deformation of their substrate. Ann Biomed Eng. 26, 409-416 (1998).
- Lammerding, J. Lamins A and C but not lamin B1 regulate nuclear mechanics. J Biol Chem. 281, 25768-25780 (2006).
- Lammerding, J. Abnormal nuclear shape and impaired mechanotransduction in emerin-deficient cells. J Cell Biol. 170, 781-791 (2005).
- Lammerding, J. Lamin A/C deficiency causes defective nuclear mechanics and mechanotransduction. J Clin Invest. 113, 370-378 (2004).
- Verstraeten, V. L., Ji, J. Y., Cummings, K. S., Lee, R. T., Lammerding, J. Increased mechanosensitivity and nuclear stiffness in Hutchinson-Gilford progeria cells: effects of farnesyltransferase inhibitors. Aging Cell. 7, 383-393 (2008).
- Brooks, S. V., Zerba, E., Faulkner, J. A. Injury to muscle fibres after single stretches of passive and maximally stimulated muscles in mice. J Physiol. 488, 459-469 (1995).
- Maniotis, A. J., Chen, C. S., Ingber, D. E. Demonstration of mechanical connections between integrins, cytoskeletal filaments, and nucleoplasm that stabilize nuclear structure. Proc Natl Acad Sci. 94, 849-854 (1997).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados