Method Article
Um método de genotipagem baseados em PCR para distinguir entre variedades do tipo selvagem e Ornamental de Imperata cylindrica
Neste Artigo
Resumo
Nós fornecer um protocolo de genotipagem custo-eficácia e rápida molecular que emprega iniciadores variedade-PCR específicos que as diferenças de sequência alvo de ADN dentro da região espaçadora cloroplasto trnL-F para diferenciar entre as variedades de Imperata cylindrica (Cogongrass) que não podem ser distinguidos pela morfologia sozinho. Estas variedades incluem a erva federal listado nocivo, cogongrass e estreitamente relacionada, a variedade difundida ornamental, eu. cylindrica Var. Koenigii (Grama sangue japonês).
Resumo
De tipo selvagem I. cylindrica (cogongrass) é um dos melhores piores dez plantas invasoras do mundo, impactando negativamente os recursos agrícolas e naturais em 73 países diferentes na África, Ásia, Europa, Nova Zelândia, da Oceania e das Américas 1-2. Cogongrass forma rápida propagação, estandes monodominantes que deslocam uma grande variedade de espécies de plantas nativas e, por sua vez ameaçam os animais nativos que dependem dos deslocados espécies de plantas nativas para forragem e abrigo. Para aumentar o problema, uma variedade ornamental [I. cylindrica var. koenigii (Retzius)] é amplamente comercializado sob os nomes de 'Rubra' Imperata cylindrica, Barão Vermelho, e grama sangue japonês (JBG). Esta variedade é supostamente estéril e não-invasivo e é considerado um ornamental desejável para suas folhas de cor vermelha. No entanto, nas condições corretas, JBG pode produzir sementes viáveis (Carol Holko, 2009 comunicação pessoal) e pode reverter para um verde iforma nvasive que muitas vezes é indistinguível da cogongrass à medida que assume as características distintivas da variedade de tipo selvagem invasiva 4 (Figura 1). Isto torna a identificação usando morfologia uma tarefa difícil mesmo para taxonomistas bem treinados plantas. Reversão de JBG a um fenótipo agressivo verde também não é uma ocorrência rara. Usando a comparação de seqüências de codificação e regiões variáveis do DNA nuclear e do cloroplasto tanto, temos confirmado que JBG foi revertido para o invasivo verde dentro dos estados de Maryland, Carolina do Sul e Missouri. JBG foi vendido e plantada no estado em quase todos os os EUA continental, onde não há uma infestação cogongrass ativo. A extensão do problema em reverter não é bem compreendido porque as plantas são revertidos em situação irregular e, muitas vezes destruída.
A aplicação deste protocolo molecular fornece um método para identificar JBG reverte e pode ajudar a manter estas variedades de co-ocorrência de umnd possivelmente hibridação. Cogongrass é um outcrosser obrigatório e, quando cruzou com um genótipo diferente, pode produzir vento dispersa sementes viáveis que se espalham sobre grandes distâncias cogongrass 5-7. JBG tem um genótipo ligeiramente diferente do que cogongrass e pode ser capaz de formar híbridos viáveis com cogongrass. Para aumentar o problema, JBG é mais frio e mais tolerantes do que cogongrass 8-10, eo fluxo gênico entre estas duas variedades é susceptível de gerar híbridos que são mais agressivas, tolerantes, e résistente frio do que do tipo selvagem cogongrass. Enquanto tipo selvagem cogongrass atualmente infesta mais de 490 milhões de hectares no mundo todo, os EUA Sudeste que infesta mais de 500.000 hectares e é capaz de ocupar mais de os EUA como se espalha rapidamente para o norte devido ao seu nicho amplo e geográfica 3,7,11 potencial. O potencial de um cruzamento genético é uma preocupação séria para o USDA-APHIS Programa Semana Federal nocivas. Atualmente, o USDA-APHIS proíbe JBG em estados where há infestações maiores cogongrass (por exemplo, Flórida, Alabama, Mississippi). No entanto, impedindo que as duas variedades da combinação pode ser mais difícil como cogongrass e JBG expandir suas distribuições. Além disso, a distribuição do JBG reverter é actualmente conhecida e sem a capacidade de identificar esses variedades por morfologia, alguns infestações cogongrass pode ser o resultado de JBG reverte. Infelizmente, os actuais métodos de identificação molecular de dependem normalmente AFLP (Polimorfismos comprimento de fragmentos amplificados) e sequenciação de ADN, ambos os quais são demorados e dispendiosos. Aqui, nós apresentamos o primeiro rentável e confiável baseada em PCR método de genotipagem molecular para distinguir com precisão entre cogongrass e JBG reverter.
Protocolo
1. Colheita e Preservação
Este método foi desenvolvido e testado usando frescos, congelados, e tecidos de folhas secas recentemente.
- Identificar cogongrass e / ou JBG tecidos com a ajuda de um taxonomista que se especializa na identificação das espécies de grama. Com os seus brilhantes folhas vermelhas, JBG ornamental é fácil de distinguir visualmente a partir de tipo selvagem e cogongrass JBG reverter, no entanto, cogongrass e JBG reverter são quase indistinguíveis um do outro. Cogongrass eo fenótipo JBG revertido tem folhas verdes, longos, rizomas consideravelmente maiores e mais longos, e mais área foliar do que JBG 12 (Figura 1).
- Tecido foliar fresco fornece DNA a qualidade mais abundante e mais alto e podem ser coletadas a partir do campo ou estufa cresceu I. plantas cylindrica. Se o tecido fresco é para ser usado, extrair DNA no prazo de 3 horas após a recolha para ajudar a prevenir a degradação. Caso contrário, preparar tecido para o armazenamento, keeping o tecido fresco e fora da luz solar direta.
- Para armazenar tecidos de extracção de ADN numa data posterior, o método mais óptima é a congelar e armazenar o tecido à temperatura de -80 ° C imediatamente. Não permitir que o tecido congelado para descongelar antes da extracção do DNA. Transferir o tecido congelado em azoto líquido antes para os passos de moagem de extracção de ADN para prevenir o descongelamento.
- Se uma temperatura de -80 ° C congelador não estiver disponível, o tecido seco imediatamente. Para armazenamento a seco, colocar o tecido em um envelope de papel e armazenar o envelope em gel de sílica desidratada ou outros dessecantes activas à temperatura ambiente. Uma pequena quantidade de sílica indicador misturado com não-indicando sílica irá assegurar que a sílica é totalmente desidratada e adequado para secagem de tecidos vegetais.
- Use pelo menos 10 vezes em gel de sílica mais do que o tecido de folha fresca, em peso. Tecido de planta deve secar no prazo de 24 horas. A qualidade ea quantidade de DNA é reduzido por meio de armazenamento seco ao longo do tempo (como no caso de espécime herbários).
2. Extração de DNA
Para extrair DNA a partir de tecido de planta, siga o DNeasy Planta Mini Kit (Qiagen, Valência, CA; Cat # 69104 ou 69106) as instruções do fabricante, com uma pequena modificação. Em vez de utilizar o tecido mg sugerido inferior a 100 fresco ou inferior a 20 mg de tecido seco para cada coluna, moer maior do que 100 mg, e, em seguida, transferir 100 mg a partir de tecido fresco ou congelado (ou> 20 mg de tecido seco) para tubos apropriados para a extração. DNA nuclear e plastidial é extraído em simultâneo.
- Antes de iniciar estes procedimentos, verificar que o etanol foi adicionado em buffers AP3 / E e AW.
- Moer> 100 mg de tecido foliar fresco ou congelado (ou> 20 mg de tecido foliar seco) a um pó fino usando três rondas de azoto líquido com trituração num almofariz e pilão refrigeradas. Perturbação insuficiente do material de partida ou lise insuficiente pode também resultará em rendimentos mais baixos de DNA. Cuidadosamente moer o tissue e não sobrecarregue as colunas com o tecido em excesso.
- Transferir 100 mg de pó a partir de tecido congelado fresco ou congelado (ou 20 mg de pó a partir de tecido seco) para um tubo de microcentrífuga de 1,5 ml contendo 400 ul de tampão de AP1 e 4 uL de RNase A. Cada tubo pode ser colocado em uma cremalheira pequena num equilibrar para monitorar o peso correcto de tecido por tubo.
- Amostra de vórtice ou de vibração (s) para misturar e incubar durante 10 min a 65 ° C, invertendo o tubo (s) de 2-3 vezes durante a incubação.
- Adicionar 130 uL de Tampão de AP2 a cada amostra. Misturar invertendo o tubo (s) várias vezes, e incubar durante 5 min em gelo.
- Pipetar lisado cada um em separado coluna de spin QIAshredder Mini, e colocar cada coluna em um tubo de recolha de 2 ml (fornecido com o kit). Coluna de centrifugação (s) durante 2 min a 20.000 xg (14.000 rpm ~), e transferir cada fluxo através fracção para um novo tubo (não fornecido com o kit), sem perturbar qualquer sedimento formado.
- Adicionar 1,5 volumes de Tampão AP3 /E, misturar e por pipetagem.
- Transferir 650 uL de mistura em uma coluna de spin DNeasy Mini em um tubo de recolha de 2 ml. Coluna de centrifugação (s) durante 1 min a 6000 xg (8000 rpm ~), e descartar o fluxo através. Repetir este passo com a mistura remanescente para cada amostra.
- Coloque a coluna de spin (s) para um novo tubo de recolha de 2 ml (s), e adicionar 500 uL de tampão de AW para o início de cada coluna. Coluna de centrifugação (s) durante 1 min a 6000 xg (8000 rpm ~), e descartar flow-through.
- Adicionar outra uL 500 de AW tampão para o início de cada coluna. Centrifugar durante 2 min a 20.000 xg (~ 14.000 rpm). Este passo irá secar a coluna, removendo assim qualquer etanol residual contido nos buffers que podem inibir a PCR.
- Transfira cada coluna de rotação para um tubo de 1,5 ml nova amostra de microcentrífuga. Adicionar 100 uL AE tampão para o início de cada coluna para a eluição, e incubar em coluna (s) durante 5 min à temperatura ambiente. Coluna de centrifugação (s) durante 1 min a 6000 xg (~ de 8.000 rpm) para recolher o ADN.
- Repita estes passos de eluição uma hora, eluindo o DNA para o mesmo tubo de microcentrífuga de 1,5 ml para se obter 200 uL de amostra. As amostras de DNA armazenar a -20 ° C até à utilização. Concentrações de ADN dependem do tipo de tecido e as condições de armazenagem. Rendimentos óptimos são obtidos quando eluindo DNA com um total de 200 uL de tampão de AE, no entanto, as concentrações podem ser aumentadas se volumes de eluição são reduzidos a tão pouco quanto 50 uL.
3. Verificação da qualidade e quantidade de DNA
- Testar a qualidade ea quantidade de DNA extraído antes da PCR configuração usando um espectrofotómetro ou fluorómetro e electroforese em gel. Isso ajudará a garantir o sucesso das etapas subseqüentes.
- Utilizando um espectrofotómetro, a qualidade do DNA de teste e quantidade. Rendimento de ADN boas deve estar entre 50 e 150 ng / uL com 260/280 e 230/280 rácios cerca de 2,0. Como um exemplo, a Figura 2 mostra os resultados de boa qualidade usando o espectrofotómetro NanoDrop (ThermoScientific, Wilmington, DE).
- Conduzir electroforese utilizando um padrão de 1% de gel de agarose. Verificar a presença de bandas relativamente grande (> 10 Kb), sem a estrias pouco de contaminação RNA (Figura 3).
- Se a concentração de ADN é elevada, amostras de DNA diluir até 70 ng / uL de passos subsequentes.
4. Primers de PCR
Os iniciadores de PCR utilizados neste protocolo são baseados em diferenças de sequência entre a região espaçadora plastidial trnL F-de genótipos cogongrass e JBG. Estas diferenças surgem na forma de SNPs (polimorfismos de um único nucleótido) e indels (inserções e deleções) que permitiram o desenvolvimento de uma variedade primers específicos, localizando os iniciadores nos locais de sequências únicas (Figura 4).
- A qualidade dos iniciadores podem ter um impacto significativo sobre os resultados da PCR. Iniciadores da ordem de uma companhia respeitável. Nós ordenamos nossos primers de Oblique Bio, Inc. (:/ / Www.obliquebio.com/web/ "target =" _blank "> http://www.obliquebio.com/web/, Huntsville, AL), solicitando apenas procedimentos padrão de dessalinização.
- trnL-F iniciadores de controlo positivo: Este conjunto de iniciadores amplifica a região espaçadora plastidial trnL F-de taxa mais grama 11-13 e serve como um bom controle positivo (resultando em uma banda pb 890).
Nome Seqüência trnF (GAA)-F 5'-ATTTGAACTGGTGACACGAG-3 ' trnL (5 'do exão)-C 5'-CGAAATCGGTAGACGCTACG-3 ' - Iniciadores de tipo selvagem cogongrass: Este conjunto iniciador é específico para cogongrass e não amplificar a região F-trnL de JBG genótipos. Os resultados conjuntos em uma banda que é 595 bp.
Nome Sequência trnLF-C-F1 5'-TCCACTTTTTTGAAAAAACAAGTGCAA-3 ' trnLF-C-R1 5'-GCCGATACTCTAATAAATAAAAAAAAAAAAGAAAT-3 ' - JBG e JBG Reverter iniciadores: Este conjunto de iniciadores é específico para JBG genótipo e não amplificar a região F-trnL de cogongrass. Os resultados conjuntos em uma banda que é 594 bp.
Nome Seqüência trnLF-R-F2 5'-CCAAATCCACTTTTTTGAAAAAACAAGTGGTT-3 ' trnLF-R-R2 5'-CGAGATTCCTTGCCGATACTCTAATAAAA-3 ' - Ressuspender cada iniciador, em volume suficiente de nuclease livre de ddH2O para se obter um stock de 100 mM de soluções que podem ser armazenados a -20 ° C, a longo prazo.
- Diluir cada ação primário a 12 mm antes das etapas de instalação da PCR.
5. PCR Setup
DNA extracções são amplificados utilizando cada um dos conjuntos de iniciadores acima em reacções de PCR. Incluir um controlo positivo para assegurar que todos os reagentes de PCR estão a funcionar bem e pode gerar uma banda. Incluir um controlo negativo para assegurar que nenhum dos reagentes estão contaminados com DNA indesejado. O controlo negativo não contém nenhum-molde e deve resultar em nenhuma produção banda.
- Preparar todas as reacções em tubos de paredes finas de PCR para permitir uma melhor transferência de calor entre o bloco de termociclador ea amostra. É recomendável usar comercialmente disponíveis pontas de aerossol livres de pipeta para ajudar a evitar a contaminação.
- Para cada amostra de ADN isolado, configurar 50 uL reacções de PCR utilizando cada um dos conjuntos de iniciadores acima em 0,2 ml tubos de paredes finas de PCR adicionando os seguintes reagentes em ICE na ordem listada abaixo. Se as amostras múltiplas estão a ser preparadas, fazer um cocktail contendo todos os reagentes com a excepçãodo molde de ADN para estabelecer as condições uniformes entre todas as reacções.
Reagente PCR Volume Usado Concetration final Livre de nuclease ddH2O 40,5 ul Vantagem 2 10X PCR Buffer (Clonetech, CA) 5,0 ul 10% (v / v) Vantagem UltraPure PCR dNTP Mix (10 mm cada, Clonetech, CA) 1,0 ul 0,2 mM Primer 1 (12μM em ddH2O) 1,0 ul 0,24 mM Primer 2 (12μM em ddH2O) 1,0 ul 0,24 mM Vantagem Mix Polimerase 2 (Clonetech, CA) 0,5 ul 1% (v / v) Extração de DNA(70 ng / reação; ajustar DDH O 2 º volume, conforme necessário) 1,0 ul 1,4 ng / uL Total: 50,0 uL - Para garantir que todos os reagentes da PCR estão funcionando bem, configurar o controle positivo utilizando os primers de controlo positivo. Este conjunto de primers funciona igualmente bem para cogongrass, JBG, JBG reverter e outras gramíneas e resultará em uma banda que é 890 bp.
- Configurar o controlo negativo utilizando o iniciador mesmo controle definido como o controlo positivo, usando ddH2O em vez de a extracção do DNA. Se todos os reagentes estão livres de DNA contamina, esta reacção irá resultar em nenhuma banda.
- Se a concentração de DNA é baixa, mais DNA pode ser adicionado a cada reacção, ajustando a quantidade de Nuclease livre de ddH2O utilizado para levar o volume total de reacção de 50 uL. Não utilizar mais do que 5 ul de DNA (10% do o volume total) por reaction, impurezas como possíveis contidas nas amostras de DNA pode inibir reacções de PCR.
6. PCR bicicleta
- Efectuar amplificações por PCR em um termociclador equipado com uma tampa aquecida usando os parâmetros de ciclagem seguintes PCR. Usamos o Mastercycle pro S termociclador (Eppendorf, Hauppauge, NY) configurado para operar com as condições de rampa padrão de temperatura. Qualquer termociclador qualidade deve ter um bom desempenho.
Ciclo Desnaturação Recozimento Polimerização 1 2 min a 95 ° C 2 30 seg a 95 ° C 30 seg a 61 ° C 90 seg a 68 ° C 35 ciclos 3 5 min a 68 ° C Mantenha a 4 ° C até que a amostra é removida - Optimizar as condições para o PCR (incluindo a temperatura de recozimento iniciador, os tempos de extensão, eo número de ciclos), conforme necessário, dependendo da qualidade do ADN, os iniciadores, Taq polimerase ou tipo de termociclador utilizado. Sugerimos utilizando um termociclador de gradiente capaz quando a determinação do óptimo recozimento temperaturas.
- Se o termociclador a ser utilizado não tem uma tampa aquecida, adicionar 1 gota de óleo mineral para o início de cada amostra para evitar a evaporação durante o ciclismo PCR.
7. Electroforese em gel de produtos de PCR
Para visualizar os resultados da análise, os produtos de PCR separadas sobre um gel de agarose 1%, usando electroforese padrão.
- Combinar 2 ul de um tampão padrão de ADN de carga (tipicamente uma solução 5x ou 6x) com 5 uL de cada produto de PCR amplificado.
- Carregar amostras sobre um gel de agarose a 1% contendo EtBr (brometo de etídio para a coloração do ADN) e feita comither TAE ou SB (borato de sódio) sistemas tampão 16. Usamos 1 ul de uma solução estoque de 10 mg / ml EtBr por 100 ml de agarose a 1% (0,1 ug / ml).
- Executar amostras em ~ 120V até que a frente de corante atinge ¾ do comprimento total do gel.
- Sob a luz UV (por exemplo, uma onda curta caixa de luz UV), inspecionar as bandas resultantes para ver se um fragmento apropriado foi amplificado.
- Documentar o gel e as bandas resultantes usando um sistema de foto-documentação disponível ou câmera.
8. Os resultados representativos
Após a visualização dos produtos de PCR, cogongrass tem um padrão único de bandas comparada com a de JBG ou revertido JBG (Figura 5). Para cada amostra de DNA, o trnL-F conjunto de iniciadores de controlo positivo deve resultar numa banda de alta intensidade em ~ 890 pb. Isso verifica se todos os reagentes da PCR estão funcionando bem. Do mesmo modo, o controlo negativo (nenhum modelo) reacção não deve conter bandas para qualquer s iniciadoret utilizado. Isto verifica que nenhum dos reagentes foram contaminadas.
Se a amostra de DNA é derivado do tipo selvagem cogongrass, uma reacção de PCR usando o conjunto de iniciadores cogongrass específico irá resultar em uma única banda a ~ 595 pb, enquanto os iniciadores específicos do JBG irá resultar em nenhuma banda. Da mesma forma, se a amostra de DNA é derivado do JBG ou revertido JBG, uma reacção de PCR usando o conjunto de iniciadores JBG específico irá resultar em uma única banda a ~ 594 pb, enquanto os primers específicos cogongrass irá resultar em nenhuma banda. Porque JBG e reverteu JBG ter seqüência de ácido nucléico idênticos, eles vão, portanto, têm padrões idênticos de bandas. Se muitas amostras devem ser comparados com um gel ao mesmo tempo, é recomendável a execução de todas as amostras derivadas de cada iniciador situado ao lado um do outro, tornando assim mais fácil para digitalizar as amostras para resultados positivos.
Diferenças morfológicas entre JBG e reverte JBG são bastante óbvios (cor vermelha por exemplo, das folhas e menor estatura de JB G vs a coloração verde, maior estatura e crescimento agressivo do JBG reverter), de modo enquanto os resultados da PCR será o mesmo, as variedades JBG são fáceis de distinguir usando morfologia da planta.

Figura 1. Comparação de estufa cresceram Imperata cylindrica var. Koenigii (grama sangue japonês), revertidas I. cylindrica var. koenigii (JBG Revert) e I. cylindrica (tipo selvagem cogongrass).
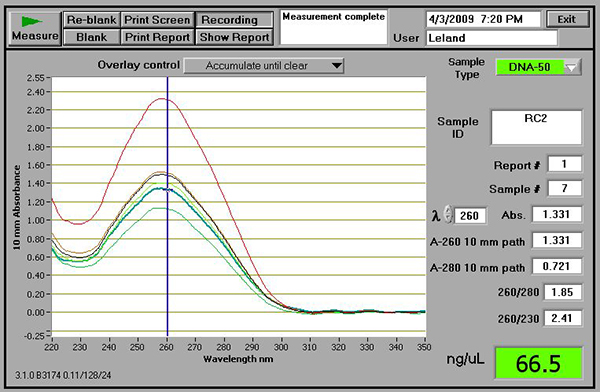
Figura 2. Um exemplo de amostras de ADN verificada utilizando um espectrofotómetro NanoDrop. Note-se que, independentemente do espectrofotómetro utilizado, o rácio 260/280 deve ser próximo de 1,8 eo rácio 260/230 deve fechar a 2,0.
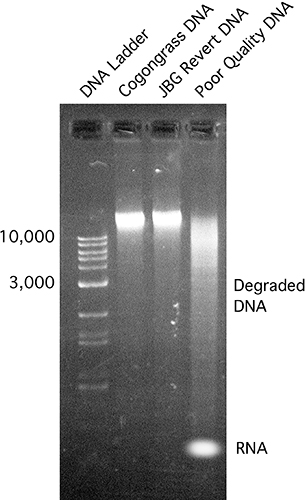
As amostras de DNA Figura 3. Verificada usando electroforese em gel de padrão em um gel de agarose a 1%. Um marcador de ADN comercial foi utilizado para análise de tamanho. Lane # 4 é um exemplo de amostra pobre qualidade do DNA, mostrando manchas e alguma contaminação RNA.
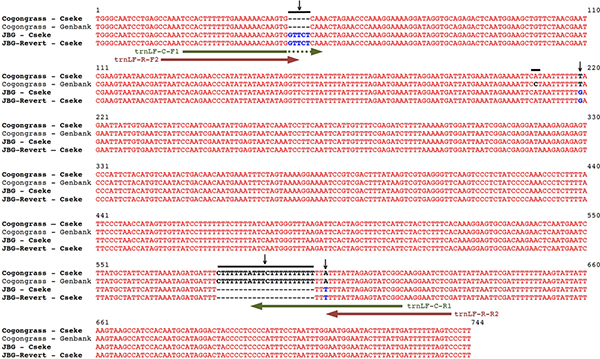
Alinhamentos Figura 4. Seqüência das regiões trnL-F de Imperata cylindrica var. Koenigii (grama sangue japonês), revertidas I. cylindrica var. koenigii (JBG Revert) e I. cylindrica (tipo selvagem cogongrass). Vertical setas pretas indicam diferenças nas sequências resultantes de SNPs e indels. Horizontais setas verdes indicam as posições dos iniciadores de tipo selvagem cogongrass utilizados para cogongrass-PCR específica. Horizontais setas vermelhas indicam a posições da JBG e JBG Reverter iniciadores utilizados para JBG-PCR específica.
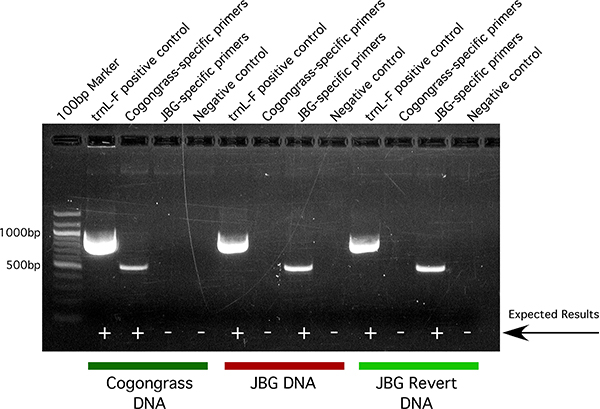
Figura 5. Resultado representativas de electroforese em gel dos produtos de PCR derivados de cogongrass, JBG e JBG reverter amostras de DNA combinados com cogongrass-e JBG primers específicos, bem como a trnL-F de controlo positivo e um controle de modelo não negativo.
Discussão
O viveiro EUA e indústrias paisagem prosperar no cultivo e venda de espécies de plantas exóticas e romance. Isso, juntamente com a crescente globalização do comércio, aumenta as chances de que uma espécie de planta invasora entrarão, estabelecer e difundir os EUA em A capacidade de governo federal regular tais plantas depende de informações que muitas vezes não é disponível, incluindo o potencial para se tornar invasiva , taxonomia correta e distinção genética da taxa nativos e naturalizados. Porque o nosso conhecimento de plantas invasoras é muitas vezes limitado, plantas importadas, com características ocultas invasivas têm sido introduzidos voluntariamente para descobrir depois que eles invadem nossos recursos agrícolas e naturais. Este protocolo visa resolver esses problemas associados I. variedades cylindrica, proporcionando o primeiro método simplificado molecular que pode distinguir com precisão entre tipo selvagem cogongrass e reverteu a forma da sua contraparte JBG ornamentais.
> jove_content "Para o desenvolvimento deste protocolo, tipo selvagem cogongrass foram coletados a partir de populações naturalizadas na Unidade Florestal Pond Creek, em Santa Rosa County perto de Jay, FL, em junho de 2008. JBG foi obtido a partir de um viveiro comercial (Bluebird Nursery, Inc ..) em junho de 2008, bem como da coleção de um proprietário em Columbia, MO JBG reverte foram obtidos a partir do pátio do Museu Campbell Geológica na Universidade de Clemson, SC, em junho de 2008; da Universidade Park, em Riverdale, MA, em junho de 2009; a partir da frente estaleiro de um proprietário em Columbia, MO em 2009 (identificados por Leland Cseke). Todas as plantas foram mantidas em uma estufa localizado na Universidade do Alabama em Huntsville (Huntsville, AL).A sequência genética de DNA recolhidos a partir destas plantas incluiu o em comparações profundidade de 9 regiões de DNA independentes comumente usados para plantas de código de barras 2. Em todos os casos, as sequências de JBG eram um fósforo de 100% para os do JBG reverter, ajudando assim averificar que JBG, de fato, reverter para um formulário, verde invasivo. Apenas o ITS nuclear e os cloroplastos trnL F-regiões têm diferenças que podem ser utilizados para distinguir entre geneticamente cogongrass e JBG. A região ITS tem um total de 3 SNPs (polimorfismos de nucleotídeo único) entre cogongrass e JBG, enquanto a região trnL F-tem 2 SNPs e 2 indels (inserções e deleções). Estas diferenças genéticas permitiu variedade primers específicos de PCR para ser desenvolvido que pode distinguir entre tipo selvagem cogongrass e JBG reverte. Os resultados mais fiáveis ter vindo de iniciadores derivados da região trnL F-plastidial. Assim, este protocolo é baseada nas diferenças de sequência entre as regiões F-trnL do genoma do cloroplasto de cogongrass, JBG e reverteu JBG (Figura 4).
Para ajudar a gerar primers que são mais específicos para as variedades em questão e para evitar falsos positivos de espécies estreitamente relacionadas, all conhecidos trnL F-seqüências de I. variedades cylindrica foram comparados com trnL-F seqüências de espécies de gramíneas relacionados (43 seqüências independentes de 29 espécies, por exemplo, Cymbopogon citratus, Sorghastrum incompletum e Coix lacryma-jobi sinensis Miscanthus, Saccharum officinarum, Sorghum halepense). Embora, examinámos sequências variedade de iniciadores específicos através 29 espécies de relva, a especificidade dos iniciadores específicos variedade não tenha sido exaustivamente examinados para a sua capacidade para amplificar DNA da maioria das outras espécies de gramíneas. Consequentemente, o tecido usado para extração de DNA devem ser cuidadosamente identificados como I. cylindrica antes de iniciar este protocolo. Se a grama não pode ser identificada como a cogongrass ou JBG, então nós sugerimos o seqüenciamento do produto de PCR para se certificar de que as seqüências são uma correspondência exata para cogongrass ou JBG. Actualmente, o método mais preciso para verificar a identidade de um espécime grama dada é a realização de PCR em ambos os trnl-F e das suas regiões, seguido por verificação sequência de produtos de PCR e comparação das sequências para sequências conhecidas de taxa identificado com precisão. O ADN pode ser amplificado utilizando iniciadores de controlo descritos no presente protocolo (para a região trnL-F) ou iniciadores outros disponíveis em outras publicações 13-15. A sequenciação é muito mais trabalho e dispendiosa do que usar o nosso procedimento simplificado.
A qualidade dos iniciadores utilizados para a PCR é crítica para o sucesso do procedimento. Fizemos os iniciadores para esse procedimento disponível a partir de Bio oblíqua, Inc. ( http://www.obliquebio.com/web/ , Huntsville, AL). A vantagem de ordenação dos iniciadores a partir de Bio oblíqua é que eles armazenar grandes números de alíquotas de cada iniciador a partir da série idêntica de amostra de produção como os iniciadores foram utilizados para a optimização no nosso laboratório. Consequentemente, não só primers têm a mesma sequência, mas they vêm do lote de produção exatamente o mesmo que foi usado neste protocolo. Usando iniciadores a partir do mesmo lote, pode ajudar a evitar variáveis estranhas no procedimento que pode resultar de diferenças na qualidade dos iniciadores de PCR. Da mesma forma, enquanto outras polimerases Taq deve funcionar bem para PCR, a qualidade da polimerase Taq usado terá um impacto sobre a qualidade dos resultados de PCR. Para permitir uma melhor consistência na PCR reagentes, temos otimizado o protocolo utilizando reagentes de Clontech. A vantagem 2 Polimerase (Clontech, Mountain View, CA, Cat. # 639201 ou 639202) é uma mistura de um sólido, a polimerase Taq de arranque a quente e uma enzima prova de leitura de que ajuda a proporcionar alta especificidade e ampliações mais precisos.
Como este protocolo baseia-DNA do cloroplasto, que é herdada da mãe em gramíneas, eventos de hibridação entre genótipos JBG cogongrass e não pode ser capturado com o nosso processo de identificação molecular. Nos casos em que é hypridization suspeCTED, recomendamos o uso de regiões nucleares que são herdadas de ambos os pais. O mais comumente utilizado região não plastid variável a considerar na genotipagem das plantas é a região ITS nuclear ribossomal 13-15,17. Actualmente, estão a avançar em direcção a multiplexação a amplificação do cloroplasto região trnL-F com a da região ITS nuclear no tubo de PCR mesmo. Multiplexação plastid com regiões do DNA nuclear seria potencialmente contornar as limitações do uso sozinho, no entanto, estes métodos requerem otimização e avaliação adicional para determinar a viabilidade caso a caso. O uso de métodos quantitativos PCR em tempo real (qPCR) e novas tecnologias, tais como faróis molecular (sondas fluorescentes primers), também estão sendo avaliados como métodos de genotipagem não-seguras e precisas de plantas.
O protocolo apresentado aqui proporciona uma abordagem rápida e fiável para distinguir o JBG reverter da de tipo selvagem cogongrass. Encorajamos a utilizaçãors deste protocolo em contactar-nos para relatar os resultados derivados do uso deste protocolo. Tal informação compartilhada ajudará a fornecer informações sobre a distribuição de JBG reverte. Isso também irá ajudar os reguladores no USDA tomar decisões informadas sobre as ações que podem ser necessárias para contornar a propagação e hibridação potencial das variedades altamente invasivos cogongrass.
Divulgações
Não temos nada a divulgar.
Agradecimentos
Agradecemos Alan Tasker (USDA-APHIS, Riverdale, MD), Stephen Compton (Clemson), Sherry Aultman (Clemson), Craig Ramsey (USDA-APHIS, Fort Collins, CO), e Betty Marose (UMD) para a assistência na obtenção de amostras . Agradecemos aos alunos Andrew Adrian (UA-Huntsville) e Derek Thacker (UA-Huntsville) por sua assistência em testar este protocolo, e José Herdy por seu trabalho nas filmagens do vídeo. Este trabalho foi financiado pelo National Peixe e Wildlife Foundation.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| DNeasy Usina Mini Kit | Qiagen, Valencia, CA | 69104 ou 69106 | Qualquer respeitável genômica kit DNA da planta que produz DNA de boa qualidade deve funcionar bem para estes procedimentos. |
| trnF (GAA)-F | Oblique Bio, Inc. | 3-0578 | Cartilha controle positivo para a frente |
| trnL (5 'do exão)-C | Oblique Bio, Inc. | 3-0579 | Reverter cartilha controle positivo |
| trnLF-C-F1 | Oblique Bio, Inc. | 3-0864 | Atacante do tipo selvagem cartilha cogongrass |
| trnLF-C-R1 | Oblique Bio, Inc. | 3-0865 | Revertertipo selvagem cartilha cogongrass |
| trnLF-R-F2 | Oblique Bio, Inc. | 3-0866 | Encaminhar JBG e JBG reverter cartilha |
| trnLF-R-R2 | Oblique Bio, Inc. | 3-0867 | Reverter JBG e JBG reverter cartilha |
| Vantagem UltraPure PCR dNTP Mix | Clontech, Mountain View, CA | 639125 | |
| Polimerase vantagem de 2 | Clontech, Mountain View, CA | 639201 ou 639202 | Uma boa leitura de prova, hot-start Taq polimerase |
Referências
- Holm, L. G., Pancho, J. V., Herberger, J. P., Plucknett, D. L. A Geographical Atlas of World Weeds. , Krieger Publishing Company. Malabar, Florida, U.S. (1979).
- CABI, Crop Protection Compendium. Commonwealth Agricultural Bureau International (CABI). , Wallingford, UK. (2007).
- MacDonald, G. E. Cogongrass (Imperata cylindrica) - Biology, ecology, and management. Critical Reviews in Plant Sciences. 23, 367-380 (2004).
- Talley, S. M., Cseke, L. J. Molecular diagnostic technologies for invasive plants. CPHST Fort Collins Laboratory 2009 Annual Report. Zink, R. , USDA-APHIS-PPQ-CPHST. Fort Collins, Colorado. 27-28 (2009).
- Dozier, H., Gaffney, J. F., McDonald, S. -K., Johnson, E. R. R. L., Shilling, D. G. Cogongrass in the United States: History, ecology, impacts, and management. Weed Technology. 12, 737-743 (1998).
- Chikoye, D., Ekeleme, F. Weed flora and soil seedbanks in fields dominated by Imperata cylindrica in the moist savannah of West Africa. Weed Research. 41, 475-490 (2001).
- Weber, E. Invasive Plant Species of the World: A Reference Guide to Environmental Weeds. , CABI Publishing. Wallingford, UK. (2003).
- Patterson, D. T. Shading effects on growth and partitioning of plant biomass in Cogongrass (Imperata cylindrica) from shaded and exposed habitats. Weed Science. 28, 735-740 (1980).
- Cole, J. T., Cole, J. C. Ornamental grass growth response to three shade intensities. Journal of Environmental Horticulture. 18, 18-22 (2000).
- Bryson, C. T., Koger, C. H., Byrd, J. D. Effects of temperature and exposure period to heat on cogongrass (Imperata cylindrica) viability. Weed Technology. 21, 141-144 (2007).
- Capo-chichi, L. J. A., Faircloth, W. H., Williamson, A. G., Patterson, M. G., Miller, J. H., van Santen, E. Invasion Dynamics and Genotypic Diversity of Cogongrass (Imperata cylindrica) at the Point of Introduction in the Southeastern United States. Invasive Plant Science and Management. 1 (2), 133-141 (2008).
- Talley, S. M., Ramsey, C. L. Experimentally assessing the invasive potential of plants. CPHST Laboratory 2009 Annual Report. Zink, R. , USDA-APHIS-PPQ-CPHST. Fort Collins, Colorado. 29-30 (2009).
- Hodkinson, T. R., Chase, M. W., Lledó, M. D., Salamin, N., Renvoize, S. A. Phylogenetics of Miscanthus, Saccharum and related genera (Saccharinae, Andropogoneae, Poaceae) based on DNA sequences from ITS nuclear ribosomal DNA and plastid trnLintron and trnL-F intergenic spacers. J. Plant Res. 115, 381-392 (2002).
- Kress, W. J., Wurdack, K. J., Zimmer, E. A., Weigt, L. A., Janzen, D. H. Use of DNA barcodes to identify flowering plants. Proc. Natl. Acad. Sci. U.S.A. 102 (23), 8369-8374 (2005).
- Roodt-Wilding, R., Spies, J. J. Phylogenetic relationships in southern African chloridoid grasses (Poaceae) based on nuclear and chloroplast sequence data. Systematics and Biodiversity. 4, 401-415 (2006).
- Brody, J. R., Kern, S. E. Sodium boric acid: a Tris-free, cooler conductive medium for DNA electrophoresis. BioTechniques. 36, 214-216 (2004).
- Chou, C. -H., Tsai, C. C. Genetic variation in the intergenic spacer of ribosomal DNA of Imperata cylindrica (L.) Beauv. var. major (Cogongrass) populations in Taiwan. Botanical Bulletin of Academia Sinica (Taipei). 40, 319-332 (1999).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados