É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Detecção de multiplex de bactérias em amostras clínicas e Complexo Ambiental utilizando Oligonucleotídeos-coupled microesferas fluorescentes
Neste Artigo
Resumo
Nós descrevemos um método multiplex para a detecção de microorganismos em uma amostra usando oligonucleotídeos-coupled partículas fluorescentes. Amplicon de todos os organismos dentro de uma amostra é hibridizada com um painel de sonda acoplada contas. Um instrumento Luminex ou Bio-Plex é usado para consultar cada grânulo para o tipo de grânulo e sinal de hibridização.
Resumo
Vaginose bacteriana (BV) é uma síndrome recorrente polimicrobiana que se caracteriza por uma mudança na microbiota "normal" de Lactobacillus dominado a uma microbiota dominado por um número de espécies de bactérias, incluindo Gardnerella vaginalis, Atopobium vaginae, e outros 1-3. Esta condição está associada com uma série de resultados negativos de saúde, incluindo HIV aquisição 4, e pode ser difícil de gerir clinicamente 5. Além disso, o diagnóstico de BV tem contado com o uso de manchas Gram de esfregaço de swab vaginal que são pontuados em vários critérios numéricos 6,7. Embora este diagnóstico é simples, barato e bem adequado para ambientes de recursos limitados, que podem sofrer de problemas relacionados com interpretações subjetivas e não dar um perfil detalhado da composição da microbiota vaginal 8. Recentes esforços de seqüenciamento de profundidade revelaram uma rica, diversificada microbiota vaginal comdiferenças claras entre as amostras retiradas de indivíduos que são diagnosticados com BV em comparação com aqueles indivíduos que são considerados normais 9,10, o que resultou na identificação de um número de alvos potenciais para o diagnóstico molecular da BV 11,12. Esses estudos têm fornecido uma riqueza de informação útil, mas seqüenciamento profunda ainda não é prática como um método de diagnóstico em um ambiente clínico. Recentemente, descreveu um método para rapidamente perfil da microbiota vaginal em um formato multiplex usando oligonucleotídeos-coupled partículas fluorescentes com detecção em uma plataforma Luminex 13. Este método, como o atual Gram métodos baseados, é rápido e simples, mas acrescenta a vantagem adicional de explorar os conhecimentos moleculares decorrentes de estudos de seqüenciamento no projeto da sonda. Este método, portanto, fornece uma maneira de perfil dos microorganismos mais importantes que estão presentes em um swab vaginal que podem ser usadas para diagnosticar BV, com alta especificidade e sensibilidade compared para Gram, fornecendo informações adicionais sobre a presença e abundância de espécies de uma forma semi-quantitativa e rápida. Este método multiplex é expansível bem além do alcance dos atuais ensaios de PCR quantitativo para os organismos particular, que é actualmente limitado a 5 ou 6 ensaios diferentes em uma única amostra 14. Importante, o método não se limita à detecção de bactérias em swabs vaginal e pode ser facilmente adaptado para rapidamente perfil quase qualquer comunidade microbiana de interesse. Por exemplo, temos recentemente começaram a aplicar esta metodologia para o desenvolvimento de ferramentas de diagnóstico para uso em estações de tratamento de águas residuais.
Protocolo
Este método foi utilizado na pesquisa, publicada na Dumonceaux et al. J. Clin. . Microbiol 47, 4067-4077, doi: 10.1128/jcm.00112-09 (2009).
Um diagrama esquemático representando o processo geral é apresentado na Figura 1.
1. Talão de acoplamento
Este descreve os métodos a serem utilizados para o acoplamento de sondas de oligonucleotídeos esferas de poliestireno Luminex (ver Tabela 2). Volumes são adaptados ligeiramente para a avaliação de sondas de captura de novos em uma base experimental, estes volumes são indicados entre parênteses.
- Retire 1-etil-3-(3-dimethylamiopropyl) carbodiimida HCl pó (EDC) de -20 ° C e dessecador aquecê-lo à temperatura ambiente.
- Ressuspender as microesferas por sonciation em um banho de água sonicador por 20 segundos, então vórtex aproximadamente 20 segundos.
- Transferir 400 mL (100 L) de microesferas de um tubo de Eppendorf (isto corresponde a 5x10 6 ou 1,25 x10 6 microesferas). Luminex sugere o uso de baixa proteína tubos de microcentrífuga de ligação (por exemplo, tubos Eppendorf LoBind Protein, número de catálogo 0030 108,094) para evitar que as contas desacoplado de degola para os tubos e interferindo com a recuperação do grânulo. Não encontramos este é um problema usando tubos de polipropileno padrão de microcentrífuga, que normalmente usam (por exemplo, número de catálogo VWR 87003-298).
- Pelotização em 14.000 xg por 1 minuto. Remova e descarte o sobrenadante.
- Ressuspender as microesferas em 50 ul (12,5 L) de temperatura ambiente 0,1 M 2 - (N-Morfolino) etanosulfónico (MES) pH 4.5.
- Prepare uma nova solução de EDC em 10 mg / ml em água.
- Prepare menos de 1 ml desta solução, pesando 50-10 mg de EDC em uma escala de análise, em seguida, adicionar água a 10 mg / ml.
- Adicione 1 nmol de 5'-amino oligonucleotídeo capturar C12-modificado (Tabela 2) para micro e misturar em vortex. 1 nmol é de 5 mL de 200 M oligonucleotídeos.
- Adicionar 2,5 ml de solução EDC fresco para as micro e misturar em vortex por 5 segundos.
- Incubar à temperatura ambiente por 30 minutos no escuro.
- Descartar a solução EDC (passo 1.6) e preparar uma nova amostra de 10 mg / ml em água EDC como acima (passo 1.6).
- Adicionar outro 2,5 mL de solução EDC fresco para as micro e vortex por 5 segundos.
- Incubar à temperatura ambiente por 30 minutos no escuro.
- Lave as contas adicionando 1 ml de 0,02% Tween 20. Vortex (opcionalmente sonicate por 20 segundos também) para voltar a suspender as contas.
- Centrifugar 14000 xg 1 minuto. Remova e descarte o sobrenadante.
- Lave as contas novamente, adicionando 1 ml de sulfato de sódio 0,1% dodecil (SDS). Vortex (opcionalmente sonicate por 20 segundos também) para voltar a suspender as contas.
- Centrifugar 14000 xg 1 minuto. Remova e descarte o sobrenadante.
- Volte a suspender as contas em 100 l (25 L) de Tris-EDTA (TE) buffer [10 mM Tris-Cl pH 8.0, 1 mM EDTA, pH 8,0].
- Enumerar as contas em um hemocitómetro ou contador Coulter para determinar a concentração de ações.
- Prepare um Mix Master Microesfera diluindo cada grânulo para uma concentração final de 100 pérolas / mL em tampão TE. Piscina acoplada contas correspondente ao complexo desejado do ensaio (por exemplo, para um ensaio de 10-plex, mix de 10 diferentes contas acoplado a uma concentração final de 100/μl de cada talão).
- Armazenar o Mix Master Microesfera a 4 ° C no escuro. A mistura pode ser armazenada por meses se mantido sob estas condições.
2. Chaperonin 60 universal alvo (cpn60 UT) amplicon de produção e geração de cadeias simples.
- Gerar reação em cadeia da polimerase produto (PCR) para cada amostra. Inclua o fosforotioato e set-biotina modificado 5 'primer (Tabela 1). Veja a Tabela 3 para os volumes a misturar e concentrações.
- Imediatamente após a PCR está completa, adicione 2 mL (20 unidades) de ex T7onuclease a cada tubo de PCR (PCR buffer será suficiente para a reação T7). Incubar a reação à temperatura ambiente (~ 22-25 ° C) por 40 minutos.
- No final desta incubação, adicionar 12,5 mL de 0,5 M de etileno diamina ácido tetraacetic (EDTA) pH 8,0 e misturar. Isto dá um total de ~ 64.5μl de single-stranded produto da PCR.
3. Hibridação de single-stranded do produto de PCR para contas de oligonucleotídeo-coupled poliestireno.
- Pré-aquecer o instrumento a 60 ° C para manter a temperatura de hibridização durante a análise. Ligar o aquecedor plataforma usando o software do instrumento e não se esqueça de usar o bloco de aquecimento de latão que se encaixa a placa de PCR-estilo que contém a mistura hybridized talão.
- Ressuspender Microesfera Mix Master (passo 1.20) com uma pipeta, dispensar uma quantidade adequada em um tubo Eppendorf, cap do tubo, e sonicate em um banho de água sonicador por 2 minutos. Como alternativa, para assegurar a coerência absoluta no talão resuspension, o master mix microesfera pode ser ressuspenso em vórtice e pipetagem, então sonicação como acima, então dispensar a quantidade desejada em um tubo Eppendorf de polipropileno.
- Dispense 17 mL de single-stranded produto da PCR (passo 2.2) nos poços apropriados de um perfil de baixa de 96 poços Poço PCR placa (Tabela 2). Adicionar 33 mL de sedimento, mistura sonciated talão para cada poço. Cubra com capa de silicone (Tabela 2) e bata suavemente.
- Coloque a placa no termociclador com um programa de: 95 ° C por 5 min, 60 ° C por 10 min, 60 ° C segurar, 60 ° C por 5 min, fim. Iniciar o programa.
- Faça novos estreptavidina-ficoeritrina (SA-PE) solução; você precisará de 25 mL por poço (fazer vários poços extra). Faça SA-PE solução diluindo estoque SA-PE (1 mg / ml) 01:50 a 20 mcg / ml com 1x cloreto de tetrametilamónio (TMAC) buffer (3 M TMAC, 0,1% Sarkosyl, 50 mM Tris-HCl, pH 8,0 ; 4 mM EDTA, pH 8,0).
- Quando o termociclador chega ao 60 passo segurar ° C,abrir a tampa, retire a tampa de silicone e adicione SA-PE solução diretamente a cada poço (NÃO ter a placa fora do termociclador). Recoloque a tampa do silicone, feche a tampa do termociclador e retomar o programa.
- Quando o programa estiver concluído, pegar a placa para fora e rapidamente transferi-lo para a máquina BioPlex de ler. Placa deve ser lido em 10 minutos. Leia a 60 ° C; garantir que o BioPlex foi pré-aquecido a esta temperatura. Esteja certo de que a altura da sonda foi ajustado para acomodar a placa utilizada, conforme descrito no manual do usuário do instrumento utilizado.
- Para talão precisas e detecção de sinal, as configurações da porta na BioPlex deve ser definida de acordo com o tipo de microesferas a ser utilizado. Esferas de poliestireno BioRad exigem uma configuração porta de 4,335-10,000 enquanto microesferas magnéticas exigem um ajuste de 5,000-25,000. Há também a opção de executar o prato usando a configuração PMT alta o que aumenta o valor do ganho repórter. Isto pode increase a intensidade dos sinais de menor entanto, é importante incluir um controle apropriado negativos como sinal de fundo também será aumentado.
4. Resultados representativos:
Um dos nossos objetivos no desenvolvimento e implementação deste ensaio foi para torná-lo o mais simples e aerodinâmico possível. Por isso, otimizou a crítica pós-amplificação etapas, incluindo o tempo de tratamento T7 exonuclease e tempo de hibridização, a fim de desenvolver um teste que pode ser concluído em um prazo razoável. Conforme mostrado na Figura 2A, T7 tratamento do amplicon é essencial para a geração do sinal, como a maioria das sondas tinha pouco ou nenhum sinal de curto ou nenhum tratamento T7. O sinal de aumento linear até aproximadamente 40 minutos, momento em que o aumento do sinal retardado. Nenhuma degradação do sinal foi observada mesmo em 2 horas de tempo de tratamento T7, indicando que a modificação fosforotioato dos primers é altamente eficaz na prevenção target degradação vertente, tal como descrito 15. Escolhemos 40 minutos para T7 tempo de tratamento para minimizar o tempo de protocolo geral, mas é claro da Figura 2A que o tratamento T7 pode continuar por muito mais tempo. Determinou-se também o efeito do tempo de hibridização na geração de sinal (Figura 2B) e descobriu que 10 minutos era suficiente para a máxima do sinal, como nenhum novo aumento de sinal foi observada mesmo após 4 horas de hibridização. Portanto, um passo de hibridação de 10 minutos foi escolhido, novamente para minimizar o tempo de ensaio geral. Com estes resultados em mente, e com as técnicas de extração de DNA rápida como InstaGene (Bio-Rad), o ensaio geral, que inclui a extração de DNA, PCR, análise e Luminex pode ser concluído em 4-5 horas.
Em uma nota de advertência, o nível de agregação das esferas de poliestireno durante uma corrida ou Luminex BioPlex pode ter um impacto importante sobre a eficiência do ensaio. O software BioPlex irá exibir o nível de agregação de talão, que ocorre quandomais de um talão é detectado no caminho de laser e resulta na exclusão do sinal de hibridização do agregado. Agregação de talão aparente também pode ser causada por qualquer partícula que não é o tamanho adequado para um cordão simples, e pode até mesmo ser causada por bolhas de ar. Em qualquer um desses eventos, o sinal do agregado talão, bolha de ar, ou partícula é descartado. Na maioria dos casos, descobrimos que a agregação de talão é mínima (Figura 3A) e do nível-alvo de 100 eventos de contagem para cada grânulo é facilmente realizável. Ocasionalmente, no entanto, as contas mostram uma moderada (Figura 3B) ou grave nível (Figura 3C) de agregação. Nestes casos, já que a maioria dos dados é descartado, o instrumento pode ter dificuldade para chegar a 100 eventos por tipo de contas e os resultados podem ser questionáveis. A etapa de sonicação (passo 3.2) é utilizado para minimizar a agregação de contas. Além disso, armazenar as contas como uma mistura diluída micro master (passo 1.20) pode ajudar. Temos notado que TMAC excluindo do hybritampão dization - adicionando-o ao diluente SA-PE vez (passo 3.5) - ajuda a minimizar a agregação de contas. Além disso, enquanto nós não testei isso, as contas mais recentes magnética disponível a partir de Luminex ou BioRad são pensados para mostrar menos de uma tendência para agregar.
Os resultados da aplicação de uma matriz 5 Luminex-plex segmentação G. vaginalis, A. vaginae, L. iners, L. crispatus, e L. gasseri juntamente com correspondentes Gram esfregaços vaginais corados swab são mostrados na Figura 4. Estas amostras foram tomadas a partir de um único indivíduo em momentos diversos. No tempo 0, o indivíduo foi diagnosticado com BV com base na coloração de Gram (Figura 4A) e os resultados Luminex a partir desta mesma amostra (Figura 4B) mostram que dos organismos representados na matriz, G. vaginalis foi mais prevalente, enquanto A. vaginae e L. iners também foram positivos. Nove dias depois, o indivíduo ainda era BV positivo eo signal para G. vaginalis tinha aumentado substancialmente, enquanto A. vaginae e L. iners ainda eram positivas. Importante, após esse tempo o indivíduo começou a transição para uma microbiota normal, como bacilos Gram positivos se tornou detectável nos esfregaços (Figura 4A), enquanto o sinal para G. vaginalis e diminuiu o sinal para L. iners aumentou (Figura 4B). Pela nossa definição original do BV com este método (amostras positivas para G. vaginalis e / ou A. vaginae foram considerados positivos BV) 13, esse indivíduo era BV positiva em todos os momentos, embora o Gram falhou em detectar G. vaginalis nos dois últimos pontos do tempo. Com o método descrito aqui Luminex, as tendências podem ser facilmente comparada com métodos baseados em seqüenciamento, e os resultados do ensaio Luminex normalmente corroboram manchas Gram 13 ao fornecer informações adicionais sobre a identidade organismo e abundância.

Figura 1. Diagrama esquemático do protocolo Luminex para determinar o perfil microbiota de uma amostra clínica complexa ou ambientais. O protocolo começa com DNA modelo que foi extraído da amostra de interesse. (1) Gerar produto da PCR de DNA modelo usando fio específico biotinilado, fosforotioato modificados cpn60 primers UT PCR juntamente com inalterado cpn60 UT PCR (Tabela 1), (2) Isto gera um pool de produtos PCR representando o microbiota com a biotina- fosforotioato modificação em uma fita (3); Digest o double-stranded produto de PCR com T7 exonuclease, que não pode degradar a vertente fosforotioato modificados e, portanto, gera single-stranded DNA que é modificado no terminal 5 'com biotina; (4) Couple esferas de poliestireno a espécie-específicos cpn60 sondas UT - cada conta tem um endereço exclusivo espectral (indicado pela cor pérola) que is discernível pela Luminex ou Bio-Plex instrumento; (5) Combine o single-stranded produto de PCR gerados a partir da amostra de interesse para o conjunto de espécies específicas de oligonucleotídeos-coupled contas; (6) Adicionar estreptavidina-ficoeritrina conjugada que se liga a biotinilado o single-stranded produto de PCR e atua como um indicador da hibridação, (7) Determine o endereço espectral (talão de identidade) e intensidade de sinal de hibridização utilizando um Luminex ou Bio-Plex instrumento. Pelo menos 100 contas são contados para cada identidade de contas e da intensidade mediana de fluorescência (MFI) do sinal de ficoeritrina é relatada como a saída. Até 100 diferentes tipos do grânulo podem ser avaliados simultaneamente, mas um ensaio mostrando a discriminação de três tipos do grânulo é ilustrado. (8) Quando o MFI de PCR replica gerada a partir da mesma amostra é significativamente maior do que o controle negativo para um cordão de dado (unicaudal aluno t-test, p <0,05), a amostra é considerada positiva para o organismo.
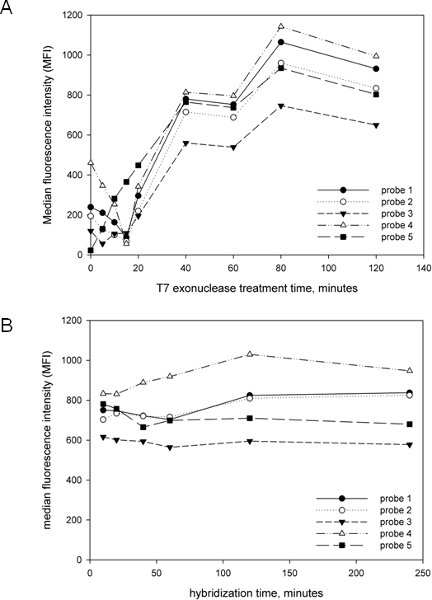
Figura 2. Otimização dos parâmetros de ensaio Luminex. Cinco sondas diferentes direcionados para anaerobius Peptostreptococcus foram usados com amplicons gerados a partir de um modelo misto, composto por plasmídeos contendo o clonado cpn60 UT de 20 bactérias conhecidas para ser associado com a vagina, incluindo P. anaerobius. (A). Efeito do tempo de tratamento T7 exonuclease. O protocolo descrito acima foi seguido, mas o tempo de tratamento com exonuclease T7 foi variado antes de hibridação e da intensidade mediana de fluorescência (MFI) foi determinada para todas as sondas. (B). Efeito do tempo de hibridização. O protocolo descrito acima foi seguido com uma variedade de tempos de hibridização. Os mesmos conjuntos de sondas foram usadas com um amplicon gerado a partir do mesmo modelo como em A ea intensidade mediana de fluorescência (MFI) foi determinada para todas as sondas.
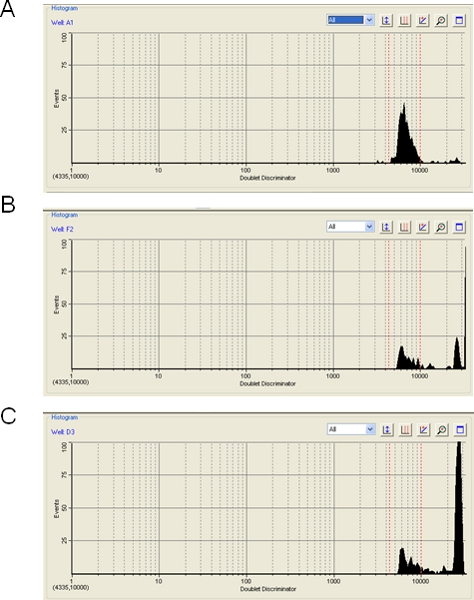
Figura 3. Determinação de agregação talão usando software BioPlex. Três exemplos são dados de pistas BioPlex em que o nível de talão de agregação é (A) aceitável (5%); (B) borderline (50%), e (C) inaceitável (80%).
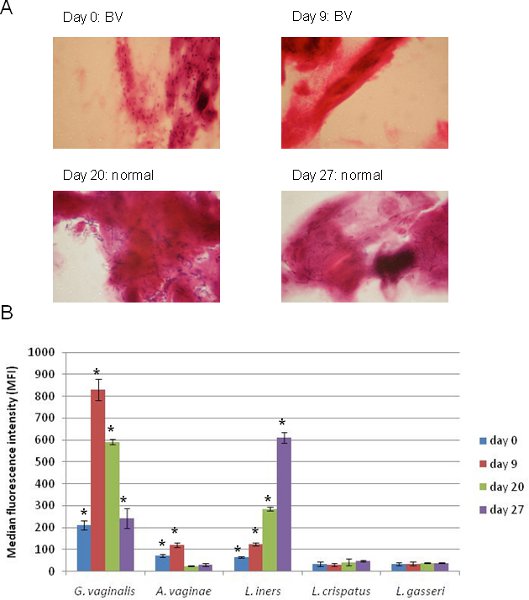
Figura 4. Aplicação de uma matriz 5 Luminex-plex a BV diagnóstico em amostras seqüenciais retirado do mesmo indivíduo. (A). Gram slides mostrando o diagnóstico tradicionais utilizados para avaliar cada amostra de BV. (B). Aplicação de uma matriz 5 Luminex-plex preparado e executado, conforme descrito neste protocolo para as mesmas quatro amostras mostrado em (A). Alvos bacteriana cujo sinal MFI foi significativamente positiva por nossa definição (unicaudal teste t de Student, p <0,05) são indicados por um asterisco (*).
Discussão
A especificidade da geração do sinal é de fundamental importância, você deve estar confiante de que o sinal observado reflecte verdadeiramente a detecção de amplicon gerado a partir desse organismo. Software, tais como PrimerPlex (Premier Biosoft) pode ajudar a projetar as sondas que hibridizam de forma eficiente, mas eles podem ou não podem cruzar-hibridizar espécies não-alvo. Quando primers universais PCR são utilizados, como descrito neste protocolo, é importante ter em mente que o amplicon gerado represe...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Agradecemos a Alberto Severini e Vanessa Goleski para ajudar no desenvolvimento de ensaios e comentários críticos sobre este manuscrito. Este trabalho foi financiado pela Agência Canadense de Saúde Pública e do Programa de Assistência de Pesquisa Industrial (National Research Council of Canada). Apoio adicional foi obtida a partir da Universidade de Saskatchewan Fundo de Publicação.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome oligonucleotídeo | Companhia | Sequência 1 | |
| H279BP | Invitrogen, IDT, ou outros | Biotina-OEFO GAIIIIGCIGGIGAYGGIACIACIAC | |
| H280 | YKIYKITCICCRAAICCIGGIGCYTT | ||
| H1612BP | Biotina-OEFO GAIIIIGCIGGYGACGGYACSACSAC | ||
| H1613 | CGRCGRTCRCCGAAGCCSGGIGCCTT | ||
| 1O, fosforotioato-C, E, fosforotioato-G, F, fosforotioato-A; I, inosina, Y, C ou T; R, A ou G, K, T ou G, S, C ou G. | |||
Tabela 1. Seqüências de oligonucleotídeos modificados para o universal cpn60 PCR.
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| 1-etil-3-(3-dimethylamiopropyl) carbodiimida HCl (EDC) | Perfurar | 22980 | |
| Fluorescentes esferas de poliestireno: Microplex microesferas (Luminex), ou Bio-Plex COOH Bead (Bio-Rad) | Luminex ou Bio-Rad | Bio-Rad: 171-506xxx onde xxx corresponde ao identificador de talão | Esferas magnéticas estão se tornando disponíveis para o acoplamento de oligonucleotídeos e podem oferecer algumas vantagens. Que não tenham sido julgados por esses autores. |
| Oligonucleotídeos de captura (5 'amino C12 modificados) | Invitrogen, IDT, ou outros | vários | Grau de pureza dessalinizada é aceitável. Seqüências das sondas de captura usado para caracterizar swabs vaginal são fornecidos no manuscrito em que este protocolo é baseado em 13. |
| T7exonuclease | New England Biolabs | M0263S | |
| Estreptavidina-R-ficoeritrina (SA-PE) | Invitrogen | S-866 | Tenha o cuidado de obter alta pureza SA-PE, este número de catálogo é recomendado |
| Thermowell 96 placas bem PCR | Pescador | CS006509 | se encaixam em ambos os termociclador de 96 poços e máquina BioPlex |
| Thermowell vedação mat | Pescador | CS006555 | Pode ser re-utilizados; lavar com água e sabão, enxágüe bem e seque |
| TMAC 5M | Sigma | T3411 | |
| Bio-Plex ou Luminex instrumento | Bio-Rad ou Luminex | Bio-Rad: 171-000201 | |
| PrimerPlex software para o projeto da sonda | Premier Biosoft | www.premierbiosoft.com | Sugerido para Luminex sonda desinal, apesar de outras plataformas de software pode ser usado |
Tabela 2. Reagentes específicos e equipamentos.
| componente | mL / ensaio | ensaios μl/100 | concentração final |
| 10x PCR buffer (Invitrogen) | 5 | 500 | 1x |
| 50 mM MgCl 2 (Invitrogen) | 2,5 | 250 | 2,5 mM |
| 10 mM dNTP | 1 | 100 | 0,2 mM de cada |
| H279BP, 25 mM | 0,25 | 25 | 375 nM |
| H1612BP, 25 mM | 0,75 | 75 | 125 nM |
| H280, 25 mM | 0,25 | 25 | 375 nM |
| H1613,25 mM | 0,75 | 75 | 125 nM |
| Água | 34 | 3400 | --- |
| Totais | 44,5 | 4450 |
Tabela 3. Sugerida misturas de PCR com primers modificada cpn60 UT (Tabela 1). O ensaio está configurado para 5 mL de DNA modelo e 0,5 mL (2.5U) de Taq DNA polimerase (Invitrogen). Normalmente, grandes volumes são preparados (por exemplo, suficiente para 100 testes) e armazenadas a -20 ° C.
Referências
- Hale, L. P., Swidsinski, A., Mendling, W. Bacteria associated with bacterial vaginosis. N. Engl. J. Med. 354, 202-203 (2006).
- Hay, P. Life in the littoral zone: lactobacilli losing the plot. Sex. Transm. Infect. 81, 100-102 (2005).
- Morris, M., Nicoll, A., Simms, I., Wilson, J., Catchpole, M. Bacterial vaginosis: a public health review. Brit. J. Obstet. Gynecol. 108, 439-450 (2001).
- Myer, L., Kuhn, L., Stein, Z. A., Wright, T. C., Denny, L. Intravaginal practices, bacterial vaginosis, and women's susceptibility to HIV infection: epidemiological evidence and biological mechanisms. Lancet. Infect. Dis. 5, 786-794 (2005).
- Wilson, J. Managing recurrent bacterial vaginosis. Sex. Transm. Infect. 80, 8-11 (2004).
- Nugent, R. P., Krohn, M. A., Hillier, S. L. Reliability of diagnosing bacterial vaginosis is improved by a standardized method of gram stain interpretation. J. Clin. Microbiol. 29, 297-301 (1991).
- Ison, C. A., Hay, P. E. Validation of a simplified grading of Gram stained vaginal smears for use in genitourinary medicine clinics. Sex. Transm. Infect. 78, 413-415 (2002).
- Money, D. The laboratory diagnosis of bacterial vaginosis. Can. J. Infect. Dis. Med. Microbiol. 16, 77-79 (2005).
- Schellenberg, J. Pyrosequencing of the chaperonin-60 universal target as a tool for determining microbial community composition. Appl. Environ. Microbiol. 75, 2889-2898 (2009).
- Spear, G. T. Comparison of the diversity of the vaginal microbiota in HIV-infected and HIV-uninfected women with or without bacterial vaginosis. J. Infect. Dis. 198, 1131-1140 (2008).
- Brotman, R. M., Ravel, J. Ready or not: the molecular diagnosis of bacterial vaginosis. Clin. Infect. Dis. 47, 44-46 (2008).
- Fredricks, D. N., Fiedler, T. L., Marrazzo, J. M. Molecular identification of bacteria associated with bacterial vaginosis. N. Engl. J. Med. 353, 1899-1911 (2005).
- Dumonceaux, T. J. Multiplex detection of bacteria associated with normal microbiota and with bacterial vaginosis in vaginal swabs by use of oligonucleotide-coupled fluorescent microspheres. J. Clin. Microbiol. 47, 4067-4077 (2009).
- Molenkamp, R., van der Ham, A., Schinkel, J., Beld, M. Simultaneous detection of five different DNA targets by real-time Taqman PCR using the Roche LightCycler480: Application in viral molecular diagnostics. J. Virol. Meth. 141, 205-211 (2007).
- Nikiforov, T. T., Rendle, R. B., Kotewicz, M. L., Rogers, Y. H. The use of phosphorothioate primers and exonuclease hydrolysis for the preparation of single-stranded PCR products and their detection by solid-phase hybridization. PCR. Methods. Appl. 3, 285-291 (1994).
- Hill, J. E., Penny, S. L., Crowell, K. G., Goh, S. H., Hemmingsen, S. M. cpnDB: A Chaperonin Sequence Database. Genome. Res. 14, 1669-1675 (2004).
- Bradshaw, C. S. The association of Atopobium vaginae and Gardnerella vaginalis with bacterial vaginosis and recurrence after oral metronidazole therapy. J. Infect. Dis. 194, 828-836 (2006).
- Dumonceaux, T. J. Enumeration of specific bacterial populations in complex intestinal communities using quantitative PCR based on the chaperonin-60 target. J. Microbiol. Meth. 64, 46-62 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados