Method Article
Profiling Proteome Redox tiol Usando Isótopos Espectrometria de Massa Tagging
Neste Artigo
Resumo
Nível de espécies reativas de oxigênio é elevado quando as células encontrar condições de estresse. Aqui mostramos o exemplo de 3'-3 'diaminobenzidina coloração, bem como cysTMT rotulagem e espectrometria de massa para traçar o perfil do proteoma redox em Pseudomonas syringae Tratada folhas de tomateiro.
Resumo
Pseudomonas syringae pv DC3000 estirpe. Tomate não só provoca a doença de pinta bacteriana em Solanum lycopersicum, mas também sobre as espécies de Brassica, bem como em Arabidopsis thaliana, um 1,2 planta geneticamente tratável hospedeiro. O acúmulo de espécies reativas de oxigênio (ROS) em cotilédones inoculados com DC3000 indica um papel de ROS na morte celular modulando necrótico durante a doença mancha bacteriana do tomateiro, 3. O peróxido de hidrogénio, um componente de ROS, é produzida após a inoculação de plantas de tomate com Pseudomonas 3. O peróxido de hidrogénio pode ser detectada usando um histoquímica mancha 3'-3 'diaminobenzidina (DAB) 4. Coloração DAB reage com o peróxido de hidrogénio para produzir uma mancha marrom no 4 tecido foliar. ROS tem um papel regulador do ambiente celular redox, o que pode alterar o estado redox de certas proteínas 5. A cisteína é um aminoácido importante sensível àalterações redox. Sob oxidação suave, a oxidação reversível de grupos de cisteína sulfidrilo serve como sensores de redox e transdutores de sinal que regulam uma variedade de processos fisiológicos 6,7. Massa em tandem tag (TMT) reagentes permitir a identificação simultânea e multiplexada quantificação de proteínas em amostras diferentes, usando espectrometria de massa 8,9. As cisteína-reactivas TMT (cysTMT) reagentes permitir rotulagem selectiva e quantificação relativa de cisteína contendo péptidos de até seis amostras biológicas. Cada etiqueta cysTMT isobárica tem o mesmo pai massa nominal e é composto de um grupo sulfidrilo reactivo, um braço espaçador MS-neutro e um repórter MS / MS 10. Após a marcação, as amostras foram sujeitas a digestão de protease. Os péptidos-cisteína marcadas foram enriquecidos utilizando uma resina contendo anti-TMT anticorpo. Durante MS / MS, análise de uma série de iões repórter (isto é, 126-131 Da) emergir na região de baixa massa, fornecendo informação sobre a quantificação relativa.O fluxo de trabalho é eficaz para reduzir a complexidade da amostra, melhorando a gama dinâmica e estudar modificações de cisteína. Aqui apresentamos uma análise proteômica redox do tomate DC3000 Pst tratado (Rio Grande) deixa usar a tecnologia cysTMT. Este método de alto rendimento tem o potencial de ser aplicado para estudar outras redox-regulados processos fisiológicos.
Protocolo
1. Produção de mudas e bactérias Preparação
- As plântulas são germinadas em Metromix mistura de solo 500 (Companies IBW) numa câmara de crescimento (160 umol fotões m -2 s -1 com um fotoperíodo de 8 horas de luz a 22 ° C e 16 horas escuro a 20 ° C durante 1 semana. Relativa humidade foi ajustada para 70%. As plântulas foram regadas com água da torneira, conforme necessário para manter o solo de secarem.
- Duas mudas de tamanho semelhante são então transplantadas para vasos de 4 "de diâmetro contendo o solo 500 Metromix e cultivadas durante mais 3 semanas nas mesmas condições que 1,1.
- Pseudomonas syringae (PST) é inicialmente T-listado na Kings B Médio (KBM) placas (por litro: 20 g peptona proteose, 0,1975 g MgSO4, Glicerol 1%, 1,5 g de K 2 HPO 4, 18 g Ágar) e crescidas durante dois dias a 28 ° C. Uma única colônia é coletado, misturado em 300 l Reis líquido B Médio (20 g Proteose peptona, 0,1975 g MgSO 4 , glicerol a 10 mL, 10 mL de 100x 2 HPO 4 K de stock (1,5 g de K 2 HPO 4 em 10 mL de H2O), e espalhada sobre uma placa de Reis B Médio. Este é cultivada durante a noite a 28 ° C.
- O inoculo de bactérias é preparado por raspagem das bactérias cultivadas a partir da placa KBM em 20 mL de H2O Uma DO600 de 0,03 (~ 10 6 ufc / ml) é preparado em 1 L de tampão de inoculação (10 mM de MgCl2 + 400 uL Silwett-70). Tampão de inoculação menos bactérias foi preparado para a solução de controlo.
2. A inoculação de Tomate com Pst e H 2 O 2 histoquímica Stain
- Quatro semanas de idade plantas foram inoculadas por meio de imersão na solução de inoculação acima durante 30 segundos. Sacos de plástico foram colocados sobre as plantas imediatamente após a inoculação.
- 3'-3 'diaminobenzidina (DAB) a mancha 11 foi preparado (1 mg / ml DAB em H2O, pH 3,8 (HCl)) três days antes de usar para atingir a solubilização completa. A solução foi sacudido para misturar à temperatura ambiente.
- Vinte e quatro horas após o tratamento, a folha totalmente expandida foi removido, colocado em DAB mancha lado epidérmico acima, e sob vácuo infiltrada durante 15 min. As folhas foram então colocadas no escuro à temperatura ambiente durante a noite para coloração.
- As folhas foram fervidas em etanol a 95% durante 10 min e, em seguida mantidos em etanol a 75%.
3. Extração de Proteínas
- Após a colheita, as folhas do tomate foram moídos em pó fino com nitrogênio líquido utilizando almofariz e pilão.
- Para 0,5 g de tecido, adicionar 1,25 mL de Tris pH 8,8 tamponada de fenol e 1,25 mL / 0,5 g de tampão de extracção (0,1 M Tris-HCl pH 8,8, EDTA 10 mM, 0,9 M de sacarose), em seguida continuam de moagem para um min mais alguns num coifa.
- Transferir o extracto a Oakridge tubo de centrifugação (Thermo Fisher Scientific Companhia Nalgene) e agitar durante 2 horas à temperatura ambiente.
- Centrifugar a 5.000 xge 15 ° C durante 10 min. Transferir a fase superior fenol, para um novo tubo limpo.
- Back-extrair a fase aquosa com um volume igual de tampão extraído com fenol; agitar num agitador durante 30 min. Centrifugar e transferir o extracto para um tubo Falcon de novo.
- Repita o passo anterior mais uma vez e transferir o extrato para novo tubo Oakridge.
- Precipitar fenol proteínas extraídas pela adição de 5 volumes de acetato de amónio 0,1 M em metanol a 100% (armazenada a -20 ° C). Vortex e incubar à temperatura de -20 ° C durante a noite.
- Recolher o pelete de proteína por centrifugação a 20.000 xg, 4 ° C durante 20 min.
- Lavar o pellet de 2 vezes com o acetato de frio de amónio 0,1 M em metanol e 2 vezes com 80% de acetona fria. Adicionar 1,5 mL de etanol frio a 70% para sedimentar e suspender. Transferir para um tubo de microcentrífuga de 2 mL (EUA Scientific) e centrifugar a 14.000 rpm, 4 ° C durante 20 min. Remover o etanol.
- Seca-se a pelete brevemente num concentrador Speedvac e dissolver em uma extr proteínatampão de acção, por exemplo, Proteína ReadyPrep Reagente extraction kit 3 (8 M de ureia, CHAPS 4%, 40 mM de Tris-base, 2 M Tioureia) (Bio-Rad). Centrifugar a 14.000 rpm e 20 ° C durante 30 min para formar uma pelota. Recolher o sobrenadante.
- Medir a concentração de proteína usando um ensaio de proteína CB-X de acordo com o manual do fabricante (Geno Technology).
4. Preparação de Amostras e Rotulagem Peptide com cysTMTs
- Preparar amostra de proteína a uma concentração de 2-5 ug / uL. Aqui nós usamos 100 mg de proteína para cada marca de massa, 20 ul-50 amostras microlitro. Para esta experiência todos os 6 etiquetas foram utilizados.
- Adicionar volume igual de tampão de alquilação (100 mM Tris-HCl pH 7,5, 200 mM de iodoacetamida) para a amostra de proteína para bloquear o grupo tiol livre. Tampão deve ser preparado fresco. A alquilação é realizado a 37 ° C durante 1 hora.
- Precipitar a proteína por adição de 1 ml de acetona a 80% frio a -20 ° C durante a noite. Pelete de proteína do por centrifugação a 14.000rpm, 4 ° C durante 20 min. Lavar o pellet 3 vezes com acetona a 80% vórtice de cada vez. Remover o sobrenadante e deixar secar ao ar, enquanto seca sobre gelo.
- Ressuspender o sedimento em 50 uL de tampão de lise (6 M de ureia, 50 mM de base Tris, 1 mM de EDTA). 0,5 ul de 100 mM de Tris (2-carboxietil) fosfina (TCEP) é então adicionado para reduzir as pontes dissulfeto por incubação durante uma hora à temperatura ambiente.
- Preparar etiquetas por adição de 20 uL de acetonitrilo para os tubos de reagentes para solubilizar as etiquetas. Vortex e girar para o fundo.
- Lavar a TCEP extra usando uma centrífuga Microcon 3KD dispositivo de filtro (Millipore). Adicionar 50 uL de amostra para Microcon coluna 3KD, e 50 uL de tampão de lise para a coluna. Centrifugar durante 15 min a 10.000 xg e 4 ° C. Repetir este passo para um total de três vezes. Remover coluna e inverter para um tubo limpo. Centrifugar durante 5 min a 1000 xg e 4 ° C.
- Verifique o pH. Se necessário, ajustar o pH a 7,0-8,0 com 1 M de HCl.
- Adicionar 5 uL da cysTMO reagente de T para cada amostra (O kit de reagentes cysTMT fornece 20 uL etiquetas para até 500 ug de proteína. Este método utiliza 100 ug de proteína, por conseguinte, usamos um quinto da marca.). Permitir que a reacção prosseguir durante 2 horas à temperatura ambiente.
- As amostras foram combinadas com Tampão de amostra Laemmli (Bio-Rad) em uma razão de 1:1.
5. A remoção de não-fracionamento da amostra reagiu Tag
- As amostras foram fervidas durante 5 min e separados num gel de poliacrilamida pré-fundido 12% (Bio-Rad). O gel foi lavado três vezes em intervalos de 5 minutos com filtrada H2O e coradas com Azul de Coomassie (Bio-Rad) durante 1 hora. O gel foi corado de-dia para o outro com H2O
- Doze frações foram coletadas de cada pista gel. As fracções foram determinados por concentrações banda de proteína. As fracções foram cortadas em pedaços de 1 mm e recolhido num tubo de 1,5 ml.
- Pedaços de gel foram coradas utilizando de-acetonitrilo 50% e 0,1 M de bicarbonato de amónio em H 2 O. Este passo deve ser repetido até que a cor azul é removido a partir de pedaços de gel (~ de 3-4 vezes, 15 min de cada vez).
- Peças de-gel corado foram secos usando acetonitrilo a 100% (cobrir pedaços de gel) durante 15 min, o sobrenadante removido, e os pedaços secos usando um Speedvac.
- Dissolve-se tripsina (Promega) a 1:10 (tripsina: proteína) ratio no mesmo volume (como amostras combinadas rotulada) de bicarbonato de amónio 25 mM. Por exemplo, 600 ug de proteína da amostra com um volume de 500 uL precisa 60 ug de tripsina dissolvido em 500 uL de 25 mM de bicarbonato de amónio.
- Adicionar a solução de tripsina para os pedaços de gel e definir em gelo para re-hidratar. Se as peças não estão cobertas adicionar 50 mM de tampão de bicarbonato de amónio. Depois de re-hidratação, incubar a 37 ° C durante 12-16 hr.
- Remover o extracto de proteína de líquido a partir das amostras incubadas.
- Parar a reacção e remover qualquer digerir a proteína restante cobrindo pedaços de gel com 5% de ácido fórmico, acetonitrilo 50% .. Agitar pedaços de gel durante 20 min a sala de TEmperature. Transferir o sobrenadante para os tubos de amostra extraída de proteína a partir de 5,7. Repita o procedimento de extração duas vezes. Seca-se os péptidos extraídos utilizando o Speedvac.
6. Enriquecimento amostra de cysTMT rotuladas de peptídeos
- Adicionar um gradiente de anti-TMT resina para tubos de 0,5 ml para uma lama de 50%. (Gradiente foi determinada a partir da concentração banda em gel de fraccionamento. Se uma recolha de fracções consiste de uma banda escura, mais resina é necessária. As fracções com bandas mais fracas requerem menos resina como há menos proteína.)
- A resina é então lavada três vezes com um volume de coluna de Tris-1x solução salina tamponada com (TBS) (25 mM de Tris, 0,15 M NaCl, pH 7,2) (Thermo Scientific Pierce Research Products de proteína).
- Adicionar 200 uL 1xTBS para cada amostra. As amostras são então adicionados aos anti-TMT resina (Thermo Scientific Pierce Research Products de proteína) e agitado à temperatura ambiente durante 2 hr seguido por balançar durante a noite a 4 ° C.
- Adicionar amostra a coluna (ARMO Scientific Protein Pierce Produtos de Pesquisa).
- Lava-se cada coluna três vezes com TBS 1x 200 uL. Esta é seguida com lavagem três vezes com CHAPS 0,05% (dissolvido em 1x TBS).
- A coluna é então lavada três vezes com 4 M ureia em 1x TBS. Duzentos microlitros de H2O é então usada para lavar a coluna três vezes.
- Cada amostra é eluída três vezes com 200 uL de tampão de eluição de 50% (acetonitrilo a 50%, TFA a 0,4%).
- As amostras são então secas num concentrador de vácuo.
7. Por espectrometria de massas
- Ressuspender as amostras em 12 uL de acetonitrilo 3% com 0,1% de ácido fórmico e injectar 5 uL directamente sobre uma coluna de pressão elevada Eksigent nanoLC-1D cromatografia líquida (AB SCIEX, EUA).
- Os péptidos serão separados sobre uma coluna C18 Proteopep ID iM II 75 x 20 cm (Novo Objectivo, EUA) utilizando um gradiente de 4-60% (A: acetonitrilo 3%, 0,1% ácido fórmico; B: acetonitrilo 97%, fórmico 0,1% ácido)em 3 mL / min ao longo de 60 min.
- Um Thermo Scientific LTQ Orbitrap XL espectrómetro de massa foi utilizado para detectar péptidos utilizando um topo 2 x experimento 3 consistindo de uma única fase MS seguido por aquisição de 3 MS / MS espectros com fragmentação dissociação maior energia C-armadilha (HCD) seguido por 3 MS / MS com dissociação induzida por colisão (CID) para identificação de proteínas. Parâmetros neste modo foram: largura Isolamento: 3,0 m / z; energia de colisão: 50% (10% em dois passos). Peptídeos Apenas duplamente e triplamente carregadas foram selecionados para a fragmentação. Parâmetros de exclusão dinâmicos foram definido como: Repita count = 1; Duração Repeat = 60; tamanho Exclusão list = 500; Exclusão duração = 28. Os valores alvo são as seguintes: MS = 5 e 5; MS / MS (HCD) = 1 e 5. Tempo de transferência de íon foram definidos para 500 para FTMS e 300 para MS / MS (HCD). Dois microscans foram necessários para espectros HCD.
8. Buscando banco de dados e quantificação
- Os dados adquiridos CID e HCD eram umaanalisados: usando Thermo Scientific software Discoverer Proteome 1,2 (Thermo Scientific Pierce Produtos pesquisa de proteína) através de um fluxo de trabalho ramificada que implementou um Reporter Ion Quantizer (20 ppm de tolerância de massa de íons fragmento) para quantificar a proporção. Um segmento separado processou a MS 2 espectros através da Spectrum Selector, Normalizer Spectrum, e nós Garoupa Spectrum.
- Os dados foram então comparados com o motor de busca Sequest. A 20 tolerância massa ppm foi utilizado. Modificações estáticas foram definidas para o reagente cysTMT (304,18 Da) e alterações dinâmicas incluiu fosforilação e oxidação da metionina (15,99 Da). Um banco de dados personalizado para Solanum lycopersicum foi composta usando RNA de dados (com cerca de 350.000 entradas da Universidade de Harvard) 13 foi utilizado em ambos os casos.
9. Os resultados representativos
Uma imagem representativa de uma folha da planta de controlo de tomate e uma folha Pseudomonas inoculado émostrada na Figura 1. Uma diferença entre o controlo tratado e folhas de Pseudomonas tratados é observada. Depois de as folhas são removidos e corados usando DAB, o processo de coloração-permite a mancha histoquímica para mostrar sinais de ROS no tecido foliar (Figura 2). Figura 2A é representativa de uma folha de controle sem coloração. Figura 2B é representativa de uma folha tratada com Pseudomonas e positiva para a coloração de H2O 2 produção. Um exemplo de saída de dados Proteome Discover de uma proteína diferencialmente redox regulada é mostrado na Figura 3. Esta proteína é um ferredoxina-1 proteína conhecida redox regulamentado 14 e tem sido demonstrado que desempenham um papel na defesa contra Pseudomonas syringae pv tomate 15. Intensidade de pico entre o controlo e as amostras inoculadas é usado para obter quantificação relativa, que mostrou alterações significativas na ferredoxem-1 regulação redox (p <0,05). Picos de alta intensidade sugerem que esta proteína é oxidado em resposta ao tratamento do patógeno. A Figura 4 é um exemplo de saída de dados Proteome Discover de uma proteína que tem regulação redox de uma proteína semelhante entre uma amostra de controlo e inoculada. Os picos de intensidade semelhante sugerem a presença de ligações dissulfureto não regulada por uma alteração do tratamento. O método vai revolucionar a forma como os cientistas a detectar redox cisteínas responsivas e dissulfetos 10.

Figura 1. Uma imagem representativa de tomate inoculadas folhas com a solução de controlo (A) e Pseudomonas (B).

Figura 2. Uma imagem representativa de DAB coloração de tomate inoculadas folhas com a solução de controlo (A) e Pseudomonas (B). As folhas foram coradas utilizando 3'-3 'diaminobenzidina. A clorofila foi removido a partir de folhas por ebulição em etanol a 95%. A coloração escura indica a presença de H2O 2. Apenas deixa inoculado com cultura de bactérias apresentaram coloração escura.
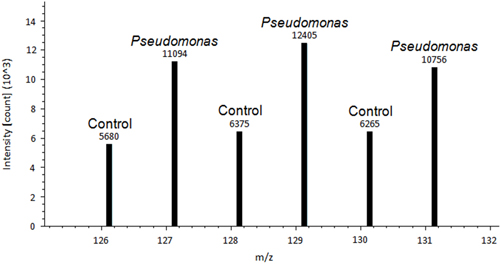
Figura 3. Um exemplo de Proteome saída de dados de Discover ferredoxina-1, diferencialmente redox proteína regulada 14. Intensidade do pico acima de cada pico é usado para a quantificação absoluta. Intensidade de pico entre o controlo e as amostras inoculadas é usadopara realizar a quantificação relativa.

Figura 4. Um exemplo de saída de dados Proteome Discover de uma proteína que tem intensidade de pico semelhante entre uma amostra de controlo e inoculada. Os picos de intensidade semelhante sugerem a presença de ligações dissulfureto não regulada por uma alteração do tratamento.
Discussão
Este protocolo fornece informações sobre a execução de coloração DAB, bem como cysTMT quantificação de cisteína marcado redox. Estes procedimentos são benéficos em examinar a produção de ROS, bem como o efeito sobre a regulação da proteína quando Solanum lycopersicum é inoculado com Pseudomonas syringae. Os métodos apresentados neste protocolo fornecer uma forma de examinar ROS em amostras de folhas inteiras de uma maneira que faz com que a menor quantidade de danos ao tecido foliar. O procedimento de etiquetagem fornece uma maneira para examinar potencialmente redox proteínas reguladas por utilizando um método de marcação de cisteína. Isto é benéfico quando se examina uma fase precoce da resposta ao stress.
Métodos tais como isótopo-codificado marcador de afinidade (ICAT) e cysTMT pode ser usado para examinar o potencial redox proteínas reguladas em amostras biológicas. ICAT permite rotulagem e comparação de duas amostras de 12. Ambos os métodos rotular cisteínas livres e pode ser usado para a proteína Quantification 10,12. No entanto, o método cysTMT permite uma diminuição da variação experimental, bem como a multiplexação 10. O número de marcas disponíveis permite que os pesquisadores de incluir repetições ou várias amostras em seu projeto experimental. Tendo em amostras mais oferece o potencial para um maior número de proteínas identificadas. Uma grande desvantagem da técnica de cysTMT é que compromete a qualidade global da identificação de proteínas por causa das etapas de enriquecimento selectivo para cysTMT rotulados-péptidos (6,5-6,6). O número de péptidos para a identificação de proteínas depende em grande medida o número de resíduos de cisteína na sequência de proteína. Este problema pode ser superado através da apresentação de parte da amostra antes tríptica de enriquecimento para a identificação de espectrometria de massa de proteína.
Devido à natureza do desenho experimental, bem como o mecanismo de etiquetagem de que o método cysTMT utiliza, certos passos são críticos. Durante a execução de proteína precipitation e pellet lavagens (3,9) é importante para manter as amostras arrefecida em gelo para reduzir a degradação de proteínas. Durante a etiquetagem cysTMT, a remoção do reagente redutor (4,6) é importante porque as amostras podem ser submetidos a rotulagem inversa. Rotulagem inversa é possível se a redução do reagente permanece na amostra. Se as amostras forem reduzidos após rotulagem, a etiqueta cysTMT pode ser removido. Uma vez que as etiquetas são adicionados às amostras, o nível de pH deve ser verificada (4,7), a fim de ter eficiência de marcação óptima. Além disso, a análise dos dados é dependente do que é exigido do pesquisador eo objetivo final na utilização do protocolo. Também é dependente do software a ser utilizado como cada software tem algoritmos diferentes.
Este experimento utiliza um patógeno como eliciador para aumento da produção de espécies reativas oxidantes em tomate, no entanto, outros redox respostas regulamentados pode ser medido em conformidade. Este projeto experimental é adaptável a outras plantas e sistemas animais.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Os autores gostariam de agradecer ao Dr. Greg Martin (Cornell University) e seu grupo para fornecer a tensão DC3000, sementes de tomate, e aconselhamento. Eles também gostariam de agradecer ao Dr. Zhonglin Mou para obter ajuda com o protocolo DAB e da Divisão de Proteômica na UF Centro Interdisciplinar de Pesquisa de Biotecnologia para a assistência no desenvolvimento do método. O protocolo para a extracção de proteína foi modificado a partir do Hurkman e Tanaka 16. O protocolo em cysTMT rotulagem, os passos 4 a 6 foi adaptada de acordo com o Thermo Pierce inicial Fisher Scientific produto manual 17. Este trabalho foi financiado pela National Science Foundation (MCB 0818051 para S Chen).
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
| Metromix 500 | Empresas IBW | TX-500 | |
| 3,3 '-diaminobenzidina | Sigma-Aldrich | D8001 | |
| ReadyPrep Reagente kit Sequential Extraction 3 | Bio-Rad | 163-2104 | |
| CB-X ensaio de proteína | Geno Tecnologia | 786-12x | |
| cysTMT reagentes | Thermo Scientific Research Pierce produtos de proteína | 90071 | |
| Tampão de amostra Laemmli | Bio-Rad | 161-0737 | |
| Bio-Safe Comassie (G-250 mancha) | Bio-Rad | 161-0786 | |
| Microcon coluna 3KD | Millipore | 42403 | |
| Imobilizado resina Anti-TMT | Thermo Scientific Research Pierce produtos de proteína | 90076 | |
| Coluna centrífuga | Thermo Scientific Research Pierce produtos de proteína | 89896 | |
| Proteopep coluna II C18 | Objetivo Novo | PFC7515-PP2-10 | |
| NanoLC-1D HPLC | AB Sciex | 90389 | |
| LTQ Orbitrap XL | Thermo Scientific | 0020137580 | |
| Speedvac | Labconco | 7812013 | |
| Proteome Discoverer 1,2 software | Thermo Scientific Research Pierce produtos de proteína | ||
| Tripsina | Promega | V5111 | |
| Oakridge tubo de centrífuga | Thermo Scientific NalgeEmpresa ne | 3139-0050 | |
| Tubo de microcentrífuga (2 ml) | EUA Científico | 1620-2700 | |
| 12% Mini-PROTEAN TGX Precast Gel | Bio-Rad | 456-1043 | |
| Top of Form > Bio-Safe Coomassie StainBottom do formulário | Bio-Rad | 161-0786 | |
| TMT kit enriquecimento | Thermo Scientific Research Pierce produtos de proteína | 90077 |
Referências
- Almeida, N. F. A draft genome sequence of Pseudomonas syringae pv. tomato T1 reveals a type III effector repertoire significantly divergent from that of Pseudomonas syringae pv. tomato DC3000. Mol. Plant Microbe Interact. 22, 52-62 (2009).
- Preiter, K. Novel virulence gene of Pseudomonas syringae pv. tomato strain DC3000. J. Bacteriol. 187, 7805-7814 (2005).
- Apostol, I., Heinstein, P. F., Low, P. S. Rapid stimulation of an oxidative burst during elicitation of cultured plant cells: role in defense and signal transduction. Plant Physiol. 90, 109-116 (1989).
- Orozco-Cardenas, M., Ryan, C. A. Hydrogen peroxide is generated systemically in plant leaves by wounding and systemin via the octadecanoid pathway. Proc. Natl. Acad. Sci. U.S.A. 96, 6553-6557 (1999).
- Tonks, N. K. Redox redux: revisiting PTPs and the control of cell signaling. Cell. 121, 667-670 (2005).
- Alvarez, S., Wilson, G. H., Chen, S. Determination of in vivo disulfide-bonded proteins in Arabidopsis. J. Chromatogr. B. 877, 101-104 (2009).
- Apel, K., Hirt, H. Reactive oxygen species: metabolism, oxidative stress, and signal transduction. Annu. Rev. Plant Biol. 55, 373-399 (2004).
- Dayon, L. Relative quantification of proteins in human cerebrospinal fluids by MS/MS using 6- plex isobaric tags. Anal. Chem. 80, 2921-2931 (2008).
- Thompson, A. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Anal. Chem. 75, 1895-1904 (2003).
- Rosenblatt, M. A novel cysteine-reactive tandem mass tag reagent for subproteome labeling, enrichment and quantitation. , TP169 (2010).
- Clark, J. Phenotypic analysis of Arabidopsis mutants: diaminobenzidine stain for hydrogen peroxide. Cold Spring Harb. Protoc. , (2009).
- Sethuraman, M. Isotope-Coded affinity tag (ICAT) approach to redox proteomics: identification and quantification of oxidant-sensitive cysteine thiols in complex protein mixtures. J. Proteome Res. 3, 1228-1233 (2004).
- Asso, M. EPR and redox characterization of ferredoxins I and II from Desulfovibrio vulgaris Miyazaki. Biochem. Biophys. Res. Commun. 211, 198-204 (1995).
- Huang, H. e. Disease resistance to bacterial pathogens affected by the amount of ferredoxin-I proteins in plants. Mol. Plant Pathol. 8, 129-137 (2007).
- Hurkman, W., Tanaka, C. Solubilization of plant membrane proteins for analysis by two-dimensional gel electrophoresis. Plant Physiol. 81, 802-806 (1986).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados