É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Detecção MicroRNA em tumores de próstata por Quantitative PCR em tempo real (qPCR)
Neste Artigo
Resumo
Quantitativa em Tempo Real reacção em cadeia da polimerase (qPCR) é um método rápido e sensível para investigar os níveis de expressão de vários microRNA (miRNA) moléculas em amostras de tumor. Usando este método de expressão de centenas de moléculas miRNA diferentes pode ser amplificado, quantificado e analisado a partir do mesmo modelo cDNA.
Resumo
MicroRNAs (miRNAs) são de cadeia simples, 18-24 nucleótidos de comprimento, moléculas de RNA não codificantes. Eles estão envolvidos em praticamente todos os processos celulares, incluindo o desenvolvimento 1, 2 apoptose e regulação do ciclo celular 3. MiRNAs Estima-se que regulam a expressão de 30% a 90% de genes humanos 4 por ligação aos seus RNAs alvo mensageiro (ARNm) 5. Desregulação generalizada de miRNAs tem sido relatada em várias doenças cancerosas e subtipos 6. Devido à sua prevalência e estrutura única, estas moléculas pequenas são susceptíveis de ser a próxima geração de biomarcadores, agentes terapêuticos e / ou alvos.
Os métodos utilizados para investigar a expressão de miRNA incluem SYBR green eu à base de corantes, bem como Taqman-sonda qPCR base. Se miRNAs estão a ser efetivamente utilizado na prática clínica, é imperativo que a sua detecção, frescos e / ou arquivados amostras clínicas ser preciso, reprodutível e specific. qPCR tem sido amplamente utilizado para validar a expressão de miRNAs em análises do genoma inteiro, tais como estudos microarray 7. As amostras utilizadas neste protocolo eram de pacientes que foram submetidos a prostatectomia radical para o cancro da próstata clinicamente localizado, no entanto outros tecidos e linhas celulares pode ser substituído pol espécimes de próstata foram snap-congelado em azoto líquido após a ressecção. As variáveis clínicas e follow-up de informação para cada paciente foram coletadas para análise posterior 8.
Quantificação dos níveis de miRNA em amostras de tumores de próstata. As etapas principais da análise qPCR de tumores são: a extracção de RNA total a síntese de cDNA, e detecção de produtos de qPCR usando miRNA primers específicos. O RNA total, que inclui mRNA, miRNA, e outros pequenos RNAs foram extraídos a partir de amostras utilizando reagente Trizol. Sistema da QIAGEN miScript foi utilizado para sintetizar ADNc e realizar qPCR (Figura 1). MiRNAs endógenos não são polyadenylated, portanto, durante o processo de transcrição reversa, uma polimerase de (A) poli polyadenylates o miRNA. O miRNA é utilizado como um modelo para sintetizar ADNc utilizando oligo-dT e transcriptase reversa. Uma sequência de etiqueta universal sobre a extremidade 5 'de oligo-dT iniciadores facilita a amplificação de cDNA no passo de PCR. A amplificação por PCR do produto é detectado pelo nível de fluorescência emitida pelo SYBR Green, um corante que intercala em DNA de cadeia dupla. Primers específicos miRNA, juntamente com um Primer Universal que se liga à seqüência tag universal irá amplificar sequências específicas de miRNA.
Os ensaios Primer miScript estão disponíveis para mais de mil humano miRNAs específicos e centenas de murino específicos miRNAs. Método de quantificação relativa foi usado aqui para quantificar a expressão de miRNAs. Para corrigir para a variabilidade entre diferentes amostras, níveis de expressão de um miRNA alvo é normalizado para os níveis de expressão de um gene de referência. A escolha de um geem que ne para normalizar a expressão de alvos é crítica na método de quantificação relativa de análise. Exemplos de genes de referência tipicamente usados nesta capacidade são o RNU6B pequenos RNAs, RNU44, e RNU48 como eles são considerados ser estavelmente expressos na maior parte dos exemplos. Neste protocolo, RNU6B é usado como o gene de referência.
Protocolo
1. Coleta de Amostras de próstata
- Recolher as amostras da próstata, no momento da prostatectomia. A amostra é orientada usando marcos anatômicos. A próstata e as vesículas seminais são pintadas como segue: verde do lado direito, azul lado esquerdo.
- Um midsection aleatória transversal da próstata é tomada perpendicularmente à superfície rectal, congelados em azoto líquido e armazenado a -80 ° C 9.
- Bancados fatias de espécimes são fotocopiadas, orientado (anterior, posterior, direita e esquerda), quadrisected. As secções são cortadas utilizando o criostato.
- As seções são corados com H & E e revisadas por um patologista para determinar e delinear áreas de tumor versus normal nas lâminas coradas e uma imagem correspondente. As áreas marcadas são usadas como um guia para indicar as áreas a partir da qual para extrair o tecido do tumor a partir do qual o RNA será extraído nos passos subsequentes.
2. Isolando RNA totais, incluindo miRNA, a partir de amostras
- Colocar as amostras congeladas de próstata em gelo seco e referindo-se à fotocópia delineada, cortada uma pequena porção do tumor da próstata (entre 50 a 100 mg).
- Homogeneizar o tecido do tumor da próstata em 1 mL de reagente Trizol. As quantidades em os seguintes passos são baseadas no uso de 1 mL de reagente Trizol.
Nota: Aqui temos utilizado reagente Trizol para RNA de extracção, no entanto outros kits que isolam pequenos ARN contendo RNA total pode também ser usado.
- Incubar as amostras homogeneizadas durante 5 minutos à temperatura ambiente.
- Adicionar 0,2 mL de clorofórmio às amostras e agitar vigorosamente por 15 segundos. Incubar as amostras durante 3 minutos a temperatura ambiente, em seguida, centrifugar a 12.000 xg durante 15 minutos a 4 ° C.
- Transferir a fase superior incolor aquosa para tubos frescos e adicionar 0,5 mL de álcool isopropílico.Incubar as amostras durante 10 minutos à temperatura ambiente, em seguida, centrifugar a 12.000 xg durante 10 minutos a 4 ° C.
- Aspirar cuidadosamente o sobrenadante sem perturbar o sedimento contendo o RNA. Lava-se a pelete de RNA com 1 mL de etanol a 75%. Vórtice a amostra e re-sedimento por centrifugação durante 5 minutos a 7500 xg, a 4 ° C.
- Aspirar cuidadosamente o sobrenadante e secar o pellet de RNA por 5-10 minutos, certificando-se o pellet de RNA não está completamente seca. Dissolver em água livre de nuclease adequada ao tamanho da pelota. Medir a concentração de RNA usando o espectrofotómetro 1000 NanoDrop (absorvância medida a 260 nm e 280 nm).
- Verifique a qualidade ea integridade das amostras de RNA usando Agilent Bioanalyzer.
3. Transcrição Reversa de RNA
- A transcrição reversa do RNA foi realizada utilizando Kit de Transcrição Reversa miScript de acordo com as instruções do fabricante (Qiagen). Este kit inclui umtranscriptase reversa e um poli-polimerase (A). O miScript tampão RT inclui Mg 2 +, dNTPs, primers oligo-dT, e iniciadores aleatórios.
- Use entre 10 pg e 1 ug de RNA para sintetizar cDNA. Se utilizar mais do que 1 ug de RNA, dimensionar-se a reacção linearmente para o volume apropriado.
- Prepara-se uma mistura mestra que contém 5X tampão RT miScript (4 ul), miScript Mix Transcrição Reversa (1 uL), e livre de RNase água para trazer reacções volume final de 20 uL. Também incluem RNA modelo (até 1 mg) na mistura principal.
- Incubar as amostras durante 60 minutos a 37 ° C, seguida imediatamente por uma incubação durante 5 minutos a 95 ° C. Este passo pode ser realizado numa máquina de PCR, o aquecimento de banho de bloco, ou água. Termocicladores são o método mais adequado e preciso. Armazenar o ADNc em gelo durante curto prazo, e -20 ° C para armazenamento a longo prazo.
4. Gerando uma curva padrão
- Antes de experiment com miRNAs alvo, uma curva padrão é gerada usando cDNAs de concentrações conhecidas contra os seus pontos de cruzamento (CP) (Figura 2).
- Prepara-se uma série de diluições de 2 vezes, 10 vezes, 50 vezes, 250 vezes, e 1250 vezes o ADNc original é de uma amostra que é conhecido por ter uma expressão substancial do seu gene de interesse.
- Execute o PCR, conforme especificado na Seção 5 "Real-time PCR para a detecção de miRNA", com a modificação que é cDNA em diluições seriadas não diluição estática 40x.
- Realizar a análise utilizando o software RelQuant (Roche) para gerar a curva padrão.
Nota: uma nova curva padrão devem ser gerados para cada gene de interesse.
5. Real-time PCR para a detecção de miRNA
- PCR em tempo real para miRNAs foi realizada utilizando miScript SYBR Green PCR Kit e miScript Assay Primer de acordo com as instruções do fabricante (Qiagen). Prepare uma mistura master contendo2x QuantiTect SYBR Green PCR Master Mix, Primer Universal miScript 10x, 10x Ensaio Primer miScript, e RNase água. Preparar um master mix para uma reação volume de 20 l.
- O Ensaio Primer é específico para o miRNA de interesse. Para reconstituir Assay 10x miScript Primer, centrifugar o frasco de forma breve, e adicionar 550 uL de tampão TE, pH 8,0. Frasco Vortex brevemente para misturar, primers alíquotas para volumes menores, e armazenar a -20 ° C. Dois iniciadores são necessários:. Iniciadores para o gene alvo eo gene de referência RNU6B é usedas o gene de referência.
- Diluir o cDNA 40x e armazenamento de alíquotas adicionais a -20 ° C.
- ADNc serve como molde para a PCR. Use 2 ul de cDNA 40x diluída e dispensar para o reciclador 20 uL de luz capilares (Roche).
- Adicionar 18 ul da mistura principal para cada capilar, e centrífuga, usando um adaptador capilar.
- Coloque os vasos capilares no capilar termociclador em tempo real baseado, como LightCycler 3.5 Real-Time PCR System com um formato de carrossel 32-capilar.
- Executar o programa de ciclos de PCR como se segue:
Para activar Polimerase HotStarTaq que está no 2x QuantiTect SYBR Green PCR Master Mix, pré-incubar a 95 ° C durante 15 minutos.
Seguido por 50 ciclos de:
Desnaturação, 15 s, 94 ° C;
Recozimento, 30 s, 55 ° C;
Extensão, 30 s, 70 ° C. - Selecione uma amostra para ser o calibrador, e definir seu valor alvo normalizado para 1. Comparar a expressão relativa da miRNA em todas as outras amostras para o calibrador.
Nota: Dentro de um estudo, a mesma amostra de calibração deve ser usado para manter a consistência dos resultados.
6. Análise de Dados
- As curvas de amplificação para as reacções de PCR são representados graficamente e numericamente por Molecular Software versão Biochemicals LightCycler 3,5 (Roche). Quantificar reações na "quantificação" guia, umnd exportar os dados para um arquivo de texto.
- Importe os dados para o software de análise RelQuant (Roche) para gerar resultados de quantificação. Importar ficheiros separados para o gene alvo, o gene de referência, e os dados da curva padrão.
- Especifique a posição do calibrador para ambos alvo e gene de referência. Também indicar as posições das amostras. Os dados são expressos como o alvo para fazer referência a proporção de diferentes amostras dividido pelo alvo para relação de referência do calibrador. A curva padrão gerada anteriormente para um miRNA particular e do gene de limpeza é utilizado como um padrão de referência para extrapolação dos dados quantitativos para alvos miRNA de concentrações desconhecidas.
- Três repetições de amostras são analisadas como um grupo e as concentrações médias e desvios-padrão da triplicata é calculado. Se um dos triplicados é inconsistente com o resto do conjunto, ele irá ser excluídos pelo programa.
7. Os resultados representativos
Um exemplo de qPCR análise de amostras da próstata é mostrado na Figura 3. Os resultados são representados numericamente, bem como graficamente. Os gráficos que mostram os níveis de expressão do gene de referência, U6, começam a amplificação exponencial menos cerca de ciclo 20, enquanto que a expressão do gene alvo, miR-98, mostrou amplificação retardada aproximadamente no ciclo 25. Os dados deste experimento foi exportado como arquivo de texto e analisados pelo software de análise RelQuant. Posições dos capilares que contêm o calibrador e as amostras são especificados. Figura 4 ilustra a forma como o calibrador é ajustado para ser 1, ea expressão de outras amostras em relação ao calibrador.
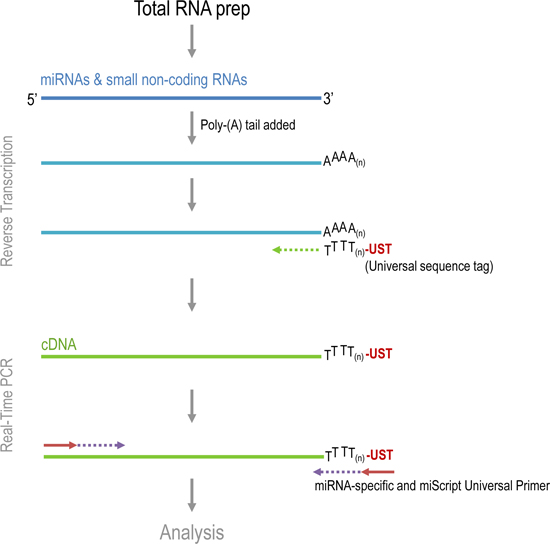
Figura 1. Várias etapas miScript transcrição reversa e PCR em tempo real.

Figura 2. Uma curva padrão é gerada por meio de uma série de diluições de 2 vezes, 10 vezes, 50 vezes, 250 vezes, e 1250 vezes amostra original de cDNA.

Figura 3. Roche Molecular Software LightCycler Biochemicals mostra toda a informação do experimento graficamente e pelo texto. Quantitativas em tempo real parcelas de amplificação por PCR mostram aumento na fluorescência a partir de amostras diferentes.

Dados Figura 4. Foram quantificados utilizando o software de análise RelQuant LightCycler. Normalmente, três repetições de amostras são analisadas como um grupo e amostras que produzem resultados claramente inconsistentes são excluídos e as concentrações médias e desvios-padrão da triplicata é calculado.
Access restricted. Please log in or start a trial to view this content.
Discussão
Expressões aberrantes de alguns miRNAs foram consistentemente encontrados em tumores de próstata quando comparado com o tecido normal 10, e alguns destes miRNAs foram nomeados como potenciais novos agentes terapêuticos contra o cancro da próstata 11. Assim, os níveis de expressão aberrante de miRNAs podem ser úteis biomarcadores de diagnóstico e / ou prognóstico. A metodologia qPCR Real-Time aqui apresentado proporciona um ensaio para a quantificação precisa dos níveis de miRNA em tecid...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi financiado pela Canadian Cancer Society Research Institute, conceder nenhuma. 019038.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do o reagente de | Companhia | Número de catálogo | |
| Reagente de TRIzol | Invitrogen | 15596 | |
| miScript Kit de Transcrição em reverssa | Qiagen | 218061 | |
| miScript Assays Primer | Qiagen | Experiment específico | |
| miScript SYBR Green PCR Kit | Qiagen | 218073 | |
| LightCycler 3,5 Real-Time Sistema de PCR | Roche | ||
| Luz Cycler Capilares | Roche | 04929292001 | |
| NanoDrop 1000 espectrofotômetro | Thermo Scientific | 2538 | |
| Agilent 2100 Bioanalyzer | G2943CA |
Referências
- Reinhart, B. J. MicroRNAs in plants. Genes Dev. 16, 1616-1616 (2002).
- Xu, P. The Drosophila microRNA Mir-14 suppresses cell death and is required for normal fat metabolism. Curr. Biol. 13, 790(2003).
- Bueno, M. J., Perez, deC. astro, I,, Malumbres, M. Control of cell proliferation pathways by microRNAs. Cell Cycle. 7, 3143-3143 (2008).
- Friedman, R. C. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19, 92(2009).
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116, 281-281 (2004).
- Croce, C. M. Causes and consequences of microRNA dysregulation in cancer. Nat. Rev. Genet. 10, 704(2009).
- Coppola, V., De, M. R., Bonci, D. MicroRNAs and prostate cancer. Endocr. Relat. Cancer. 17, 1-1 (2010).
- Gordanpour, A. miR-221 Is down-regulated in TMPRSS2:ERG fusion-positive prostate cancer. Anticancer Res. 31, 403-403 (2011).
- Nam, R. K. Expression of the TMPRSS2:ERG fusion gene predicts cancer recurrence after surgery for localised prostate cancer. Br. J. Cancer. 97, 1690(2007).
- Ambs, S. Genomic profiling of microRNA and messenger RNA reveals deregulated microRNA expression in prostate cancer. Cancer Res. 68, 6162(2008).
- Liu, C. The microRNA miR-34a inhibits prostate cancer stem cells and metastasis by directly repressing CD44. Nat. Med. 17, 211(2011).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados