Method Article
Abordagem de processamento paralelo multi-alvo para Gene-a-estrutura Determinação da polimerase da gripe PB2 Subunidade
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Concepção de fármacos com base Estrutura desempenha um papel importante no desenvolvimento de medicamentos. Prosseguindo alvos múltiplos em paralelo aumenta a chance de sucesso para a descoberta de chumbo. O seguinte artigo destaca como o Seattle Center Structural Genomics para Doenças Infecciosas utiliza uma abordagem multi-alvo para determinação da influenza A subunidade PB2 gene-to-estrutura.
Resumo
Surtos de pandemia de cepas de influenza altamente virulentas podem causar morbidade e mortalidade generalizada nas populações humanas em todo o mundo. Só nos Estados Unidos, uma média de 41.400 mortes e hospitalizações 1.860.000 são causadas por infecção pelo vírus da gripe em cada ano 1. As mutações pontuais na polimerase subunidade da proteína básica 2 (PB2) têm sido associados à adaptação da infecção virai em seres humanos 2. Os resultados de tais estudos revelaram a importância biológica da PB2 como um fator de virulência, destacando, assim, o seu potencial como um alvo da droga antiviral.
O programa de genômica estrutural estendeu pelo Instituto Nacional de Alergia e Doenças Infecciosas (NIAID) oferece financiamento para Emerald Bio e outras três instituições Pacific Northwest que, juntos, compõem o Seattle Center Structural Genomics para Doenças Infecciosas (SSGCID). O SSGCID é dedicado a fornecer à comunidade científica threestruturas de proteínas e-dimensionais de NIAID categoria patógenos AC. Fazendo essa informação estrutural disponível para a comunidade científica serve para acelerar o desenho de drogas baseado em estrutura.
Concepção de fármacos com base Estrutura desempenha um papel importante no desenvolvimento de medicamentos. Prosseguindo alvos múltiplos em paralelo aumenta a chance de sucesso para a nova descoberta de liderança, visando um caminho ou uma família de proteínas inteiro. Emerald Bio desenvolveu um pipeline de processamento paralelo de alto rendimento, multi-target (MTPP) para gene-to-estrutura de determinação de apoiar o consórcio. Descrevemos aqui os protocolos usados para determinar a estrutura da subunidade PB2 de quatro estirpes de gripe A diferente.
Protocolo
Uma visão geral do protocolo é apresentado na Figura 1.
Biologia Molecular
1. Construir projeto
Use software Composer Gene para projetar construção de proteínas e seqüências de genes sintéticos projetados códon. O uso de software Compositor genética tem sido oferecido em detalhe noutro local 3.
- Use o módulo Visualizador de Alinhamento e construir módulo de desenho para comparar alinhamentos de seqüência de proteínas e definir a construção de proteínas. Alinhar a sequência de aminoácidos alvo para ambos os elementos estruturais primárias e 3D de homólogos do Protein Data Bank (PDB), se estiverem disponíveis (Figura 2).
- Use as informações de alinhamento para fazer desenhos constroem estrutura guiadas por escolha de novos terminais baseados na conservação da estrutura primária e estruturas 3D de homólogos.
- Projeto de inserção PCR (IPCR) e vetor de PCR (VPCR) amplímeros (terminal primers).
- Utilizando Gene Calgoritmo de proteína e ADN da omposer, volta-traduzir a sequência de aminoácidos em constructo codão sequência de ácido nucleico de engenharia.
- Use a tabela de uso códon adequada (CUT) para otimizar a seqüência de expressão em E. coli.
- Virtualmente clonar inserção em pET28 vector modificado para incorporar uma tag de Histidina N-terminal e 6x Smt3/SUMO proteína de fusão que permite a fácil purificação.
- Coloque ordem gene sintético com DNA 2.0 e primers ordem de Tecnologias Integradas de DNA.
2. Polymerase incompleta Extensão Primer (PIPE) Clonagem
- Prepare Primers e Genes
- Centrifugar as placas disponibilizados pelos fornecedores, contendo primers a 1.000 rpm por 1 min.
- Levar a concentração dos iniciadores para 100 uM e adicionar 50 ul de tampão TE.
- Dilui-se iniciadores a 10 uM, com água desionizada (Dl) em uma placa de fundo em V de 96 poços.
- Centrifugar o gene fornecido pelo fornecedor em um tubo de 1,5 ml a 1.300 rpm por 1 min.
- Usando tampão TE, trazer a concentração de ADN de cada tubo a 50 ng / mL.
- Em tubos de 1,5 ml, fazer diluições de cada primer a 10 ng / mL.
- Loja de primers e genes a -20 ° C, quando não estiver em uso.
- Prepare Insert PCR (IPCR)
- Descongelar um frasco de Pfu Master Mix em gelo; manter genes e iniciadores à temperatura ambiente.
- Criar um mapa da placa atribuindo poços para um conjunto de primers e construir.
- Adicionar 13 mL de água Dl para cada poço de uma placa de PCR de 96 poços.
- Adicionar 5 uL de frente e 5 ul de iniciador reverso de cada reacção na placa de 96 cavidades de acordo com o mapa da placa, assegurando a mudar dicas entre cada poço.
- Adicionam-se 2 jil de cada gene de comprimento total para a sua bem adequado de acordo com o mapa do prato.
- Adicionar 25 ul de Pfu mistura principal para cada poço, assegurando a mudar dicas entre cada poço.
- Ciclo as reacções utilizando os seguintes condies de PCR:
- 95 ° C 2 min
- 95 ° C 30 seg
- 50 ° C 45 seg
- 68 ° C 3 min
- 4 ° C ∞
- Repita os passos bd para 25 ciclos.
- Transferem-se 10 ul de cada reacção de IPCR para uma nova placa de PCR de 96 poços.
- Adiciona-se 3 ul de corante de carga 6X a cada amostra.
- Separa-se cada amostra numa% de gel de agarose TAE EtBr 1 a 110 V ao lado de uma escada de 100-500 pb do ADN para confirmar a amplificação do fragmento.
- Loja IPCR produto a -20 ° C quando não estiverem em uso (evitar o congelamento descongelamento, tanto quanto possível).
3. Prepare Vector PCR (VPCR)
- Iniciar a cultura durante a noite de E. transformado coli com pET28 vector plasmídeo.
- Inocular dois tubos de 5 mL de caldo de 2 YT com 50 ug / ml de canamicina.
- Crescer as culturas durante a noite a 37 ° C, em agitador rotativo a 220 rpm.
- Girar as culturas após crescimento durante a noite por centrifugação a 3.000 rpm durante 15 min.
- Use um Qiagen QIAprep rotação Miniprep Kit para extrair pET28 vector de pelotas de bactérias de acordo com as instruções do fabricante.
- Restrição de configuração digestões enzimáticas de extraído pET28 plasmídeo.
- Adicionar 2,2 mL de tampão BamHI 10x e 1 ul de BamHI e Hindi II e 20 pi de vector pET28.
- Incubar a reacção durante 1 hora a 37 ° C.
- Produto da digestão separada em um gel.
- Consulte o passo 2.2.10.
- Cortar banda do vector a partir do gel e purificá-lo utilizando o QIAquick Gel Extraction Kit de acordo com as instruções do fabricante.
- Usando um NanoDrop, quantificar a concentração de ADN.
- Diluir cortar vector a 10 ng / mL. Armazenar a -20 ° C quando não estiverem em uso.
- Prepare primers VPCR.
- Centrifugar IDT oligonucliotides fornecidos para 1 min a 1.300 rpm.
- Levar a concentração de 100 uM com água Dl.
- Prepare-se 10 mM de diluição de ambos os primers para a frente e para trás em um tubo de 1,5 ml.
- Loja de primers e diluições de primers a -20 ° C.
- Descongelar Pfu Master Mix em gelo e degelo molde e os iniciadores à temperatura ambiente.
- Reações VPCR de configuração em uma placa PCR de 96 poços:
- Na primeira fila de uma placa de 96 poços 60 ul de combinar ambos os iniciadores VPCR frente e reversa e 24 ul de pET28 digerido molde (10 ng / mL).
- Usando uma pipeta de múltiplos canais 12 da ponta, transferir 12 ul da mistura de iniciador e molde mestre para cada restante das cavidades da placa. Isto deve resultar em 12 ul de iniciador e molde mestre de mistura em cada poço da placa.
- Adicionar 13 ul de água desionizada a cada poço.
- Adicionar 25 ul de Pfu Master Mix para cada poço.
- Ciclo as reacções através das condições de PCR utilizadas no passo 2.2.7.
- Piscina todas as reações VPCR em um tubo Falcon de 15 ml.
- Verifique amplificação fragmento separando 10 ul de produto de PCR reunidos num gel (comprimento esperado de vector digerido pET28é de aproximadamente 6 Kb).
- Consulte o passo 2.2.10.
- Prepare placas de fusão.
- Alíquotas de 3 mL de produto VPCR em cada poço de uma placa de fundo em V de 96 poços.
- Loja de placas a -20 ° C até fusão com o produto IPCR.
4. Mesclar IPCR e VPCR produtos
- Produtos IPCR descongelar e pré-VPCR alíquota de 96 poços fundir placa à temperatura ambiente.
- Adiciona-se 3 uL de cada produto de IPCR à sua respectiva cavidade da placa de junção.
- Transformar placa fundir Top Ten células competentes quimicamente.
- Adicione 2 mL de cada reação de fusão em um único tubo de 50 ml de células quimicamente competentes fornecedor fornecidos e prosseguir com o protocolo fornecido pelo fabricante.
- Prepare culturas durante a noite para cada construção de chapa de transformação.
- Alíquotas de 5 ml de caldo TB (com 50 ug / ml de canamicina) a partir de um reservatório de 25 ml esterilizado, em cada poço de um bloco de poços profundos.
- Usando sterile técnica, escolher uma colônia isolada de cada placa transformação e inocular o bem apropriado do bloco de poço profundo.
- Cubra o bloco com uma cobertura Airpore.
- Agitar bloco a 220 rpm a 37 ° C durante a noite.
- Células pelota por centrifugação do bloco durante 30 min a 4000 rpm.
- Decantar o sobrenadante e pat o topo do bloco seco com uma toalha de papel.
- Mini-prep da Qiagen utilizando um aparato de 96 poços de vácuo de acordo com as instruções do fabricante.
5. Preparando-se Stocks de glicerol de construções clonados com sucesso
- Transform clonado com sucesso seqüência de DNA validados em BL21 (DE3), as células quimicamente competentes de acordo com as instruções do fabricante.
- Para cada constructo, escolher uma única colónia isolada a partir da (DE3) de transformação e inocula BL21 em 1 ml de caldo 2-YT (com 50 ug / ml de canamicina).
- Agitar culturas a 220 rpm durante 3-4 horas a 37 ° C.
- Rotular um 1,5 mlparafuso tubo com tampa com o número único de identificação de construto, a estirpe de células, e a data. Adicionar 500 ul de glicerol a 50% e 500 mL de cultura de células e gire várias vezes. Imediatamente armazenar estoque de glicerol em gelo seco ou num congelador -80 ° C.
6. Teste de expressão
| Lise Buffe r | Tampão de Lavagem | Tampão de eluição |
| 50 mM de NaH 2 PO 4, pH 8,0 300 mM de NaCl Imidazole a 10 mM 1% de Tween 20 MgCl2 2 mM 0,1 ul / ml de Benzonase 1 mg / ml de lisozima | 50 mM de NaH 2 PO 4, pH 8,0 300 mM de NaCl Imidazole 20 mM Tween 20 0,05% | 25 mM Tris, pH 8,0 300 mM de NaCl Imidazole 250 mM Tween 20 0,05% |
* Adicionar Benzonase, lisozima,e inibidores da protease imediatamente antes da lise.
- Causadas uma amostra a partir de estoque de glicerol em canamicina agar selectiva e incubar durante a noite a 37 ° C.
- Iniciar uma pré-cultura não indutoras em um bloco de 96 poços de fundo redondo; inocular 1,2 ml de caldo TB (com 50 mg / ml de canamicina), suplementado com 0,5% de glucose com um E. recentemente crescido coli isoladas. Crescer durante a noite com agitação a 220 rpm a 37 ° C.
- Após o crescimento durante a noite, iniciar culturas de indução por inoculação de 1,2 ml de caldo TB (com 50 mg / ml de canamicina), suplementado com Novagen noite expresso Sistema 1 (de acordo com o protocolo do fabricante) com 40 ul de pré-cultura.
- Crescer as culturas em pequena escala de indução a 20 ° C durante 48 horas, agitando a 220 rpm.
- Colheita de células por centrifugação a 4.000 rpm durante 15 minutos, deitar fora o sobrenadante e armazenar a -20 ° C durante pelo menos 1 hora antes do processamento.
- No bloco 96, bem, ressuspender as pastilhas de células em 300 ul de tampão de lise.
- Incubar as células em tampão de li se à temperatura ambiente durante 30 min seguido por lise mecânica por agitação vigorosa durante 30 minutos à temperatura ambiente.
- Clarificar o lisado em bruto por centrifugação durante 30 min a 4000 rpm a 4 ° C.
- Usar uma pipeta multi-canal para transferir 200 ul do ligado clarificado (fracção solúvel) para uma bandeja de 96 poços de fundo plano (Qiagen). Para cada uma das cavidades contendo uma amostra, adicionar 40 pérolas de Ni-NTA (Qiagen magnéticos ul).
- Agitar suavemente a placa por um balancim durante 1 hora a 16 ° C.
- Coloque a placa em uma placa de pós magnética (Qiagen) e remova a fração solúvel não ligado. Tome cuidado para não pipetar qualquer das esferas Ni-NTA.
- Remova a placa do prato de pós e ressuspensos suavemente as pérolas em 200 ul de tampão de lavagem. Pipeta cima e para baixo por 30 segundos e, em seguida, colocar a placa de volta no prato post.
- Retire o tampão de lavagem e repita o passo 6.12.
- Remova a placa de placa de pós e eluir o Ni-NTA obrigado taproteína rPegue por lavagem com 50 ul de tampão de eluição durante 5 min.
- Placa de fundo plano de retorno para a chapa de pós magnéticos e transferir a eluição para uma placa de 96 poços de fundo em V fresco.
- Transferir 20 uL da eluição a uma placa de 96 poços de fundo em V fresco e reagir com 1 ul ULP1 protease.
- De acordo com o protocolo do fabricante, analisar a + fracção eluida e eluída Ulp1 por electroforese capilar utilizando um LabChip 90.
- Alternativamente, todas as fracções a partir de ensaios de expressão pode ser analisado através de SDS-PAGE.
7. Large Scale Fermentação
- Usar uma pipeta estéril para obter uma raspagem a partir de um estoque de glicerol, inocular 100 ml de caldo TB (com 50 mg / ml de canamicina) e crescer durante a noite. Agitar a 220 rpm e 37 ° C.
- Após o crescimento durante a noite, expandir pré-cultura por inoculação de 1 L de caldo TB com soluções autoindução Merck (ver o protocolo do fabricante) (com 50 mg / ml de canamicina) num balão de 2 confundido com 10 ml dapré-cultura (diluição 1:100).
- Agitar as culturas expandidas de 1 L, a 37 ° C, a mudança da temperatura da incubadora com agitação a 20 ° C quando uma densidade óptica de 0,6 (OD 600) for atingido.
- Após o crescimento durante a noite, ter um representante alíquota de 10 ml cada construto para testes de expressão.
- Colheita de células colar por centrifugação a 5.000 rpm durante 15 minutos e descartar o sobrenadante.
- Célula congelamento cole a -80 ° C.
PURIFICAÇÃO DA PROTEÍNA
Buffers:
| Tampão de Lise | Tampão A (Equilíbrio) | Tampão B (eluição) | Dimensionamento tampão de coluna |
| 25 mM Tris, pH 8,0 200 mM de NaCl 0,5% de Glicerol 0,02% de CHAPS Imidazole a 10 mM 1 mM TCEP 50 mM de Arginina 5 Benzonase mL 100 mg lisozima 3 inibidor da protease Tablets (EDTA-free) | 25 mM Tris, pH 8,0 200 mM de NaCl Imidazole a 10 mM 1 mM TCEP 50 mM de Arginina 0,25% Glicerol | 25 mM Tris, pH 8,0 200 mM de NaCl Imidazole 200 mM 1 mM TCEP | 25 mM Tris, pH 8,0 200 mM de NaCl 1% Glicerol 1 mM TCEP |
* Adicione Benzonase, lisozima e comprimidos inibidores de protease para cada 150 ml de amostra imediatamente antes da lise.
8. Lise Celular
- Adicione 2 L de tampão de lise; não adicionar lisozima, comprimidos inibidores de protease ou benzonase (cada amostra serão lisadas separadamente em 150 ml de tampão de lise).
- Descongelar e ressuspender pasta de células em tampão de lise a uma massa 01:05: relação de volume por agitação vigorosa durante 30 min a 4 ° C. Quebre os pedaços soltos de lados do copo com uma espátula limpa. Durante este período de tempo preparar Ni e Dialysis Buffers
- No gelo, lisar as células usando um sonicadora Misonix (70% de potência, 2 sec on / off 1 seg pulsos para 3 min) e gentilmente recipiente agite para evitar o superaquecimento. Salvar um pequeno (200 mL), alíquota de lisado bruto para análise futura.
- Clarificar o lisado em bruto por centrifugação a 18.000 xg durante 35 min a 4 ° C, recolhe-se o sobrenadante e guardar um pequeno (200 mL) alíquota para análise futura. Loja pelete a 4 ° C até que seja confirmada a proteína tenha sido lisadas na fracção solúvel.
9. Setup Criador Protein pré-run
- Com a máquina de proteína ligado e abrir o software, inicialize o instrumento.
- Uma vez inicializado, coloque uma coluna de 5,0 ml de níquel-quelato GE Healthcare HisTrap FF (Ni coluna) em uma linha separada do pórtico para cada uma das amostras.
- Executar 3-4 volumes de coluna (CV) de tampão de equilíbrio através de cada coluna.
- Preparar o equilíbrio e as linhas de tampão de eluição.
- EquilibrComeram as colunas o tampão A por aspiração através da coluna uma vez.
10. Níquel 1 (Ni1) Coluna
- Lava-se cada coluna com 20 ml de água Milli-Q para remover o tampão de armazenamento. Run 5 ml de tampão B a 25 ml de tampão A para o equilíbrio.
- Carregar o lisado clarificado contendo proteínas solubilizadas para as colunas, a uma taxa de 2 ml / min e depois seguem por uma lavagem de 15 ml com tampão A.
- Eluir a proteína ligada com um gradiente passo com tampões A e B pelas seguintes razões respeitosamente: 5 ml de 95:5, 5 ml de 60:40, 10 ml de 0:100. Recolher separadamente cada fracção de eluição.
- Analise: fracções eluídas, ligado em bruto, ligado clarificado, e por escoamento por SDS-PAGE. Piscina fracções contendo a proteína e usar um Nanodrop para medir A 280 para determinar aproximadamente a quantidade de proteína presente.
11. ULP1 Cleavage
- Manter uma pequena alíquota (250 ul) da piscina Ni1 coluna para análise em gel subsequente. Traga o restoda piscina Ni1 a 10 ml e adicionar a protease semelhante a ubiquitina 1 (ULP1) a 1 mL / 5 mg de proteína total para remover a marca de afinidade His-Smt.
- Dializar a piscina Ni1 + ULP1 contra 2 L de tampão A durante 4 horas a 4 ° C numa solução de 10 kDa de corte de peso molecular (MWCO) de uma placa de agitação, a 4 ° C.
- Após diálise, execute SDS-PAGE de Ni1 piscina e Ni1 piscina + ULP1 para determinar se ULP1 clivagem foi bem sucedida.
12. Níquel 2 (Ni2) Coluna
- Carga de proteína clivada pela mesma coluna Ni e repetir o passo 9.3, a uma taxa de fluxo reduzida de 1 ml / min. A marcação fora clivada ligarão à coluna e a proteína alvo irá agora tagless flow-through. Recolher o flow-through em um recipiente fresco.
- Lavar a coluna de Ni com 3 ml de tampão A seguido por 5 ml de tampão B para eluir todas as proteína marcada com His e não especificamente ligado. Recolhe cada fracção separadamente.
- Executar SDS-PAGE de Ni2 flow-through, lave e Ni2 frações de eluição para verificar ULP1 clivagem e que protein está presente no escoamento. Usar um Nanodrop para medir A 280 para determinar grosseiramente a presença de proteína.
13. Concentrando
- Concentra-se o fluxo através de Ni2 (e Ni2 eluição se a proteína estiver presente) até 5 ml com um Ultra tubo de centrífuga de 10 kDa MWCO Amicon. Girar em intervalos de 10 min a 4000 rpm a 4 ° C. Misture com uma pipeta entre cada rodada para evitar o excesso de proteína de concentração ao longo da membrana.
14. Cromatografia por exclusão de tamanho (SEC)
- Configurar uma S-100 10/30 GL coluna Sephacryl (GE Healthcare) foram equilibradas com 200 ml de tampão de SEC com um caudal de 0,5 ml / min num sistema ÄKTApurifier (GE Healthcare).
- Preparar 10 ml superloops para utilização na coluna SEC de acordo com as instruções do fabricante.
- Usando uma seringa de 5 ml, carregar amostras para superloops e começar a SEC prazo.
- Monitorar o traço UV-absorvência a 280 nm durante a coleta de pequeno volume frações.
- Executar frações SEC via SDS-PAGE.
- Reunir as frações SEC mostrando as maiores bandas de intensidade.
- Concentre-se frações SEC pool. Consulte o passo 13.1.
- Proteína alíquota em 100 amostras il, congelamento de flash em nitrogênio líquido e armazenar a -80 ° C.
CRISTALIZAÇÃO
15. Cristalização de proteínas
- Pré-preencher cada reservatório de Jr placa de cristalização de 96 poços Compact (Emerald Bio) com 80 ml de tela cristalização (Emerald Bio) de escolha.
- Dilui-se a proteína com tampão de dimensionamento para 2-20 mg / ml e armazenar no gelo.
- Pipetar 0,4 mL de proteína e 0,4 ul da tela cristalização em cada um dos 96 poços e cobrir com fita de vedação cristalina (Manco).
- Guarde a placa a 16 ° C durante a verificação de cristalização de proteínas periodicamente ao longo das próximas semanas sob um microscópio de dissecação.
16. Cristal colheita
- Criar um crioprotector a partir do licor mãe e etileno-glicol. Cortar a fita transparente que cobre o poço com o cristal da proteína alvo. Para um poço vazio, adicionar 1,6 mL da condição de cristalização correspondente e combinam-se com 0,4 mL de etileno-glicol originando uma concentração final de 20% de etileno-glicol e 80% de condição de cristalização. Nota: para optimizar difracção cristal experimentar diferentes agentes crioprotectores, tais como: glicerina, óleos, polietileno-glicóis de baixo MW, e / ou em diferentes percentagens do crioprotector.
- Antes da colheita esfriar um disco de estilo ALS em um Dewar preenchido com nitrogênio líquido e cubra com a tampa.
- Recolher os cristais, colocando um CryoLoop com o diâmetro interno correspondente ao tamanho do cristal sobre uma varinha de cristal magnético (Hampton Research) e colher-lo directamente a partir da solução bem.
- Mergulhar imediatamente o CryoLoop com o cristal colhidas no cryoprotectant seguida submergir no disco de estilo ALS a piscar congelar o crystal. Repetir para um número desejado de cristais.
17. Cristal Coleção Triagem / Data
- Uma vez que a colheita está completa usar uma varinha disco para colocar o disco crio tampa magnética no ALS disco. Com pinças dobrados, vire o disco de cabeça para baixo.
- Transfira o disco a um Rigaku ATOR Dewar, um parafuso Pusher Puck para o disco, e perfurar a tampa deixando-o na Dewar com os pinos voltados para cima.
- Usando software JDirector, cada tela de cristal sob os seguintes parâmetros: fenda feixe definido para 0,5 graus, a distância detector ajustado a 50 mm, passo imagem a 70 graus, e duração de exposição definido para 30 segundos.
- Executar Mosflm nas imagens de teste que você tiro com JDirector para determinar qual a melhor estratégia é cristalina e para a coleta de dados.
- Coletar um conjunto de dados completo com base em seus resultados de Mosflm.
18. Processamento de Dados / determinação da estrutura
- Executar XDS / XSCALE 4 para processar o conjunto de dados.
- Abra o software suíte CCP4.
- Executar Phaser 5 para calcular uma solução de substituição molecular, utilizando um modelo de pesquisa de homologia elevado, quando disponíveis. Neste caso, usamos o 3CW4 PDBID como um modelo de pesquisa 6.
- Executar Refmac 7 para refinar o modelo molecular contra o reflexo observado coletadas no conjunto de dados. Resolução final deve ser baseado fora do maior shell e determinada pelos seguintes parâmetros: R fator de> 50%, I / sigma> 2, e integralidade> 90%.
- Construir um modelo de densidade de elétrons 3-Dimensional com o software gráfico molecular COOT 8.
- Antes de depositar a estrutura no APO validar com MolProbity 9 software para verificar se a qualidade da estrutura é adequado para a deposição.
Resultados
Os resultados seguintes ilustram os resultados esperados se o protocolo descrito, e, no caso de PB2, os resultados observados.
Usando Composer Gene, cinco seqüências de aminoácidos alvo full-length do vírus influenza subunidade da polimerase PB2 foram concebidos (Figura 2). As seqüências foram PB2 volta traduzido e submetido a várias etapas de engenharia 3, resultando em seqüências harmonizadas códons otimizados para expressão em E. coli a partir dos produtos. IPCR (Figura 3b), um total de trinta e quatro constructos foram clonados com sucesso num sistema de vector pET28 modificado 10 com um N-terminal de 6x His-tag Smt fusão usando clonagem TUBO 3 como mostrado na Figura 3a. Um resumo do fluxo de trabalho de clonagem é apresentado na Figura 4.
Após a clonagem bem sucedida, a expressão de proteínas em micro-escala de cada construção foi testada em células BL21 (DE3) de E.células de E. coli. As células foram cultivadas em meio TB suplementado com Novagen noite expresso um meio (de acordo com o protocolo do fabricante) durante 48 horas a 20 ° C numa incubadora com agitação conjunto a 220 rpm. Após crescimento, as células foram colhidas e testadas quanto à expressão da proteína solúvel usando eletroforese capilar com um compasso de calibre 90 LabChip. Catorze dos trinta e quatro PB2 construções levaram à proteína alvo solúvel e entrou fermentação em larga escala. Culturas em larga escala de cada construção foram cultivadas em meio TB suplementado com 1 noite expresso meio de Novagen de acordo com o protocolo do fabricante. Após crescimento, as células foram colhidas através de centrifugação e armazenadas a -80 ° C. A expressão da proteína em larga escala de cada cultura foi confirmada através de análise de SDS-PAGE (Figura 5) antes de continuar com a purificação em grande escala.
A proteína Maker foi usado para realizar a purificação paralelo dos catorze construções PB2. Os lisados esclarecidas de all catorze construções foram executadas através de uma coluna de níquel-quelato. Depois de determinar que fracções continham proteína alvo por SDS-PAGE, as fracções correspondentes foram reunidas para cada amostra e a concentração de cada foi determinada por uma leitura de A 280. A remoção da etiqueta de His-Smt 6x foi realizado pela adição de ULP1 seguido de diálise durante a noite e uma segunda coluna de níquel. Confirmação da remoção da etiqueta de His-Smt foi realizada por SDS-PAGE (Figura 6), e cada amostra foi concentrada com um tubo de centrífuga kDa Amicon Ultra-10. Após a concentração, utilizando os tubos de centrífuga Ultra Amicon, cada amostra foi corrido numa coluna de dimensionamento de atingir pureza cristalográfica. Uma segunda concentração foi efectuada para aumentar a concentração de proteína a um nível necessário para a cristalização. Todos os quatorze construções foram purificados e entraram em ensaios de cristalização com sucesso.
A cristalização foi iniciada por descongelação do anteriormente frproteína ozen. A cristalização foi realizado numa sala de clima controlado a 16 ° C, com placas especialmente concebidos (Emerald Bio) para estar gota de difusão de vapor (Figura 7). Triagem inicial foi realizado com quatro telas de matriz esparsa; JCSG +, Pacto, Assistente completa e CryoFull (Emerald Bio), seguindo uma estratégia de Newman estendida. 0,4 ul de solução de proteína foi então misturada com 0,4 ml de crystallant (ou solução de reservatório) do reservatório correspondente utilizando placas de cristalização de 96 poços Jr compactas (Emerald Biológicos). Dos catorze amostras purificadas nove deles produziram cristais adequados para estudos de di fracção (Figura 8). Uma in-house difração conjunto de dados foi coletado em cinco das nove construções cristalizadas em Cu Ka comprimento de onda usando um Rigaku SuperBright FR-E + rotativa-ânodo gerador de raios X equipado com Osmic VariMAX HF óptica e um Saturn 944 + detector CCD (Figura 9 ). Cada conjunto de dados foi processada com XDS / XSCALE 4 < / Sup> e escalado para uma resolução final. As tentativas para resolver as estruturas por substituição molecular foram realizadas com 5 Phaser do pacote CCP4 7. Os modelos finais foram obtidos após refinamento na REFMAC 7 e reconstrução manual com Galeirão 11. As estruturas foram avaliadas e corrigida para a geometria e aptidão com MolProbity 9. Um total de quatro estruturas da subunidade PB2 foram determinados (Figura 10) e depositado no PDB. Figura 11 ilustra o resultado total em cada fase na tubagem MTPP.

Figura 1. Visão geral da via gene-to-estrutura SSGCID para processamento paralelo Multi-alvo em Emerald Bio.
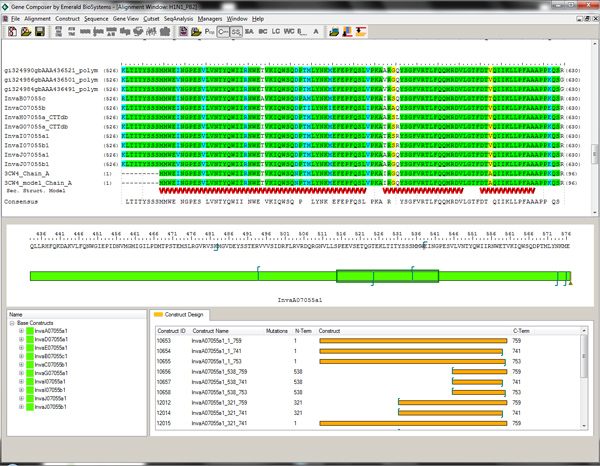
Figura 2. Visualizador de alinhamento e Proteína Construir Módulo de Design, em software Composer Gene. Construção do destino da base de aminoácidos é mostrada em verde (janela do meio) ea estrutura truncations guiadas de construções alternativas são mostradas em ouro (janela inferior). Um alinhamento de múltiplas sequências de gripe viral PB2 é mostrado em relação à seqüência e elementos de estrutura secundária do domínio C-terminal a partir 3CW4 PDBID. O conhecimento da estrutura de domínio e elementos de estrutura secundária permite truncations N-terminal a ser escolhido dentro do Módulo Projeto Composer Gene clicando no resíduo de aminoácido desejado. Clique aqui para ver a figura maior .
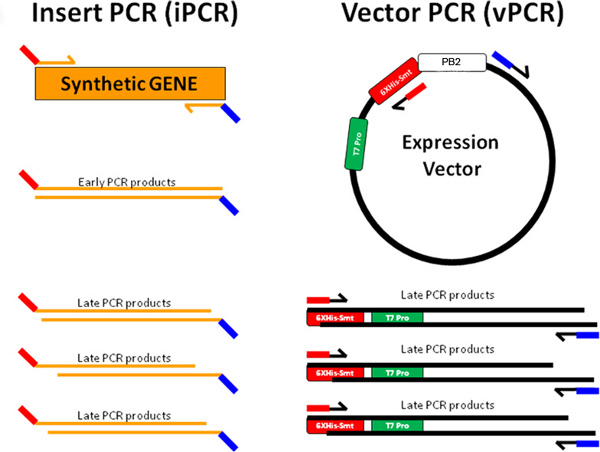
Figura 3A. Clonagem tubo é ilustrado em que a inserção de gene sintético (laranja) é amplificada pela projetado para frente (linhas vermelho-alaranjado) e primers reversos (linhas laranja-azul) para gerar inserir material de PCR. Vector de expressão é amplificado com linhas inversa (rubro-negras ) e para frente (linhas azul e preto), primers para gerar material de vetor PCR. As sequências de produtos IPCR terminais são complementares às seqüências terminais de produtos VPCR (vermelho de IPCR complementa vermelho de VPCR e azul de IPCR complementa azul VPCR). Isto permite que os produtos de IPCR e VPCR recozer para formar os plasmídeos que são replicados a transformação em hospedeiro BL21 (DE3) de E. quimicamente competente células de E. coli.

Figura 3B. Análise em gel de agarose de IPCR produção ts da subunidade PB2. IPCR falhas podem ser vistos como bandas ténues ou manchada, enquanto os produtos de IPCR bem sucedidas são representadas por bandas robustos. a qualidade do produto pode geralmente ser IPCR correlacionada com sucesso a clonagem. Marcadores de peso molecular são em kDa. A figura é reproduzida a partir de Raymond et al., 2011 12.

Figura 4. Gene passos de engenharia de proteínas PB2 alvo foram realizadas utilizando o software Compositor Gene. Depois foi estabelecida a sequência de ácido nucleico de engenharia para cada destino, 6-7 construções alternativas de proteínas foram desenhados para cada um. Processamento paralelo multi-alvo nas etapas iniciais do projeto gene e clonagem resultou em 34 construções, das quais 14 eram alvos viáveis que produzem proteínas solúveis em E. coli.
re 5 "src =" / files/ftp_upload/4225/4225fig5.jpg "/>
Figura 5. Representativas de análise de SDS-PAGE de fermentação em grande escala mostrando a expressão da proteína robusta (tamanho esperado de 25,76 kDa), cerca de 50% solúvel em água (pista 4) e cerca de 50% de clivagem da etiqueta 6x His-Smt de proteína eluida (pista 7).
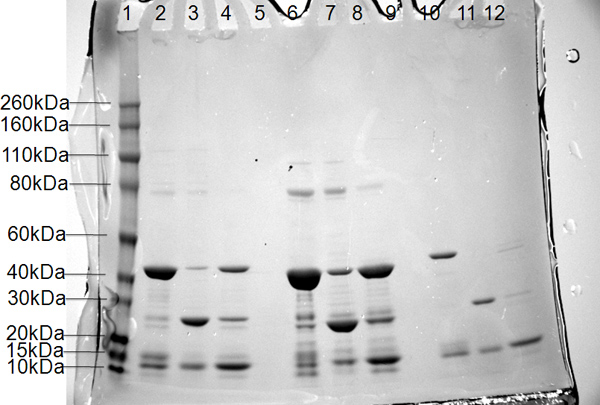
Figura 6. Resultados de SDS-PAGE de três construções da polimerase subunidade PB2 pista 1, os marcadores de peso molecular (rotuladas à esquerda em kDa),. Pistas 2, 6 e 10, proteínas agrupados a partir de uma coluna de níquel; faixas 3, 7 e 11, fluxo através da proteína clivada em tampão A a partir de 2 de níquel; pistas 4, 8 e 12, a remoção de etiqueta 6x His-Smt em tampão B a partir de 2 de níquel.
d/4225/4225fig7.jpg "/>
Figura 7. Um esquema de difusão de vapor pelo método da gota sentada. Método queda sentado por cristalização de proteínas cai sob a categoria de difusão de vapor. Este método baseia-se uma amostra purificada de proteína precipitante e equilibrar-se com um reservatório maior contendo condições semelhantes em uma concentração mais elevada. Como a água se vaporiza a partir da amostra de proteína e transfere para o reservatório, a concentração do precipitante aumenta para um nível óptimo para a cristalização de proteínas.

Figura 8. A proteína cristalina de polimerase PB2 subunidade de uma estirpe do vírus da gripe.

Figura 9. Imagem da polimerase a partir de uma subunidade de PB2 de difracção de raios-Xestirpe do vírus da gripe.

Figura 10. Fita diagramas das moléculas na unidade assimétrica cristalográfica de quatro estruturas. PB2 estruturas secundárias no padrão colorido arco-íris com códigos correspondentes. PDB (a) 3K2V (A/Yokohama/2017/2003/H3N2) (b) 3KHW (A / México / InDRE4487/2009/H1N1) (c) 3KC6 (A/Vietnam/1203/2004/H5N1) (d) 3L56 (A/Vietnam/1203/2004/H5N1).

Figura 11. Análise de resultado para gripe PB2 alvos pelos métodos descritos. The structure determinação do gasoduto é ilustrada em cinco passos: clonagem, a solubilidade, a purificação, a cristalização e determinação de estrutura.
Discussão
Multi-Alvo Processamento Paralelo
Concepção de fármacos com base Estrutura desempenha um papel importante na descoberta de drogas. O SSGCID é dedicado a fornecer a comunidade científica com estruturas de proteínas em três dimensões a partir de NIAID categoria patógenos AC. Fazendo essa informação estrutural amplamente disponível acabará por servir para acelerar o desenho de drogas baseado em estrutura.
O primeiro passo crítico da abordagem MTPP é o projeto da construção. Várias construções de cada proteína alvo aumenta a probabilidade de a determinação da estrutura e aumenta o retorno bem sucedido. É inevitável que algumas construções de proteína irá falhar durante os estágios do pipeline. Aplicação do método de clonagem TUBO suporta o método MTPP, permitindo a geração de muitas construções em formato de 96 poços, sem passos de purificação de trabalho intensivo. Emparelhamento clonagem tubo com a capacidade de analisar a expressão de proteína com o mesmo formato de 96 poços (Caliper LabChip 90) agiliza ainda mais o fluxo total. O emparelhamento destes métodos permite a rápida identificação de construções que produzem a proteína solúvel, que garante o sucesso da produção de proteína em larga escala e purificação.
Um aspecto essencial para o sucesso da MTPP alto rendimento é o fabricante de proteínas (Patente dos EUA No. 6.818.060, Emerald Bio) instrumento. A proteína Maker é um canal paralelo 24 sistema de cromatografia líquida desenvolvida especificamente para aumentar a eficiência da produção de proteína de alto rendimento e as aplicações de investigação gasoduto relacionados estruturais genômicas. Utilizando o protocolo anteriormente descrito para a proteína de chá, as vantagens são evidentes, em comparação com um único sistema de FPLC linha. Uma única pessoa pode purificar até 48 alvos em paralelo dentro de um período de oito horas. Em contraste, uma única pessoa que utiliza um único sistema de FPLC linha só pode purificar a um máximo de quatro alvos dentro do mesmo período de tempo. Os altos níveis de pureza para cada alvoobtida com a proteína Maker são um factor crítico no sucesso posterior do crescimento de cristais de proteínas para análise da estrutura.
Limitações e problemas
Solução de estruturas tridimensionais por cristalografia de raios-x é um esforço de multi-estágios com vários desafios, uma das quais é a incapacidade de obter grandes quantidades de proteína alvo solúvel. Uma estratégia que possa ser aplicado para ultrapassar o problema de solubilidade é o uso de um hospedeiro de expressão alternativo como E. células coli são incapazes de realizar vários importantes eucarióticas modificações pós-traducionais. Expressão em várias leveduras, linhas celulares de insecto e de mamífero que são capazes de realizar estas modificações pós-traducionais são frequentemente uma alternativa adequada. Proteínas alvo são por vezes expressos mas completamente insolúvel nas condições padrão de lise. A proteína Maker pode ser um recurso valioso para o teste rápido de condições de lise celular alternativastal como descrito em Smith et ai. 2011 13. Esta estratégia é muitas vezes necessário para manter alvos em movimento através do pipeline. Em qualquer gasoduto genômica estrutural, protocolos padronizados pode não ser adequado para todos os alvos que vem através do gasoduto e as metas podem precisar de otimização individual. Por exemplo, optou-se por utilizar 20% de etileno glicol para cada crioprotetor. No caso de esta condição não é adequado, os crioprotectores ou concentrações alternativas podem precisar de ser testado.
Devido à natureza única de cada proteína alvo individual, a taxa de limitação e imprevisível passo na determinação de uma estrutura é a cristalização. Os deslocamentos gasoduto MTPP o comumente baixa taxa de sucesso de cristalização de proteínas com a otimização das telas iniciais matriz esparsas. Cada cristal inicial bater comercialmente disponíveis a partir de telas de matriz esparsa é ainda mais otimizado com um Construtor de E-Screen (Emerald Bio). A tela de otimização é projetado around o estado do cristal de acerto inicial, alterando a concentração dos sais, tampões e outros aditivos. Telas de otimização de sucesso produzir cristais adequados para estudos de difração e determinação estrutura.
O programa de genômica estrutural estendeu pelo Instituto Nacional de Alergia e Doenças Infecciosas (NIAID) oferece financiamento para Emerald Bio e outras três instituições Pacific Northwest que, juntos, são a SSGCID (Emerald Bio, SeattleBiomed, da Universidade de Washington e Pacific Northwest National Laboratory) . Cada membro do consórcio foi escolhido por sua experiência em aplicação de tecnologias state-of-the-art necessários para realizar os objetivos do programa de genômica estrutural NIAID. Até à data, SSGCID tenha depositado 461 estruturas para o ranking PDB como o sétimo maior contribuinte no mundo e, em 2011, o mais produtivo. Os protocolos e metodologias da SSGCID são fornecidos com a intenção de beneficiar ocomunidade científica e perpetuar a pesquisa de doenças infecciosas.
Divulgações
Os autores são funcionários da Emerald Bio, Inc.
Agradecimentos
Os autores gostariam de agradecer a todos os membros do consórcio SSGCID. Consecução dos objetivos da SSGCID é possível graças as enormes esforços de todos os membros da equipe em Emerald Bio. Esta pesquisa foi financiada no âmbito do contrato Federal n º HHSN272200700057C do Instituto Nacional de Alergia e Doenças Infecciosas, dos Institutos Nacionais de Saúde e do Departamento de Saúde e Serviços Humanos.
Materiais
| Name | Company | Catalog Number | Comments |
| Primers | IDT | ||
| Genes | DNA 2.0 | ||
| TE buffer | Qiagen | provided in kit | |
| 96-well half skirt PCR plates | VWR | 10011-248 | |
| PFU Master Mix | |||
| 6X Orange Loading Dye | Fermentas | R0631 | |
| 10X TAE | Teknova | T0280 | |
| Agarose | Sigma-Aldrich | A9414-10G | |
| pET28 vector | |||
| 2-YT Broth | VWR | 101446-848 | |
| Kanamycin | Teknova | K2151 | |
| Restriction Enzymes | Fermentas | ||
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Top 10 chemically comp cells | Invitrogen | C4040-06 | |
| Disposable Troughs (Sterile, 25 ml) | VWR | 89094-662 | |
| Airpore covers (Rayon films for bio cultures) | VWR | 60941-086 | |
| 24-well blocks | VWR | 13503-188 | |
| QIAvac 96 | Qiagen | 19504 | |
| BL21(DE3) cells chemcomp (phageR) | NEB | C2527H | |
| 50% Glycerol | VWR | 100217-622 | |
| TB Media | Teknova | T7060 | |
| IPTG | Sigma-Aldrich | ||
| 1 M Tris pH 8.0 | Mediatech | 46-031-CM | |
| 5 M NaCl | Teknova | S0251 | |
| Glycerol | Aldrich | G7893-4L | |
| CHAPS | JT Baker | 4145-01 | |
| Imidazole | Sigma | 56749-1KG | |
| TCEP | Amresco | K831-10G | |
| L-arginine | Amresco | 0877-500G | |
| Benzonase | EMD | 70746-3 | |
| Lysozyme | USB | 1864525GM | |
| 10 kDa MWCO dialysis tubing | Thermo | 68100 | |
| Amicon Ultra 10 ka MWCO concentrators | Millipore | UFC901024 | |
| HisTrap FF columns | GE | 17-5255-01 | |
| HiTrap Chelating columns | GE | 17/0408-01 | |
| Compact Jr crystallization plates | Emerald Bio | EBS-XJR | |
| Crystalization screens | Emerald Bio | ||
| Ethylene Glycol 100% | Emerald Bio | EBS-250-EGLY | |
| Crystal Wand Magnetic Straight | Hampton Research | HR4-729 | |
| Mounted CryoLoop 0.1-0.2 mm | Hampton Research | HR4-955 | |
| ALS style puck | |||
| Puck Wand | |||
| Bent Tongs | |||
| Puck Pusher |
Referências
- Lowen, A. C., Mubareka, S., Steel, J., Palese, P. Influenza virus transmission is dependent on relative humidity and temperature. PLoS Pathogens. 3 (10), 1470-1476 (2007).
- Yamada, S., et al. Biological and structural characterization of a host-adapting amino acid in influenza virus. PLoS Pathog. 6, e1001034 (2010).
- Lorimer, D., Raymond, A., Walchli, J., Mixon, M., Barrow, A., Wallace, E., Grice, R., Burgin, A., Gene Stewart, L. Composer: database software for protein construct design, codon engineering, and gene synthesis. BMC Biotechnol. 9, 36 (2009).
- Kabsch, W. Integration, scaling, space-group assignment and post-refinement. Acta Cryst. D. 66, 125-132 (2010).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. J. Phaser crystallographic software. Appl. Cryst. 40, 658-674 (2007).
- Kuzuhara, T., Kise, D., et al. Structural basis of the influenza A virus RNA polymerase PB2 RNA-binding domain containing the pathogenicity-determinant lysine 627 residue. J. Biol. Chem. 284, 6855-6860 (2009).
- Murshudov, G. N., Skubàk, P., Lebedev, A. A., Pannu, N. S., Steiner, R. A., Nicholls, R. A., Winn, M. D., Long, F., Vagin, A. A. REFMAC5 for the refinement of macromolecular crystal structures. Acta Cryst. D. 67, 355-367 (2011).
- Cowtan, K. Recent developments in classical density modification. Acta Cryst. D. 66, 470-478 (2010).
- Chen, V. B., Arendall, W. B., Headd, J. J., Keedy, D. A., Immormino, R. M., Kapral, G. J., Murray, L. W., Richardson, J. S., Richardson, D. C. MolProbity: all-atom structure validation for macromolecular crystallography. Acta Cryst. D. 66, 12-21 (2010).
- Mossessova, E., Lima, C. D. Ulp1-SUMO crystal structure and genetic analysis reveal conserved interactions and a regulatory element essential for cell growth in yeast. Mol. Cell. 5, 865-876 (2000).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Cryst. D. 66, 486-501 (2010).
- Raymond, A. C., Haffner, T. E., Ng, N., Lorimer, D., Staker, B. L., Stewart, L. J. Gene design, cloning and protein-expression methods for high-value targets at the Seattle Structural Genomics Center for Infectious Disease. Acta Cryst. F. 67, 992-997 (2011).
- Smith, E. R., Begley, D. W., Anderson, V., Raymond, A. C., Haffner, T. E., Robinson, J. I., Edwards, T. E., Duncan, N., Gerdts, C. J., Mixon, M. B., Nollert, P., Staker, B. L., Stewart, L. J. The Protein Maker: an automated system for high-throughput parallel purification. Acta Cryst. F. 67, 1015-1021 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados