É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Uma ferramenta analítica que quantifica Alterações morfologia celular de imagens tridimensionais de fluorescência
Neste Artigo
Resumo
Nós desenvolvemos uma plataforma de software que utiliza Imaris Neuroscience, ImarisXT e MATLAB para medir as alterações na morfologia de uma forma indefinida retirado tridimensional fluorescência confocal de células individuais. Esta nova abordagem pode ser utilizada para quantificar as alterações na forma da célula após a activação do receptor e, portanto, representa um instrumento adicional possível para a descoberta de drogas.
Resumo
As ferramentas de software mais comuns de análise disponíveis para a medição de imagens de fluorescência são para dados bidimensionais (2D) que dependem de ajustes manuais para inclusão e exclusão de pontos de dados, e auxiliado por computador de reconhecimento de padrão para apoiar a interpretação e as conclusões da análise. Tornou-se cada vez mais importante ser capaz de medir imagens de fluorescência construídos a partir de conjuntos de dados tridimensionais (3D) a fim de ser capaz de captar a complexidade da dinâmica celular e compreender a base de plasticidade celular dentro de sistemas biológicos. Instrumentos sofisticados de microscopia permitiram a visualização de imagens 3D de fluorescência através da aquisição de imagens de fluorescência multiespectrais e software analítico poderoso que reconstrói as imagens a partir de pilhas confocal que, então, fornecem uma representação em 3D das imagens recolhidas 2D. Avançados métodos de projeto baseados em estereologia evoluíram a partir da aproximação e suposições da original estereologia modelo baseado em um mesmo em cortes de tecidos complexos 2. Apesar destes avanços científicos em microscopia, permanece a necessidade de um método automatizado analítico que explora plenamente os dados intrínsecos 3D para permitir a análise e quantificação das mudanças complexas na morfologia celular, localização de proteínas e tráfico receptor.
Técnicas correntes disponíveis para quantificar as imagens de fluorescência incluem meta-Morph (Molecular Devices, Sunnyvale, CA) e Image J (NIH), que fornecem uma análise manual. Imaris software (Andor Technology, Belfast, Irlanda do Norte) fornece o recurso MeasurementPro, que permite a criação manual de pontos de medição que podem ser colocados em uma imagem de volume ou desenhados em uma série de fatias de 2D para criar um objeto 3D. Este método é útil para a medição de um único clique de ponto para medir a distância da linha entre dois objectos, ou para criar um polígono que inclui uma região de interesse, mas é difícil de aplicar a complex celulares estruturas de rede. Filamento Tracer (Andor) permite a detecção automática do neuronal 3D filamento como no entanto, este módulo foi desenvolvido para medir estruturas definidas, como neurônios, que são compostas de dendritos, axônios e espinhas (árvore-como estrutura). Este módulo foi engenhosamente utilizado para fazer medições morfológicas células não neuronais 3, no entanto, os dados de saída de informação de uma extensa rede celular por meio de um software que depende de uma forma da célula em vez de ser definido um modelo em forma amorfa celular. Para superar o problema da análise amorfo em forma de células e fazendo com que o software mais adequado para uma aplicação biológica, Imaris desenvolvido celular Imaris. Este foi um projeto científico com Eidgenössische Technische Hochschule, que tem sido desenvolvido para o cálculo da relação entre as células e organelas. Enquanto o software permite a detecção de restrições biológicas, forçando um núcleo por célulae usando membranas de células para as células do segmento, ele não pode ser utilizado para analisar os dados de fluorescência que não são idealmente contínuo porque ele cria na superfície celular, sem espaços vazios. Para nosso conhecimento, no momento nenhuma abordagem modificável pelo usuário automatizado que fornece informações morfométrica de 3D imagens de fluorescência foi desenvolvido que atinge informação espacial celular de uma forma indefinida (Figura 1).
Nós desenvolvemos uma plataforma analítica usando o módulo de software Imaris núcleo e Imaris XT interface com MATLAB (Mat Works, Inc.). Estas ferramentas permitem a medição 3D de células sem uma forma pré-definida e com os componentes da rede de fluorescência inconsistentes. Além disso, este método vai permitir aos pesquisadores que estenderam experiência em sistemas biológicos, mas não familiaridade com aplicativos de computador, para realizar a quantificação de alterações morfológicas na dinâmica celulares.
Protocolo
1. Tridimensional Análise morfométrica de uma única célula alterações fenotípicas
- De rim embrionário humano (HEK293) as células foram transfectadas com a hemaglutinina (HA), factor de libertação da corticotropina marcado receptor-2 (CRF-R2), um receptor acoplado à proteína G (GPCR), como descrito anteriormente 4, 5.
- As células foram deixadas sem tratamento (sem tratamento, NT), estimuladas com o ligando endógeno do CRF-R2, factor de libertação de corticotropina, CRF (1 uM, 30 min), ou pré-tratados com um antagonista de CRF-R2 selectiva, anti-sauvagina 30 (AS -30, 1 uM, 30 min) antes do tratamento com o agonista.
- As células foram então fixadas, permeabilizadas e tratados com anti-HA. CRF-R2 foi visualizada utilizando Alexa 594 nm conjugado anti-ratinho (IgG 1) anticorpo. DAPI foi utilizado para visualizar a fase mitótica núcleos.
- Para limitar a subjectividade experimentador, as condições experimentais não foram conhecidos depois as imagens foram obtidas e analisadas.
- Adquirimos imagems fixos a partir de células HEK293, utilizando um plano-Apochromat 63x/1.4 óleo objectivo DIC e Zeiss LSM 510 META microscópio confocal ligado a um sistema de laser Coherent integrado de dois fotões compreende um laser Verdi-V5 e um 900-F Mira sistema laser.
- Durante o processo de aquisição de dados, as células foram tanto compartimentado por seccionamento multiespectral, 488 nm, e 790 nm (350 nm ~ Ex 2PH.) E particionamento de z-(em incrementos de 0,5 ^ M) de modo a incluir os dados a partir da membrana nuclear para o receptor externo extracelular extremidades.
- Os dados de fluorescência foram primeiro tratados com Imaris, que permite a visualização e segmentação do conjunto de dados de microscopia 3D, e um modelo 3D composto por voxels cúbicos foi criado para análise morfométrica.
Então, Imaris XT módulo foi utilizado para Imaris interface com MATLAB linguagem de programa de computador para determinar o ponto de coordenadas das extensões de GPCR.
Para levar em conta a variabilidade celular, que obteve e analisou as imagens de fluorescência taken das 22 células: sem tratamento (NT) (n = 7), o tratamento com agonista (CRF) (n = 8) e um pré-tratamento com o antagonista (AS-30) antes de tratamento com o agonista (n = 7) (Figura 2) .- A região de interesse (ROI) deve incluir uma célula que não está em fase mitótica ativa e não perto de outras células. Deste modo, a análise inclui as células com apenas um núcleo e as extensões do receptor não são perturbados pela proximidade de outras células.
- A estrutura 3D celular foi reconstruída a partir de dados de fluorescência utilizando multiespectrais Imaris (v.7.1.1).
- Seguindo o algoritmo desenvolvido por Imaris, primeiro processamento da superfície foi utilizada para representar a membrana nuclear. Imaris irá determinar se existe mais do que um núcleo na ROI.
- Em seguida, o algoritmo de criação de pontos foi utilizada para localizar a extensão CRF-R2. Detecção manchas foi utilizada porque compensa o ruído de fundo e da intensidade da irregularrede complexa de células em forma amorfa.
- Para maximizar a inclusão de cada unidade de detecção de fluorescência de CRF-R2, o diâmetro dos locais foi fixado a 0,2 um, o que é a unidade mais pequena dentro da imagem para extrapolar informações distintas sob a forma de uma intensidade medida utilizando um filtro de Gauss. Filtragem local foi incorporado no processo de criação de pontos automatizado. O software, no entanto, dá ao usuário a flexibilidade de usar filtros para definir os parâmetros.
- Para evitar o truncamento de dados, o conjunto de dados foi convertido a partir de 8 bits (unsigned) ponto fixo, a 32-bit decimal.
- Os dados voxel intensidades foram trocadas para detectar dados de coordenadas usando Imaris módulo XT interface com MATLAB e a localização exacta de cada ponto espacial foi determinada através da realização de uma transformação distância utilizando a membrana nuclear, tal como um ponto de referência (Figura 3).
- Os dados resultantes podem ser quantificados e apresentados em formato gráfico para sanálise tatistical. As comparações entre os grupos foram realizadas utilizando ANOVA two-way e Bonferroni pós-teste. Os dados são apresentados como média ± DP. As diferenças são consideradas significativas quando p * <0,05. Os cálculos foram feitos com o programa GraphPad Prism 5.02 (Figura 4).
2. Resultados representativos
Para demonstrar o poder da nossa abordagem, quantificou as alterações celulares que resultam da interacção da proteína G receptores acoplados (GPCRs) e factor de libertação da corticotropina receptor-2 (CRF-R2) com o seu ligando endógeno de CRF em células HEK293 transfectadas.
Mostra-se que o CRF-R2 Os receptores estão localizados na membrana plasmática e do projecto de regiões finitas da membrana das células (Figura 2A e Filme 1). Usando a análise 2D convencional, é possível detectar este subconjunto de extracelulares receptores CRF-R2 apenas se analisar o receptor de adesão points no vidro cobre. Consequentemente perdemos qualquer outra informação derivada de z-empilhados dados multiespectrais (Figura 5).
Quando as células são tratadas com CRF, os receptores extracelulares são muito reduzidos, tal como demonstrado pela diminuição da distância dos pontos da membrana plasmática. Eles também são redistribuídas principalmente a partir de locais em um número finito de locais discretos (Figura 2B e Filme 2).
O efeito do CRF sobre a distribuição dos receptores da membrana é evitada por pré-tratamento com o antagonista de CRF-R2 específico, antisavagine 30 (AS-30) e descobrimos que as extensões de CRF-R2 não mudam (Figura 2C e Filme 3).
A distribuição de pontos distal, representada nas 5 um espectro de cores codificadas com intervalos, é utilizada para visualizar a distância dos voxels da membrana nuclear. Não pretreatm tratamento e antagonistaent (AS-30, 1 ^ M, 30 min) antes de tratamento com o agonista (CRF, 1 uM, 30 min) não mostram diferenças (ns) significativa na contracção GPCR. O tratamento das células com o agonista (CRF, 1 uM, 30 min) reduz progressivamente o número de CRF-R2 contendo voxels quando comparado com qualquer outro tratamento, 0-5 ^ m (ns), 6-15 uM (** p < 0,01) e> 15 ^ m (*** p <0,005) ou em comparação com o tratamento AS-30, 0-10 ^ m (ns), 11-15 ^ m (** p <0,01) e> 15 ^ m (*** p <0,005) (Figura 4).
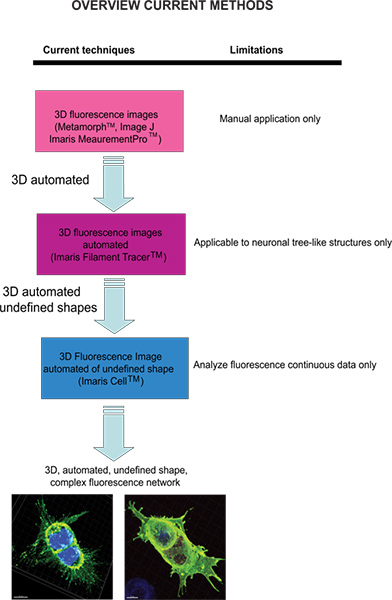
Figura 1. Diagrama das técnicas atuais disponíveis e sua limitação para analisar imagens de fluorescência. Clique aqui para ver maior figura .
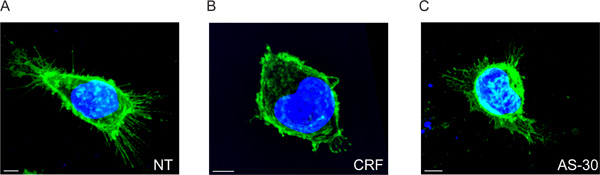
Figura 2. 3D multiespectrais imagens de fluorescência das células HEK293 transfectadas com HA-CRF-R2, antes e depois do tratamento agonista e antagonista. Imagens fundidas mostrando HA-CRF-R2-sondadas com anti-HA e visualizados utilizando Alexa 488 de anticorpo anti-ratinho (IgG 1) conjugado secundário; DAPI foi utilizado para visualizar os núcleos. As imagens foram obtidas com microscópio confocal de varredura a laser (CLS). Escala da barra 5 um.
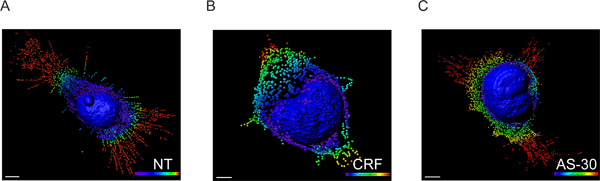
Figura 3. Modelo 3D do HEK293 células transfectadas com HA-CRF-R2 reconstruído a partir de imagens usando o software CLS Imaris. Rendering superfície do núcleo e da criação de pontos que descreve extensões GPCR convertidos em pequenas vesículas. Os dados de fluorescência foram primeiro tratados com Imaris que permite a visualização e segmentação do conjunto de dados 3D de microscopia. Então, Imaris XT foi utilizado para Imaris interface com MATLAB. As intensidades de voxel foram trocadas emlocal coordena. Os pontos do espectro de cores codificadas (azul 0-5 pm, verdes, 6-10 uM, amarelas e vermelhas uM 11-15> 15 uM) representam a forma de distância da membrana nuclear. Escala de 5 ^ m.
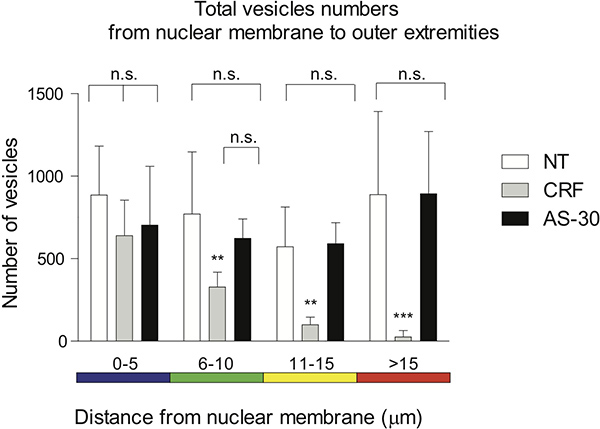
Figura 4. Representação gráfica da distribuição distal dos pontos traçados no espectro de cores codificadas em intervalos de 5 uM é utilizada para visualizar a distância dos voxels da membrana nuclear. Nenhum tratamento e pré-tratamento com um antagonista (AS-30, 1 uM, 30 min) antes de tratamento com o agonista (CRF, 1 uM, 30 min) não mostram diferenças (ns) significativa na contracção GPCR. O tratamento das células com agonista (CRF, 1 uM, 30 min) reduz progressivamente a distância entre o número de CRF-R2 contendo voxels quando comparado com nenhum tratamento 0-5 ^ m (ns), 6-15 uM (** p <0,01) e> 15 mm (*** p <0,005), ou AS-30 do tratamento 0-10 fim (ns), 11-15 uM (** p <0,01) e> 15 mm (*** p <0,005).
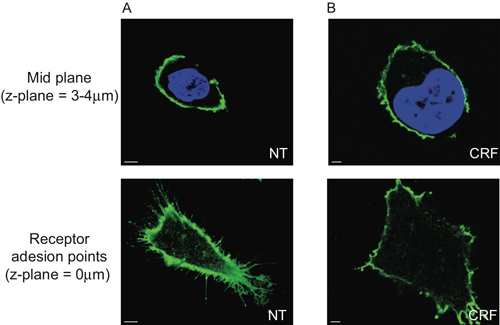
Figura 5. Limitação da análise morfométrica 2D de células HEK 293 transfectadas com HA-CRF-R2. A secção do plano médio de células (3-4μm acima da lamela de vidro) que mostra o centro dos núcleos visualizado com DAPI e HA-CRF-R2 sondadas com anti-HA e visualizados utilizando Alexa 488nm conjugado anti-ratinho (IgG 1) secundário anticorpo e adquiridas com CLS não mostra nenhuma diferença entre (A) sem tratamento (NT) e (B) agonista (CRF, 1 uM, 30 min), enquanto que os pontos de aderência dos receptores são muito diferentes.
Filme 1. Livremente rotativo modelo 3D em "Surpass" modo de células HEK 293 transfectadas com HA-CRF-R2, nenhum tratamento, para avaliar as diferenças entre o fenótipo celular proteínas receptoras. Barra de escala de 5-20 ^ m./ Files/ftp_upload/4233/4233movie1.avi "target =" _blank "> Clique aqui para ver filme.
Filme 2. Livremente rotativo modelo 3D em "Surpass" modo de células HEK 293 transfectadas com HA-CRF-R2, o tratamento com o agonista, CRF (CRF, 1 uM, 30 min) para avaliar diferenças entre os fenótipos celulares proteínas receptoras. Barra de escala 5-20 m. Clique aqui para ver filme.
Filme 3. Livremente rotativo 3D em "Surpass" modo de células HEK 293 transfectadas com HA-CRF-R2, o pré-tratamento com um antagonista (AS-30, 1 uM, 30 min) antes de tratamento com o agonista (CRF, 1 uM, 30 min ) para avaliar diferenças entre os fenótipos celulares proteínas receptoras. Barra de escala 5-20 m. Clique aqui para ver filme.
Discussão
Nós mostramos que o tratamento CRF induz uma alteração significativa na morfologia e localização do CRF-R2. A mudança de CRF-R2 foi inibida pelo tratamento com o antagonista selectivo. Nós demonstramos que as modificações do receptor não foi detectada e não pode ser medida usando as técnicas padrão de 2D multiespectrais. A capacidade para estudar as imagens 3D complexas é essencial para incorporar a complexidade de parâmetros biológicos para análise morfométrica. Fomos capazes de fazer medições em 3D...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Agradecemos ao Centro de Desenvolvimento de Imagens Biológicas (BIDC) da Universidade da Califórnia, San Francisco para o uso do Imaris, XT Imaris e Matlab. Agradecemos a V. Kharazia para a assistência técnica e para Henry, LK Floren, L. Daitch por suas contribuições para a edição do manuscrito. Este trabalho foi financiado por fundos do Estado da Califórnia Medical Research em Álcool e Abuso de Substâncias através UCSF a SEB, dos Institutos Nacionais de Saúde: 1R21DA029966-01 e NIH prêmio Fast Track para a tela a coleção MLSMR a SEB, UCSF Faculdade de Farmácia ( Escritório de Dean e Farmácia Clínica) e da Faculdade de Medicina (Farmacologia Clínica e Terapêutica Experimental) para CLHK.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários (opcional) |
| Rim Embrionário Humano (HEK293) | American Type Culture Collection | CRL-1573 | |
| Dulbecco modificado por Eagle (DMEM) | Invitrogen | 11965118 | |
| Soro fetal bovino (FBS) | Invitrogen | SH30070.03 | |
| AlexaFluor-488 (IgG2b) | Invitrogen | A-11001 | |
| monoclonal anti-HA.11 (IgG 1) | Covance | 16B12 | |
| DAPI | Vector Laboratories | H-1200 | ; |
| CRF | Sigma | C2917 | |
| Antisauvagine-30 (AS-30) | Sigma | A4727 |
Referências
- West, M. J. Design-based stereological methods for counting neurons. Prog, Brain Res. 135, 43-51 (2002).
- Burke, M., Zangenehpour, S., Mouton, P. R., Ptito, M. Knowing what counts: unbiased stereology in the non-human primate brain. J. Vis. Exp. (27), e1262 (2009).
- Sugawara, Y., Ando, R., Kamioka, H., Ishihara, Y., Honjo, T., Kawanabe, N., Kurosaka, H., Takano-Yamamoto, T., Yamashiro, T. The three-dimensional morphometry and cell-cell communication of the osteocyte network in chick and mouse embryonic calvaria. Calcif. Tissue Int. 88, 416-424 (2011).
- Vickery, R. G., von Zastrow, M. Distinct dynamin-dependent and -independent mechanisms target structurally homologous dopamine receptors to different endocytic membranes. J. Cell Biol. 144, 31-43 (1999).
- Bartlett, S. E., Enquist, J., Hopf, F. W., Lee, J. H., Gladher, F., Kharazia, V., Waldhoer, M., Mailliard, W. S., Armstrong, R., Bonci, A. Dopamine responsiveness is regulated by targeted sorting of D2 receptors. Pro.c Natl. Acad. Sci. U.S.A. 102, 11521-11526 (2005).
- Gordon, A., Colman-Lerner, A., Chin, T. E., Benjamin, K. R., Yu, R. C., Brent, R. Single-cell quantification of molecules and rates using open-source microscope-based cytometry. Nat. Methods. 4, 175-181 (2007).
- Schock, F., Perrimon, N. Molecular mechanisms of epithelial morphogenesis. Annu. Rev. Cell Dev. Biol. 18, 463-493 (2002).
- Pincus, Z., Theriot, J. A. Comparison of quantitative methods for cell-shape analysis. J. Microsc. 227, 140-156 (2007).
- Spiller, D. G., Wood, C. D., Rand, D. A., White, M. R. Measurement of single-cell dynamics. Nature. 465, 736-745 (2010).
- Loo, L. H., Wu, L. F., Altschuler, S. J. Image-based multivariate profiling of drug responses from single cells. Nat Methods. 4, 445-453 (2007).
- Yarrow, J. C., Totsukawa, G., Charras, G. T., Mitchison, T. J. Screening for cell migration inhibitors via automated microscopy reveals a Rho-kinase inhibitor. Chem. Biol. 12, 385-395 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados