É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Iterativo Otimização de Duplexos de DNA para Cristalização de SEQA-Complexos de DNA
Neste Artigo
Resumo
Estrutura cristalina de complexos DNA-proteína pode fornecer informações sobre a função da proteína, mecanismo, assim como, a natureza da interacção específica. Aqui, nós relatamos como otimizar o comprimento, a sequência e as extremidades do DNA duplex para co-cristalização com Escherichia coli SEQA, um regulador negativo da iniciação da replicação.
Resumo
Escherichia coli SEQA é um regulador negativo da replicação de ADN que impede eventos reinício prematuras por sequestrantes hemimethylated GATC aglomerados dentro da origem de replicação 1. Para além da origem, SEQA é encontrado nos garfos de replicação, onde organiza DNA recentemente replicado em maiores estruturas ordenadas 2. SEQA associados apenas fracamente com únicas seqüências GATC, mas forma complexos de alta afinidade com duplex de DNA contendo vários sites GATC. A unidade estrutural e funcional mínimo de SEQA é um dímero, explicando assim a exigência de pelo menos duas sequências GATC, para formar um complexo de alta afinidade com o DNA hemimethylated 3. Além disso, a arquitectura SEQA, com a oligomerização de ligação de ADN e domínios separados por um ligante flexível, permite a ligação de repetições GATC separados por até três voltas helicoidais. Portanto, a compreensão da função de SEQA em um nível molecular requer a ana estruturallise de SEQA ligada a várias sequências GATC. Na cristalização da proteína-DNA, o DNA pode ter nenhum efeito para uma excepcional sobre as interacções de embalagem, dependendo dos tamanhos relativos e da arquitectura da proteína e DNA. Se a proteína é maior do que o DNA ou pegadas maioria do DNA, o empacotamento cristalino é primariamente mediada por interacções proteína-proteína. Por outro lado, quando a proteína é do mesmo tamanho ou menor do que o DNA ou que abrange apenas uma fracção das interacções de DNA, DNA-DNA e DNA-proteína dominar empacotamento cristalino. Por conseguinte, a cristalização do DNA-proteína complexos requer o rastreio sistemático de 4 ADN de comprimento e as extremidades de ADN (sem corte ou saliência) 5-7. Neste relatório, nós descrevemos como projetar, otimizar, purificar e cristalizar duplex de DNA hemimethylated contendo repetições em tandem GATC em complexo com uma variante dimérica SEQA (SeqAΔ (41-59)-A25R) para obter cristais adequados para a determinação da estrutura.
Protocolo
1. Protein Purification
O ligante flexível que liga o. N-(oligomerização) e C-terminal (de ligação ao ADN), domínios de SEQA auxilia o reconhecimento de hemimethylated repete GATC separadas por 1-3 voltas sobre o DNA Para este estudo, utilizou-se uma variante de SEQA dimérica (SeqAΔ (41-59)-A25R) com um ponto de mutação no domínio N-terminal que impede ainda mais a oligomerização e um linker encurtado que restringe a ligação a DNA em tandem GATC repete separada exclusivamente pela uma vez no DNA (figura 1) de 2,8.
- Transformar BL21 (DE3) células com o plasmídeo que codifica SEQA sob o controle do promotor T7,
- Placa da reacção de transformação em placas de agar LB incluindo 100 fig / ml de ampicilina,
- Escolha colónias mistas para inocular uma cultura de pequena escala durante a noite (meio LB com 100 ug / ml de ampicilina),
- Na manhã seguinte, inocula um L 1 de suporte, utilizando uma diluição de 1:100 do overnight cultura,
- Cultivar as células a uma OD 600 de ~ 0,7 e induzir a produção de proteínas por adição de isopropil β-D-1-tiogalactopiranósido (IPTG) para uma concentração final de 1 mM,
- Continuar a incubação durante 3 horas a 37 ° C com agitação orbital e depois a colheita das células por centrifugação (10 min a 3300 g),
- Ressuspender o sedimento de células em tampão de purificação A e lise por sonicação,
- Limpar o lisado por centrifugação (40 min a 39.000 g) e carregar o sobrenadante numa coluna de heparina, equilibrada com tampão de purificação,
- Eluir SEQA utilizando um gradiente linear de NaCl a 1 M (SEQA elui a ~ 0,7 M de NaCl),
- Reunir as fracções contendo SEQA juntos, dilua para reduzir a força iónica da amostra e de carga em uma coluna de cromatograf ia de troca catiónica equilibrada com tampão de purificação,
- Usando um gradiente de sal linear, SEQA pura elui a ~ 0,4 M de NaCl,
- Reunir as fracções contendo SEQA juntos, concentrandoe (3 mg / ml) em tampão de armazenamento e o armazenamento.
2. Purificação de DNA
- Encomendar complementares oligonucleotídeos unmethylated e metilados de sua empresa favorita,
- Dissolver 1 mol de cada DNA de cadeia única liofilizado em 800 ul de autoclavado DDH 2 O, vórtice e deixe descansar por 10-20 min,
- Adicionar 800 ul de tampão de carga pré-aquecida 2X a cada oligonucleótido,
- Durante 20-30 oligonucleótidos nucleótidos de comprimento, preparar um gel desnaturante a 10% grande (160 x 250 x 1 mm):
* Misture bem 80 ml de mistura de PAGE a 10%, 80 ul de TEMED e 800 ul de persulfato de amónio por gel e derrame,
* Uma vez polimerizado, retire o pente e lavar os poços com DDH 2 O fundo,
* Reúna os géis no elenco gel incluindo placa de arrefecimento e depois preencher os reservatórios superior e inferior com tampão de corrida (1X TBE),
* Pré-executar o gel em 700-750 V para aquecer o gel até 55 ° C,
* Pare a execução e lavar a thoro poçosughly com tampão de corrida. - Aquecer os oligonucleótidos a 90 ° C durante 2 min,
- Vortex e girar as amostras e imediatamente antes de colocar o gel,
- Execute o gel em ~ 700 V e pará-lo uma vez que seu oligonucleotídeo migrou meio. (Note-se que em um gel de poliacrilamida a 10%, azul de bromofenol a co-migra com oligonucleótidos ~ 20 bases de comprimento e xileno cianol FF com ~ 60 bases de comprimento),
- Pare o gel, desmontá-la da caixa de gel e remover os espaçadores,
- Sobre uma superfície plana, remova uma placa de vidro e cobrir o gel com película de plástico,
- Vire o gel ao redor, retire a placa de outro vidro e cubra com filme plástico,
- Marcar as bandas com luz UV e uma placa fluorescente por trás do gel para ver a sombra DNA,
- Cortar a banda com uma lâmina de barbear em pedaços pequenos e transferi-las para um tubo estéril de 15 ml,
- Adicionar 9 ml de tampão de eluição e eluir a noite a 37 ° C, com agitação,
- Transferir cuidadosamente a soluçãopara um tubo de centrífuga de autoclavado utilizando uma ponta de pipeta de carregamento de gel, para evitar a transferência de peças de acrilamida e adicionar 1 ml de acetato de sódio 3 M, pH 7 (diluição 1:10), mais 25 ml de etanol a 100% gelado (2,5 volumes),
- Incubar a -20 ° C durante pelo menos 3 horas,
- Girar e transferir o sobrenadante para um tubo separado,
- Seque a pelota no speed-vac em fogo médio,
- Ressuspender o sedimento em 400 ul de autoclavado DDH 2 O e transferir para um tubo fresco,
- Adicionar 40 ul de acetato de sódio 3 M, a pH 7 e 1 ml de etanol a 100% e misturar bem (vortex) e incubar 30 min à temperatura ambiente, seguido de 30 min a -20 ° C,
- Girar durante 15 min a 18000 g e descarte do sobrenadante,
- Enxaguar o sedimento com 100 ul de etanol a 70% frio para remover o sal residual a partir do sedimento e centrifugação durante 6 min a 18.000 g. Descartar o etanol e secar o sedimento no speed-vac,
- Ressuspender o sedimento em um total de 100 ul de autoclavado DDH 2 O. Medir a concentração do oligonucleótido,
- Para emparelhar as cadeias duplas de ADN hemimethylated, misturar concentrações equimolares de as cadeias complementares individuais e aquecer as misturas a 95 ° C num banho de água durante 5 minutos e, em seguida, deixar arrefecer lentamente até à temperatura ambiente dentro do banho de água.
3. Proteína-DNA Formação de Complexos e Análise
- Mistura de volumes iguais de SeqAΔ purificada (41-59)-A25 (81 uM) e DNA hemimethylated (81 uM),
- Incubar à temperatura ambiente por 15 minutos e armazenar a 4 ° C até que esteja pronto para usá-lo,
- Tela para condições de cristalização usando comerciais esparsas de matriz telas,
- Uma vez que leva a cristalização iniciais terem sido identificados, optimizar as condições de crescimento de cristais de qualidade de difração,
- Cryoprotect os resultantes cristais SEQA-DNA, seja aumentando a quantidade de PEG 400 presente na solução de cristalização para uma concentração final de 25% (v / v) ou a adição de glicerol a 20% (v / v) à solução de cristalização,
- Colher cristais individuais com um laço de nylon, e flash-congelá-los em nitrogênio líquido,
- Teste o limite de difração de cada cristal em 100 K.
4. Resultados representativos
Para obter a estrutura de cristal de SeqAΔ (41-59)-A25R ligado ao DNA hemimethylated, nós consecutivamente três parâmetros optimizados no DNA: (i) a separação entre hemimethylated sequências GATC, (ii) o comprimento do duplex e (iii) o ausência / presença de 5 'saliências.
Os ensaios de deslocamento da mobilidade electro-indicam que SeqAΔ (41-59)-A25R liga preferencialmente repete GATC separadas por 9-10 pares de bases (Figura 1). Portanto, inicialmente peneirado duplex 23-24 pares de base (bps) de comprimento contendo duas seqüências hemimethylated GATC separados por 9 ou 10 bps. Três duplexes produziu cristais bem perfilada (Figura 2). Alembora nenhum dos cristais difractados de alta resolução, o 23 bps duplex longo com os dois sítios GATC separados por 9 bps difractado raios X melhor do que o resto, o que indica que a separação GATC de 9 pb foi preferida para a cristalização. Por isso, fixo o espaçamento inter-GATC a 9 bps para todas as telas subseqüentes.
Geralmente, a cristalização do DNA é favorecido para comprimentos exactos correspondentes a duplex voltas helicoidais, porque as moléculas de ADN múltiplas pode empilhar cabeça-a-cauda, para formar uma contínua B-DNA dentro do cristal 9. Portanto, é reduzido o comprimento total dos duplexes a 21 bps (ou seja, duas voltas helicoidais). Enquanto um 21 bps duplex com extremidades rombas não produziram cristais de difração de qualidade (dados não mostrados), um 21 bps duplex com uma saliência 5 'de um único nucleótido em cada extremidade deu cristais que difractado a 5 Å em nossa fonte de origem. A melhoria em relação ao limite de difracção sugerido que end-to-end duplex associação foi efectivamente favorecering empacotamento cristalino.
Desde SEQA interage com sequências GATC que estão na mesma face do DNA, a face oposta do duplex de ADN devem ser expostos ao solvente. Para melhorar ainda mais os cristais do complexo, então a sequência modificada de modo a incluir o duplex um dinucleótido CG entre os dois sítios GATC promover ranhura backbone interacções com moléculas de DNA adjacentes no cristal por meio da face oposta do duplex, um método que tem sido utilizado para melhorar a cristalização do DNA entre os 10 passado. No entanto, o limite de difracção dos cristais crescidos com o duplex contendo CG era idêntica às cultivadas com um duplex de ADN similares que não contêm o dinucleótido CG (Figura 3). Este resultado indicou que a ranhura backbone interacções não são importantes neste caso. Nós posteriormente optimizada do comprimento das saliências, comparando os cristais crescidos com um duplex de ADN que tinha dois nucleótidos adicionais em cadaExtremidade 5 '. Esta alteração não teve um efeito drástico sobre a morfologia do cristal, bem como, o limite de difracção, indicando que as sequências de nucleótidos adicionais mudou dramaticamente os contactos moleculares e organização de cristal aumentada (Figura 3).
A estrutura cristalina da SeqAΔ (41-59)-A25R ligado a este último duplex de ADN confirmou que a face livre do duplex de ADN não se envolve em contactos de cristais, tal como esperado a partir do efeito limitado da introdução de um dinucleotídeo CG. Apesar do tamanho relativo das proteínas e de DNA, a maioria das interacções entre os companheiros de simetria são mediados por interacções proteína-proteína e proteína-DNA (Figura 4). É interessante notar que, neste caso particular, o efeito benéfico de uma saliência 5 'dinucleótido não é devido à formação de um pseudo-DNA contínua. Em vez disso, a extremidade 5 'dos projectos de partículas metiladas distância dos eixos de DNA e interage com a molécula SEQA proximal do complexo, o que explica os nucleótidos dois w ere estritamente necessária para mudar a embalagem cristal de 8,11.
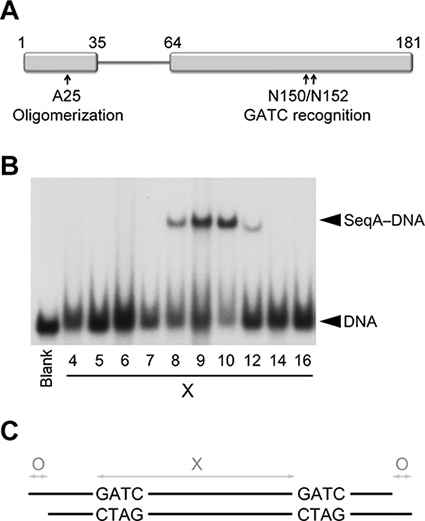
Figura 1. A ligação do SeqAΔ (41-59)-A25R ao DNA hemimethylated. (A) Representação esquemática dos domínios do SEQA que medeiam a oligomerização de proteínas e ligação ao ADN. (B) ensaio de desvio da mobilidade electroforética de SeqAΔ (41-59)-A25R com DNAs contendo dois hemimethylated sequências GATC, separados por um número crescente de pares de bases ( X). A pista mais à esquerda (rotulada em branco) contém uma mistura equimolar de DNAs com 5, 7, 12, 21, 25 e 34 pares de bases entre as duas sequências GATC, na ausência de SeqAΔ (41-59)-A25R. (C) diagrama representando as três variáveis optimizados sobre os dúplices de ADN para se obter cristais de qualidade de difração.
upload/4266/4266fig2.jpg "fo: conteúdo largura =" 6in "fo: src =" files/ftp_upload/4266/4266fig2highres.jpg / "/>
Figura 2. Efeito da variação da inter-GATC Resumo distância. Dos oligonucleótidos utilizados e os cristais obtidos com 23-24 pb de comprimento duplexes contendo dois sítios GATC hemimethylated separados por 9 ou 10 pares de bases. Todas as imagens de cristais foram tiradas com a mesma ampliação e a barra de escala indica 100 mm. O limite de resolução de cada SeqAΔ (41-59)-A25R-DNA de cristal é baseada em imagens de difração coletados em um RU-300 Rigaku sistema gerador de raios-X. Clique aqui para ver maior figura .

Figura 3. Efeito da variação do DNA termina Resumo. De tele oligonucleótidos utilizados e os cristais obtidos com 21 duplexes bps longos contendo dois sítios GATC hemimethylated separados por nove pares de bases, incluindo 0, 1 ou 2 nucleótidos saliências na extremidade 5 '. Todas as imagens de cristais foram tiradas com a mesma ampliação e a barra de escala indica 100 mm. O limite de resolução de cada SeqAΔ (41-59)-A25R-DNA de cristal é baseada em imagens de difração coletados em linhas de luz X12C e X29 (NSLS, BNL). Clique aqui para ver maior figura .
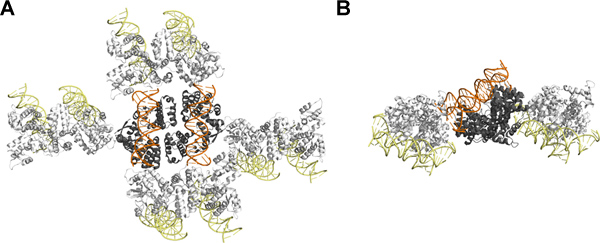
Figura 4. Empacotamento cristalino de SeqAΔ (41-59) A25R-ligado ao DNA com saliência dinucleótido: Vista de cima (A) e lateral (B). A unidade assimétrica contém dois SeqAΔ (41-59)-A25R-DNA complexos onde a proteína é mostrado a cinzento, enquanto o DNA é mostrada na laranja. Symmetry relacionado SeqAΔ (41-59)-A25R-ADN moléculas são mostrados em branco para a proteína e amarelo para o DNA. Esta figura foi preparada utilizando PyMOL 12. Este valor está relacionado ao Filme 1. Clique aqui para ver maior figura .
Filme 1. Clique aqui para ver filme .
Discussão
Um dos maiores desafios em cristalografia macromolecular de raios-X é a obtenção de cristais de qualidade de difração. No caso de complexos de proteínas ou proteína-DNA, este desafio é agravado devido às variáveis adicionais que devem ser optimizadas. Acredita-se que o comprimento do DNA e a presença de saliências pegajosas para melhorar associação de moléculas de DNA adjacentes a mais longo pseudo-duplex são os principais parâmetros para optimizar. No entanto, mostraram que a natureza e extensão...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Os autores gostariam de agradecer ao pessoal PXRR no NSLS (Brookhaven National Laboratory) para a assistência durante a coleta de dados e Pillon Monica para ajuda com a purificação do DNA. Este trabalho foi financiado pelos Institutos Canadenses de Pesquisa em Saúde (MOP 67.189).
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Catálogo # | Comentários (opcional) |
| TRIS | Bioshop | TRS003.5 | |
| Ácido etilenodiaminotetra-acético (EDTA) | Fisher Scientific | E478-500 | |
| Dithiotheitrol (TDT) | Bio Básico Inc. | DB0058 | |
| NaCl | Bioshop | SOD002.10 | |
| Glicerina | Caledon | 5350-1 | |
| Sacarose | Sigma-Aldrich | S5016-500G | |
| Dodecilsulfato de sódio (SDS) | Bioshop | SDS001.500 | |
| Uréia | Bioshop | URE001.5 | |
| 40% Bis 29:1 / acrilamida | Bio Básico Inc. | A0007-500ml | Armazenar a 4 ° C |
| Ácido bórico | EMD | BX0865-1 | |
| Xileno cianol FF | Bio-Rad | 161-0423 | |
| Azul de bromofenol | Bioshop | BR0222 | |
| Sistema de Gel dupla ajustável Vertical | CBC Scientific Company Inc. | DASG-250 | |
| Tela de cristalização índice | Hampton Research | HR2-144 | Armazenar a 4 ° C |
| Assistente de tela cristalização I | Esmeralda BioSystems | EBS WIZ-1 | Armazenar a 4 ° C |
| Assistente de tela cristalização II | Emerald BioSystems | EBS WIZ-2 | Armazenar a 4 ° C |
| Clássicos tela cristalização | Qiagen | 130701 | Armazenar a 4 ° C |
| Bandejas Intelliplate | Arte Robbins Instruments | 102-0001-00 | |
Soluções Tampão de purificação de proteína: 100 mM de TRIS, pH 8, EDTA 2 mM, DTT 2 mM e glicerol a 5%. Tampão de proteínas de armazenamento: Tris 20 mM pH 8, NaCl 150 mM, DTT 5 mM, EDTA 0,5 mM e glicerol a 5%. Gel mistura de carga: Adicionam-se 20 g de sacarose, 25 mg de azul de bromofenol, 25 mg de xileno cianol FF, 1 ml de 10% w / v de SDS e 10 ml de TBE 10X e 70 ml de DDQ autoclavado 2 O. Agita-se com aquecimento suave até que a sacarose é dissolvido e ajustar a finalo volume a 100 ml com autoclavado DDH 2 O. Armazenar a 4 ° C. 2X tampão de carregamento: Adicionar 11 g de ureia em 10 ml de mistura de carregamento de gel. Agita-se sobre uma placa quente até que a ureia dissolve-se. Alíquota em tubos de 2 ml e armazenar a 4 ° C. Mistura PAGE 10X: Misturar 420,4 g de ureia, 100 ml de TBE 10X (autoclavada), 250 ml de 40% de 29:1 Bis / Acrilamida em DDH 2 O. Agitar até totalmente dissolvido e ajustar o volume para 1 litro. Armazenar em frascos escuros a 4 ° C. TBE 10X: Dissolver 108 g de TRIS, 55 g de ácido bórico e 9,3 g de EDTA em 1 litro de DDH 2 O. Autoclave e armazenar à temperatura ambiente. Tampão de eluição: Diluir 8 ml de NaCl 5 M, 2 mL de 1 M de Tris pH 7,5, 0,4 ml de EDTA 0,5 M pH 8 em 200 ml de DDQ 2 O. Autoclave e armazenar à temperatura ambiente. |
Referências
- Campbell, J. L., Kleckner, N. E. coli oriC and the dnaA gene promoter are sequestered from dam methyltransferase following the passage of the chromosomal replication fork. Cell. 62, 967-979 (1990).
- Guarné, A. Crystal structure of a SeqA-N filament: implications for DNA replication and chromosome organization. Embo. J. 24, 1502-1511 (2005).
- Brendler, T., Austin, S. Binding of SeqA protein to DNA requires interaction between two or more complexes bound to separate hemimethylated GATC sequences. Embo. J. 18, 2304-2310 (1999).
- Jordan, S. R., Whitcombe, T. V., Berg, J. M., Pabo, C. O. Systematic variation in DNA length yields highly ordered repressor-operator cocrystals. Science. 230, 1383-1385 (1985).
- Tan, S., Hunziker, Y., Pellegrini, L., Richmond, T. J. Crystallization of the yeast MATalpha2/MCM1/DNA ternary complex: general methods and principles for protein/DNA cocrystallization. J. Mol. Biol. 297, 947-959 (2000).
- Rice, P. A., Yang, S., Mizuuchi, K., Nash, H. A. Crystal structure of an IHF-DNA complex: a protein-induced DNA U-turn. Cell. 87, 1295-1306 (1996).
- Yang, W., Steitz, T. A. Crystal structure of the site-specific recombinase gamma delta resolvase complexed with a 34 bp cleavage site. Cell. 82, 193-207 (1995).
- Chung, Y. S., Brendler, T., Austin, S., Guarne, A. Structural insights into the cooperative binding of SeqA to a tandem GATC repeat. Nucleic Acids Res. , (2009).
- Anderson, J., Ptashne, M., Harrison, S. C. Cocrystals of the DNA-binding domain of phage 434 repressor and a synthetic phage 434 operator. Proc. Natl. Acad. Sci. U.S.A. 81, 1307-1311 (1984).
- Timsit, Y., Moras, D. DNA self-fitting: the double helix directs the geometry of its supramolecular assembly. Embo J. 13, 2737-2746 (1994).
- Chung, Y. S., Guarne, A. Crystallization and preliminary X-ray diffraction analysis of SeqA bound to a pair of hemimethylated GATC sites. Acta Crystallogr Sect F Struct Biol Cryst Commun. 64, 567-571 (2008).
- DeLano, W. L. . The PyMOL Molecular Graphic Systems. , (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados