Method Article
Visualização e análise de moléculas de mRNA Usando Fluorescência
Neste Artigo
Resumo
Este protocolo descreve o procedimento experimental para a realização de Fluorescência In situ Hybridization (FISH) para a contagem de mRNAs em células individuais em resolução de uma única molécula.
Resumo
A hibridação in situ fluorescente (FISH), o método permite a detecção de ácidos nucleicos no ambiente celular nativo. Aqui nós fornecemos um protocolo para a utilização de FISH para quantificar a quantidade de ARNm em células de levedura individuais. As células podem ser cultivadas em qualquer condição de interesse e, em seguida, fixado e tornado permeável. Posteriormente, vários deoxyoligonucleotides single-stranded conjugado com corantes fluorescentes são usados para rotular e visualizar mRNAs. Difracção de fluorescência limitada a partir de moléculas de mRNA individuais é quantificada utilizando um algoritmo de detecção de ponto de identificar e contar o número de mRNAs por célula. Enquanto os métodos de quantificação mais padrão de manchas de Northern, RT-PCR e microarrays de expressão do gene de informação sobre os mRNAs da população média em grandes quantidades, PEIXE facilita tanto a contagem e localização destes ARNm em células individuais com uma resolução de uma única molécula.
Introdução
Utilizando técnicas de medição de massa, não é possível ensaiar a quantidade de transcritos ou atividade transcricional dentro de células individuais 1. Usando proteínas fluorescentes dirigidos por promotores de interesse como repórteres de expressão do gene pode resolver este problema, em certa medida, mas o tempo necessário para as proteínas fluorescentes para dobrar obscurece dinâmica precoces. Proteínas fluorescentes de longa duração também não pode relatar vidas de mRNA. O método de peixe pode ser usado para ensaio de ARNm durante o seu ciclo de vida completo, desde o início da transcrição no núcleo para a maturação e posterior deterioração das células individuais, com a resolução de uma única molécula.
O original experimentos in situ para a visualização de ácidos nucléicos utilizadas sondas de RNA radiomarcados para sondar elementos de DNA. Estes incluíram visualizando ADN ribossomal em ovários da rã Xenopus laevis e 2 de ADN em tecido de rato por satélite 3. O primeiro fluorescente in situ em experiment usada uma molécula de ARN marcado com um fluoróforo para sondar as sequências de ADN específicas 4. A primeira aplicação de sondas fluorescentes para a visualização de RNA in situ foi a visualização da expressão do gene de actina de galinha em cultura de tecido muscular 5. Mais recentemente, na levedura de brotamento, FISH tem sido utilizada para investigar as oscilações na transcrição durante o ciclo metabólico levedura 6, o decaimento do ARNm durante a progressão do ciclo celular 7, e localização espacial dos transcritos de ARNm durante a mitose 8. FISH tem sido utilizado em fermento para mostrar que as flutuações não correlacionadas em genes constitutivamente transcritos, que constituem mais de metade de todos os genes de levedura, surgem uncorrelated iniciação da transcrição 9. Nas espécies não-levedura, FISH foi usado para identificar os marcadores de células-tronco no intestino do rato 10 e para determinar que a penetrância incompleta dos destinos de células pode resultar de flutuações de expressão de genes em C. estocásticoselegans embriões 11.
O método descrito aqui funciona FISH por hibridização marcados com corante, as sondas de ADN de cadeia simples com o ARNm mensagens. As células são fotografadas e os mRNAs são contadas usando um algoritmo local de detecção. Sondas de cadeia simples podem ser gerados com um sintetizador de ADN e, em seguida rotulado (aqui referida como sondas de Singer) ou ordenados comercialmente como sondas de pré-marcadas (Stellaris sondas) 12,13. Uma diferença importante entre o cantor e Stellaris sondas é que as sondas são mais Cantor (~ 50 pb), e são multi-rotulado, enquanto as sondas são curtas Stellaris (~ 20 pb), com apenas uma etiqueta por sonda, tal como descrito por Raj et al 14. Além disso, a abordagem Stellaris utiliza sondas por muitos mais genes do que a de Cantor (~ 30 contra 5 sondas por gene, respectivamente). Abaixo é proporcionado um protocolo que descreva o uso de qualquer tipo de sonda. Na seção 2, apresentamos um protocolo para a rotulagem de amino-alil sondas thymidine contendo wom um corante Cy escolhido. Uma visão geral dos passos computacionais necessários para identificar pontos de mRNA individuais é fornecida na Seção 7.
Protocolo
Figuras 1 e 2 são esquemas dos procedimentos experimentais peixes e dutos de análise de imagem utilizado para quantificar as imagens dos peixes.
1. Soluções para preparar
* As soluções abaixo são para uso com sondas cantor. Se estiver usando sondas Stellaris, substituir "40% formamida" com "10% de formamida" tanto Tampão de Hibridização e tampão de lavagem. Alterações adicionais Tampão de Hibridização quando utilizando sondas Stellaris são: (1) adicionar 1 g de sulfato de dextrano e (2) não incluem 10 mg ssDNA.
Tampão B
8 ml de 1 M de KH 2 PO 4
41,5 ml de 1 M de K 2 HPO 4
109,3 g Sorbitol
Tampão Spheroplasting
890 mL de buffer B
VRC 100 mL
10 ul de 25.000 U / ml de liticase
2 ul β-mercaptoetanol
tampão de hibridação (10 ml de volume final)
10 mg de E. coli tRNA
10 mg de ssDNA *
100 l mM estoque 200 VRC
40 ul de 50 mg / ml de BSA
1 ml de SSC 20X
4 ml de formamida a 40% *
Nuclease de água livre (a 10 ml de volume final)
1 g de sulfato de dextrano *
* Tampão de hibridização pode ser mantido em alíquotas de 0,5 ml a -20 ° C por conveniência.
Tampão de Lavagem (50 ml de volume final)
5 ml de 20X SSC
20 ml de formamida a 40% *
Nuclease de água livre (a 50 ml de volume final)
Rotulagem de buffer
1,06 g de carbonato de sódio
100 ml de água DEPC
pH 9
2. Sonda-rotulagem (Sondas Cantor Only)
Nós obtemos estas sondas por síntese em casa usando um aparelho síntese de oligonucleotídeos ABI. Distróficoaliado, 4-5 ~ 50 pb são sintetizados oligonucleótidos que são homólogos ao gene de interesse, substituindo-alil-amino-timidina por vários thymidines espaçadas de pelo menos 8, de preferência de + 10 pb separadas. Por causa de sua sensibilidade ao ozônio, nós trabalhamos em uma instalação livre de ozono ao usar tinturas CY.
- Obter ~ 5 sondas e ressuspender em 100 mL de água - as concentrações de seleção na Nanodrop.
- Dependendo de quantas sondas / gene, combine total de 10 mg / oligonucleotídeos gene (por exemplo, se tiverem 5 sondas / gene, então quero 2 mg / sonda).
- Use colunas QIAquick para purificar as sondas de acordo com o Kit QIAquick Nucleotide remoção protocolo.
- Adicionar 10 volumes de tampão PN ao volume total de sondas combinadas e misture.
- Aplicar a amostra para a coluna QIAquick - se o volume total é superior a 750 mL, spin para baixo duas vezes com metade do volume em cada rodada
- Deixe repousar por 1 min.
- Centrifugar 1 min a 6000 rpm.
- Lavar com 750 mL de tampão PE.
- Centrifugador1 min a 6000 rpm.
- Elimine o fluxo através da coluna e re-centrifugação durante 1 min a 13.000 rpm a secar.
- Coluna QIAquick lugar no novo tubo de microcentrífuga e DNA eluir com 50 mL H 2 O - Certifique-se de H 2 O pH está dentro de 7.0 e 8.5 e é colocado diretamente na membrana.
- Deixe repousar por 1 min.
- Centrifugar durante 1 minuto a 13000 rpm, para eluir o DNA.
- Liofiliza-se o ADN a 45 ° C.
- Ressuspender o sedimento em 10 ul de tampão de marcação e adicionar ao tubo do corante sem tocar no corante.
- Repita com outro buffer rotulagem 10 ml.
- Vortex e spin para baixo tubo de tinta e DNA.
- Cubra os tubos com papel alumínio e manter no escuro, a RT O / N para rotular.
- Repita QIAquick Nucleotide Removal Kit protocolo. Execute com duas diferenças:
- Adicionar 200 mL de tampão PN sondas e colocar sondas marcadas através de colunas de 2x.
- Realizar três lavagens com tampão PE, a fim de lavar qualquer corante solto antes de eluição. - After eluição obter concentração via NanoDrop. A eficiência de marcação é tipicamente ~ 0,25 pmol / ng de ADN de cadeia simples.
3. Lamela Preparação
- Coloque lamelas em slides em Plasma-preen câmara de vácuo ( http://www.plasmapreen.com/ ) (o mais próximo do centro, melhor).
- Coloque câmara de vácuo no microondas e verifique se ele está selado.
- Ligue a bomba primeiro e depois ligar a bomba de vácuo apenas uma vez é iniciado.
- Ligue o forno de microondas, e parar de 5 segundos após plasma é visível.
- Desligue o vácuo depois da bomba.
- Retire a câmara de vácuo e remover lamelas com uma pinça (aqueles que caíram precisam ser limpos novamente).
- Lugar Lamelas lado limpo em placas de 12 poços.
4. Procedimento de fixação
- Levedura crescer a uma DO600 de cerca de 0,1-0,2 em meio mínimo. 10 ml de células produz suficiente for ~ 10 hibridações separadas.
- Adicione 1/10 volume 37% de formaldeído diretamente para a mídia de crescimento (10 ml de cultura + 1 ml de 37% de formaldeído) e deixe descansar por 45 min.
- Lavar 2x com 1 ml de tampão B gelado num tubo de microcentrífuga (pode girar a 13.000 rpm durante 1 min.)
- Adicionar 1 ml de tampão spheroplasting.
- Incubar a 37 ° C durante 15 min. Confira as células a cada poucos minutos, sob microscópio até a maioria das células são negros (ou seja, não phase-brilhante).
- Lavar 2x com tampão B gelado, girando a uma velocidade baixa (~ 3500 rpm).
- Adicione 1 ml de EtOH a 70%, ressuspender cuidadosamente e deixar durante a noite a 4 ° C (pode armazenar indefinidamente a -20 ° C).
5. Procedimento de Hibridação
- Preparar a solução de hibridização: para 100 ul de tampão de hibridação, adicionar 1-3 mL de detecção, depois vórtice e centrifuga-se. Sondas Cantor usar 8-10 total de ng por conjunto da sonda (ou seja, por gene). Temos fotografada até três genes simultaneamente com três difsonda marcada rente define.
Certifique-se de aquecer a solução de hibridização à temperatura ambiente antes de abri-lo.
Para sondas Stellaris, recomenda-se iniciar quatro reacções de hibridação em separado pela adição de 1 ul de cada uma de 1:10, 1:20, 1:50 e 1:100 diluições de trabalho de sondas para ver qual é óptima. Diluições de trabalho de sondas Stellaris são preparadas em tampão de hibridação.
- Centrífuga (para todos os passos subsequentes, 3.500 rpm, 5 min) as células fixas (por exemplo, 200 ul) e aspirar para fora do etanol.
- Suavemente ressuspender em 1 ml de tampão de lavagem que contém a mesma percentagem de formamida como tampão de hibridização. Deixe repousar por 2-5 min.
- Centrifugar amostra e tampão de lavagem aspirado, em seguida, adicionar solução de hibridização. Incubar no escuro, com agitação suave, O / N a 37 ° C.
Nota: o procedimento a seguir é para a aplicação de células / Imagem em lamelas. Para wcalcinação / células de imagens em placas de 96 poços, incluindo limpador solução alternativa espécies reativas de oxigênio ver http://www.biosearchtech.com/stellarisprotocols.
- No dia seguinte, ou antes, limpo (ver procedimento) e tratar lamelas com 150 ul de 0,01% de poli-L-lisina, durante 5 min. Aspirar, deixe secar, lave 3x com dH 2 O e deixe secar.
- Na manhã seguinte, adicionar 1 ml de tampão de lavagem para a amostra, ressuspender cuidadosamente, de centrifugação e aspiração, ressuspender em seguida mais 1 ml de tampão de lavagem e incubar a 37 ° C durante 30 min.
- Lava-se com 2X SSC + 0,1% de Triton X-100 à temperatura ambiente no agitador de 15 min.
- Tome meio de montagem de freezer para permitir que ele venha até RT antes da montagem.
- Lava-se com 1X SSC a RT 15 min, em agitador.
- Diluir DAPI em PBS (0,1 ug / ml final) e ressuspender as células em 150 ul.
- Solução local para limpeza / poli-Lys-tesouroted tampa desliza na placa de 12 poços, de pelo menos 30 minutos sem ser perturbado.
- Remova a solução (você pode colocá-lo em uma lamínula de reposição como um backup) e lavar 3x com 1 ml de 1X PBS.
- Coloque 3 ml de prolongar meio de montagem de ouro em um slide (Invitrogen P36934). Faça isso um de cada vez se você tiver vários deslizamentos de cobertura para evitar a secagem do meio de montagem.
- Adicionar ~ 0,5 ml de etanol a lamela na placa de 12 poços, remover a lâmina de cobertura e de ar seco, mantendo, com uma pinça.
- Coloque a tampa do lado da cela de deslizamento para baixo em meio de montagem e deixar endurecer várias horas ou O / N no escuro.
- Selar as bordas com unha polonês e proceder à imagem.
6. Imagem de células com Olympus IX-81 Visão geral microscópio invertido
- Para a aquisição de imagens que utilizam software Slidebook (inteligente-imaging.com) e 100X, 1,45 NA, objetivo petróleo TIRFM. Para a série GFP, DAPI, Cy3, Cy5 Cy3.5 e imagem: conjuntos de filtros Chroma (ver reagentes).
- Use filtro DAPIpara encontrar e concentrar células.
- Definir isso como ponto de referência.
- Tome uma z-stack de imagens ao redor do ponto de referência onde a distância total é de 5 mm com tamanho de 0,2 mM (25 aviões). Repita para cada canal de corante (ou seja, conjunto de cada sonda).
- Exportar como um TIFF de 16 bits para análise de imagem.
7. Resumo Análise de Imagem (Sondas cantor)
Abaixo fornecemos um esboço de métodos computacionais que usamos para a análise de imagens de peixes em MATLAB. As funções do MATLAB relevantes utilizados estão entre colchetes à direita. Os algoritmos e limites estão ajustados para dados de sondas estilo cantor. Usando sondas estilo Stellaris exigirá algum ajustamento, particularmente para o passo final de filtragem (7.8).
Identificação de células 15
- Celas separadas de fundo usando um limiar global sobre DAPI imagens de 16 [graythresh].
- Identificar núcleos usando a função maxima estendida [imextendedmax].
- Células segmento usando núcleos como sementes para um algoritmo de watershed [divisor de águas].
Encontrar pontos em cada canal de fluorescência
- Executar a transformação cartola para normalizar fundo e melhorar a relação sinal-ruído [bwmorph].
- Encontrar máximos para identificar a camada de focagem (no plano z) para cada mancha [imregionalmax].
- Filtrar pontos potenciais usando um modelo de ajuste linear a um gradiente radial.
Intensidade da mancha medida e sinais de filtro simples vs múltipla sonda
- Montar um perfil gaussiano 2D para o local na camada de focagem previamente identificados e intensidade de estimativa 17.
- Filtrar os pontos fracos que utilizam um limiar (baseado em um histograma). Para o tipo de cantor sondas isso é importante, mas é menos para as sondas Stellaris.
- Contagem de pontos em cada célula.
Resultados
A figura 3 mostra histogramas típicos calculado a partir de imagens FISH e utilizada para determinar a quantidade de ARNm presente nas células individuais. Uma vantagem importante da microscopia baseado quantificação de ARN é que se pode obter informação sobre a localização de transcritos. Por exemplo, foi utilizado para identificar mRNAs FISH em células individuais com uma indutível CBF1 alelo (Figura 4). Como muitas moléculas de mRNA estão presentes no local da transcrição, que são capazes de identificar a presença e localização de sítios de transcrição no núcleo.
Através da utilização de diferentes corantes de etiquetas de mRNAs de genes diferentes, pode-se quantificar a múltiplas espécies de mRNA nas mesmas células. Para demonstrar isto, as células de levedura foram incubadas na presença de factor-α e sorbitol. FUS1 transcrição (corante Quasar670, vermelho) é induzida por α-fator. Transcrição STL1 (tintura 570 Quasar, verde) é induzida pelo aumento da osmolaridade extracelular ( Figura 5). Figura 4 é um exemplo de FISH com as sondas cantor. Figura 5 é um exemplo de FISH com as sondas Stellaris.
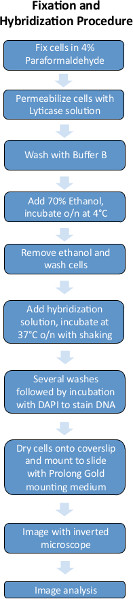
Figura 1. Esquema de procedimento experimental FISH. Clique aqui para ver a figura maior .

Figura 2. Esquemático do gasoduto de análise de imagem. Os pontos finais são determinados na figura da direita.
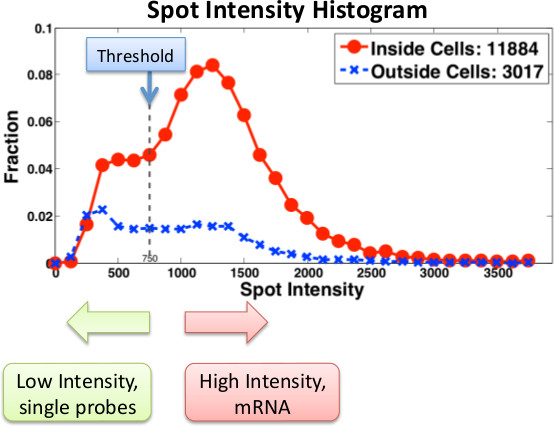
Figura 3. Histograma de intensidades local para um gene em particular usando sondas cantor . Intensidades de sonda são computados dentro (vermelho) e fora das células (azuis). Pontos de baixa intensidade são ou ruído ou sondas individuais. Mensagens de mRNA reais são rotulados com várias sondas. Ao usar sondas Stellaris, sondas individuais são menos detectáveis e, portanto, os limites e filtragem deve ser ajustada em conformidade.
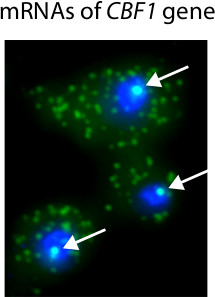
Figura 4. Os resultados representativos do procedimento FISH cantor. Nesta experiência, CBF1 transcrição é activada com um promotor indutível 18. Núcleos estão manchadas azul com DAPI. CBF1 mRNAs são marcados com sondas Cy3-marcadas. As setas brancas, evidenciar a presença de CBF1 sítios de transcrição no núcleo. Transcritos de mRNA individuais são visíveis no citoplasma.
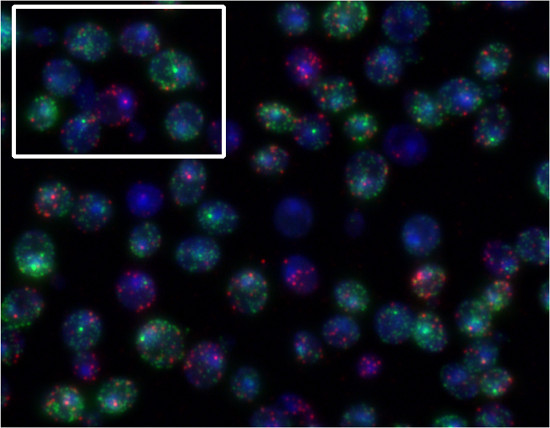
Figura 5. Os resultados representativos do procedimento FISH Stellaris. As células de levedura foram expostas simultaneamente Mata a 30 ng / ml de factor-α e 0,75 M de sorbitol durante 10 minutos e, simultaneamente, foram sondadas para FUS1 (Quasar 670, vermelho) e STL1 (Quasar 570, verde) transcrições. Na caixa de destaque, podemos ver uma célula responder apenas ao feromônio (FUS1 local de início, vermelho) e outro predominantemente respondendo ao sorbitol (local de início STL1, verde).
Discussão
Até à data, FISH tem sido principalmente um método de baixo rendimento. O uso de Cy3, Cy3.5, Cy5 e corantes limita o número de genes pode-se investigar em células individuais ou três de cada vez. Algumas sondas adicionais têm sido desenvolvidos (Stellaris), mas o número de sondas distinguíveis é ainda, no máximo sete. Para ultrapassar esta limitação, as estratégias de rotulagem combinatórias usando vários fluoróforos têm sido usados para criar os códigos de barras para as diferentes espécies de ARNm 19,20. Mais recentemente, Lubeck e Cai usado barcoding óptica e espectral para quantificar 32 espécies diferentes em simultâneo com FISH em células de levedura único 19. Uma das limitações dessa abordagem combinatória recente é que ele requer a utilização de microscopia de super-resolução. A análise necessária para distinguir as sondas de código de barras é também bastante complexa.
Descobrimos que Cy3 e Cy3.5 são preferíveis para Cy5 para experiências de FISH. Uma das limitações do corante Cy5 é a sua sensibilidadea fotodegradação. No entanto, Stellaris desenvolveu recentemente Cy5 variantes que são anunciados como mais resistentes à fotodegradação, e pode aliviar este problema técnico. É importante notar também que o peixe é um método caro de implementar e que tanto o cantor e Stellaris sondas normalmente custam US $ 700 - US $ 1.000 por conjunto da sonda, embora os preços para sondas disponíveis no mercado devem diminuir no futuro. Poupadores de reagentes e rotulagem eficiente traz sondas cantor até a faixa mais baixa de preço.
Um dos grandes desafios técnicos é a separação dos pontos de sonda única versus múltiplas, o que requer a implementação de sofisticados algoritmos de ponto-determinantes. Isso pode levar extensa revisão manual para ajustar os parâmetros de análise de imagem para montagens experimentais específicos. Um esboço de nosso pipeline computacional com funções MATLAB relevantes é fornecida na Seção 7 do protocolo. Esta questão é um pouco aliviado pelas sondas Stellaris que apenas têm uma etiqueta por sonda. É, portanto, requer a co-localização de várias sondas para ver um sinal.
Porque FISH requer a fixação de células, isso não facilitar o rastreamento de células individuais ao longo do tempo. Anteriormente, foram utilizados dados instantâneos peixe para reconstruir a dinâmica da expressão gênica em metabolicamente populações de leveduras de ciclismo individuais 6. Ciclismo metabólica é observada no pré-esfomeados, culturas contínuas, e é caracterizada por em toda a população oscilações coletivas no consumo de oxigênio. Essas oscilações estão associadas com o genoma de toda oscilações de transcrições que ocorrem por meio de todos os genes de leveduras em diferentes fases do consumo de oxigênio. Buscou-se determinar se ciclismo metabólica esteve presente em culturas de leveduras não sincronizadas contínuas. Se estiverem presentes, os transcritos que são anti-correlacionados em populações síncronas também deve ser anti-correlacionados em células individuais não sincronizadas, e vice-versa para as transcrições de correlacionadas.
ent "> Para reconstruir dinâmica da produção de ARNm de tempo, os dados observados instantâneo deve ser comparado com o que é esperado a partir de um modelo de comportamento subjacente. existem limitações teóricas ao quando tais" instantâneos "de dados de expressão de genes podem ser usadas para determinar a dinâmica de expressão de genes subjacentes e que tipos de modelos podem ser distinguidos 21. Pelos dados do ciclo metabólicos, ao invés de mostrar diretamente a presença de oscilações temporais, medidas estatísticas foram implementadas para comprovar que realmente existe uma célula de programa oscilatório autônoma consistente com microarray massa medições.Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Esta pesquisa foi suportada por concessões GM046406 (a DB) e pelo Instituto Nacional de General Medical Sciences Center para Quantitative Biology (GM071508). RSM reconhece financiamento do NSF Graduate Research Fellowship. MNM é apoiado por um Lewis-Sigler Fellowship. Gostaríamos de agradecer os membros do laboratório Botstein para discussões úteis e ex-membros Allegra Petti e Nikolai Slavov por suas contribuições para o projeto ciclo metabólico. Agradecemos a Daniel Zenklusen e Robert Singer para obter nós começou com o método FISH.
Materiais
| Name | Company | Catalog Number | Comments |
| Vanadyl Ribonucleoside Complex | NEB | S1402S | |
| Lyticase | Sigma | L5263 | |
| E. coli tRNA | Roche | 1010954001 | |
| BSA (RNase free) | Ambion | ||
| Beta-mercapt–thanol | Fisher | 03446l | |
| DAPI, dilactate | Sigma | D9564 | |
| PBS 10X (RNase free) | Ambion | AM9624 | |
| Triton X-100 | Shelton Scientific | ||
| Dextran sulfate | Sigma | D6001 | Or equivalent |
| Saline-sodium citrate (SSC) 20X | VWR | 82021-484 | |
| Formamide (deionized) | Ambion | AM9342 | |
| Nuclease-free water | Ambion | AM9932 | |
| Alpha-D-glucose | Sigma | 158968 | For GLOX solution |
| 1 M Tris-HCl, pH 8.0 | Ambion | AM9855G | |
| 100% Ethanol | |||
| Glucose oxidase | Sigma | G0543 | For GLOX solution |
| Catalase | Sigma | C3155 | For GLOX solution |
| Concanavalin A | MP Biomedicals | 150710 | |
| Polylysine (0.01%) | Sigma | P8920 | |
| Coverslips | Warner Instruments | Cs-18R15 | |
| Prolong Gold Mounting Medium | Invitrogen | P36934 | |
| QIAquick Nucleotide Removal Kit | QIAGEN | 28304 | |
| FISH Probes | Biosearch Technologies | Custom order for your desired mRNA sequence | |
| Glass bottom 96-well plates | Nunc | 265300 | Alternative to coverslips |
| 12-well plates | BD Falcon | 351143 | |
| Cy3, Cy3.5, Cy5 dyes | GE Healthcare | monofunctional NHS-ester | |
| EQUIPMENT | |||
| Plasma-Preen I Cleaner | Terra Universal | 9505-00 | Controller (Cat #9505-17 optional) |
| Vacuum Pump | Alcatel | 205SDMLAM | For operating Plasma-Preen |
| Widefield Fluorescence Microscope | Olympus | IX81 | Or equivalent |
| 100X objective | Olympus | 1-UB617R | |
| Light Source | X-Cite | XCT 10-A | Or equivalent |
| Filter Sets | Chroma | U-NSP100V2-SPR, U-NSP101V2-SPR, U-NSP102V2-SPR, U-NSP103V2-SPR,U-NSP104V2-SPR. | |
| Cooled CCD or EMCCD Camera | Hamamatsu | C4742-98-24ER | |
Referências
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of single RNA transcripts in situ. Science. 280, 585-590 (1998).
- Gall, J. G., Pardue, M. L. Formation and Detection of Rna-DNA Hybrid Molecules in Cytological Preparations. Proceedings of the National Academy of Sciences of the United States of America. 63, 378 (1969).
- Jones, K. W. Chromosomal and Nuclear Location of Mouse Satellite DNA in Individual Cells. Nature. 225, 912 (1970).
- Bauman, J. G., Wiegant, J., Borst, P., van Duijn, P. A new method for fluorescence microscopical localization of specific DNA sequences by in situ hybridization of fluorochromelabelled RNA. Exp Cell Res. 128, 485-490 (1980).
- Singer, R. H., Ward, D. C. Actin gene expression visualized in chicken muscle tissue culture by using in situ hybridization with a biotinated nucleotide analog. Proc. Natl. Acad. Sci. U.S.A. 79, 7331-7335 (1982).
- Silverman, S. J., et al. Metabolic cycling in single yeast cells from unsynchronized steady-state populations limited on glucose or phosphate. Proc. Natl. Acad. Sci. U.S.A. 107, 6946-6951 (2010).
- Trcek, T., Larson, D. R., Moldon, A., Query, C. C., Singer, R. H. Single-molecule mRNA decay measurements reveal promoter- regulated mRNA stability in yeast. Cell. 147, 1484-1497 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Mol Cell. 2, 437-445 (1998).
- Gandhi, S. J., Zenklusen, D., Lionnet, T., Singer, R. H. Transcription of functionally related constitutive genes is not coordinated. Nat. Struct. Mol. Biol. 18, 27-34 (2011).
- Itzkovitz, S., et al. Single-molecule transcript counting of stem-cell markers in the mouse intestine. Nature Cell Biology. 14, 106-U193 (2012).
- Raj, A., Rifkin, S. A., Andersen, E., van Oudenaarden, A. Variability in gene expression underlies incomplete penetrance. Nature. 463, 913-U984 (2010).
- Raj, A., Tyagi, S. Detection of individual endogenous RNA transcripts in situ using multiple singly labeled probes. Methods Enzymol. 472 (10), 365-386 (2010).
- Trcek, T., et al. Single-mRNA counting using fluorescent in situ hybridization in budding yeast. Nature Protocols. 7, 408-419 (2012).
- Raj, A., vanden Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5, 877-879 (2008).
- Otsu, N. A Tlreshold Selection Method from Gray-Level Histograms. IEEE Transactions on Systems, Man and Cybernetics. 9, 62-66 (1979).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophys J. 82, 2775-2783 (2002).
- McIsaac, R. S., et al. Fast-acting and nearly gratuitous induction of gene expression and protein depletion in Saccharomyces cerevisiae. Molecular biology of the cell. 22, 4447-4459 (2011).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nature Methods. 9, 743-U159 (2012).
- Levsky, J. M., Shenoy, S. M., Pezo, R. C., Singer, R. H. Single-cell gene expression profiling. Science. 297, 836-840 (2002).
- Wyart, M., Botstein, D., Wingreen, N. S. Evaluating Gene Expression Dynamics Using Pairwise RNA FISH Data. Plos Computational Biology. 6, (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados