É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
A lignina Down-regulação da
Neste Artigo
Resumo
A interferência de RNA fita dupla (dsRNAi) técnica é empregada para baixo-regular a coenzima A redutase cinamoil milho gene (ZmCCR1) para diminuir o teor de lignina da planta. A lignina sub-regulação da parede da célula é visualizada por meio de análises microscópicas e quantificada pelo método de Klason. São analisadas as mudanças de composição em hemicelulose e celulose cristalina.
Resumo
Para facilitar a utilização de biomassa lignocelulósica como um recurso bioenergia alternativa, durante os processos de conversão biológica, um passo de pré-tratamento é necessário para abrir a estrutura da parede celular das plantas, o aumento da acessibilidade dos hidratos de carbono de parede celular. A lignina, um material de polifenóis presente em muitos tipos de parede celular, é conhecida por ser um obstáculo significativo para a enzima de acesso. Redução no teor de lignina para um nível que não interfira com a integridade estrutural e a defesa do sistema da planta pode ser um passo importante para reduzir os custos de produção de bioetanol. Neste estudo, foram geneticamente regulada para baixo um dos genes relacionados com a biossíntese da lenhina, cinamoil-CoA redutase (ZmCCR1) por meio de uma técnica de cadeia dupla de RNA de interferência. O constructo ZmCCR1_RNAi foi integrado no genoma do milho utilizando o método de bombardeamento de partículas. As plantas de milho transgénicas cresceram normalmente, em comparação com as plantas de controlo do tipo selvagem, sem noterfering com o crescimento da biomassa ou de mecanismos de defesa, com exceção da exibição de marrom-coloração em transgênicos plantas de folhas meados de costelas, cascas e caules. As análises microscópicas, em conjugação com o ensaio de histológica, revelou que as fibras esclerenquimáticas foliares foram desbastadas, mas a estrutura e tamanho de outros componentes principais do sistema vascular não foi alterada. O teor de lignina no milho transgénico foi reduzida em 7-8,7%, o teor de celulose cristalina foi aumentada em resposta a uma redução da lignina, hemicelulose e manteve-se inalterada. As análises podem indicar que o fluxo de carbono podem ter sido desviadas da biossíntese de lignina para a biossíntese de celulose. Este artigo traça os procedimentos utilizados para regular o teor de lignina em milho através de tecnologia RNAi, e as análises de composição da parede celular utilizada para verificar o efeito das modificações sobre a estrutura da parede celular.
Introdução
A produção de biocombustíveis a partir da biomassa lignocelulósica é altamente desejável, devido à sua abundância presente em os EUA 1, e, no caso de a colheita sustentável de resíduos agrícolas e florestais, a capacidade de não competir diretamente para terras agrícolas utilizadas para a produção de alimentos e rações animais. No entanto, ao contrário dos grãos de milho, que é a principal fonte de biocombustível momento gerado em os EUA, materiais lignocelulósicos são significativamente mais complexo e difícil de quebrar. Além do hidratos de carbono de cadeia longa, a celulose e hemicelulose, que são as principais fontes de açúcares durante a fermentação de materiais lignocelulósicos, muitos tipos de paredes de células de planta também contêm lenhina, um polímero que proporciona a força de fenilpropanóide, a defesa contra o ataque patogénico, e hidrofobicidade as paredes celulares. Embora necessários para o crescimento e sobrevivência da planta, lignina também apresenta uma barreira significativa para a conversão enzimática de sucesso da celulose e hemicelluperder para açúcares solúveis. Materiais com alto teor de lignina são geralmente materiais menos desejáveis, tanto para o biocombustível (através de vias de conversão biológica) e as indústrias de papel e celulose, devido aos impactos negativos sobre as características de processamento e qualidade do produto. Assim, a manipulação genética dos materiais vegetais para a redução de lignina a um nível que não interfira com a resistência estrutural das culturas e sistemas de defesa poderia ser importante para a redução de custos de produção, tanto para o biocombustível lignocelulósico e as indústrias de papel e celulose.
No milho (Zea mays), a lenhina é covalentemente reticulada a hemicelulose na parede celular primária através de ácido ferúlico e pontes diferulate 2. O complexo de lignina-hemicelulose se liga a microfibrilas de celulose por meio de ligações de hidrogénio, formando uma matriz complexa que confere integridade e resistência da parede celular secundária. A resistência mecânica das paredes das células de planta é fortemente determinada pelo tipo de lignin subunidades 3-5. Em estudos anteriores, alterando as proporções de subunidades lignina mostrou nenhuma tendência clara sobre a digestibilidade enzimática 6-11. No entanto, reduzir o teor de lignina apresentam geralmente uma melhoria em conversões 12,13 e pode ser a chave para o aumento da digestibilidade do material vegetal por enzimas hidrolíticas incluindo endocellulases, celobiohidrolases, e β-glucosidases 14.
A engenharia genética para regular o nível de transcritos de expressão tem sido amplamente praticado para melhorar as características de cultura. Técnicas avançadas, incluindo anti-sense 15 e co-supressão de 16 tecnologias, permitir eficaz infra-regulação de genes alvo. Completa gene knock-out também foi alcançado usando construções de genes que codificam RNA emendados-intron com uma estrutura hairpin 17. Além disso, uma dupla interferência de RNA encalhado (dsRNAi) técnica, ou seja, um poderoso e eficaz mídia expressão gênicator que funciona por um ou outro visando a degradação transcrição ou tradução repressão, fornece um meio potente para induzir uma ampla gama de efeitos de supressão do mRNA alvo 18. Técnicas de silenciamento de genes mostram várias limitações. Estas técnicas não são reguladas com precisão o nível de transcrição e pode causar efeitos de silenciamento inesperadas em outros genes homólogos.
Neste método, utilizou-se o bombardeamento de partículas para realizar a dsRNAi construções no genoma de milho. Até à data, uma vasta gama de espécies de plantas têm sido transformadas com sucesso usando bombardeamento de partículas, transformação mediada por Agrobacterium, electroporação, microinjecção e métodos. Na transformação genética de milho, o método de bombardeamento de partículas é vantajoso sobre todos os outros métodos, pois é o mais eficiente. O bombardeamento com partículas não é dependente de bactérias, de modo que o método é livre de restrições biológicas, tais como o tamanho dos genes, espécies de gene ouigin, ou o genótipo da planta. O sistema de entrega de transgene física permite alta de DNA de peso molecular e de múltiplos genes a ser introduzido no genoma da planta e, em certos casos em cloroplastos de alta eficiência de transformação 19. A redução de lignina no sistema vascular da folha no meio da nervura podem ser visualizados por microscopia electrónica de varrimento (SEM), o que é benéfico para a análise da topografia e composição das amostras.
Em plantas de milho, dois dos cinamoil-CoA redutase (ZmCCR1: X98083 e ZmCCR2: Y15069) os genes foram encontradas no genoma de milho 20. Cinamoil-CoA redutase catalisa a conversão dos ésteres hidroxicinamoil-CoA em aldeídos cinamilo. Escolhemos o gene ZmCCR1 para regular esta enzima, porque o gene é expresso em todos os tecidos lignifying. Os 523 nucleótidos no terminal 3 'do gene ZmCCR1 foram escolhidos para um dsRNAi construir porque as sequências parecia sermais diversificada em comparação com aqueles da ZmCCR2. Assim, a construção dsRNAi seria precisamente ligar apenas para ZmCCR1, evitando off-alvo silenciando 21. Um construto ZmCCR1_RNAi foi manipulada no ImpactVector1.1-tag sistema de expressão citoplasmática (IV 1,1) contendo o promotor específico de tecido verde, a ribulose-1, 5-carboxilase oxigenase-bisfosfato (RuBisCO).
Para estudar os efeitos da dsRNAi construção de plantas transgénicas, o teor de lenhina foram quantificadas. A medição de lenhina de Klason (ácido insolúvel) é conhecido por ser mais preciso em comparação com os métodos de quantificação de ácidos detergente lignina que solubilizam alguma da lenhina 22. Portanto, a lenhina de Klason foi medido em caules de milho transgénicas. Este procedimento consiste de uma hidrólise ácida de dois passos que converte os hidratos de carbono poliméricos solúveis em monossacarídeos 23. A biomassa hidrolisada foi então fracionado em materi solúveis e insolúveis ácidoals e da lignina insolúvel em ácido foi medida de acordo com estudos anteriores 23,24. Idealmente, a análise de lenhina deve incluir extracções com água e etanol antes do passo de hidrólise, a fim de remover materiais solúveis que podem interferir com os resultados, e uma combustão pós-hidrólise do resíduo de lenhina para contabilizar quaisquer cinzas presentes no resíduo. Sem estes passos, o teor de lenhina da amostra pode ser artificialmente. O método integral é aqui apresentado, no entanto, para as nossas experiências fomos capazes de realizar ambos os passos devido à pequena quantidade de material disponível para testes
Dois outros componentes da parede celular, celulose e hemicelulose foram também analisadas na lignina regulada linhagens de milho transgénico. Tem sido relatado que as plantas transgénicas que foram regulados negativamente em ambos os seus fenilalanina amónia-liase (PAL) 25, 4-cumarato: CoA ligase (4CL) 26, ou um cinamilolcohol desidrogenase (CAD) 27 mostram um aumento em outros componentes estruturais da parede celular. Como um primeiro passo para os nossos estudos, celulose cristalina foi medida usando o método Updegraff 28. Este método foi originalmente concebido para a determinação de celulose em um grande número de bactérias celulolíticas e fungos. Resumidamente, os estoques de milho moídos foram tratados com Updegraff reagente (ácido acético: ácido nítrico: água) para remover a hemicelulose, lignina, e xylosans. A celulose cristalina foi completamente hidrolisada em glicose através Saeman hidrólise por adição de H 2 SO 4. A celulose cristalina foi então analisado utilizando o método colorimétrico de antrona 29. Para verificar se o conteúdo de hemicelulose foram alterados, os extractos de monossacárido de talos moídos foram hidrolisadas utilizando ácido trifluoroacético, derivatizados utilizando o método de acetato de alditol e, em seguida, analisado por cromatografia gasosa (GC) de 30. Os procedimentos detalhados para cel cristalinaanálises de composição de conteúdo lose e polissacáridos de matriz são descritos em Foster et al. (2010) 31.
Aqui, descrevemos os procedimentos utilizados para a lignina baixa regulação no milho por meio de uma tecnologia de RNAi, transformação bombardeamento de partículas e análise de lignina para a desconstrução acelerada de milho biomassa lignocelulósica em açúcares fermentáveis para biocombustíveis.
Protocolo
1. Preparação de dsRNAi construções usadas para o Down-regulação da ZmCCR1
- Primers específicos do gene projeto, incluindo sítios de enzimas de restrição necessários para fazer uma dsRNAi construir a knock-out do gene ZmCCR1. Dois conjuntos de iniciadores foram concebidos para amplificar dois segmentos de fragmentos de cDNA ZmCCR1:. Um fragmento 523 bp a partir de nucleótidos 748-1271, e um fragmento de 285 pb a partir de nucleótidos 986-1271 O ZmCCR1 cDNA foi fornecido pelo Instituto Genoma Arizona (AGI). Mais detalhes são descritos na Figura 1.
- Amplificar o fragmento grande de polimerase de reacção em cadeia (PCR) a partir do modelo de cDNA usando os ZmCCR1 ZmCCR1_748F_BglII iniciadores (5'-AGATCTACATCCTCAAGTACCTGGAC-3 ') e ZmCCR1_1271R_NcoI (5'-CCATGGTTTACACAGCAGGGGAAGGT-3'). Amplificar o fragmento menor (285 pb), utilizando os iniciadores ZmCCR1_986F_BglII (5'-AGATCTGGAAGCAGCCGTACAAGTTC-3 ') e um ZmCCR1_1271R_SacI (5R17; GAGCTCTTTACACAGCAGGGGAAGGT--3 ').
- Individualmente ligar os fragmentos de em pGEM-T Easy, seguindo as instruções do fabricante.
- Realizar mini-preparação de plasmídeo de isolamento de ADN a partir de transformantes individuais, cada uma contendo o pGEM-T constrói usando um kit comercial plasmídeo mini-prep.
- Digerir tanto o pGEM-T :: ZmCCR1 (523 pb) e ImpactVector (IV) -1.1 (expressão citoplasma vetor) com tanto BglII e Ncol.
- Ligar o grande digerido purificado em gel ZmCCR1 fragmento (523 bp) em gel digerido purificado IV-1.1.
- Digerir o pGEM-T :: ZmCCR1 (285 pb) e IV-1.1 :: ZmCCR1 (523 pb) com ambos BglII e Saci, a fim de inserir o pequeno fragmento no IV-1.1 :: ZmCCR1 (523 pb).
- Ligar o pequeno fragmento purificado em gel digerido ZmCCR1 (285 bp) em gel digerido purificado IV-1.1 :: ZmCCR1 (523 pb).
- Clone ambos 523 pb e 285 pb fragmentaçãots em IV-1.1 para fazer o ZmCCR1 RNAi construção, que tem uma sequência de repetição invertida 285 pb 238 pb com um espaçador no meio dos fragmentos de repetição invertida (ver Figura 1).
- Transferir esta construção em Escherichia coli (E. coli), cultivá-los e efectuar um tamanho de isolamento de DNA de plasmídeo midi-prep para obter DNA de plasmídeo suficiente para a transformação genética de milho.
2. Milho Transformação Genética
- Preparação de Partículas de tungstênio
- Colocar 60 mg de esferas de tungsténio (M10) em um tubo de 1,5 ml e lava-se com 1 ml de etanol a 70% em vortex durante 2 min. Incubar durante 10 minutos a 23 ° C e depois centrifugar a 18,894 xg durante 2 minutos e descarta-se o sobrenadante.
- Lavar 3x com 1 ml de etanol 100%, centrifugando durante 2 min e rejeitando o sobrenadante. Adicionar 1 ml de glicerol a 50% estéril para levar a concentração de micropartículas até 60 mg / ml.
- Preparação de DNA para bombardeamento
- Place os 50 ul (3 mg) de esferas de tungsténio preparadas, em glicerol a 50% em um tubo de 1,5 ml. Adicionam-se 5 ul (1 ug) de IV-1.1 :: ZmCCR1 RNAi plasmídeo de DNA, 50 uL de 2,5 M de CaCl2, e 20 pi de espermidina 0,1 M. Vortex brevemente entre cada adição dos reagentes acima.
- Vortex o tungstênio-DNA talão mistura brevemente e centrifugar a 18.894 xg por 30 segundos. Decantar o sobrenadante e ressuspender as pelotas em 140 ul de etanol a 70%. Remover o líquido e descartar. Adicionar 140 ul de etanol a 100%. Remover o líquido e descartar.
- Adicionar 48 ul de etanol a 100%. Use imediatamente ou armazenar no gelo por até 4 horas antes da bombardeio.
- Bombardeamento
- Colocar 3-5 cm de diâmetro Hi-II de calos de milho embriogénico (fornecida a partir de milho Centro transformação de Iowa State University) no meio de 100 mm em placas de Petri contendo meios de comunicação N6OSM 32 (como osmotium) pelo menos 4 horas antes do bombardeamento.
- Prepare o dispositivo de entrega de partículas PSD-1000/He de acordo com as instruções do fabricante 33.
- Esterilizar parede da câmara com etanol 70%. Carregar estéril disco de ruptura de 650 psi em cap retenção estéril. Espalhe 5-6 ul da solução de M10-ADN para a superfície de um microtransportador, brevemente seco. Carga microtransportador e parando tela em microcarregador montagem lançamento.
- Coloque microcarregador montagem lançamento e calos de milho em câmara a uma distância selecionada a partir da tela de parada (L2 = 6 cm) e feche a porta. Acelerar a um vácuo de 27 psi contra uma tela de malha de arame.
- Pressione o botão de fogo até rajadas de disco de ruptura e pressão de hélio indicador cai para zero. Solte o botão de fogo.
- Incubar os calos bombardeados numa placa de Petri contendo N6OSM (meio osmótico) 32 durante 16 horas no escuro a 27 ° C. Quebre o calos em cerca de dez peças e transferir para N6E (meio de indução de calos) 32 em placas de Petri e incubar durante 5 dias in escuro a 27 ° C.
- Seleção
- Depois de 5 dias em N6E, transferir os calos em meio N6S (mídia seleção) 32. Subcultura todos os calos em meio de selecção, a cada 30 dias durante 8-12 semanas, sem perturbar a estrutura de formação de calos.
- Depois de cerca de 8-10 semanas, branco sectores de rápido crescimento vai crescer fora da não-proliferação e calos mãe parcialmente necrótica. Especial sobre o consumo dos tecidos de crescimento rápido brancos e subcultura-los a médio seleção fresco (N6S) 32 e continuar a incubar como acima.
- Regeneração
- Transferir os calos embrionário branco e de crescimento rápido em meio de regeneração e incubar 32 como acima, durante 1 semana. Desligue o calos embriogênicos regenerar a um período de 16 horas luz do dia e 8 horas escuro, a 25-27 ° C
- Transfira a regeneração atira para o enraizamento médio de 32 em um tubo de ensaio de vidro após 3-4 semanas, continuam a incubar como acima. Após substancial rdesenvolvimento oot aparece, lavar cuidadosamente as raízes sob a água da torneira, depois transplantar as mudas para 4 "vasos com terra. Cubra os vasos com sacos plásticos para manter úmida. Após 2 dias fazer pequenos furos os sacos de plástico. Depois de 5-6 dias remover os sacos de plástico. Continue-se a incubar conforme acima por mais 5-6 dias.
- Estufa
- Transfira as mudas em vasos de 18 "com o solo e manter sob a luz solar ou à luz verão cheio de efeito estufa. As plantas regeneradas iniciais são chamados t 0, enquanto as primeiras sementes pertencem à geração T 1.
3. Ensaio histológica
- Corrigir os de milho folha meados da década de costelas em 5 ml de 10% formalina tamponada neutra.
- Processo e vácuo infiltrar com parafina com um processador de tecido usando um processador de tecidos.
- Incorporar os tecidos em parafina usando uma estação de incorporação HistoCentre III.
- Remover o excesso de parafina a partir das bordas, uma vez blocoks são arrefecidos.
- Seção amostra em 4-5 mM com um micrótomo usando um micrótomo.
- Coloque secções sobre lâminas de microscópio e seco em uma incubadora a 56 ° C durante 2-24 horas. Fazer seções certeza são totalmente aderida ao slide.
- Retire a parafina secções em duas mudanças de xileno durante 5 min a 23 ° C.
- Hidrato desliza através de duas mudanças de etanol a 100% durante 2 min e duas mudanças de etanol a 95% durante 2 minutos a 23 ° C.
- Lavar as seções em água corrente por 2 min.
- Mancha com 0,05% de toluidina O azul para 1-2 minutos e enxaguar rapidamente com DDH 2 O.
- Coloque uma lamela sobre as amostras com óleo de imersão e visualização com microscopia de luz.
- Microscopia Eletrônica de Varredura (MEV)
- Fixar as folhas de milho meados nervuras transversas em 4% de glutaraldeído e 0,1 M de tampão fosfato de sódio (pH 7,4) a 4 ° C durante 1-2 horas.
- Resumidamente enxaguar as amostras do tampão, desidratou-los numaséries de etanol (25%, 50%, 75% e 95%) durante 10-15 minutos em cada gradação e etanol a 100% durante 10 min, 3x.
- Seque as folhas de milho meados costelas seccionadas-cross desidratadas em um secador de ponto crítico com dióxido de carbono líquido como fluido de transição.
- Monte as amostras secas em topos de alumínio usando guias de carbono de alto vácuo
- Revista as folhas de milho meados costelas montadas em bases de alumínio com ouro (cerca de 20 nm de espessura) em um coater pulverização catódica purgado com gás argônio.
- Examine as amostras revestidas em um JEOL JSM-6400V (lantânio hexaboreto emissor de elétrons) microscópio eletrônico de varredura.
- As imagens digitais foram fotografados usando um software de análise Pro (versão 3.2).
4. Klason lignina Medição
- Moinho das amostras através de uma tela de 2 mm.
- Use um analisador de umidade para determinar o conteúdo de cada amostra de umidade e gravar o valor.
- Pesar ~ 1,5 g de cada amostra e registrar a massa. Extrair as amostras usando água para a primeira extracção, seguida de etanol para a segunda extracção usando um solvente de extracção automática (3 ciclos por extracção, ~ 14 minutos por ciclo) ou aparelho de Soxhlet (8 horas por extracção). (Nota: Esta etapa remove extrativos que podem condensar durante a hidrólise ácida e interferir com a medição precisa de lignina, aumentando o teor de lignina Klason aparente.)
- Seque as amostras extraídas a 45 ° C durante a noite, em seguida, deixe esfriar no dessecador, e pesar novamente.
- Definir uma incubadora a 30 ° C. Meça 0,3 g de cada amostra seca, extraído em tubos de alta pressão com tampa (triplicatas por amostra é recomendado) e registrar os pesos para a aproximação de 0,1 mg. Adicionar 3 ml de 72% de H 2 SO 4 a cada tubo de pressão.
- Misturar a amostra usando um copo ou vara Teflon mexa. Deixe a vara agitação no tubo até que a água é adicionado após incubação.
- Coloque os frascos em uma incubadora set para 30 ° C e 150 rpm durante 60 min. Depois de 1 hora adicionar 84 ml de água desionizada, para diluir a concentração de ácido de 4% e misturar-se com a vareta de agitação. Ter cuidado para não deixar grandes quantidades de amostra sobre os lados do frasco acima da linha de água.
- Fortemente selar as rolhas em todos os frascos e colocá-los em um rack de metal ou de grandes copos. Autoclave a 121 ° C por meio de um ciclo de esterilização líquido, durante 1 hora. Deixar arrefecer à temperatura ambiente antes de abrir.
- Pré-ash os cadinhos de filtragem em um forno a 575 ° C durante pelo menos 4 horas. Permitir cadinhos a arrefecer-se num exsicador durante pelo menos uma hora.
- Aspirador de filtrar a solução a partir de cada tubo através de um cadinho separado, usando um adaptador de borracha para fixar o cadinho. Use água deionizada para enxaguar todas as partículas restantes do tubo.
- Seca-se o resíduo de lenhina a 105 ° C durante um mínimo de 4 h. Registar o peso do cadinho e seco resíduo.
- Se estiver usando um 575 ° C, pré-Fogo as amostras mais de um bico de Bunsen até que não haja fumaça ou cinzas e, em seguida, colocar no forno por 24 horas, ou se estiver usando um forno programável, não pré-cinzas e usam o seguinte programa:
- Rampa desde a temperatura ambiente até 105 ° C e manter por 12 minutos.
- Rampa até 250 ° C a 10 ° C / min e mantenha durante 30 minutos.
- Rampa até 575 ° C a 20 ° C / min e manter durante pelo menos 180 min.
- Retirar os cadinhos a partir do forno e arrefece-se num exsicador. Pesar o cadinho e cinzas.
- Calcula-se o resíduo insolúvel ácido usando a seguinte equação:
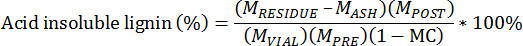
M PRE = Massa da biomassa pré-extraído
M POST = Massa da biomassa pós-extraído
M VIAL = Massa de biomassa extraída adicionada ao frasco
M = Massa RESÍDUO de resid cadinho e ligninaue
M = Massa ASH de cadinho e cinzas
MC = teor de umidade da biomassa pré-extraído, base de peso total
5. Análise Carboidratos
- Realize parede celular de carboidratos analisa baseado no Foster et al. (2010) Protocolo de 31. Em resumo, Prepare álcool resíduo insolúvel de liofilizado material vegetal. Em seguida hidrolisar o material com ácido trifluoroacético e combinar os derivados de monossacáridos solubilizados para os seus acetatos de alditol correspondentes. Analisar estes derivados voláteis por cromatografia gasosa (GC) conectada a um espectrômetro de massa quádruplo.
Resultados
Nós demonstramos uma redução do teor de lignina em plantas de milho via RNAi. O método de transformação de bombardeamento de partículas produziram cerca de 30% de eficiência trnasformation. O silenciamento de genes de ZmCCR1 foi observado de forma consistente em gerações T0-T2. A lignina reduzida transgênicos cresceu de forma semelhante a plantas de milho de tipo selvagem, exceto para a exibição de coloração marrom na folha mid-rib, casca e tronco. O ensaio histológico mostrou que as linhas muta...
Discussão
A acessibilidade de celulases microbianas para plantar polissacáridos da parede celular está em grande parte dependente do grau ao qual estão associados com polímeros fenólicos 23. A taxa de conversão de biomassa lignocelulósica para fermentáveis açúcar está negativamente correlacionada com o teor de lignina depositado na parede celular secondadry plantas. Esta correlação é atribuída às propriedades físicas da lignina tais como hidrofobicidade 24, heterogeneidade química, e ...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
A imagem microscópica foi realizada através dos serviços do Estado Centro Universitário Michigan for Advanced Microscopia. Milho calo foi comprado a partir do Centro de Transformação de milho de Iowa State University. Os autores gostariam de agradecer Jeffrey R. Weatherhead do Laboratório de Pesquisa de Plantas MSU para a sua assistência técnica na análise de carboidratos. Esta pesquisa foi generosamente financiada pelo Programa Milho de Marketing de Michigan (CMPM) eo Consórcio para Plant Biotechnology Research (CPBR).
Materiais
| Name | Company | Catalog Number | Comments |
| N6OSM (Osmotic medium) | Made in-house | ||
| N6E (Callus induction) | Made in-house | ||
| N6S media (Selection media) | Made in-house | ||
| Regeneration medium | Made in-house | ||
| Rooting medium | Made in-house | ||
| 10% Neutral buffered formalin (1 L) | Made in-house | ||
| Bio-Rad PSD-1000/He Particle Delivery device | Hercules, CA, United States | ||
| Zeiss PASCAL confocal laser scanning microscope | Carl Zeiss, Jena, Germany | For brightfield microscopy, the images were recorded using a Zeiss (Jena, Germany) PASCAL confocal laser scanning microscope with a 488 nm excitation mirror, a 560 nm emission filter, and a 505-530 nm emission filter. Image analysis was performed using Laser scanning microscope PASCAL LSM version 3.0 SP3 software. | |
| Excelsior ES Tissue Processor | Thermo Scientific, Pittsburgh, PA, United States | ||
| HistoCentre III Embedding Station | Thermo Scientific, Pittsburgh, PA, United States | ||
| Microtome Model Reichert 2030 | Reichert, Depew, NY, United States | ||
| Emscope Sputter Coater model SC 500 | Ashford, Kent, England | ||
| JEOL JSM-6400V Scanning Electron Microscope | JEOL Ltd., Tokyo, Japan | ||
| Fitzpatrick JT-6 Homoloid mill | Continental Process Systems, Inc., Westmont, IL | ||
| MA35 Moisture Analyzer | Sartorius | ||
| Critical point dryer, Balzers CPD | Leica Microsysstems Inc, Buffalo Grove, IL, United States | ||
| Screw-top high pressure tubes | Ace Glass, Vineland, NJ | #8648-27 | |
| Screw-top high pressure tube plugs | Ace Glass, Vineland, NJ | #5845-47 |
Referências
- Ralph, J., Grabber, J. H., Hatfield, R. D. Lignin-ferulate cross-links in grasses - Active incorporation of ferulate polysaccharide esters into ryegrass lignins. Carbohydrate research. , 275-178 (1995).
- Park, S. -. H. . Expediting cellulosic biofuels agenda: Production of high value-low volume co-products and lignin down-regulation of bioenergy crops [Ph.D. thesis]. , (2011).
- Boerjan, W., Ralph, J., Baucher, M. Lignin biosynthesis. Annual review of plant biology. 54, 519-546 (2003).
- Gibson, L. J. The hierarchical structure and mechanics of plant materials. Journal of the Royal Society, Interface / the Royal Society. 9, 2749-2766 (2012).
- Dien, B. S., et al. Enhancing alfalfa conversion efficiencies for sugar recovery and ethanol production by altering lignin composition. Bioresource technology. , 102-6486 (2011).
- Fu, C. X., et al. Downregulation of Cinnamyl Alcohol Dehydrogenase (CAD) Leads to Improved Saccharification Efficiency in Switchgrass. Bioenerg Res. 4, 153-164 (2011).
- Grabber, J. H., Ralph, J., Hatfield, R. D., Quideau, S. p-hydroxyphenyl, guaiacyl, and syringyl lignins have similar inhibitory effects on wall degradability. Journal of agricultural and food chemistry. 45, 2530-2532 (1997).
- Li, X., et al. Lignin monomer composition affects Arabidopsis cell-wall degradability after liquid hot water pretreatment. Biotechnology for biofuels. 3, (2010).
- Mansfield, S. D., Kang, K. Y., Chapple, C. Designed for deconstruction--poplar trees altered in cell wall lignification improve the efficacy of bioethanol production. The New phytologist. 194, 91-101 (2012).
- Studer, M. H., et al. Lignin content in natural Populus variants affects sugar release. Proceedings of the National Academy of Sciences of the United States of America. 108, 6300-6305 (2011).
- Chen, F., Dixon, R. A. Lignin modification improves fermentable sugar yields for biofuel production. Nature. 25, 759-761 (2007).
- Ziebell, A., et al. Increase in 4-coumaryl alcohol units during lignification in alfalfa (Medicago sativa) alters the extractability and molecular weight of lignin. The Journal of biological chemistry. 285, 38961-38968 (2010).
- Park, S. -. H., et al. The quest for alternatives to microbial cellulase mix production: corn stover-produced heterologous multi-cellulases readily deconstruct lignocellulosic biomass into fermentable sugars. Journal of Chemical Technolog., and Biotechnology. 86, 633-641 (2011).
- Mol, J. N., et al. Regulation of plant gene expression by antisense RNA. FEBS letters. 268, 427-430 (1990).
- Adamo, A., et al. Transgene-mediated cosuppression and RNA interference enhance germ-line apoptosis in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 109, 3440-3445 (2012).
- Smith, N. A., et al. Total silencing by intron-spliced hairpin RNAs. Nature. 407, 319-320 (2000).
- Park, S. -. H., et al. Downregulation of Maize Cinnamoyl-Coenzyme A Reductase via RNA Interference Technology Causes Brown Midrib and Improves Ammonia Fiber Expansion-Pretreated Conversion into Fermentable Sugars for Biofuels. Crop Sci. 52, 2687-2701 (2012).
- Altpeter, F., et al. Particle bombardment and the genetic enhancement of crops: myths and realities. Mol Breeding. 15, 305-327 (2005).
- Pichon, M., Courbou, I., Beckert, M., Boudet, A. M., Grima-Pettenati, J. Cloning and characterization of two maize cDNAs encoding cinnamoyl-CoA reductase (CCR) and differential expression of the corresponding genes. Plant molecular biology. 38, 671-676 (1998).
- Mansoor, S., Amin, I., Hussain, M., Zafar, Y., Briddon, R. W. Engineering novel traits in plants through RNA interference. Trends in plant science. 11, 559-565 (2006).
- Hatfield, R. D., Jung, H. -. J. G., Ralph, J., Buxton, D. R., Weimer, P. J. A comparison of the insoluble residues produced by the Klason lignin and acid detergent lignin procedures. J Sci Food Agr. 65, 51-58 (1994).
- Sluiter, J. B., Ruiz, R. O., Scarlata, C. J., Sluiter, A. D., Templeton, D. W. Compositional analysis of lignocellulosic feedstocks. 1. Review and description of methods. Journal of agricultural and food chemistry. 58, 9043-9053 (2010).
- Sluiter, A., Hames, B., Ruiz, R., Scarlata, C., Sluiter, J., Templeton, D., Crocker, D. Determination of structural carbohydrates and lignin in biomass. Laboratory Analytic Procedure. , (2008).
- Bate, N. J., et al. Quantitative Relationship between Phenylalanine Ammonia-Lyase Levels and Phenylpropanoid Accumulation in Transgenic Tobacco Identifies a Rate-Determining Step in Natural Product Synthesis. Proceedings of the National Academy of Sciences of the United States of America. 91, 7608-7612 (1994).
- Hu, W. J., et al. Repression of lignin biosynthesis promotes cellulose accumulation and growth in transgenic trees. Nature. 17, 808-812 (1999).
- Lapierre, C., et al. Signatures of cinnamyl alcohol dehydrogenase deficiency in poplar lignins. Phytochemistry. 65, 313-321 (2004).
- Updegraff, D. M. Semimicro determination of cellulose in biological materials. Anal Biochem. 32, 420-424 (1969).
- Yemm, E. W., Willis, A. J. The estimation of carbohydrates in plant extracts by anthrone. The Biochemical journal. 57, 508-514 (1954).
- Filomena, A. P., Cherie, W., Geoffrey, B. F., Antony, B. Determining the polysaccharide composition of plant cell walls. Nature. 7, 1590-1607 (2012).
- Foster, C. E., Martin, T. M., Pauly, M. Comprehensive Compositional Analysis of Plant Cell Walls (Lignocellulosic biomass) Part II: Carbohydrates. J. Vis. Exp. (e1837), (2010).
- Department of Agronomy, Iowa State University. Particle bombardment of Hi II immature zygotic embryos and recovery of transgenic maize plants. , (2005).
- Cano-Delgado, A., Penfield, S., Smith, C., Catley, M., Bevan, M. Reduced cellulose synthesis invokes lignification and defense responses in Arabidopsis thaliana. The Plant journal : for cell and molecular biology. 34, 351-362 (2003).
- Boudet, A. M., Kajita, S., Grima-Pettenati, J., Goffner, D. Lignins and lignocellulosics: a better control of synthesis for new and improved uses. Trends in plant science. 8, 576-581 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados