É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Rotulagem específica de seqüência de ácidos nucléicos e proteínas com Metiltransferases e cofator Analogues
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
DNA e as proteínas são seqüência especificamente marcado com afinidade ou grupos repórter fluorescentes de DNA ou proteína methyltransferases e análogos sintéticos cofactor. Dependendo da especificidade do cofactor de enzimas, aziridina ou duplas análogos de cofactores activados são empregues para marcação de uma ou duas etapas.
Resumo
-Adenosyl S-L-metionina (AdoMet ou SAM) metiltransferases dependentes de (MTAse) catalisam a transferência do grupo metilo de AdoMet activado a posições específicas no ADN, ARN, proteínas e pequenas biomoléculas. Esta reacção de metilação pode ser natural, expandida para uma grande variedade de reacções de alquilação usando análogos sintéticos de cofactor. A substituição do centro reactivo da AdoMet sulfónio com um anel aziridina leva à co-factores que podem ser acoplados com ADN por vários MTases ADN. Estes cofactores de aziridina pode ser equipado com grupos repórter em diferentes posições a porção de adenina e usado para S-specific equence M ethyltransferase- I nduced ing L abel de DNA (DNA de sorriso). Como um exemplo típico, damos um protocolo para a biotinilação de DNA de plasmídeo pBR322 na sequência 5'-ATCG A T-3 'com o ADN MTase M.BseCI e o cofactor aziridina em 6BAzum passo. Extensão do grupo metil ativado com insaturados grupos alquilo resulta em uma outra classe de análogos AdoMet que são utilizados para dirigida por ethyltransferase m T ransferência de A ctivated rupos G (mTAG). Uma vez que as cadeias laterais estendidas são activados pelo centro de sulfónio e a ligação insaturada, estes cofactores são chamados análogos AdoMet dupla activada. Estes análogos não só funcionam como co-factores para MTases ADN, como os cofactores de aziridina, mas também para ARN, proteínas e pequenas MTases molécula. Eles são tipicamente usados para a modificação enzimática da MTAse substratos com grupos funcionais únicos que são marcados com grupos repórter num segundo passo químico. Isso é exemplificado em um protocolo para a rotulagem de fluorescência da proteína histona H3. Um pequeno grupo propargilo é transferido do SeAdoYn cofactor análogo à proteína pela lisina histona H3 4 (H3K4) MTase SET7 / 9 seguido por clique rotulagem dohistona H3 alkynylated com TAMRA azida. Rotulagem MTase mediada com análogos cofactor é uma tecnologia capacitadora para muitas aplicações interessantes, incluindo a identificação e estudo funcional de substratos MTase bem como genotipagem de DNA e detecção de metilação.
Introdução
Rotulagem específica de ácidos nucléicos e proteínas 1,2 3,4 é de grande interesse para caracterizações funcionais, diagnóstico médico e (nano) biotecnologia. Aqui apresentamos um método de marcação enzimática para esses biopolímeros que é baseada no S -adenosyl-l-metionina (AdoMet ou SAM) methyltransferases -dependentes (MTases). Esta classe de enzimas (EC 2.1.1.) Tem como alvo as posições individuais nucleofílicos (azoto, oxigénio, enxofre e átomos de carbono) no interior resíduos específicos de ácidos nucleicos e proteínas e naturalmente transfere o grupo metilo activado da AdoMet cofactor (Figura 1A) 5. Além disso, pode utilizar MTases análogos sintéticos de cofactor para marcação específica com fluoróforos, marcadores de afinidade ou outras etiquetas (Figura 1B) 6. Duas classes de análogos da AdoMet foram desenvolvidos: cofactores aziridina para M ethyltransferase- específico equence S-I Nduced L abel ING (sorrindo) 7 e duplos análogos ativados AdoMet para dirigida por ethyltransferase m T ransferência de A ctivated rupos G (mTAG) 8.

Figura 1: As reacções catalisadas por metiltransferases (MTases) de transferência de grupo A. Metil do cofactor naturais AdoMet (SAM) a vários substratos, incluindo ADN, ARN, proteínas e pequenas biomoléculas B. Etiquetagem / funcionalização de ácidos nucleicos e proteínas (NNNNN. =. pares de bases de DNA, nucleotídeos de RNA para ácidos e aminoácidos das proteínas; XXXXX = sequência de reconhecimento do MTase com resíduo alvo em verde) com análogos cofactor sintéticos. Cofatores Aziridine contendo um grupo repórter (esfera azul)ligado ao anel de adenina são especificamente sequência acoplado com o resíduo alvo (esquerda) e análogos dupla activada AdoMet conduzir à transferência de cadeias alquilo que transportam um repórter estendidos química Y (direita), que podem ser marcados por reacção bioorthogonal clique num segundo passo. Por favor, clique aqui para ver uma versão maior desta figura.
Cofatores Aziridine trabalhar melhor com DNA MTases. Eles contêm um anel de três membros, com um átomo de azoto, 9 (ou um seu N mostarda 10,11) em vez do centro do grupo de sulfónio como reactivo. A protonação do átomo de azoto presente no anel de aziridina activa para o ataque nucleofílico pelo nucleotídica alvo que leva para o acoplamento covalente de todo o co-factor com ADN. Ao ligar-se grupos repórter para o anel de adenina os cofactores de aziridina pode ser utilizado em combinação com DNA para rotular MTases ADN em um passo ( g> Figura 1B, à esquerda) 7,12. Isto é demonstrado em detalhe para a biotinilação de ADN com 6BAz 13-15 (cofactor aziridina com biotina ligada à posição 6 do anel de adenina) e a adenina-specific DNA MTase de Bacillus stearothermophilus (M.BseCI) 16 (Figura 2, consulte a seção de protocolo 2: rotulagem de uma etapa de DNA via aziridina cofatores). Além M.BseCI ('sequência de reconhecimento, o MTases ADN de Thermus aquaticus (M.TaqI, 5'-TCG Um -3 5'-ATCG A T-3)'), a partir de Haemophilus heamolyticus (M.HhaI, 5 '-G C GC-3') e de Spiroplasma (M.SssI, 5'-G C-3 ') foram usados com sucesso para biotinilar ADN com 6BAz 17. Além disso, co-factores de aziridina pode ser empregue para um passo de ADN de fluorescência rotulagem 18,19.
ontent "fo: manter-together.within-page =" always ">
Figura 2:. Sequência de biotinilação específica de um passo de ADN com M.BseCI e 6BAz O ADN MTase M.BseCI reconhece a sequência de ADN de cadeia dupla 5'-ATCG A T-3 'e naturalmente metila o grupo amino da segunda adenina resíduo (verde) usando AdoMet. Com o co-fator aziridine 6BAz o curso da reacção é alterada e M.BseCI leva para sequenciar biotinila�o DNA específico acoplando todo o cofator incluindo biotina (azul) com a adenina alvo. Por favor, clique aqui para ver uma versão maior desta figura.
Duplos análogos AdoMet activados contêm estendido cadeias laterais insaturadas em vez de um grupo metilo no centro de sulfónio (Figura 1B , direita) 20. A ligação dupla ou tripla insaturada em β-position para o centro da sulfonium compensa eletronicamente efeitos estéricos desfavoráveis no interior do estado de transição de estabilização conjugative. Uma vez que tanto o centro de sulfonium ea ligação insaturada ativar a cadeia lateral para a transferência enzimática, estes cofatores foram nomeados análogos AdoMet double-ativadas. Tipicamente, eles são utilizados para transferir as cadeias laterais com grupos repórteres únicos químicos (químicos), como grupos amino, alquino e azida, para rotulagem quimio-selectiva numa segunda etapa 8,21. Em geral, os análogos dupla ativada AdoMet pode não só funcionam como co-fatores para DNA MTases 8,20,21, mas também para RNA MTases 22,23 e MTases proteína 24-28 permitindo rotulagem adicional de RNA e proteínas. No entanto, as cadeias laterais estendidas estão estericamente mais exigente do que um grupo metilo e alargando os locais activos MTAse por engenharia de proteínas é frequentementeen necessária para a obtenção de taxas de transferência eficientes. Outra solução para este problema é a utilização de um análogo AdoMet com um grupo pequeno propargilico (três carbonos), onde o terminal de alcino tem duas funções: 1. Estabilização do estado de transição durante a transferência enzimática e 2. alça reativos para seguir modificações químicas por cobre- catalisada cicloadi�o azida-alcino (CuAAC) clique química. Descobriu-se que o resultante propargílica AdoMet análogo 29 é bastante instável em condições neutras ou ligeiramente básicas e somente de uso limitado. Este inconveniente pode ser corrigido, substituindo o átomo de enxofre com selênio. O cof actor resultante 5 '- [(Se) [(3 S) -3-amino-3-carboxipropil] prop-2-ynylselenonio] -5'-desoxiadenosina (SeAdoYn, Figura 3) é aceite pelo ADN de tipo selvagem, ARN e MTases proteína 30-32 que anula a necessidade de engenharia de proteínas, em muitos casos. Isto é exemplificado por fluorescência pró rotulagem tein com a lisina histona H3 4 (H3K4) MTase SET7 / 9 33 (Figura 3, ver seção de protocolo 3: marcação de proteínas em dois passos via cofatores ativados duplas).
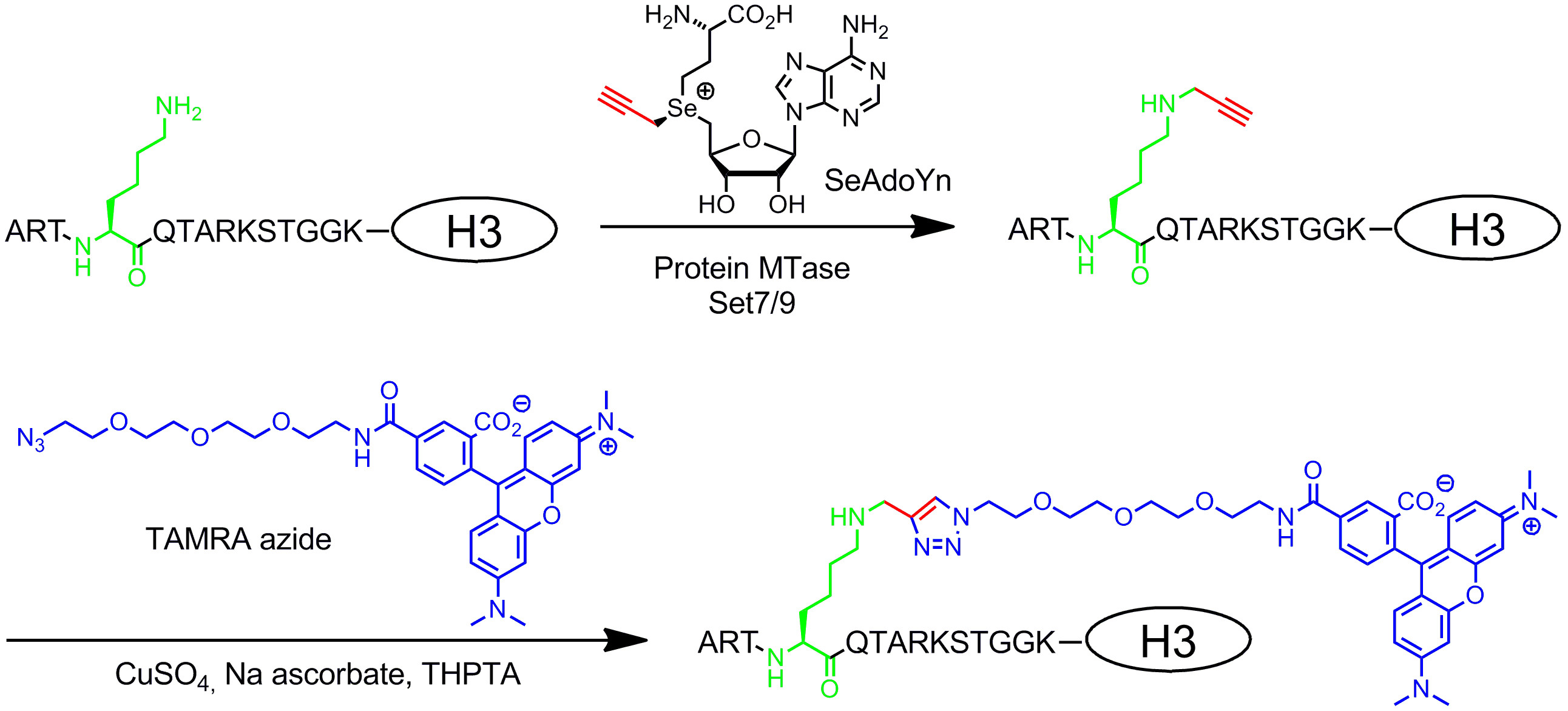
Figura 3:. Específica da sequência de fluorescência de duas etapas rotulagem de histona H3 com SET7 / 9, SeAdoYn e TAMRA azida A proteína MTase SET7 / 9 naturalmente metila o grupo amino da lisina 4 em histona H3 (H3K4, verde) usando AdoMet. Com o co-fator duplo ativado SeAdoYn o MTase transfere um grupo propargilico pequeno (vermelho) ao resíduo de lisina. A ligação tripla terminal conectado é então modificada seletivamente em uma reação clique bioorthogonal (azida-alcino cicloadi�o catalisada por cobre, CuAAC) com (tetramethylrhodamine, azul) fluorophore derivado-azida TAMRA.carga / 52014 / "target =" _ 52014fig3highres.jpg blank "> Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Instruções Gerais
- Aziridina loja cofactor 6BAz (em DMSO) e proteína MTase SET7 / 9 a -80 ° C e todos os outros reagentes, incluindo co-factor activado SeAdoYn dupla e ADN MTase M.BseCI (em glicerol a 50%) à temperatura de -20 ° C.
- Determinar a concentração de 6BAz e SeAdoYn via UV / Vis utilizando os coeficientes de extinção ε 269nm (6BAz) = 16,000 centímetros -1 M-1 e ε 260nm (SeAdoYn) = 15,400 centímetros -1 M-1 em água deionizada. Determinar a concentração de MTases pelo ensaio de Bradford ou, se o coeficiente de extinção está disponível, por meio de absorção directa a 280 nm.
- Tentar evitar a criação de bolhas por pipetagem intensivo ou em vortex para evitar a perda de actividade da enzima. Em vez disso, misture cuidado pipetando para cima e para baixo.
- Quando a adição de co-factores de aziridina partir de soluções mãe em DMSO a certificar-se de que a concentração final de DMSO no ensaio é de menosdo que 5%. Sempre incluir 10 mM de iões de magnésio no tampão de ensaio para evitar reacções não específicas com o ADN.
- Quando a adição de co-factores activados duplas a partir de soluções de estoque ácidas utilizar pequenos volumes (soluções de reserva altamente concentrada) para evitar a variação do pH e certificar-se de que o pH da solução de ensaio não se altera significativamente. Evitar, por exemplo, tióis, β-mercaptoetanol ou ditiotreitol (DTT), em tampão de ensaio, porque eles podem interferir com a reacção de cliques por complexação dos iões de cobre necessários.
2. Um passo Rotulagem de DNA via Aziridine Cofactores
- Específica da sequência Induzido por Metiltransferase Etiqueta ing (sorrindo) de DNA plasmídeo com M.BseCI DNA MTase e aziridine cofator 6BAz.
- Descongelar o cofactor solução a 20 ° C e preparar as misturas de reacção em gelo.
- Além de realizar um ensaio de controlo "cofactor", para visualizar as modificações não específicas, e um & #8220; enzima "controle, para se certificar de que a preparação MTase está livre do AdoMet cofator natural.
- Para a mistura do ensaio 2 ul de tampão de modificação 10x (contendo 100 mM de Tris-HCl, 100 mM de MgCl 2, 20 mM de β-mercaptoetanol, pH 7,4), 2 uL do pBR322 (0,5 ug / uL), 10 eq. M.BseCI por sequência de reconhecimento no ADN (sequência de reconhecimento em pBR322 1) e o cofactor aziridina 6BAz para uma concentração final de 60 pM num volume total de 20 ul. Adicionar cofator e DNA MTase passada.
NOTA: β-mercaptoetanol é tóxico, corrosivo e prejudicial para o ambiente. - Para o controle "cofator" adicionar água deionizada em vez de M.BseCI e para o controle de "enzima" adicionar água deionizada em vez de 6BAz.
- Misture as soluções cuidado pipetando para cima e para baixo.
- Incubar os tubos a 55 ° C durante 1 h.
- Centrifugar brevemente para recolher todo o líquido na parte inferior dos tubos.
- Ensaio restrição-modificação para verificar modificação DNA.
- Prepara-se uma solução por mistura de 10 ul de tampão 10x R.TaqI (contendo 100 mM de Tris-HCl, 50 mM de MgCl 2, 1 M de NaCl, 1 mg / ml de albumina de soro bovino, pH 8,0), 80 ul de água desionizada e 3,3 mL da endonuclease de restrição (Rease) a partir de Thermus aquaticus (R.TaqI, 10 U / ul). Certifique-se de adicionar o Rease na última etapa.
- Para cada tubo de 2.1.7 adicionar 2 ul de tampão Taq 10x R. e 28 ul da solução a partir de acima (2.2.1).
- Misture as soluções cuidado pipetando para cima e para baixo.
- Incubar os tubos a 65 ° C durante 30 min.
- Centrifugar brevemente para recolher todo o líquido na parte inferior dos tubos.
- Teste de desvio de Electromobility (EMSA) com estreptavidina para verificar modificação funcional.
- Remover 25 mL de cada tubo (2.2.5) e adicionar 2,4 mL de uma solução de estreptavidina (1 mM com respeito a estreptavidina monomer estreptavidina em tampão contendo 100 mM de Na 2 HPO 4, NaCl 100 mM, pH 7,5; 4 equivalentes de biotina total). Adicionar 2,4 mL de tampão de estreptavidina para os restantes tubos.
- Incubar todos os tubos a 37 ° C durante 1 h.
- Análise através de electroforese em gel de agarose.
- Adiciona-se 5 mL de tampão de carregamento 6x (0,25% de azul de bromofenol, 30% de glicerol) a cada tubo.
- Misture delicadamente as soluções.
- Carga de 10 ul de cada amostra para os poços de um gel de agarose (1% de agarose em tampão TBE 0,5x contendo 1x GelRed a partir de uma solução estoque 10.000x).
- Execute o gel em tampão TBE 0,5x com 80 V por aprox. 1 hr.
- Visualizar as bandas de ADN numa mesa de UV (312 nm) com uma câmara CCD equipado com um filtro (540 ± 50 nm).
NOTA: a luz UV é prejudicial para os olhos e pele.
3. Duas fases de marcação de proteínas via casal Cofactores activados
- MethyltransfTransferência de Grupos ativado (mTAG) com SET7 / 9 e cofator double-ativado SeAdoYn para histona H3 lisina 4 rotulagem (etapa de modificação) apagar-dirigida.
- Descongelar os componentes e preparar as misturas de reacção em gelo. NOTA: Sempre mantenha SeAdoYn resfriado para evitar a degradação.
- Além de realizar um ensaio de controlo "cofactor", para visualizar as modificações não específicas, e um controlo de "enzima", para excluir as reacções não específicas da sonda fluorescente.
- Prepara-se uma solução de ensaio (20 ul) contendo tampão de modificação (50 mM Tris-HCl, 5% de glicerol, pH 8,5), 10 mM de histona H3, 10 uM SET7 / 9 e 600 uM SeAdoYn (mistura de ambos os epímeros em selénio). Nas últimas etapas adicionar cofator e depois MTase.
- Para o controlo "cofactor" preparar uma solução de ensaio como em 3.1.3 e adicionar AdoMet 60 mM para competir com o cofactor sintético. Para o controle de "enzima" adicionar água deionizada em vez de SeAdoYn.
- Misture as soluções por lentamente pipetagem cima e para baixo. Verificar o pH por adição de 1 ul de cada solução para o campo superior de uma faixa de pH (gama de pH de 5 - 10).
- Incubar a 37 ° C durante 2 horas.
- No gel entretanto preparar um gel de SDS-poliacrilamida a 12% (em execução: 357 mM Bis-Tris pH 6,5-6,8, 0,1% (w / v) de APS, 0,04% (v / v) e TEMED a 12% de acrilamida / bisacrilamida 37,5: 1 ; carregamento do gel: 357 mM Bis-Tris pH 6,5-6,8, 0,1% (w / v) de APS, 0,04% (v / v) e TEMED a 5% de acrilamida / bisacrilamida 37,5: 1).
NOTA: A acrilamida / bisacrylamide é tóxico e nocivo à saúde. Usar luvas durante este procedimento.
- Rotulagem Chemical de lisina alkinylated 4 em histona H3 via cicloadi�o azida-alcino (CuAAC) (etapa de rotulagem) catalisada por cobre.
- Pouco antes do fim da reacção de modificação preparar uma mistura 5x clique contendo 3 mM de CuSO 4, 3 mM de tris (3-hidroxipropil-triazolilmetil) amina (THPTA), ascorbato de sódio 250 mM e TAMRA 6 mM de azida com umvolume total de 20 ul.
- Adiciona-se 5 uL da mistura de 5x clique recentemente preparada a cada tubo para iniciar a CuAAC e extingue-se a reacção de modificação.
- Misture delicadamente por pipetagem cima e para baixo.
- Proteja todos os tubos com folha de alumínio da luz para evitar a foto-branqueamento do fluoróforo.
- Incubar a 20 ° C durante 1 h.
- Precipitação de proteínas para remover o excesso de livre fluorophore TAMRA.
- Para evitar resplendor da marcação fluorescente histona H3 por intensivo fluorescência in-gel de livre fluorophore TAMRA, retire o excesso fluorophore por precipitação de proteínas (3.3.2 - 3.3.4) 34.
- Adicionar 75 mL de metanol, 18,8 mL de clorofórmio e 50 ul de água desionizada a cada tubo e vortex brevemente após cada adição. Centrifuga-se a 16000 xg durante 5 min. Remover a fase superior sem perturbar a camada de interface, que contém a proteína.
- Adicionar 56,3 mL de metanol à fase restante in cada tubo, vortex e centrifuga-se a 16.000 xg durante 5 min para sedimentar a proteína. Remover o sobrenadante. Repetir esta etapa para lavar o sedimento.
- Cubra os tubos abertos com um tecido que não solte fiapos e deixe-os secar por 15-30 min.
- Análise via SDS PAGE.
- Dissolve-se as proteínas precipitadas a partir de 3.3.4 em 20 ul de tampão de carga SDS (50 mM Tris-HCl, 2,5% (w / v) de SDS, 10% (v / v) de glicerol, 320 mM β-mercaptoetanol e 0,05% (w / v) azul de bromofenol, pH 6,8). Certifique-se para dissolver completamente o sedimento, por lavagem das paredes dos tubos com uma pipeta.
- Incubar as amostras a 95 ° C durante 10 min e deixa-os arrefecer até 20 ° C.
- Centrifugar brevemente para recolher todo o líquido na parte inferior dos tubos.
- Coloque toda a quantidade de cada amostra para os poços de um gel de poliacrilamida SDS (3.1.7). Use 50 mM de MOPS, 50 mM de Tris-X (Tris-base), EDTA 5 mM, 0,1% (w / v) de SDS como tampão de corrida para electroforese.
- Execute o gel com 120 V por aprox. 90 min.
- Visualizar a fluorescência em gel sobre uma mesa de UV (312 nm) com uma câmara CCD equipado com um filtro (540 nm ± 50 nm).
NOTA: a luz UV é prejudicial para os olhos e pele.
Resultados
Um passo-a Rotulagem de DNA via Aziridine Cofactores
Esta reacção é levada a cabo exemplo com o ADN MTase M.BseCI, que modifica o resíduo de adenina segunda dentro do 5'-ATCG A T-3 'sequência de cadeia dupla e tem um local de reconhecimento no plasmídeo pBR322 (Figura 4A). Para testar rotulagem plasmídeo, pBR322 é desafiado com a endonuclease de restrição (Rease) R.TaqI (5'-TCGA-3 '). R.TaqI tem sete sites na pBR322, um dos...
Discussão
Rotulagem de uma etapa de DNA com MTases DNA e cofatores aziridina (DNA sorrindo) é um método robusto, mas alguns aspectos devem ser considerados quando se planeja o experimento.
Cofactor aziridina: A concentração 6BAz para marcação de ADN com M.BseCI foi de 60 uM. Ao usar outros MTases DNA a concentração cofator deve ser otimizada, por exemplo, concentrações tão baixas como 20? M têm sido empregadas com o DNA MTase M.TaqI 19. Baixas concentrações 6...
Divulgações
The authors disclose the following competing financial interest: E.W. is inventor on related patents.
Agradecimentos
The authors thank Kerstin Glensk for preparing the MTases M.BseCI and Set7/9 and gratefully acknowledge funding by the Excellence Initiative of the German Federal and State Governments and RWTH Aachen University. The authors are happy to provide 6BAz and SeAdoYn or other cofactor analogues for collaborative research.
Materiais
| Name | Company | Catalog Number | Comments |
| 6BAz | Synthesized according to Weinhold et al., Patent number US 8,129,106, published March 6, 2012. | ||
| β-Mercaptoethanol | Serva | 28625 | |
| Acetic acid | Fisher Scientific | 10304980 | |
| Acrylamide/Bis Solution, 37.5:1 | Serva | 10688 | |
| UltraPure Agarose | Invitrogen | 16500100 | |
| Ammonium persulfate (APS) | Serva | 13375 | |
| Bis-Tris | Gerbu | 1304 | |
| Boric acid | Gerbu | 1115 | |
| Bromophenol blue Na salt | Serva | 15375 | |
| Copper(II) sulfate | Aldrich | C1297 | |
| Chloroform | Fisher Scientific | 10020090 | |
| Coomassie Brilliant Blue | Serva | 17525 | |
| EDTA disodium salt | Gerbu | 1034 | |
| Ethanol | Merck | 100983 | |
| GelRed (10,000x in water) | Biotium | 41003 | |
| Glycerol (99.5%) | Gerbu | 2006 | |
| FastRuler Low Range DNA Ladder | Thermo Scientific | SM1103 | |
| Histone H3 | Expression plasmid obtained from Dr. Philipp Voigt and Prof. Danny Reinberg; expression and isolation according to T. J. Richmond et al., J. Mol. Biol. 1997, 272, 301-311. | ||
| M.BseCI | Expression plasmid obtained from Dr. Michael Kokkinidis; expression and isolation according to Kapetaniou et al., Acta Cryst. 2006, F63, 12-14. | ||
| Methanol | Fisher Scientific | 10675112 | |
| Magnesiumchloride hexahydrate | J.T. Baker | 4003 | |
| MOPS | Gerbu | 1081 | |
| Sodium chloride | Gerbu | 1112 | |
| pH strip (Neutralit) | Merck | 1,095,330,001 | |
| pBR322 | Thermo Scientific | SD0041 | |
| R.TaqI (10 u/µl) | Thermo Scientific | ER0671 | |
| SeAdoYn | Synthesized according to Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| Set7/9 | Expression plasmid obtained from Prof. Danny Reinberg, expression and isolation according to D. Reinberg et al., Genes Dev.2002, 16, 479-489. | ||
| Streptavidin | Gerbu | 3058 | |
| (+)-Sodium L-ascorbate | Sigma Life Science | A7631 | |
| SDS Granular | Gerbu | 1833 | |
| di-Sodium hydrogenphosphate | Merck | 106,586 | |
| TAMRA azide | Synthesized according to reference 30: Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| TaqI buffer (10x) | Thermo Scientific | B28 | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) | Acros Organics | 42058 | |
| Tris-HCl | Gerbu | 1028 | |
| Tris-X (TRIS-base) | Gerbu | 1018 | |
| Tris(3-hydroxypropyltriazolyl-methyl)amine (THPTA) | Sigma-Aldrich | 762342 |
Referências
- Gottfried, A., Weinhold, E. Sequence-specific covalent labelling of DNA. Biochem. Soc. Trans. 39, 623-628 (2011).
- Zohar, H., Muller, S. J. Labeling DNA for single-molecule experiments: methods of labeling internal specific sequences on double-stranded DNA. Nanoscale. 3, 3027-3039 (2011).
- Hinner, M. J., Johnsson, K. How to obtain labeled proteins and what to do with them. Curr. Opin. Biotechnol. 21, 766-776 (2010).
- Wua, Y. -. W., Goody, R. S. Probing protein function by chemical modification. J. Pept. Sci. 16, 514-523 (2010).
- Struck, A. -. W., Thompson, M. L., Wong, L. S., Micklefield, J. S-Adenosyl-methionine-dependent methyltransferases: Highly versatile enzymes in biocatalysis, biosynthesis and other biotechnological applications. ChemBioChem. 13, 2642-2655 (2012).
- Klimasauskas, S., Weinhold, E. A new tool for biotechnology: AdoMet-dependent methyltransferases. Trends Biotechnol. 25, 99-104 (2007).
- Pljevaljcic, G., Schmidt, F., Weinhold, E. Sequence-specific Methyltransferase-Induced Labeling of DNA (SMILing DNA). ChemBioChem. 5, 265-269 (2004).
- Lukinavicius, G., Lapiene, V., Stasevskij, Z., Dalhoff, C., Weinhold, E., Klimasauskas, S. Targeted labeling of DNA by methyltransferase-directed Transfer of Activated Groups (mTAG). J. Am. Chem. Soc. 129, 2758-2759 (1021).
- Pignot, M., Siethoff, C., Linscheid, M., Weinhold, E. Coupling of a nucleoside with DNA by a methyltransferase. Angew. Chem. Int. Ed. 37, 2888-2891 (1998).
- Weller, R. L., Rajski, S. R. Design, synthesis, and preliminary biological evaluation of a DNA methyltransferase-directed alkylating agent. ChemBioChem. 7, 243-245 (2006).
- Du, Y., Hendrick, C. E., Frye, K. S., Comstock, L. R. Fluorescent DNA Labeling by N-Mustard Analogues of S-adenosyl-l-methionine. ChemBioChem. 13, 2225-2233 (2012).
- Pljevaljcic, G., Schmidt, F., Scheidig, A. J., Lurz, R., Weinhold, E. Quantitative labeling of long plasmid DNA with nanometer precision. ChemBioChem. 8, 1516-1519 (1002).
- Wilkinson, S., et al. Molecular scale architecture: engineered three- and four-way junctions. Bioconjugate Chem. 19, 470-475 (2008).
- Braun, G., et al. Enzyme-directed positioning of nanoparticles on large DNA templates. Bioconjugate Chem. 19, 476-479 (2008).
- Kim, S., et al. Enzymatically incorporated genomic tags for optical mapping of DNA binding proteins. Chem. Int. Ed. 51, 3578-3581 (2012).
- Rina, M., Bouriotis, V. Cloning purification and characterization of the BseCI DNA methyltransferase from Bacillus stearothermophilus. Gene. 133, 91-94 (1993).
- Weinhold, E., Meier, T., Düfel, H., Markert-Hahn, C., Schmuck, R. Sequence-specific detection of methylation in biomolecules. US Patent. , (2012).
- Pljevaljcic, G., Pignot, M., Weinhold, E. Design of a new fluorescent cofactor for DNA methyltransferases and sequence-specific labeling of DNA. J. Am. Chem. Soc. 125, 3492-3410 (2003).
- Schmidt, F. H. -. G., Hüben, M., Gider, B., Renault, F., Teulade-Fichou, M. -. P., Weinhold, E. Sequence-specific Methyltransferase-Induced Labelling (SMILing) of plasmid DNA for studying cell transfection. Bioorg. Med. Chem. 16, 40-48 (2008).
- Dalhoff, C., Lukinavicius, G., Klimasauskas, S., Weinhold, E. Direct transfer of extended groups from synthetic cofactors by DNA methyltransferases. Nat. Chem. Biol. 2, 31-32 (2006).
- Lukinavicius, G., Tomkuviene, M., Masevicius, V., Klimasauskas, S. Enhanced chemical stability of AdoMet analogues for improved methyltransferase-directed labeling of DNA. ACS Chem. Biol. 8, 1134-1139 (2013).
- Motorin, Y., et al. Expanding the chemical scope of RNA:methyltransferases to site-specific alkynylation of RNA for click labeling. Nucleic Acids Res. 39, 1943-1952 (1943).
- Schulz, D., Holstein, J. M., Rentmeister, A. A chemo-enzymatic approach for site-specific modification of the RNA cap. Angew. Chem. Int. Ed. 52, 7874-7878 (2013).
- Peters, W., et al. Enzymatic site-specific functionalization of protein methyltransferase substrates with alkynes for click labeling. Angew. Chem. Int. Ed. 49, 5170-5173 (2010).
- Islam, K., Zheng, W., Yu, H., Deng, H., Luo, M. Expanding cofactor repertoire of protein lysine methyltransferase for substrate labeling. ACS Chem. Biol. 6, 679-684 (2011).
- Wang, R., Zheng, W., Yu, H., Deng, H., Luo, M. Labeling substrates of protein arginine methyltransferase with engineered enzymes and matched S-adenosyl-l-methionine analogues. J. Am. Chem. Soc. 133, 7648-7651 (2011).
- Islam, K., et al. Bioorthogonal profiling of protein methylation using azido derivative of S-adenosyl-l-methionine. J. Am. Chem. Soc. 134, 5909-5915 (2012).
- Islam, K., et al. Defining efficient enzyme-cofactor pairs for bioorthogonal profiling of protein methylation. Proc. Natl. Acad. Sci. U.S.A. 110, 16778-16783 (2013).
- Binda, O., Boyce, M., Rush, J. S., Palaniappan, K. K., Bertozzi, C. R., Gozani, O. A chemical method for labeling lysine methyltransferase substrates. ChemBioChem. 12, 330-334 (2011).
- Willnow, S., Martin, M., Lüscher, B., Weinhold, E. A selenium-based click AdoMet analogue for versatile substrate labeling with wild-type protein methyltransferases. ChemBioChem. 13, 1167-1173 (2012).
- Bothwell, I. R., et al. Se-Adenosyl-l-selenomethionine cofactor analogue as a reporter of protein methylation. J. Am. Chem. Soc. 134, 14905-14912 (2012).
- Tomkuviene, M., Clouet-d’Orval, B., Cerniauskas, I., Weinhold, E., Klimasauskas, S. Programmable sequence-specific click-labeling of RNA using archaeal box C/D RNP methyltransferases. Nucleic Acids Res. 40, 6765-6773 (2012).
- Nishioka, K., et al. Set9, a novel histone H3 methyltransferase that facilitates transcription by precluding histone tail modifications required for heterochromatin formation. Genes Dev. 16, 479-489 (2002).
- Clark, P. M., et al. Direct in-gel fluorescence detection and cellular imaging of O-GlcNAc-modified proteins. J. Am. Chem. Soc. 130, (2008).
- Lukinavicius, G., Lapinaite, A., Urbanaviciute, G., Gerasimaite, R., Klimasauskas, S. Engineering the DNA cytosine-5 methyltransferase reaction for sequence-specific labeling of DNA. Nucleic Acids Res. 40, 11594-11602 (2012).
- Neely, R. K., Dedecker, P., Hotta, J., Urbanaviciute, G., Klimasauskas, S., Hofkens, J. DNA fluorocode: A single molecule, optical map of DNA with nanometre resolution. Chem. Sci. 1, 453-460 (2010).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE-a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Res. 38, 234-236 (2010).
- Petrossian, T. C., Clarke, S. G. Uncovering the human methyltransferasome. Mol. Cell. Proteomics. 10, 1-12 (2011).
- Kriukiene, E., et al. DNA unmethylome profiling by covalent capture of CpG sites. Nat. Commun. 4, 2190 (2013).
- Wang, R., et al. Profiling genome-wide chromatin methylation with engineered posttranslation apparatus within living cells. J. Am. Chem. Soc. 135, 1048-1056 (2013).
- Zhang, C., Weller, R. L., Thorson, J. S., Rajski, S. R. Natural product diversification using a non-natural cofactor analogue of S-adenosyl-l-methionine. J. Am. Chem. Soc. 128, 2760-2761 (2006).
- Stecher, H., et al. Biocatalytic Fiedel-Crafts alkylation using non-natural cofactors. Angew. Chem. Int. Ed. 48, 9546-9548 (2009).
- Lee, B. W. K., Sun, H. G., Zang, T., Kim, B. J., Alfaro, J. F., Zhou, Z. S. Enzyme-catalyzed transfer of a ketone group from an S-adenosylmethionine analogue: A tool for the functional analysis of methyltransferases. J. Am. Chem. Soc. 132, 3642-3643 (2010).
- Winter, J. M., et al. Expanding the structural diversity of polyketides by exploring the cofactor tolerance of an inline methyltransferase domain. Org. Lett. 15, 3774-3777 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados