É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
De alta resolução de ressonância magnética estrutural do Subcórtex Humano
Neste Artigo
Resumo
Here we present a protocol to determine the minimum number images that needed to be registered and averaged to resolve subcortical structures and test whether the individual layers of the LGN could be resolved in the absence of physiological noise.
Resumo
O foco deste estudo foi testar os limites de resolução da ressonância magnética estrutural de um cérebro pós-morte em comparação com cérebros humanos vivos. A resolução da ressonância magnética estrutural in vivo é finalmente limitado pelo ruído fisiológico, incluindo pulsação, respiração e movimento da cabeça. Embora o hardware de imagem continua a melhorar, ainda é difícil de resolver estruturas na escala milimétrica. Por exemplo, as vias sensoriais primários Visual sinapse no núcleo geniculado lateral (LGN), um relê de controlo visual e núcleo no tálamo que normalmente está organizado em seis camadas monoculares intercalados. Estudos de neuroimagens não foram capazes de distinguir de forma segura estas camadas, devido a sua pequena dimensão que são menos do que 1 mm de espessura.
O limite de resolução de ressonância magnética estrutural, em um cérebro pós-morte foi testada usando várias imagens em média por uma longa duração (~ 24 h). O objectivo foi testar se que era possível resolver o l indivíduoAyers da LGN na ausência de ruído fisiológico. A densidade de protões (DP) 1 sequência de pulso ponderado foi utilizado com vários outros parâmetros e resolução para determinar o número mínimo de imagens necessárias para ser registado e a média para distinguir de forma segura a LGN e outras regiões subcorticais. Os resultados foram também comparados com imagens adquiridas em cérebros humanos vivos. In vivo sujeitos foram verificados, a fim de determinar os efeitos adicionais de ruído fisiológico sobre o número mínimo de análises de PD necessários para diferenciar estruturas subcorticais, úteis em aplicações clínicas.
Introdução
O objetivo desta pesquisa foi testar os limites de resolução da RM estrutural na ausência de ruído fisiológico. Densidade (PD) imagens ponderadas de prótons foram adquiridas em um cérebro pós-morte durante um longo período (duas sessões de RH ~ 24) para determinar o número mínimo de imagens que precisavam ser registradas e média para resolver as estruturas subcorticais. Para efeito de comparação, PD imagens ponderadas também foram adquiridas em seres humanos vivos ao longo de um número de sessões. Em particular, o objetivo foi verificar se seria possível em um cenário mais favorável para resolver todas as seis camadas individuais do LGN humano, que são aproximadamente 1 mm de espessura (Figura 1).
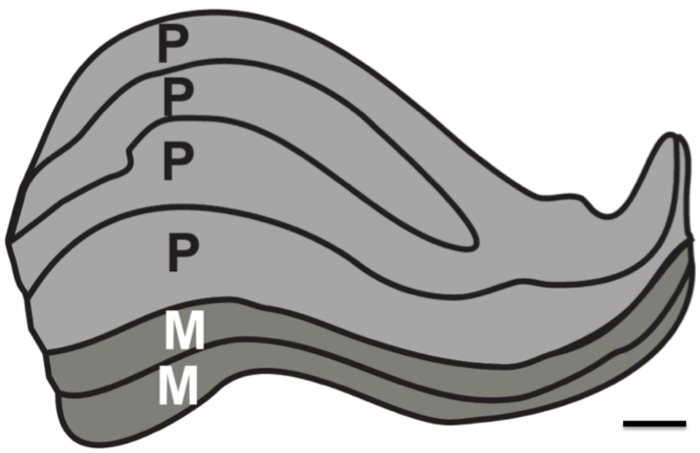
Figura 1. camadas lateral humana Geniculado núcleo. Esquemático da estrutura laminar do LGN. Magnocelulares (m) camadas são compostas de maior neuronaltamanho das células e densidade de células menores que são responsáveis para a resolução de movimento e curso contornos (camadas 1-2, descrito como cinza escuro). Camadas parvocelulares (P) são compostos de tamanho menor de células neuronais e maior densidade de células que são responsáveis pela resolução de fine-forma e cor (camadas 4-6, descrito como cinza claro). Bar escala de 1 mm. Figura baseado em coradas LGN 12 humana.
A resolução espacial em MRI é melhorada quando o tamanho da matriz aumenta, e quando de campo de visão (FOV) e espessura de corte são diminuídos. No entanto, o aumento da resolução diminui a relação sinal-ruído (SNR), que é proporcional ao volume do voxel. SNR também é proporcional à raiz quadrada do número de medições. Em seres humanos vivos, embora várias imagens pode ser adquirida ao longo de uma série de sessões de imagem separados, a resolução final é limitado pelo ruído fisiológico, como a respiração, pulsação circulatório e movimento da cabeça.
Alto-Resolução (0,35 mm voxels In-Plane) PD scans ponderados foram adquiridos. Scans PD melhorar o contraste cinza e branco no tálamo 1, e resultam em imagens que minimizem T 1 e T 2 efeitos. A sua imagem é dependente da densidade de protões, sob a forma de água e as macromoléculas, tais como proteínas e gordura no volume de imagem. Os números aumentados de protões num tecido resulta num sinal mais brilhante na imagem devido ao componente longitudinal superior da magnetização 2.
Scans ponderadas pelo DP foram coletadas uma vez que proporcionam um maior contraste de estruturas subcorticais com o tecido circundante. Outros contrastes, como T1 e T2-ocasionar dificuldade em delinear estruturas subcorticais como a LGN devido a menores rácios de contraste-ruído, tal como determinado ƒ 1,3.
Da mesma forma, estudos anteriores descobriram que as imagens ponderadas em DP de formol fixa cérebros post-mortem redundoud em diferenças de contraste mais elevados entre matéria cinzenta e branca em comparação com T1 e imagens ponderadas em T2 que tiveram cinzentos e brancos semelhantes intensidades de imagem do assunto 3,4. Os determinantes biofísicos subjacentes podem explicar essas diferenças. T1 (longitudinal) e T2 (transversal) tempos de relaxação de prótons de hidrogênio dependerá de como a água se move dentro do tecido. Fixadores, como o trabalho de formalina por proteínas de ligação cruzada. As diferenças entre a mobilidade de água são reduzidos entre os diferentes tipos de tecidos quando fixadores são usados. Reduzido contraste tecido T1 tem sido observada após a fixação, ao passo que as diferenças de densidade relativa dos protões dentro dos tecidos do cérebro aumenta com a fixação, proporcionando uma melhor diferenciação contraste 3, 4.
Estudos anteriores já haviam identificado o LGN em exames ponderados pelo PD usando um 1.5 T 5,6,7, e pelo scanner de 3 T 8,9. É crítico para obter estas verificações para ser capaz de delinear com exactidão a extensão dao LGN. Para manter a cobertura total dos núcleos subcorticais, 18 fatias ponderada-PD foram obtidos dentro do tálamo. Cada volume foi re-amostrados para o dobro da matriz de 1024, (0,15 mm de tamanho de in-plane voxel), concatenada, movimento corrigido e média para produzir uma imagem 3D de alta resolução de estruturas subcorticais. O número ideal de imagens DP necessários para a seguinte receita fatia foi de 5, reduzindo o tempo de digitalização para menos de 15 min em seres humanos vivos. Apenas uma imagem PD foi necessário para demarcar claramente regiões subcorticais no cérebro pós-morte, reduzindo o tempo de digitalização para menos de 3 min (Figura 2 e 3).
Toda uma amostra de cérebro pós-morte fixado em formol foi feita a varredura de uma mulher que morreu de parada cardiorrespiratória na idade de 82 anos. Revisão de prontuários médicos revelou que ela tinha: doença pulmonar obstrutiva crônica, cirurgia angina, triplo bypass 8 anos antes da morte, câncer uterino tratado com histerectomia7 anos antes da morte, hiperlipidemia, glaucoma, e cirurgia de catarata. O espécime cerebral postmortem foi fixado em imersão em 10% de formalina tamponada neutra por pelo menos 3 semanas a 4 ° C. A pós-mortem de cérebro foi digitalizado com o mesmo protocolo de imagem, bem como com outros parâmetros ao longo de muitas horas para comparações de qualidade de imagem . Apenas os parâmetros optimizados será descrito para o protocolo.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Participante e Postmortem Cérebro Set-Up
Observação: Todas as imagens foram obtidas utilizando um scanner de RM 3 T com uma bobina de cabeça de 32 canais e todos ressonância magnética foi realizada a temperatura ambiente, aproximadamente 20 ° C. Todos os participantes eram destros e assinaram termo de consentimento informado. Cada participante estava com boa saúde, sem histórico de distúrbios neurológicos. O protocolo experimental foi aprovado e segue as diretrizes de participantes York University Humanos Comitê de Revisão.
- Peça a cada participante preencher e assinar um formulário de consentimento do paciente que detalha as diretrizes de segurança de ressonância magnética e o protocolo neuro-imaging.
- Para cada participante, colocar tampões em cada orelha e assegurar a sua cabeça com almofadas de cabeça para minimizar movimento.
- Para pós-mortem de imagens do cérebro, certifique-se o cérebro é fixa antes de neuroimagem e está contido dentro de um saco ou recipiente que se encaixa dentro da cabeça-coil de ressonância magnética. Coloque o cérebro pós-morte na cabeça-bobina com o eixo z (superior a inferior) alinhado com o furo do scanner. O tronco cerebral (posterior) deve estar virada para o pé da cama scanner.
- Coloque almofada de vácuo mãos ao redor do cérebro post-mortem para suporte adicional.
2. Localizando e prescrever o Subcórtex
NOTA: O tálamo é uma estrutura dupla lóbulos localizada perto do centro do cérebro situado entre o mesencéfalo e o córtex cerebral. Localizado dentro do tálamo dorsal, o LGN humana é uma pequena estrutura que se estende subcortical um máximo de ~ 10 mm.
- Para registrar um novo participante, abra o software de imagem de ressonância magnética e clique na aba Patient no canto superior esquerdo. Em seguida, clique em Register.
- Preencha as informações do paciente apropriado, e em seguida, clique na guia Exame.
- Para obter uma varredura do localizador, clique na guia Exame Explorer para criar um novo protocolo. Observe a janela de set-up na tela, clique na guia de rotina, e digite os seguintes parâmetros: a aquisiçãotempo de 28 seg, matriz de aquisição de 160 × 160, 1 fatia, o tamanho do voxel isotrópico 1,6 mm de espessura, FOV = 260 mm, fase FoV = 100%, resolução fatia = 69%, fase e cortar fase parcial Fourier = 6/8, TR = 3.15 ms, TE = 1,37 ms, Flip Ângulo = 8 °.
- Overlay caixa de seleção fatia utilizado para a aquisição das imagens de PD sobre o localizador que cobrem os núcleos subcorticais dentro do tálamo, bem como estruturas circundante (Figura 4).
3. de alta resolução parâmetros estruturais
- Criar um novo protocolo para a obtenção de alta resolução scans ponderadas pelo PD. Na janela de set-up na tela, clique na guia de rotina, e digite os seguintes parâmetros na orientação coronal: tempo de aquisição de 179 seg, matriz de aquisição de 512 × 512, 0,3 × 0,3 × 1 mm3 tamanho do voxel, TR = 3.25 seg , TE = 32 ms, ângulo aleta = 120 °, a aquisição da fatia intercalada, FoV ler = 160 mm FoV fase = 100%, imagens paralelas (GRAPPA) comum fator de aceleração de 2.
- Use uma seqüência de eco Turbo Spin, com um comprimento de eco Trem de 5. O primeiro eco em 32 ms é o eco eficaz para esta sequência. Reduzir a largura de banda (BW) para o possível, 40 Hz / pixel mínimo, para maximizar a SNR. Para reduzir a duração da verificação, escolha 18 fatias, cada um milímetro de espessura, com um FOV = 160 mm. Esta laje oferece cobertura suficiente das regiões subcorticais de interesse.
NOTA: Para identificação correcta das estruturas subcorticais, adquirir 5 corridas com os parâmetros acima. A duração total de verificação é apenas ~ 15 min (Figura 5). Fat-saturação não foi empregado.
- Use uma seqüência de eco Turbo Spin, com um comprimento de eco Trem de 5. O primeiro eco em 32 ms é o eco eficaz para esta sequência. Reduzir a largura de banda (BW) para o possível, 40 Hz / pixel mínimo, para maximizar a SNR. Para reduzir a duração da verificação, escolha 18 fatias, cada um milímetro de espessura, com um FOV = 160 mm. Esta laje oferece cobertura suficiente das regiões subcorticais de interesse.
- No post mortem de imagens do cérebro, identificação fiável de estruturas subcorticais pode ser observada em apenas um scan com a duração total de apenas ~ 3 min, seguindo o mesmo protocolo de varrimento como em 3.1 (Figura 6).
4. Análise de Imagem
NOTA: Para analisar os dados de MRI, utilize o FMRIB livremente disponível daSoftware Library (FSL) pacote disponível para download em (https://www.fmrib.ox.ac.uk/fsl/).
- Abra uma janela de terminal, e converter os arquivos DICOM matérias do scanner para cada volume PD para um formato NIfTI com DICOM ao conversor NIfTI. Um número dos quais estão disponíveis gratuitamente para download (por exemplo., Https://www.nitrc.org/projects/mricron). Na linha de comando, digite dcm2nii seguido do diretório de cada PD imagem ponderada prazo.
- Em uma janela de terminal obter os parâmetros de verificação PD inicial. Escreva fslinfo na linha de comando seguido pela verificação PD em formato NIfTI.
- Crie uma alta resolução de imagem em branco volume de destino que tem o dobro da resolução e metade do tamanho do voxel dado pelos parâmetros de fslinfo da verificação PD inicial. A ordem das entradas de dados para este comando são as seguintes:
fslcreatehd
NOTA: Por exemplo, se a varredura PD original com os seguintes parâmetros, conforme descrito no ponto 3.1 são coletadas (ou seja, de 512 × 512 matriz, 18 fatia, 0,3 × 0,3 × 1 mm3 tamanho do voxel, TR = 3,25 s), digite o seguinte na janela de comando:
fslcreatehd 1024 1024 36 1 0,5 0,15 0,15 3,25 0 0 0 4 blankhr.nii.gz - Definir a transformação utilizando uma matriz de identidade. Escreva em qualquer programa editor de texto de um arquivo de texto salvo como 'identity.mat' que se parece com isso:
0 0 0
1 0 0
0 1 0
0 0 1 - Use o comando namoradeira para aplicar a transformação, upsampling cada execução ponderada PD original para duplicar a resolução total desde um 512 para uma matriz de 1024, e reduzir pela metade o tamanho do voxel em cada dimensão, resultando em uma resolução de 0,15 × 0,15× 0,5 mm 3. Em uma janela de terminal para cada volume PD, digite o seguinte comando namoradeira mudando os nomes originais e saída por corrida:
flertar -interp sinc -em originalPD.nii.gz -rep blankhr.nii.gz -applyxfm -init identity.mat out highresPD.nii.gz
NOTA: Onde originalPD.nii.gz é o volume de origem, blankhr.nii.gz é a resolução de saída desejada, e highresPD.nii.gz é o nome do volume de saída. - Mova todas as imagens de alta resolução para uma nova pasta, e navegue até ele em uma janela de terminal.
- Para cada participante, concatenar todas as imagens PD upsampled em um único arquivo usando 4D fslmerge. Em um tipo de janela de terminal:
fslmerge -t concat_highresPD * .nii.gz
NOTA: Isso cria um arquivo chamado 4D concat_highresPD.nii.gz. - Movimento corrigir o arquivo concatenado usando mcflirt 10. Esta ferramenta permite um registro robusto automatizado para linear (afim) imagens inter e inter-modais cerebrais. Selecione umaCorrecção de 4-fase, que utiliza a interpolação de sincronismo (internamente) como mais um passo de optimização para uma maior precisão. Em um tipo de janela de terminal:
mcflirt -em concat_highresPD out mcf_concat_highresPD.nii.gz -stages 4 -plots
NOTA: Isso cria um arquivo chamado 4D mcf_concat_highresPD.nii.gz. - Finalmente, criar o 3D dizer de imagem usando fslmaths. Em um tipo de janela de terminal:
fslmaths mcf_concat_highresPD.nii.gz -Tmean mean_highresPD.nii.gz
NOTA: Isso cria um arquivo 3D chamado mean_highresPD.nii.gz que é de alta qualidade - Visualize a imagem de alta resolução resultado final 3D usando o comando fslview. No diretório de onde sua imagem é, digite o seguinte em uma janela de terminal:
fslview mean_highresPD.nii.gz. " - Inspecione perfis de intensidade de ROIs em questão. Criar um ROI utilizando fslview (esta pode ser uma linha vertical traçada através de uma região do LGN por exemplo). Em fslview carregar a imagem PD de alta resolução. Clique na guia ferramentas,em seguida, clique na guia única imagem para ampliar a imagem para desenhar ROIs. Em seguida, clique na guia Arquivo seguido pelo guia Criar Mask. Desenhar uma linha no ROI de interesse. Salve o ROI clicando em Arquivo, em seguida, Salvar como. Repita as máscaras de linha para várias áreas dentro do ROI para comparações de intensidade e outros ROIs em questão.
- Use comando 3dmaskdump de Afni para analisar a intensidade da imagem resultante. No diretório de onde as imagens são, use o seguinte comando em uma janela de terminal para extrair as intensidades e localização de imagem (dado como result_mask.txt) de sua máscara ROI:
3dmaskdump -o result_mask.txt -noijk -xyz -mask ROI_linemask.nii.gz PDaverage_image.nii.gz
Access restricted. Please log in or start a trial to view this content.
Resultados
Uma vez que o subcortex é prescrito dentro do tálamo, PD imagens ponderadas são coletados dentro da caixa de seleção fatia (Figura 4). A SNR melhorada através do aumento do número de médias em ambas post-mortem e em exames in vivo. Para determinar a qualidade de imagem, o SNR de diferentes médias de digitalização foi comparada pela divisão do sinal da região cerebral média por o desvio padrão em alguma área fora do cérebro. A SNR foi calculada como SNR = 0,655 * u
Access restricted. Please log in or start a trial to view this content.
Discussão
Este estudo descreve um protocolo otimizado na aquisição e análise técnica a fim de obter de alta resolução PD imagens ponderadas das regiões subcorticais. Um certo número de parâmetros de análise foram testados e modificado com os mais significativos relacionados com o tamanho da matriz, o tamanho do voxel, e de largura de banda para aumentar a SNR e diminuir o número de aquisições, um passo crítico no ser capaz de determinar de alta resolução estruturas subcorticais. Em conjunto com a encontrar os par?...
Access restricted. Please log in or start a trial to view this content.
Divulgações
The authors have nothing to disclose.
Agradecimentos
The authors acknowledge the following funding sources, the Natural Sciences and Engineering Research Council of Canada (NSERC), the Dorothy Pitts Research Fund (NG), and the Nicky and Thor Eaton Research Fund. The authors acknowledge Kevin DeSimone, and Aman Goyal and for their knowledge in MRI acquisition and analysis expertise.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Magnetom Trio 3T MRI | Siemens (Erlangen, Germany). | ||
| Vacuum cushion hand | Siemens | Mat No: 4765454 | Manufactured by: Johannes-Stark-Stk. 8 D-92224 Amberg |
Referências
- Devlin, J. T., et al. Reliable identification of the auditory thalamus using multi-modal structural analyses. NeuroImage. 30 (4), 1112-1120 (2006).
- Fellner, F., et al. True proton density and T2-weighted turbo spin-echo sequences for routine MRI of the brain. Neuroradiology. 36 (8), 591-597 (1994).
- Schumann, C. M., Buonocore, M. H., Amaral, D. G. Magnetic resonance imaging of the post-mortem autistic brain. J Autism Dev Disord. 31 (6), 561-568 (2001).
- Tovi, M., Ericsson, A. Measurements of T1 and T2 over time in formalin-fixed human whole-brain specimens. Acta Radiol. 33 (5), 400-404 (1992).
- Fujita, N., et al. Lateral geniculate nucleus: anatomic and functional identification by use of MR imaging. Am J Neuroradiol. 22 (9), 1719-1726 (2001).
- Bridge, H., Thomas, O., Jbabdi, S., Cowey, A. Changes in connectivity after visual cortical brain damage underlie altered visual function. Brain. 131 (6), 1433-1444 (2008).
- Gupta, N., et al. Atrophy of the lateral geniculate nucleus in human glaucoma detected by magnetic resonance imaging. Br J Opthalmol. 93 (1), 56-60 (2009).
- Dai, H., et al. Assessment of lateral geniculate nucleus atrophy with 3T MR imaging and correlation with clinical stage of glaucoma. Am J Neuroradiol. 32 (7), 1347-1353 (2011).
- McKetton, L., Kelly, K. R., Schneider, K. A. Abnormal lateral geniculate nucleus and optic chiasm in human albinism. J Comp Neurol. 522 (11), 2680-2687 (2014).
- Jenkinson, M., Bannister, P., Brady, M., Smith, S. Improved optimization for the robust and accurate linear registration and motion correction of brain images. NeuroImage. 17 (2), 825-841 (2002).
- Dietrich, O., Raya, J. G., Reeder, S. B., Reiser, M. F., Schoenberg, S. O. Measurement of signal-to-noise ratios in MR images: influence of multichannel coils, parallel imaging, and reconstruction filters. J Magn Reson Imaging. 26 (2), 375-385 (2007).
- Andrews, T. J., Halpern, S. D., Purves, D. Correlated size variations in human visual cortex, lateral geniculate nucleus, and optic tract. J Neurosci. 17 (8), 2859-2868 (1997).
- Pfefferbaum, A., Sullivan, E. V., Adalsteinsson, E., Garrick, T., Harper, C. Postmortem MR imaging of formalin-fixed human brain. NeuroImage. 21 (4), 1585-1595 (2004).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados