É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Mapeamento de todo o genoma de DNA Interações medicamentosas-em células com COSMIC (reticulação de moléculas pequenas para isolar Chromatin)
Neste Artigo
Resumo
Identificar os alvos diretos de moléculas-alvo do genoma permanece um grande desafio. Para compreender como as moléculas de ligação de ADN envolver o genoma, foi desenvolvido um método que se baseia em ligação cruzada de moléculas pequenas para isolar cromatina (cósmica).
Resumo
O genoma é o alvo de alguns dos mais eficazes agentes quimioterapêuticos, mas a maior parte destes fármacos não têm especificidade de sequência de ADN, que conduz a toxicidade dose-limitante e muitos efeitos colaterais adversos. Segmentação do genoma com pequenas moléculas específicas para a sequência pode permitir que moléculas com elevação do índice terapêutico e menos efeitos fora do alvo. -methylpyrrole N / N poliamidas -metilimidazole são moléculas que podem ser racionalmente concebidos para dirigir sequências de ADN específicos com precisão requintado. E diferentemente da maioria dos fatores de transcrição naturais, poliamidas podem se ligar ao DNA metilado e chromatinized sem perda de afinidade. A especificidade da sequência de poliamidas tem sido extensivamente estudada in vitro com a identificação do local cognato (CSI) e com abordagens bioquímicas e biofísicas tradicionais, mas o estudo de poliamida de ligação para alvos genómicos em células permanece indefinida. Aqui nós relatamos um método, a reticulação de moléculas pequenas para Isolate cromatina (COSMIC), que identifica os locais de ligação de poliamida em todo o genoma. Cósmica é semelhante a imunoprecipitação da cromatina (ChIP), mas difere em dois aspectos importantes: (1) um photocrosslinker é empregue para permitir a captura selectiva, controlada temporalmente de eventos de ligação de poliamida, e (2) a afinidade punho biotina é utilizada para purificar poliamida -ADN conjugados sob condições semi-desnaturante para diminuir o ADN que não é ligada de forma covalente. Cósmica é uma estratégia geral que pode ser usado para revelar os eventos de ligação de poliamidas e outros agentes quimioterapêuticos-alvo do genoma de todo o genoma.
Introdução
A informação para fazer cada uma das células no corpo humano é codificado no DNA. A utilização selectiva de informação que governa o destino de uma célula. Os factores de transcrição (TFS) são proteínas que se ligam a sequências específicas de ADN para expressar um subconjunto particular dos genes no genoma, e o mau funcionamento do TFS está associada ao aparecimento de uma ampla variedade de doenças, incluindo defeitos no desenvolvimento, cancro e diabetes 1,2. Estamos interessados em desenvolver moléculas que podem se ligam selectivamente ao genoma e modulam redes reguladoras de genes.
Poliamidas composto de N -methylpyrrole -metilimidazole e N são moléculas de ADN que podem atingir com especificidades e afinidades que rivalizam com factores de transcrição naturais 3-6. Estas moléculas se ligam a sequências específicas no sulco menor do ADN racionalmente concebido. 4,5,7 -11 As poliamidas têm sido empregues para ambos reprimir e activar a expressão de g específicaenes. 4,12-19 Eles também têm interessantes antivirais e anticancerígenas 20-24 12,13,25-30 propriedades. Uma característica atraente de poliamidas é a sua capacidade de aceder a sequências de ADN que são metilados 31,32 e envolvidos em torno de proteínas histonas 9,10,33.
Para medir as especificidades de ligação abrangentes de moléculas se ligam ao DNA, o nosso laboratório criou o método identificador do site cognato (CSI). 34-39 A ocorrência previsto de locais de ligação com base em in vitro especificidades (genomescapes) pode ser exibido no genoma, porque o in vitro intensidades de ligação são diretamente proporcionais constantes de associação (Ka). 34,35,37 Estes genomescapes fornecer a introspecção em poliamida ocupação em todo o genoma, mas medir poliamida obrigatório em células vivas tem sido um desafio. ADN é empacotado firmemente no núcleo, o que poderia influenciar a acessibilidade de locais de ligação. A umccessibility destas sequências de ADN chromatinized para poliamidas permanece um mistério.
Recentemente, muitos métodos para estudar as interações entre as moléculas pequenas e ácidos nucleicos têm surgido. 40-48 A captura de afinidade química e sequenciamento de DNA massivamente paralelo (chem-seq) é uma tal técnica. Chem SEQ utiliza-formaldeído para reticular moléculas pequenas para um alvo genómico de interesse e um derivado biotinilado de uma pequena molécula de interesse para capturar a interacção ligando-alvo. 48,49
Formaldeído reticulação conduz a interações indiretas que podem produzir falsos positivos. 50 Desenvolvemos um novo método, a reticulação de moléculas pequenas para isolar cromatina (COSMIC), 51 com um photocrosslinker para eliminar esses chamados picos "fantasmas". 50 Para começar, nós projetamos e sintetizados derivados trifuncionais de poliamidas. Estas moléculas continha uma ligação a DNA-polyamide, um photocrosslinker (psoraleno), e um identificador por afinidade (biotina, Figura 1). Com poliamidas trifuncionais, que podem covalentemente capturar interacções DNA-poliamida com irradiação UV a 365 nm, um comprimento de onda que não danificar o DNA ou induzir reticulação não-psoraleno-base. 51 Em seguida, fragmentar o genoma e purificar o ADN capturado sob rigorosas, semi -denaturing condições para diminuir o ADN que não é ligada de forma covalente. Assim, vemos cósmicos um método relacionado com a Chem-SEQ, mas com uma leitura mais directa de ADN de direccionamento. Importante, a fraco (K 10 3 -10 4 M -1) a afinidade para o ADN de psoraleno não detectável impacto poliamida especificidade. 51,52 Os fragmentos de ADN enriquecidos podem ser analisados por qualquer polimerase de reacção em cadeia quantitativa 51 (cósmicos de qPCR) ou por sequenciamento de próxima geração 53 (COSMIC-seq). Estes dados permitem um projeto imparcial, guiada por genoma de ligantes que a Interagir com seus loci genômica desejado e minimizar os efeitos fora do alvo.
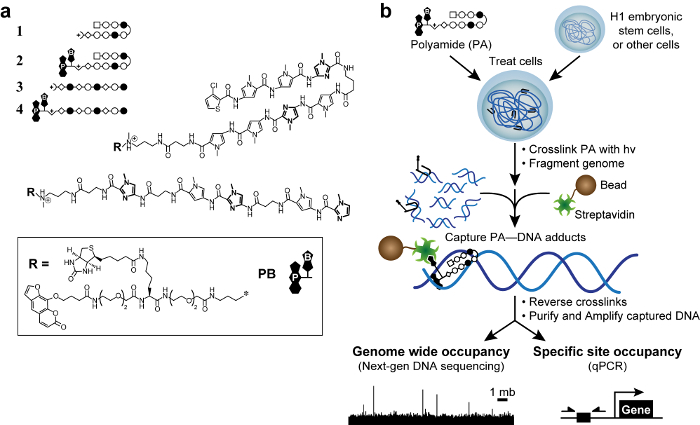
Figura 1. poliamidas bioativos e esquema cósmico. (A) poliamidas Hairpin 1 - 2-alvo a sequência de ADN 5'-WACGTW-3 '. Poliamidas lineares 3 - 4 5'-alvo AAGAAGAAG-3 '. Anéis de N-metilimidazole estão em negrito para maior clareza. Círculos abertos e cheios representam N e N -metilimidazole -methylpyrrole, respectivamente. Praça representa 3-clorotiofeno, e os diamantes representam β-alanina. Psoralen e biotina são denotados por P e B, respectivamente. (B) esquema cósmico. As células são tratadas com os derivados de poliamidas trifuncionais. Após reticulação com irradiação UV a 365 nm, as células são lADN genómico ysed e é cortado. Contas magnéticas revestidas com estreptavidina para capturar são adicionados aductos ADN-poliamida. O DNA é liberado e pode ser analisado por PCR quantitativa (qPCR) ou por sequenciamento de próxima geração (NGS). Por favor clique aqui para ver uma versão maior desta figura.
Protocolo
1. A reticulação em células vivas
- Comece com 2,5x10 ~ 7 células H1, ou outras células cultivadas.
NOTA: O número de células H1 corresponde a cinco placas de 10 cm (aproximadamente 40% de confluência). - Cultivar células em meio E8 em pratos revestidos sobre uma superfície que suporta as células estaminais pluripotentes (ver lista de materiais), e incubar a 37 ° C em atmosfera humidificada de 5% de CO 2. Células colheita enzimática (ver Lista de Materiais). Nota: Não permitir que as células H1 exceder 90% de confluência; confluência induz a diferenciação de células espontânea H1. Para contar as células, crescer um extra de 10 cm prato de células, levante as células enzimaticamente como descritos nos pontos 1.8-1.10, e contá-las com um hemocitômetro.
- Preparar os meios de cultura como descrito E8 em Chen et al. 54
- Antes da adição de poliamida, remover o meio de cultura gasto, com uma pipeta de Pasteur ligada a um sifão de vácuo. Adicionar meio fresco com comopipeta erological e pipeta distribuidor (8 mL por 10 cm de prato).
NOTA: Adicionar a mídia para o lado do prato, a fim de evitar perturbar as células. A partir deste ponto em diante proteger as células da luz para evitar prematura foto-reticulação. - Adicionar poliamida com uma pipeta (8 uL de 400 uM de poliamida em DMSO, concentração final 400 nM) directamente ao meio de cultura de cada prato. Agitar o prato para dispersar a poliamida uniformemente nos meios de comunicação.
NOTA: A concentração pode ser variada, mas assegurar que nenhuma toxicidade celular é observada para o tratamento seleccionado. - Incubam-se as células 24 h 37 ° C numa atmosfera humidificada de 5% de CO 2, e garantir que eles são protegidos da luz.
NOTA: O tempo de incubação pode ser variado para medir poliamida de ligação ao longo do tempo. - Lava-se cada prato de 10 cm com 4 ml de PBS (1,05 mM de fosfato de potássio monobásico, cloreto de sódio 155,17 mM, 2,97 mM de fosfato de sio dibico), utilizando uma pipeta e pipetas serológicas DISpenser. Aspirar PBS e adicionar 3 ml de meio de cultura E8.
- Com as luzes se apagaram, remover a tampa de 5 pratos de cultura e colocar as células em uma superfície plana do lado de fora da capa. Colocar um filtro de vidro ao longo dos 5 pratos de cultura para filtrar a luz com λ <300 nm. Coloque a fonte de UV em cima de filtro. Amostras Crosslink 30 min com 365 nm de irradiação UV (2,4 mW / cm2).
NOTA: O tempo de reticulação deve ser determinado empiricamente. - Transferência de placas de cultura de volta para a capa. Aspirar a mídia com uma pipeta Pasteur ligado a uma armadilha de vácuo. Lava-se cada prato de 10 cm com 4 ml de PBS. Aspirar o PBS. Adicionar 3 ml de enzima para a dissociação de células (ver Lista de Materiais) por 10 cm prato dissociar as células. Incubar 5 min a 37 ° C.
NOTA: Não pré-aquecer a enzima. - Extingue-se a enzima com 3 ml E8 mídia por prato. Transferência de células dissociadas em 15 ml de um tubo cónico.
NOTA: células colocar no gelo a partir deste ponto em diante. - Centrifuge células dissociadas 5 min, 500 x g a 4 ° C. Aspirar o sobrenadante para remover a enzima e mídia.
NOTA: Pausa ponto. As células podem ser congelados rapidamente em azoto líquido, armazenado a -80 ° C para subsequente processamento em um momento posterior.
2. Isolamento de cromatina
- Adicionar 1,2 ml de tampão cósmica (20 mM de Tris-HCl [pH 8,1], EDTA a 2 mM, NaCl 150 mM, 1% de Triton-X100, 0,1% de SDS) e pipeta cima e para baixo várias vezes para voltar a suspender o sedimento de células para cada amostra em 1,2 ml de tampão cósmica.
- Adicionar 133 ul de cada um dos 100 mM fluoreto de fenilmetilsulfonilo (PMSF), 100 mM de benzamidina e 150 ^ M, pepstatina inibidores da protease fresca para uma concentração final de 1 mM de PMSF e para benzamidina e 1,5 uM para pepstatina. Solução de divisão de ligado de células em duas âmbar 1,7 mL tubos de microcentrífuga.
- Sonicate com sonicator 35 min (10 seg on, 10 seg off, poder de 60%) para fragmentar o genoma para entre 100 and 500 pb.
- Manter o nível de solução da cromatina no tubo de microcentrífuga paralelamente ao nível da água no reservatório. Confirmar este nível por inspeção visual.
NOTA:. Confirmar que o DNA foi cortado com um gel de agarose a 1,5% 55 - Optimizar o tempo de sonicação empiricamente. Use uma quantidade mínima de gelo no reservatório para relaxar as amostras, e garantir o gelo não está a interferir entre as amostras eo chifre copo.
- Manter o nível de solução da cromatina no tubo de microcentrífuga paralelamente ao nível da água no reservatório. Confirmar este nível por inspeção visual.
- Centrifugar a 12000 xg amostra 10 min. Salve a solução aquosa que contém cromatina solúvel, transferindo-o para um novo tubo de microcentrífuga âmbar com uma pipeta. Descarte o sedimento.
- Transferir 110 mL (10%) de amostra para um novo tubo de microcentrífuga e rotulá-la DNA de entrada. Armazenar a -80 ° C. Guardar o resto da amostra cromatina em gelo para utilizao no passo 3.3.
3. Capturar de ligando-DNA As ligações cruzadas
- Usar uma pipeta para dispensar 60 ul streptavidin-esferas magnéticas revestidas num tubo de microcentrífuga. Adicionar 1 ml de tampão COSMIC e misture em uma nutator 5 min a RT. Coloque esferas magnéticas sobre separação magnética rack de 2 min para capturar esferas. Retirar tampão COSMIC com uma pipeta.
NOTA: Não permita que as contas a secar. Proceder imediatamente à etapa seguinte. - Adicionar amostra cromatina (~ 1 ml) a esferas (60 ul) e ressuspender as esferas. Incubar com cromatina revestidas com estreptavidina esferas magnéticas, pelo menos, 4 h em um rotativo, misturador de balanço, a 4 ° C. Incubar as amostras S / N se desejado.
4. Isolamento de ADN purificado por afinidade
- Lavar com grânulos de 7 min de intervalo à TA com os seguintes tampões de lavagem para remover as interacções não específicas. Use um rack de separação magnética para capturar grânulos após cada lavagem. Ressuspender as esferas após cada mudança de tampão de lavagem.
- Prepare tampões de lavagem em água e filtro (0,2 m) destilada deionizada antes do uso. Tampões de lavagem loja 1 e 2 a 4 ° C para several meses. Prepare Wash Buffer 3 fresco todos os dias. Adicionar e remover tampões de lavagem a partir da amostra com uma pipeta. Para 2 e 4, adicionar 1 e 3 (5 mM), respectivamente, nas lavagens. As amostras podem ser, adicionalmente, lavado duas vezes com tampão cósmica (uma vez 12 h, uma vez que 4 horas) antes das lavagens listados abaixo.
- Lavar uma vez com tampão de lavagem 1 (10 mM de Tris-Cl [pH 8,0], EDTA 1 mM, 3% de SDS). Lavar uma vez com tampão de lavagem 2 (Tris 10 mM-Cl [pH 8,0], LiCl 250 mM, EDTA 1 mM, NP40 a 0,5%, desoxicolato de sódio a 1%). Lavar duas vezes com Tampão de Lavagem 3 (4 M de ureia, 10 mM Tris-HCl [pH 7,5], EDTA 1 mM, 0,1% de NP-40). Lavar duas vezes com Tris EDTA (TE) de tampão (10 mM de Tris-Cl [pH 8,0], EDTA 1 mM).
- Ressuspender as pérolas em 200 ul de tampão TE com uma pipeta. Esta amostra de ADN capturado é referido como ADN purificado por afinidade (AP).
- Suplemento de entrada e AP ADN com 10x Crosslink Reversão Buffer (100 mM de Tris-Cl [pH 8,0], KOH 1 M, 4 mMEDTA) para concentração final de 1x. 56,57 Incubar 30 min a 90 ° C.
NOTA: 254 nm irradiação UV também pode ser usado para inverter a reticulação psoraleno, 58 mas ciclobutilo dímeros de pirimidina pode ser formado a partir de irradiação a este comprimento de onda e interferir com o processamento a jusante do ADN. - Para ADN de AP, colocar o tubo de microcentrífuga contendo a amostra sobre a separação magnética cremalheira 2 min à temperatura ambiente e líquido isolante (ADN) com uma pipeta. Transferir o DNA AP (que foi libertado a partir das pérolas) para um novo tubo de microcentrifugação âmbar.
NOTA: O DNA AP desejado está no líquido, não mais ligada às esferas. - Neutralizar de entrada e de DNA AP-se com HCl concentrado até pH 7 pela adição de cerca de 1 mL de HCl 6 N por 100 ul da amostra com uma pipeta. Confirmar as amostras são neutralizados pela adição de 0,5 ul ~ sobre papel de pH com uma pipeta ATENÇÃO:. HCl concentrado é um ácido forte corrosiva. Manusear de acordo com a sua instituição217; s diretrizes para o equipamento de proteção pessoal adequado.
- Adicionar RNase A (100 mg / mL) a 0,2 mg / mL concentração final de entrada e de ADN de AP. Incubar 1 hora a 37 ° C. Adicionar proteinase K (20 mg / ml) a 0,2 mg / mL concentração final de entrada e de ADN de AP, adicionar. Incubar 1 hora a 55 ° C.
- Purifica-se o ADN com um kit de limpeza de coluna de ADN (ver Lista de Materiais). Eluir 55 ADN em 58 ul de ADN de grau H2O Entrada da loja e AP amostras a -20 ou -80 ° C
- Analisar DNA AP por reação em cadeia da polimerase quantitativa (qPCR) com primers específicos de locos, e / ou por sequenciamento de última geração. Para qPCR, utilizar 2 ul de DNA AP por locus com os seguintes parâmetros: 1 ciclo de 95 ° C, 10 min e 40 ciclos de 95 ° C 20 seg, 54 ° C ou 56 ° C 20 seg, 72 ° C 40 seg.
NOTA: A temperatura de recozimento deve ser modificada de acordo com a temperatura de fusão dos pares de iniciadores utilizados. Selecione uma temperatu recozimentore que minimiza o ciclo de quantificação com qualquer amplificação não específica.
NOTA: Para a análise por sequenciamento de próxima geração, apontar para, pelo menos, 10 milhões mapeada lê. O número de leituras mapeados pode ser aumentada através do aumento da quantidade de DNA AP e pela combinação de menos amostras em uma corrida para o seqüenciamento. Mais lê melhorar a sensibilidade. Pacotes padrão para a análise ChIP-seq (por exemplo, local, 59 MACS, 60 e 61) SPP trabalhar com dados COSMIC-seq. Tal como acontece com outros métodos que dependem de sequenciamento de última geração, incluindo a sequenciação do genoma e ChIP-seq, regiões repetitivas criar ambiguidades em alinhamento e montagem e, assim, continuar a ser um desafio técnico.
Resultados
Para ter em conta a fragmentação genoma não uniforme e outras variáveis, o ADN purificado deve sempre ser normalizadas contra uma referência de ADN de entrada. Os iniciadores específicos para um locus de interesse pode ser utilizado. É também útil para analisar um locus em que não se espera que a molécula para se ligar, como um controlo negativo. Vemos um aumento> 100 vezes na ocupação de poliamida, após irradiação com luz de 365 nm (Figura 2).
DNA enrique...
Discussão
One of the primary challenges with conventional ChIP is the identification of suitable antibodies. ChIP depends heavily upon the quality of the antibody, and most commercial antibodies are unacceptable for ChIP. In fact, the Encyclopedia of DNA Elements (ENCODE) consortium found only 20% of commercial antibodies to be suitable for ChIP assays.50 With COSMIC, antibodies are replaced by streptavidin. Because polyamides are functionalized with biotin, streptavidin is used in place of an antibody to capture polyam...
Divulgações
A.Z.A. is the sole proprietor of Vista Motif, LLC and WINStep Forward.
Agradecimentos
We thank members of the Ansari lab and Prof. Parameswaran Ramanathan for helpful discussions. This work was supported by NIH grants CA133508 and HL099773, the H. I. Romnes faculty fellowship, and the W. M. Keck Medical Research Award to A.Z.A. G.S.E. was supported by a Peterson Fellowship from the Department of Biochemistry and Molecular Biosciences Training Grant NIH T32 GM07215. A.E. was supported by the Morgridge Graduate Fellowship and the Stem Cell and Regenerative Medicine Center Fellowship, and D.B. was supported by the NSEC grant from NSF.
Materiais
| Name | Company | Catalog Number | Comments |
| Phenylmethylsulfonyl fluoride (PMSF) | any source | ||
| Benzamidine | any source | ||
| Pepstatin | any source | ||
| Proteinase K | any source | ||
| Dynabeads MyOne Streptavidin C1 | Life Technologies | 65001 | |
| PBS, pH 7.4 | Life Technologies | 10010-023 | Other sources can be used |
| StemPro Accutase Cell Dissociation Reagent | Life Technologies | A1110501 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | We have tried other manufacturers of DNA columns with success. |
| TruSeq ChIP Sample Prep Kit | Illumina | IP-202-1012 | This Kit can be used to prepare COSMIC DNA for next-generation sequencing |
| Matrigel Basement Membrane Matrix | BD Biosciences | 356231 | Used to coat plates in order to grow H1 ESCs |
| pH paper | any source | ||
| amber microcentrifuge tubes | any source | ||
| microcentrifuge tubes | any source | ||
| pyrex filter | any source | Pyrex baking dishes are suitable | |
| qPCR master mix | any source | ||
| RNase | any source | ||
| HCl (6 N) | any source | ||
| 10-cm tissue culture dishes | any source | ||
| Serological pipettes | any source | ||
| Pasteur pipettes | any source | ||
| Pipette tips | any source | ||
| 15-ml conical tubes | any source | ||
| centrifuge | any source | ||
| microcentrifuge | any source | ||
| nutator | any source | ||
| Magnetic separation rack | any source | ||
| UV source | CalSun | B001BH0A1A | Other UV sources can be used, but crosslinking time must be optimized empirically |
| Misonix Sonicator | Qsonica | S4000 with 431C1 cup horn | Other sonicators can be used, but sonication conditions must be optimized empirically |
| Humidified CO2 incubator | any source | ||
| Biological safety cabinet with vacuum outlet | any source |
Referências
- Lee, T. I., Young, R. A. Transcriptional Regulation and Its Misregulation in Disease. Cell. 152, 1237-1251 (2013).
- Vaquerizas, J. M., Kummerfeld, S. K., Teichmann, S. A., Luscombe, N. M. A census of human transcription factors: function, expression and evolution. Nat Rev Genet. 10, 252-263 (2009).
- Meier, J. L., Yu, A. S., Korf, I., Segal, D. J., Dervan, P. B. Guiding the Design of Synthetic DNA-Binding Molecules with Massively Parallel Sequencing. J. Am. Chem. Soc. 134, 17814-17822 (2012).
- Dervan, P. B. Molecular recognition of DNA by small molecules. Bioorg. Med. Chem. 9, 2215-2235 (2001).
- Wemmer, D. E., Dervan, P. B. Targeting the minor groove of DNA. Curr. Opin. Struct. Biol. 7, 355-361 (1997).
- Eguchi, A., Lee, G. O., Wan, F., Erwin, G. S., Ansari, A. Z. Controlling gene networks and cell fate with precision-targeted DNA-binding proteins and small-molecule-based genome readers. Biochem. J. 462, 397-413 (2014).
- Mrksich, M., et al. Antiparallel side-by-side dimeric motif for sequence-specific recognition in the minor groove of DNA by the designed peptide 1-methylimidazole-2-carboxamide netropsin. Proc Natl Acad Sci U S A. 89, 7586-7590 (1992).
- Edayathumangalam, R. S., Weyermann, P., Gottesfeld, J. M., Dervan, P. B., Luger, K. Molecular recognition of the nucleosomal "supergroove". Proc Natl Acad Sci U S A. 101, 6864-6869 (2004).
- Suto, R. K., et al. Crystal structures of nucleosome core particles in complex with minor groove DNA-binding ligands. J. Mol. Biol. 326, 371-380 (2003).
- Gottesfeld, J. M., et al. Sequence-specific Recognition of DNA in the Nucleosome by Pyrrole-Imidazole Polyamides. J. Mol. Biol. 309, 615-629 (2001).
- Chenoweth, D. M., Dervan, P. B. Structural Basis for Cyclic Py-Im Polyamide Allosteric Inhibition of Nuclear Receptor Binding. J. Am. Chem. Soc. 132, 14521-14529 (2010).
- Raskatov, J. A., et al. Modulation of NF-κB-dependent gene transcription using programmable DNA minor groove binders. Proc. Natl. Acad. Sci. U.S.A. 109, 1023-1028 (2012).
- Yang, F., et al. Antitumor activity of a pyrrole-imidazole polyamide. Proc. Natl. Acad. Sci. U.S.A. 110, 1863-1868 (2013).
- Mapp, A. K., Ansari, A. Z., Ptashne, M., Dervan, P. B. Activation of gene expression by small molecule transcription factors. Proc. Natl. Acad. Sci. U.S.A. 97, 3930-3935 (2000).
- Ansari, A. Z., Mapp, A. K., Nguyen, D. H., Dervan, P. B., Ptashne, M. Towards a minimal motif for artificial transcriptional activators. Chem. Biol. 8, 583-592 (2001).
- Arora, P. S., Ansari, A. Z., Best, T. P., Ptashne, M., Dervan, P. B. Design of artificial transcriptional activators with rigid poly-L-proline linkers. J. Am. Chem. Soc. 124, 13067-13071 (2002).
- Nickols, N. G., Jacobs, C. S., Farkas, M. E., Dervan, P. B. Modulating Hypoxia-Inducible Transcription by Disrupting the HIF-1–DNA Interface. ACS Chemical Biology. 2, 561-571 (2007).
- Pandian, G. N., et al. A synthetic small molecule for rapid induction of multiple pluripotency genes in mouse embryonic fibroblasts. Sci. Rep. 2, 544 (2012).
- Pandian, G. N., et al. Synthetic Small Molecules for Epigenetic Activation of Pluripotency Genes in Mouse Embryonic Fibroblasts. Chem Bio Chem. 12, 2822-2828 (2011).
- He, G., et al. Binding studies of a large antiviral polyamide to a natural HPV sequence. Biochimie. 102, 83-91 (2014).
- Edwards, T. G., Vidmar, T. J., Koeller, K., Bashkin, J. K., Fisher, C. DNA Damage Repair Genes Controlling Human Papillomavirus (HPV) Episome Levels under Conditions of Stability and Extreme Instability. PLoS ONE. 8, e75406 (2013).
- Edwards, T. G., Helmus, M. J., Koeller, K., Bashkin, J. K., Fisher, C. HPV Episome Stability is Reduced by Aphidicolin and Controlled by DNA Damage Response Pathways. Journal of Virology. , (2013).
- Edwards, T. G., et al. HPV episome levels are potently decreased by pyrrole-imidazole polyamides. Antiviral Res. 91, 177-186 (2011).
- Dickinson, L. A., et al. Inhibition of RNA polymerase II transcription in human cells by synthetic DNA-binding ligands. Proc. Natl. Acad. Sci. U.S.A. 95, 12890-12895 (1998).
- Dickinson, L. A., et al. Arresting Cancer Proliferation by Small-Molecule Gene Regulation. Chem. Biol. 11, 1583-1594 (2004).
- Nickols, N. G., et al. Activity of a Py–Im Polyamide Targeted to the Estrogen Response Element. Molecular Cancer Therapeutics. 12, 675-684 (2013).
- Raskatov, J. A., Puckett, J. W., Dervan, P. B. A C-14 labeled Py–Im polyamide localizes to a subcutaneous prostate cancer tumor. Bioorg. Med. Chem. 22, 4371-4375 (2014).
- Jespersen, C., et al. Chromatin structure determines accessibility of a hairpin polyamide–chlorambucil conjugate at histone H4 genes in pancreatic cancer cells. Bioorg. Med. Chem. Lett. 22, 4068-4071 (2012).
- Chou, C. J., et al. Small molecules targeting histone H4 as potential therapeutics for chronic myelogenous leukemia. Molecular Cancer Therapeutics. 7, 769-778 (2008).
- Nickols, N. G., Dervan, P. B. Suppression of androgen receptor-mediated gene expression by a sequence-specific DNA-binding polyamide. Proc. Natl. Acad. Sci. U.S.A. 104, 10418-10423 (2007).
- Minoshima, M., Bando, T., Sasaki, S., Fujimoto, J., Sugiyama, H. Pyrrole-imidazole hairpin polyamides with high affinity at 5CGCG3 DNA sequence; influence of cytosine methylation on binding. Nucleic Acids Res. 36, 2889-2894 (2008).
- Warren, C. L., et al. Fabrication of duplex DNA microarrays incorporating methyl-5-cytosine. Lab on a Chip. 12, 376-380 (2012).
- Dudouet, B., et al. Accessibility of nuclear chromatin by DNA binding polyamides. Chem. Biol. 10, 859-867 (2003).
- Carlson, C. D., et al. Specificity landscapes of DNA binding molecules elucidate biological function. Proc. Natl. Acad. Sci. U.S.A. 107, 4544-4549 (2010).
- Warren, C. L., et al. Defining the sequence-recognition profile of DNA-binding molecules. Proc. Natl. Acad. Sci. U.S.A. 103, 867-872 (2006).
- Tietjen, J. R., Donato, L. J., Bhimisaria, D., Ansari, A. Z., Voigt, C. Chapter One - Sequence-Specificity and Energy Landscapes of DNA-Binding Molecules. Methods Enzymol. 497, 3-30 (2011).
- Puckett, J. W., et al. Quantitative microarray profiling of DNA-binding molecules. J. Am. Chem. Soc. 129, 12310-12319 (2007).
- Keles, S., Warren, C. L., Carlson, C. D., Ansari, A. Z. CSI-Tree: a regression tree approach for modeling binding properties of DNA-binding molecules based on cognate site identification (CSI) data. Nucleic Acids Res. 36, 3171-3184 (2008).
- Hauschild, K. E., Stover, J. S., Boger, D. L., Ansari, A. Z. CSI-FID: High throughput label-free detection of DNA binding molecules. Bioorg. Med. Chem. Lett. 19, 3779-3782 (2009).
- Lee, M., Roldan, M. C., Haskell, M. K., McAdam, S. R., Hartley, J. A. . In vitro Photoinduced Cytotoxicity and DNA Binding Properties of Psoralen and Coumarin Conjugates of Netropsin Analogs: DNA Sequence-Directed Alkylation and Cross-Link. 37, 1208-1213 (1994).
- Wurtz, N. R., Dervan, P. B. Sequence specific alkylation of DNA by hairpin pyrrole–imidazole polyamide conjugates. Chem. Biol. 7, 153-161 (2000).
- Tung, S. -. Y., Hong, J. -. Y., Walz, T., Moazed, D., Liou, G. -. G. Chromatin affinity-precipitation using a small metabolic molecule: its application to analysis of O-acetyl-ADP-ribose. Cell. Mol. Life Sci. 69, 641-650 (2012).
- Rodriguez, R., Miller, K. M. Unravelling the genomic targets of small molecules using high-throughput sequencing. Nat Rev Genet. 15, 783-796 (2014).
- Guan, L., Disney, M. D. Covalent Small-Molecule–RNA Complex Formation Enables Cellular Profiling of Small-Molecule–RNA Interactions. Angew. Chem. Int. Ed. 52, 10010-10013 (2013).
- White, J. D., et al. Picazoplatin, an Azide-Containing Platinum(II) Derivative for Target Analysis by Click Chemistry. J. Am. Chem. Soc. 135, 11680-11683 (2013).
- Rodriguez, R., et al. Small-molecule–induced DNA damage identifies alternative DNA structures in human genes. Nat Chem Biol. 8, 301-310 (2012).
- Bando, T., Sugiyama, H. Synthesis and Biological Properties of Sequence-Specific DNA-Alkylating Pyrrole−Imidazole Polyamides. Acc. Chem. Res. 39, 935-944 (2006).
- Anders, L., et al. Genome-wide localization of small molecules. Nat. Biotechnol. 32, 92-96 (2014).
- Jin, C., et al. Chem-seq permits identification of genomic targets of drugs against androgen receptor regulation selected by functional phenotypic screens. Proc. Natl. Acad. Sci. U.S.A. 111, 9235-9240 (2014).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22, 1813-1831 (2012).
- Erwin, G. S., Bhimsaria, D., Eguchi, A., Ansari, A. Z. Mapping Polyamide–DNA Interactions in Human Cells Reveals a New Design Strategy for Effective Targeting of Genomic Sites. Angew. Chem. Int. Ed. 53, 10124-10128 (2014).
- Hyde, J. E., Hearst, J. E. Binding of psoralen derivatives to DNA and chromatin: influence of the ionic environment on dark binding and photoreactivity. Biochemistry. 17, 1251-1257 (1978).
- Erwin, G. S., Bhimsaria, D., Rodríguez-Martínez, J. A., Grieshop, M. P., Ansari, A. Z. Genome-wide localization of polyamide-based genome readers reveals sequence-based binding to repressive heterochromatin. In preparation. , (2015).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nat Meth. 8, 424-429 (2011).
- Deliard, S., Zhao, J., Xia, Q., Grant, S. F. A. Generation of High Quality Chromatin Immunoprecipitation DNA Template for High-throughput Sequencing (ChIP-seq). J Vis Exp. (74), e50286 (2013).
- Shi, Y. B., Spielmann, H. P., Hearst, J. E. Base-catalyzed reversal of a psoralen-DNA cross-link. Biochemistry. 27, 5174-5178 (1988).
- Kumaresan, K. R., Hang, B., Lambert, M. W. Human Endonucleolytic Incision of DNA 3′ and 5′ to a Site-directed Psoralen Monoadduct and Interstrand. J. Biol. Chem. 270, 30709-30716 (1995).
- Cimino, G. D., Shi, Y. B., Hearst, J. E. Wavelength dependence for the photoreversal of a psoralen-DNA crosslink. Biochemistry. 25, 3013-3020 (1986).
- Heinz, S., et al. Simple Combinations of Lineage-Determining Transcription Factors Prime cis-Regulatory Elements Required for Macrophage and B Cell Identities. Mol. Cell. 38, 576-589 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biol. 9, R137 (2008).
- Kharchenko, P. V., Tolstorukov, M. Y., Park, P. J. Design and analysis of ChIP-seq experiments for DNA-binding proteins. Nat Biotech. 26, 1351-1359 (2008).
- Diamandis, E. P., Christopoulos, T. K. The biotin-(strept)avidin system: principles and applications in biotechnology. Clin. Chem. 37, 625-636 (1991).
- Martinson, H. G., True, R. J. On the mechanism of nucleosome unfolding. Biochemistry. 18, 1089-1094 (1979).
- Gloss, L. M., Placek, B. J. The Effect of Salts on the Stability of the H2A−H2B Histone Dimer. Biochemistry. 41, 14951-14959 (2002).
- Jackson, V. Formaldehyde Cross-Linking for Studying Nucleosomal Dynamics. Methods. 17, 125-139 (1999).
- Kasinathan, S., Orsi, G. A., Zentner, G. E., Ahmad, K., Henikoff, S. High-resolution mapping of transcription factor binding sites on native chromatin. Nat Meth. 11, 203-209 (2014).
- Teytelman, L., Thurtle, D. M., Rine, J., van Oudenaarden, A. Highly expressed loci are vulnerable to misleading ChIP localization of multiple unrelated proteins. Proc. Natl. Acad. Sci. U.S.A. 110, 18602-18607 (2013).
- . Phantompeakqualtools home page Available from: https://www.encodeproject.org/software/phantompeakqualtools/ (2010)
- Wang, D., Lippard, S. J. Cellular processing of platinum anticancer drugs. Nature Reviews Drug Discovery. 4, 307-320 (2005).
- Hurley, L. H. DNA and its associated processes as targets for cancer therapy. Nat. Rev. Cancer. 2, 188-200 (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados