Method Article
Análise transcricional por RNA Nascente PEIXES de
Neste Artigo
Resumo
Trophoblast giant cells (TGCs) play a key role in the placenta to ensure a healthy pregnancy. We present a protocol for assessing the transcriptional status of genes in TGCs by nascent fluorescent in situ hybridization on cryostat sections of post-implantation embryos or short-term cultures of embryonic day 7 ectoplacental cones.
Resumo
A placenta deriva de uma linhagem extra-embrionária, o trofectoderma. No blastocisto murino-implantação peri, células trofectoderma murais diferenciar em células trofoblásticas gigantes primário (CTGs) enquanto o trofectoderma polar que recobre a massa celular interna continua a proliferar depois diferenciar em CTGs secundárias. CTGs desempenhar um papel fundamental no desenvolvimento da placenta e são essenciais para uma gravidez bem sucedida. Investigação de regulação da transcrição de genes específicos durante o desenvolvimento pós-implantação pode dar insights sobre o desenvolvimento CTGs. As células do cone ectoplacental (CPE) a partir de embriões em 7-7,5 dias de gestação (E7-7.5), derivado do trofectoderma polar, diferenciam-se em CTGs secundária 1. CTGs pode ser estudada in situ, em secções de criostato de embriões em E7, embora o número de CTGs é muito baixa nesta fase. Um meio alternativo de analisar CTGs secundário é a utilização de culturas de curto prazo de EPCs individuais a partir E7 embriãos. Propomos uma técnica para investigar o estado transcricional de genes de interesse in vivo e in vitro, ao nível de uma única célula utilizando hibridização in situ fluorescente (FISH ARN) para visualizar transcritos nascentes. Esta técnica fornece uma leitura direta da expressão gênica e permite avaliar o estado cromossômica de CTGs, que são células endoreplicating grandes. Com efeito, uma característica fundamental da diferenciação terminal de CTGs é que eles saem do ciclo celular e submetidos a vários ciclos de abordagem endoreplication.This pode ser aplicado para detectar a expressão de qualquer gene expresso a partir de autossomas e / ou cromossomas sexuais e pode fornecer informações importantes no desenvolvimento mecanismos, bem como doenças da placenta.
Introdução
As células trofoblásticas gigantes mamíferos (CTGs) formar uma barreira entre os tecidos maternos e embrionárias. Eles mediar implantação e invasão do conceptus no útero e desempenham um papel crítico no desenvolvimento da formação da placenta. Eles produzem vários fatores de crescimento (citocinas) e hormônios da família e esteróides prolactina / Placentário Lactogen, necessária para o crescimento embrionário e sobrevivência. CTGs são grandes, mononucleadas e células poliplóides com um ciclo celular, o endocycle, que consiste em alternar fases S e G. Na verdade, CTGs são endoreplicating células, capazes de passar por várias rodadas de síntese de DNA, sem qualquer divisão 2. A fim de investigar o estado transcricional de genes in vivo, em CTGs em comparação com outros tipos de células embrionárias e extraembryonic, nascente PEIXES ARN pode ser realizada in vivo em secções criostáticas de 3 em estágios específicos pós-implantação. CTGs são facilmente reconhecíveis em seções devido a their grande tamanho e sua atividade transcricional gene pode ser gravado, mas o seu número em estágios pós-implante precoce é baixa. Escassez de CTGs no E7 seções embrionárias, levou-nos para realizar culturas de curto prazo de tecidos trophectodermal embrionárias para obter CTGs diferenciados, a fim de estudar a regulação da expressão de genes durante o desenvolvimento trofectoderma. Além disso, a fim de permanecer como fisiológico quanto possível, as linhas celulares estabelecidas ou seja trofectoderma células (TS) da haste não são sempre adequadas para investigar os mecanismos de desenvolvimento Geração de CTGs secundárias fornecer uma ferramenta valiosa para estudar gravidezes patológicas associadas a defeitos de CTGs devido ao gene anormal regulação em camundongos.
Células trofoblásticas do cone ectoplacental (EPC) são precursores de CTGs secundário 4. Diferenciação espontânea de EPCs cultivadas para CTGs secundário foi relatado anteriormente 5. No entanto, em contraste com CTGs primárias, estudos sobre a diferen TGC secundáriociação permaneceram limitadas, presumablydue às dificuldades de isolar explantes EPC livres de quaisquer tecidos maternos ou embrionárias. Nós adaptamos esses métodos, a fim de realizar RNA FISH em CTGs secundários derivados de embrião humano no E7, um estágio de pós-implantação de desenvolvimento, onde CTGs são muito poucos, mas poderia ser gerada a partir de precursores de EPC. PEIXES ARN para analisar transcritos primários nucleares nunca foi feito ao nível do nível de uma única célula em CTGs secundárias. Isto permite uma análise precisa da transcrição e foi usado para demonstrar a instabilidade epigenética de CTGs em fases pós-implante 3.
Um exemplo clássico da epigenética em mamíferos, a inativação do cromossomo X (ICX) é estudada no laboratório Heard 6. Neste processo, um dos cromossomas X 2 na fêmea é inactivado. O não-codificante XIST transcrição reveste o cromossoma X, a partir do qual ele é expresso em células do sexo feminino e provoca o silenciamento da maioria dos genes. Utilizando ARN FISH,transcritos nascentes de genes ligados ao X pode ser investigado como pode a acumulação de Xist ARN no cromossoma X inactivo (XI). Descrevemos aqui um procedimento para executar RNA FISH em seções de embriões pós-implantação e em culturas de curto prazo da EPC. Este protocolo adaptado a partir dos que foram utilizados para estudar ICX em diferenciação de células estaminais embrionárias fêmeas e embriões pré-implantação 7-11. Nós fornecemos exemplos de ICX em embriões femininos in vivo, assim como in vitro CTGs.
Protocolo
procedimentos com animais foram realizados de acordo com o cuidados com os animais institucional aprovado e usar comitê dos protocolos Institut Curie (CEEA-IC) (C 75-05-18). O trabalho também foi realizado sob a aprovação do Ministério do Ensino Superior e da Investigação francês para o uso de Organismos Geneticamente Modificados (número do contrato 5549CA-I).
1. Preparação de secções de criostato
- Recolha de embriões naturalmente F1 ovular murganhos C57BL / 6 x DBA ratinhos / 2J como descrito em Shea et al., 12. No dia 7 de gestação (E7), sacrificar rato 8-12 semanas de idade por deslocamento cervical. Recolha todo o concepto ou seja decídua 12.
- Isolar conceptos E7, conforme descrito em Shea et al., 12. Coloque-os em um prato de 60 milímetros Petri contendo PBS.
- Congelar o mouse concepto E7 para seções criostato.
- Prepare poços folha de alumínio adaptados 1cm de altura ( 'caseira' usando um Pasteur pip de vidroet). Depositar uma gota de meio de congelação de tecido na parte inferior deste recipiente de pequenas dimensões. Depositar o conceptus na orientação correcta (isto é, para as secções longitudinais do conceptus deve mantida na sua orientação horizontal).
- Encher o poço contendo o concepto com o tecido congelando médio. Suspendê-lo com uma pinça em vapor acima líquido N 2, a fim de permitir que ele congelar lentamente - em seguida, mergulhe o bloco no líquido N 2 durante alguns segundos até que fique branco. Subsequentemente, o bloco de transferir para um frasco de congelação e armazenar a -80 ° C (pode armazenar durante vários meses).
- Antes de crio-seccionamento, colocar o bloco congelado contendo o conceptus a -20 ° C no criostato para 30 min. Realizar criocortes de espessura de 8 um. Depósito 4 seções em um slide para permitir fixação eficiente. Seções lugar próximo o suficiente para caber sob uma lamela 18 x 18 mm 2.
- Verifique a qualidade doseções (intactos e sem arranhões) e a orientação do embrião (cortes longitudinais) com uma lupa e escolher as seções adequadas para análise posterior. O mais rapidamente possível depositá-los em uma jarra de Coplin e executar RNA FISH (Ver 3.3.2).
2. Preparação de CTGs Secundário
- Isolar a Conceptus (ver 1.1 e 1.2).
- Dissecar o E7 Ectoplacental Cone (EPC)
- Use um microscópio estereoscópico e placas de Petri contendo PBS estéril. Dissecar a decídua com uma pinça. Pierce da amostra e pinças abertas para rasgar os dois lados da decidua separados.
- Desembolsar o embrião. Utilize pontas de uma pinça fina fechados (Dumont # 5) em uma ação do tipo tesoura para separar o EPC do embrião propriamente dito. Use cuidado especial para se obter uma amostra perfeitamente limpo, separado do tecido materno, bem como a partir do cório e do saco vitelino. Lave a explantes EPC em PBS.
- Com um pouco de colher, transferir EPC dissecado indivíduo para um 4-well placa contendo uma lamela estéril em meio.
- Derivar CTGs de EPCs culturas de curto prazo.
- Preparar as placas de 4 poços contendo lamelas. Esterilizar 12 milímetros rodada lamelas de vidro de diâmetro por imersão em etanol, seguido por flame-secagem.
- Adicionar 0,5 ml de meio a cada poço de EPC (EPC meio: RPMI 1640 suplementado com 15% de FCS, 0,1 mM de 2-mercaptoetanol e antibióticos).
- único depósito, dissecaram-EPCs no centro da lamela no poço, juntamente com o meio de cultura. Use uma pinça fina para aplicar o EPC na lamela de vidro. Culturefor 3-5 dias a 37 ° C, 5% de CO 2. Explante indivíduo forma uma excrescência que se espalha como uma monocamada de CTGs achatada (Figura 2A).
3. RNA FISH
NOTA: Os protocolos são baseados naqueles descritos para células-tronco embrionárias (CES) em Chaumeil et al, 8 e Pollex e Heard 13..
- Prepa avançadaração de soluções estoque
- Preparar um tampão de acetato de sódio 3M pH 5,2.
- Preparar tampão de hibridação 2x contendo 40% (w / v) de sulfato de dextrano de sódio, 20x de BSA, 400 mM de vanadil ribonucleósido Complexo (VRC) em solução salina de 4x Citrato de sódio (SSC).
- Prepare meio de montagem contendo 90% (v / v) de glicerol, 0,1% (w / v) de p-fenilenodiamina, pH 9 em PBS.
- Soluções recentemente preparadas
- Preparar a solução fixadora composta por 3% de paraformaldeído recentemente preparada (PFA) em PBS. Preparar a solução de permeabilização que contém 0,5% de Triton-X-100 em PBS suplementado com 2 mM de VRC. Preparar o tampão de lavagem através da mistura de 50% de formamida (FA) e 2x SSC e ajustar o pH a 7,2-7,4.
- Preparar a solução de contra-coloração de ADN consistindo em 1? G / mL de DAPI (4 ', 6-diamidino-2-fenilindole dicloridrato) em 2 x SSC.
- Fixação e permeabilização em Preparação para FISH
- Para as células em lamelas, lavar as células em 1X PBS durante 5 mno. fixar as células em lamelas, ou secções embrionárias sobre as lâminas durante 10 minutos em solução fixadora (3% PFA) à TA.
- Enxaguar as células três vezes com PBS 1x. Permeabilizar as células durante 5 minutos em solução de permeabilização gelada em gelo. Lave as células três vezes com 70% de etanol. lamelas de loja em placas de 4 poços e slides na caixa do transporte 4-slide em etanol 70% a -20 ° C.
NOTA: As lamelas e as lâminas podem ser armazenados durante vários meses à temperatura de -20 ° C. As placas e caixas contendo os mesmos têm de ser selados com parafilme para evitar a evaporação do etanol.
- ADN sonda identificadora
- Rotular sondas de DNA por nick translation usando nucleótidos fluorescentes. Siga as instruções do fabricante (ver Tabela 1) 8.
- Para uma mistura de reacção de 50 ul, adicionar 1-2 ug de plasmídeo, cromossomas artificiais bacterianos (BAC) ou amplificação por deslocamento múltiplo (MDA) (para a amplificação de ADN, ver 3.4.4) de ADN de 17,5 ul de água, 2,5 ul de 0,2 mMSR-, SG-, Cy5-dUTP, 10 ul de 10 mM cada mistura dNTP (dGTP, dATP, dCTP), dTTP 5 ul 10 mM, 5 jul 10x nick tampão tradução e 8 ul nick enzima tradução.
- Incubar durante 16 horas a 15 ° C no escuro. Inactivar a reacção por congelação a -20 ° C. sondas Store para meses a -20 ° C.
- MDA
NOTA: Para DNA BAC, quantidade de DNA obtido após a preparação clássica é baixa, portanto, pode-se usar uma etapa de amplificação do DNA antes da rotulagem usando MDA. Usar um kit comercial e seguir as instruções do fabricante (ver Tabela 1). - Misture DNA de BAC 0,5 ul com 9,5 ul de tampão de amostra. Aquecer 3 min a 95 ° C. extinguir imediatamente em gelo; deixar em gelo 10 min. Prepare a mistura de tampão de reacção / enzima: (9,5 mL + 0,5 mL mistura de enzima) e manter em gelo.
- Misturar 10 ul de mistura de ADN + 10 ul de mistura de reacção tampão / enzima. Incubar a 30 ° C, pelo menos, 20 horas. Inativar a enzima por amostra de aquecimento 10 min a 65 & #176; C. Arrefecer a amostra a 4 ° C antes de armazenamento a -20 ° C.
- Verificar MDA por digestão. Adicionar 1 mL de MDA, 2 ul de tampão 10X, 2 ul de Hind III e 15 ul de H 2 O. Incubar a 37 ° C durante a noite. Executar lentamente num gel de agarose a 0,8-1% para confirmar a amplificação do DNA precisas. Os fragmentos de ADN claras de peso molecular elevado deve ser observado no gel.
- Probe Preparação
- Use 0,1 ou 1 mg sonda por lamela ou slide.
NOTA: Adicionar 2-5 ug de Cot-1 DNA se a concorrência é necessária, por exemplo, a maioria das sondas como eles podem conter sequências de repetição que hibridizam outra forma transversal e aumentar o fundo.- Para a precipitação, adicionam-se 5 ug de ADN de esperma de salmão, 1/10 do volume de 3 M acetato de sódio pH 5,2 e 3 volumes de etanol. Centrifugação a 16000 xg e 4 ° C durante 25 min. Lave pelotas com 70% de etanol e girar novamente por 5 min.
- sedimento seco durante 2 minutos num concentrador de vac / velocidade. Ressuspender em forma 100%amida em metade do volume necessário para a hibridação (por exemplo, 2,5 ul de lamela ou 7 ul de secções embrionárias na corrediça). Colocar 30 min a 37 ° C com agitação num termomisturador. Desnaturar durante 7 minutos a 75 ° C.
- Quench no gelo, ou se a concorrência for necessário colocar directamente a 37 ° C durante 30-60 minutos. Misturar a solução de sonda com um volume igual de solução de hibridação 2x.
- Use 0,1 ou 1 mg sonda por lamela ou slide.
- A hibridação e as lavagens
- Desidratar; lamelas em poços, e slides em um frasco Coplin, por lavagem sequencial em 1x 80%, 1x 95% e 2x 100% de etanol durante 5 minutos cada. lamelas secas e slides completamente.
- Para a hibridação, aplique a 5 ul mistura de hibridação da sonda (ver 3.5) sobre uma lâmina e abaixar a lamela, com células que enfrentam na mistura de hibridação. Para seções sobre slide, aplicar 14 ul mistura de hibridação da sonda (ver 3.5) e cobrir com uma lamela de 18 x 18 mm 2.
- Colocar as lâminas em câmara úmida (papel de tecido embebido em50% de FA / 2x SSC) e incubar a 37 ° C durante a noite, no escuro.
- As lavagens pós-hibridação:
- Adicionar 1 ml de 50% FA / 2x SSC para a lamela na lâmina para soltá-lo; Retirar as lamelas com cuidado a partir do slide (tomando cuidado para não raspar as células) e colocá-la do lado de células-se em uma placa de 4 poços contendo 50% FA / 2x SSC. Para seções sobre slide, remover a lamela de uma maneira similar e coloque o slide em uma jarra de Coplin contendo 50% FA / 2x SSC.
- Realizar 3 lavagens, cada uma durante 7 minutos com pré-aquecida até 50% FA / SSC 2x a 42 ° C ou 44 ° C durante lamela ou corrediça, respectivamente.
- Realizar 3 lavagens com pré-aquecido 2x SSC durante 5 min cada a 42 ° C ou 44 ° C durante lamela ou corrediça, respectivamente.
- núcleos por lavagem em contra-corante 2x SSC com 1 ug / ml de DAPI, durante 3 minutos, à TA. Lavar duas vezes com 2x SSC.
- Montagem de Lamelas e Slides
- Para as pequenas lamela de CTGs culta, aplicar 5 & #181; meio de montagem em um slide l. Coloque célula do lado da lamela para baixo no topo da gota.
- Para slides com seções, aplicar 15 ul médio no slide de montagem. Coloque um mm2 lamela de 22 x 22 em cima da gota.
- Evitar bolhas. Limpe solução de montagem excesso. Selar a lamela com uma pequena quantidade de unha polonês. Se possível, a imagem desliza imediatamente ou loja para até vários meses a -20 ° C.
4. Microscopia e Análise
- Adquirir imagens z eixo sequenciais em 3D em 0,3 mm utilizando um microscópio de fluorescência (objetiva 63X) e realizar a análise das pilhas de imagens em 3D usando o software ImageJ 14.
Resultados
CTGs podem ser identificados em secções de E7 por coloração DAPI, devido à sua localização no conceptus e o seu tamanho grande. Isto é ilustrado na Figura 1A em um corte longitudinal. ARN de FISH foi realizada em tais secções embrionárias, a fim de estudar a inactivação do cromossoma X nesta linhagem extra-embrionárias.
Várias lâminas ou lamelas podem ser processadas ao mesmo tempo. Ao utilizar diferentes corantes para rotular o ARN de diferentes genes, pode-se detectar transcritos primários diferentes no mesmo núcleo. Pelo menos 2 sondas podem ser misturados em conjunto, por exemplo, acoplado a Xist SG (sinal verde) e acoplado a ATRX SR (sinal vermelho). Um exemplo de uma TGC fêmea é mostrado na Figura 1B, onde 2 outras linhagens são representados, o embrião propriamente dito (E) e endoderma visceral (VE). Um núcleo TGC é mostrada Xist com ARN cobrindo o Chrom Xosome que é inativado (Xi), enquanto o outro cromossomo X que está ativa (Xa) exibe alguns pontinhos. Genes ATRX transcritos primários ligados ao X são expressos a partir do cromossoma X activo (não decorado por Xist) em várias cópias, devido a endoreplication. Isto ilustra monoalélicos expressão por exemplo, inactivação de ATRX em um cromossomo X.
Desde CTGs são heterogéneos no tamanho, como ilustrado na Figura 2B, só as mais importantes são registados. Três sondas podem também ser utilizadas, tal como ilustrado na Figura 2C para CTGs secundárias. Neste caso, Xist -SG (verde), dois genes ligados ao X: G6PD -SR (vermelho) e Huwe1-Cy (magenta) foram analisadas ao mesmo tempo no mesmo núcleo. Enquanto G6PD é monoallelically expressa como mostrado aqui (e previamente mostrado na CTGs secundárias 3) Huwe1 é biallelically expressa demonstrando a sua property escapar XCI. Endoreplication, com vários pontinhos seja, transcrições de nascentes cópias, também é evidente.
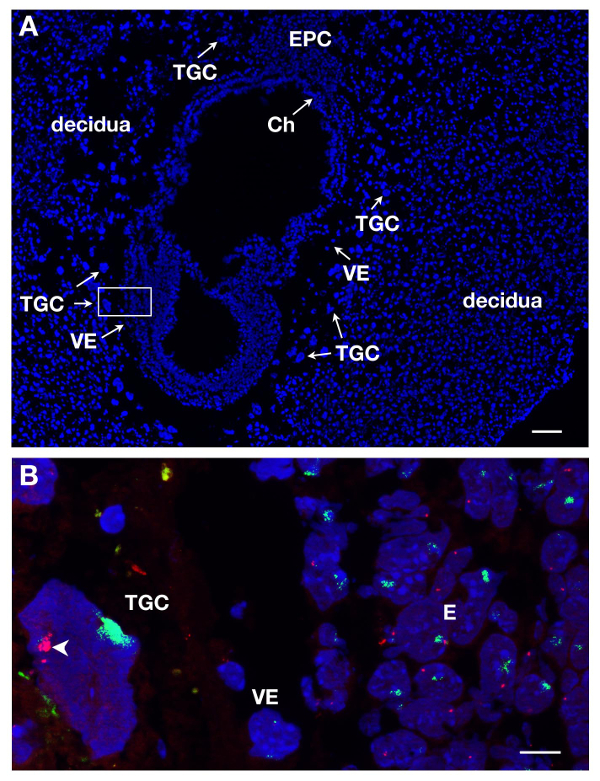
Figura 1. expressão transcrito primário em CTGs de E7 seção embrionária do sexo feminino. (A) Corte longitudinal do concepto E7 coradas com DAPI. O embrião e tecidos extra-embrionárias são cercados pelo tecido mãe, a decídua. A localização dos diferentes linhagens é feito mediante a coloração DAPI com objectiva 5X. CTGs são identificados devido ao seu grande tamanho. Ch, chorion, E, embrião, EPC, cone ectoplacental, VE, endoderme visceral, TGC, as células trofoblásticas gigantes. Barra de escala = 100 pm. (B) Maior ampliação da área de box-in em A (objetiva 63X) RNA FISH. ATRX transcrito primário em RNA vermelho e Xist em verde são visualizados no 3 linhagens = E, VE, TGC. Scale barra = 10 mm. Exemplo de um in vivo TGC onde ATRX é monoallelically expressa (Xa, ponta de seta) e em silêncio sobre o Xist -Revestido cromossomo X (Xi); vários sinais ATRX visto na Xa são devido a endoreplication. clone ATRX = ONT RP23-260I15. Por favor clique aqui para ver uma versão maior desta figura.
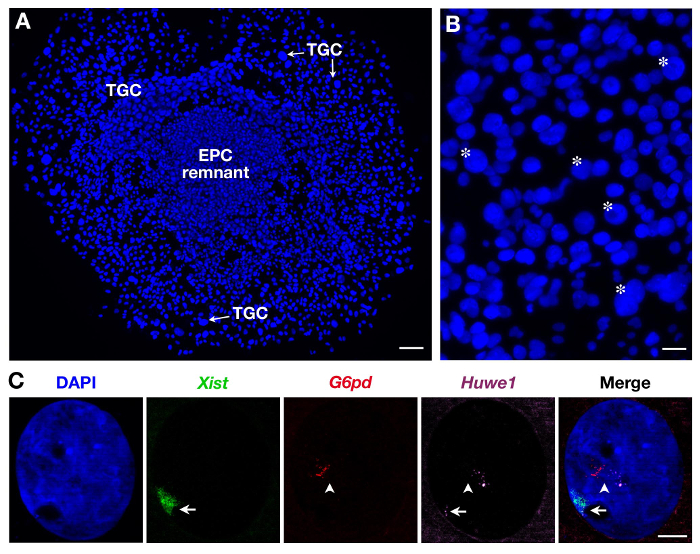
Figura 2. expressão transcrito primário em CTGs secundários derivados de EPC. (A) Vista geral de crescer CTGs secundário (objectivo x5). Barra de escala = 100 pm. (B) Asterisk indicam exemplo de CTGs de acordo com seu tamanho (objetiva de 10X). Barra de escala = 60 mm. (C) Exemplo de uma fêmea secondary TGC RNA FISH com o uso de 3 sondas (doma Xistem em verde, cobrindo o cromossomo X inativado: Xi) mostrando endoreplication da G6PD e Huwe1 transcritos primários (vários pontinhos, em vermelho e magenta). Enquanto G6PD é expressa monoallelically, expressão Huwe1 é bialélico neste núcleo Huwe1 = clone BAC RP24-157H12;. G6PD = BAC clone RP23-13D21. Xi = seta; Xa = seta (objetiva 63X). Barra de escala = 10 mm. Por favor clique aqui para ver uma versão maior desta figura.
Discussão
ARN nascente FISH é um método fácil e sensível para a análise de células isoladas de actividade de transcrição em tecidos embrionários, em diferentes estádios de desenvolvimento. O poder dessa abordagem é a capacidade de identificar diferentes linhagens embrionárias em qualquer fase particular de acordo com critérios morfológicos. No entanto, este também exige que o fundo mínimo de fluorescência está presente. Qualquer fundo torna a identificação de diferentes regiões embrionárias e tipos de células de desafio. Para assegurar fundo mínima, existem dois passos críticos neste protocolo. A primeira é a qualidade do criocorte e o segundo é a eficiência (relação sinal-ruído) do ARN de peixe, que depende do nível de expressão do gene e a qualidade da sonda. Para este último, as sondas são, por conseguinte, sempre testada em células cultivadas sobre lamelas de cobertura (células estaminais embrionárias ou células somáticas) anteriores para uso em secções.
Neste protocolo, nós fornecemos the técnicas necessárias para realizar a análise de FISH ARN em CTGs a partir de dois tipos diferentes de preparação da amostra (secções em criostato e culturas primárias de explantes embrionárias). Tal análise única célula permite mudanças dinâmicas na expressão do gene em diferentes estágios de pós-implantação a ser avaliadas.
Os métodos que descrevemos para gerar CTGs secundário (em E7-7.5 estágios pós-implantação) foram utilizados em nosso laboratório para estudar os padrões de inativação do cromossomo X de uma linhagem extra-embrionária, que é constituído de CTGs em estágios pós-implantação. desenvolvimento extra-embrionárias em roedores depende da diferenciação de CTGs. Com efeito, CTGs são essenciais para o desenvolvimento da placenta e, assim, embrionária. Defeitos na diferenciação TGC causar letalidade embrionária (para revisão ver referência 15). A atividade transcricional de CTGs usando RNA FISH nos permitiu demonstrar um estado de inativação do cromossomo X incomum de tal tipo de células durante o desenvolvimento do mouse 3.
Os métodos aqui apresentados, para obter e estudar CTGs secundárias pode ser aplicado para estudar vias moleculares no desenvolvimento das importantes tecidos extra-embrionárias que fazem parte de, tanto em ratos normais e mutantes. Estas abordagens podem ser adaptados para investigar o desenvolvimento TGC em outros mamíferos. Nossa análise envolveu o uso de embriões selvagens tipo de rato que nos permite avaliar a atividade transcricional de vários genes em in vivo CTGs e em in vitro CTGs derivados de explante EPC. Este método pode ser estendido para a análise de ratinhos transgénicos e / ou por adição de moléculas de inibidor no meio de cultura. Temos utilizado com sucesso este método de RNA FISH em outro tipo de CTGs, como CTGs primários que aparecem em um estágio inicial de desenvolvimento rato, blastocistos E3.0, que podem ser cultivados individualmente por 4-5 dias durante o qual desenvolvidos conseqüência, os ICM estar rodeado por grandes CTGs primários 16,17. transcritos nascentes para diferentesgenes, bem como Xist poderia ser visualizados e quantificados utilizando a abordagem FISH mesma RNA 3.
Imunofluorescência combinadas e ARN peixe também pode ser realizada em secções de crióstato, bem como in vitro em cultura CTGs 3. Isto demonstra que o método é bastante robusta, como transcritos primários são altamente susceptíveis à degradação e não é um requisito absoluto de RNase compostos livres. IF / RNA FISH fornece informações adicionais sobre os níveis de transcrição, localização celular e expressão da proteína, ao mesmo tempo em uma determinada célula. Embora, as secções de tecido embebidas em parafina foram previamente utilizados para detectar o ARN nascente em tumores humanos, mantendo a morfologia do tecido 18, nas nossas mãos, as secções do crióstato são mais apropriados para preservar tanto o ARN nascente e os epítopos necessários para a detecção de anticorpos durante imunofluorescência .
Em adição ao ARN de FISH, seguindo immunofluorescence, DNA cromossómico de FISH também pode ser realizada em CTGs secundárias usando sondas marcadas com fluorocromos, semelhantes aos utilizados para o ARN PEIXES 3. As sondas fluorescentes podem ser tanto plasmídeos fosmids / ou BACs, dUTPs marcados com fluorescentes como usado aqui, e descritos em Chaumeil et ai., 8. Alternativamente, os oligonucleótidos marcados com fluorescência pode ser utilizado 19. Uma vez que o ADN FISH não requer Strand-especificidade, as sondas de oligonucleótidos podem ser concebidas para atingir qualquer uma das duas cadeias complementares na região alvo. As sondas de DNA ramificado, em que a amplificação do sinal é conseguida por dois ciclos sequenciais de sondas específicas e amplificador também pode ser utilizada para melhorar a relação sinal-ruído de 20 sinais de FISH. Alternativamente, as sondas de ARN, tais como ribossondas podem ser usados, embora em nossas sondas baseadas em ADN garantir um melhor compromisso entre a qualidade do sinal, a especificidade e a facilidade de utilização.
Finalmente, acquisiti imagem 3Dem é essencial, a fim de obter a informação espacial requerida nestas células grandes, e pode ser realizada utilizando uma variedade de microscópios de fluorescência, tal como o microscópio ApoTome, ou microscópios de epifluorescência, com desconvolução, tais como o DeltaVision (GEH), ou outros microscópios adequado para secções de tecido de imagem, e grandes (> 20 mm) CTGs. Deve notar-se que, para os loci de ADN de cópia única, o sinal detectado ponteada por FISH ARN nascente ou FISH ADN pode não ser prontamente detectados por microscopia confocal.
Em conclusão, os métodos aqui descritos devem ser úteis tanto para a análise pormenorizada de TGS num contexto de desenvolvimento, mas também em outras situações de doença. Muitos genes que estão envolvidos no desenvolvimento e na função TGC em roedores são conservadas entre roedores e seres humanos, tais como factores de transcrição, proteases e moléculas de adesão celular 21. Rato CTGs são um modelo celular para genes que estudam que regulam o desenvolvimento de uma placentad, portanto, dar insights sobre doenças da placenta humana. Além disso, devido ao facto de que estas células são endoreplicating e uma vez que algumas células cancerosas envolver programas endocycle, além de processos de amplificação de genes, os métodos que descrevem deve também ser útil nas investigações de células individuais necessários para explorar mecanismos que conduzem ao genoma instabilidade em células cancerosas .
Divulgações
The authors have nothing to disclose.
Agradecimentos
Agradecemos Sophie Gournet para obter ajuda com ilustrações, Julie Chaumeil para a leitura do manuscrito, a instalação de alojamento dos animais e da plataforma de imagem da Unidade. Este trabalho recebeu apoio no âmbito do programa «Investissements d'Avenir» lançado pelo Governo francês e implementado pela ANR com as referências ANR-10-LabX-0044 e PSL ANR-10-IDEX-0001-02, o EpiGeneSys FP7 não. 257082 Rede de Excelência para EH, uma ERC Avançada Investigator adjudicação, no. 250367 e da UE FP7 SYBOSS conceder nenhuma. 242129 a EH Os autores gostariam de reconhecer o Celular e Tecidual plataforma de imagem da Genética e Departamento de Biologia do Desenvolvimento (UMR3215 / U934) do Institut Curie, membro da France-BioImaging (ANR-10-InsB-04), para obter ajuda com a luz microscopia.
Materiais
| Name | Company | Catalog Number | Comments |
| Stereomicroscope | Nikon | SMZ 1500 | |
| Stereomicroscope | Zeiss | Stemi SV6 | |
| Scissors Pascheff-Wolff | Moria | MC19 | |
| Dumont #5 forceps | Roth | PK78.1 | |
| 4-well tissue culture dishes | Nunc | 176740 | |
| 60 mm Petri dishes Falcon | Dutsher, France | 353004 | |
| 100 mm Petri dishes Falcon | Dutsher, France | 353003 | |
| Coverslips 18 mm x 18 mm | VWR | 631-1331 | |
| Coverslips 22 mm x 22 mm | VWR | 631-0125 | |
| 12 mm glass round coverslips | Harvard apparatus | 64-0712 | |
| Slides Superfrost plus | VWR | 631-9483 | |
| 4-slide Transport box Lockmailer | Dutsher, France | 40684 | |
| Cryotubes 1.8 ml Corning | Fisher Science | 10418571 | |
| Glass Coplin staining jars | Fischer Scientific | W1561L | |
| TissueTek O.C.T compound | VWR | 4583 | |
| RPMI 1640 medium | Invitrogen | 61870 | |
| Phosphate buffered saline (PBS) | Sigma-Aldrich | D1408 | 10x is used |
| Water | Sigma-Aldrich | W3500 | |
| Paraformaldehyde | Panreac Quimica, Spain | 141451 | 3% in PBS |
| Triton-X-100 | Euromedex | 2000-A | 0.5% final |
| Vanadyl ribonucleoside complex (VRC) | New England Biolabs, USA | S1402S | |
| Sodium dextran sulfate | Sigma-Aldrich | D8906 | |
| Bovine serum albumin (BSA) | New England Biolabs, USA | B9001S | |
| Formamide | Sigma-Aldrich | 47671-1L-F | aliquots kept at -20 °C |
| Illustra TempliPhi Kit Construct (Kit MDA) | Dutsher, France | 25-6400-80 | |
| Nick translation kit | Abbott, USA | 07J00-001 | |
| 20x SSC buffer concentrate | Sigma-Aldrich | S6639 | |
| Spectrum green dUTP | Abbott, USA | 02N32-050 | |
| Spectrum red dUTP | Abbott, USA | 02N34-050 | |
| Cy-5 dUTP | Dutsher, France | PA55022 | |
| Mouse Cot-1 DNA | Invitrogen | 18440016 | |
| DNA, MB grade | Invitrogen | Roche | DNA from fish sperm |
| 4′,6-diamidino-2-phenylindole dihydrochloride | Sigma-Aldrich | D9564 | DAPI |
| Glycerol | Sigma-Aldrich | G9012 | |
| p-phenylenediamine | Sigma-Aldrich | 695106 | |
| Centrifuge 5417R | Eppendorf, Germany | molecular biology grade | |
| Eppendorf concentrator plus | Eppendorf | ||
| Eppendorf Thermomixer comfort | Eppendorf | ||
| Liquiport Liquid pump | KNF Neuberger, Trenton, USA | ||
| Shake'N'Bake Hybridization oven | Boekel Scientific, USA | ||
| Cryostat | Leica | CM3050 |
Referências
- Cross, J. C. Genetic insights into trophoblast differentiation and placental morphogenesis. Sem. Cell Dev. Biol. 11, 105-113 (2000).
- Zybina, E. V., Zybina, T. G. Polytene chromosomes in mammalian cells. Int. Rev. Cytol. 165, 53-119 (1996).
- Corbel, C., Diabangouaya, P., Gendrel, A. -. V., Chow, J. C., Heard, E. Unusual chromatin status and organization of the inactive X chromosome in murine trophoblast giant cells. Development. 140, 861-887 (2013).
- Rossant, J., Tamura-Lis, W. Effect of culture conditions on diploid to giant-cell transformation in postimplantation mouse trophoblast. J. Embryol. Exp. Morphol. 62, 217-227 (1981).
- El-Hashash, A. H., Kimber, S. J. Trophoblast differentiation in vitro: establishment and characterization of a serum-free culture model for murine secondary trophoblast giant cells. Reproduction. 128, 53-71 (2004).
- Chow, J. C., Heard, E. X inactivation and the complexities of silencing a sex chromosome. Curr Opin Cell Biol. 3, 359-366 (2009).
- Chaumeil, J., Okamoto, I., Heard, E. X-chromosome inactivation in mouse embryonic stem cells: analysis of histone modifications and transcriptional activity using immunofluorescence and FISH. Methods Enzymol. 376, 405-419 (2004).
- Chaumeil, J., Augui, S., Chow, J. C., Heard, E. Combined immunofluorescence, RNA fluorescent in situ hybridization, and DNA fluorescent in situ hybridization to study chromatin changes, transcriptional activity, nuclear organization, and X-chromosome inactivation. Methods Mol. Biol. 463, 297-308 (2008).
- Okamoto, I., Otto, A. P., Allis, C. D., Reinberg, D., Heard, E. Epigenetic dynamics of imprinted XCI during early mouse development. Science. 303, 644-664 (2004).
- Okamoto, I., Arnaud, D., Le Baccon, P., Otte, A. P., Disteche, C. M., Avner, P., Heard, E. Evidence for de novo imprinted X-chromosome inactivation independent of meiotic inactivation in mice. Nature. 438, 369-373 (2005).
- Patrat, C., Okamoto, I., Diabangouaya, P., Vialon, V., Le Baccon, P., Chow, J., Heard, E. Dynamic changes in paternal X-chromosome activity during imprinted X-chromosome inactivation in mice. Proc. Natl. Acad. Sci. 106, 5198-5203 (2009).
- Geijsen Shea, K., N, Dissection of 6.5 dpc mouse embryos. J. Vis. Exp. (2), e160 (2007).
- Pollex, T., Piolot, T., Heard, E. Live-cell imaging combined with immunofluorescence, RNA, or DNA FISH to study the nuclear dynamics and expression of the X-inactivation center. Methods Mol. Biol. 1042, 13-31 (2013).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9, 671-675 (2012).
- Hemberger, M. IFPA award in placentology lecture - characteristics and significance of trophoblast giant cells. Placenta. 29, 4-9 (2008).
- Carney, E. W., Prideaux, V., Lye, S. J., Rossant, J. Progressive expression of trophoblast-specific genes during formation of mouse trophoblast giant cells in vitro. Mol. Reprod. Dev. 34, 357-368 (1993).
- Shin, J., et al. Maternal Rnf12/RLIM is required for imprinted X-chromosome inactivation in mice. Nature. 467, 977-981 (2010).
- Capodieci, P., Donovan, M., Buchinsky, H., Jeffers, Y., Cordon-Cardo, C., Gerald, W., Edelson, J., Shenoy, S. M., Singer, R. H. Gene expression profiling in single cells within tissue. Nat Methods. 9, 663-665 (2005).
- Beliveau, B. J., Joyce, E. F., Apostolopoulos, N., Yilmaz, F., Fonseka, C. Y., McCole, R. B., Chang, Y., Li, J. B., Senaratne, T. N., Williams, B. R., et al. Versatile design and synthesis platform for visualizing genomes with Oligopaint FISH probes. Proc. Natl. Acad. Sci. 109, 21301-21306 (2012).
- Kenny, D., Shen, L., Kolberg, J. A. Detection of viral infection and gene expression in clinical tissue specimens using branched DNA (bDNA) in situ hybridization. J. Histochem. Cytochem. 50, 1219-1227 (2002).
- Cross, J. C., Baczyk, D., Hemberger, M., Hugues, M., Simmons, D. G., Yamamoto, H., Kingdom, J. C. Genes, development and evolution of the placenta. Placenta. 24, 123-130 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados