É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Definindo substrato Especificidades para a lipase e fosfolipase candidatos
Neste Artigo
Resumo
Many predicted (phospho)lipases are poorly characterized with regard to their substrate specificities and physiological functions. Here we provide a protocol to optimize enzyme activities, search for natural substrates, and propose physiological functions for these enzymes.
Resumo
Microorganismos produzem um largo espectro de lipases (fosfo) que são secretadas, a fim de fazer substratos externos disponível para o organismo. Alternativamente, outros (fosfo) lipase pode ser fisicamente associado com o organismo produtor causando uma rotação de lípidos intrínsecas e frequentemente dando origem a uma remodelação das membranas celulares. Embora potenciais (fosfo) lipase pode ser previsto com um certo número de algoritmos quando a sequência do gene / proteína está disponível, não tem frequentemente sido obtida prova experimental das actividades enzimáticas, especificidades de substrato, e funções fisiológicas potenciais. Este manuscrito descreve a optimização das condições de ensaio para a (fosfo) lipase prospectivos com especificidades de substrato desconhecidos e como empregar estas condições optimizadas na pesquisa para o substrato natural de um respectivo (fosfo) lipase. Usando substratos cromogénicos artificiais, tais como derivados de p-nitrofenilo, pode ajudar a detectar uma menoractividade enzimática para um (fosfo) lipase previsto, sob condições padrão. Tendo encontrado uma actividade enzimática, tais menor, os parâmetros distintos de um ensaio de enzima podem ser variados de modo a obter uma hidrólise mais eficiente do substrato artificial. Depois de ter determinado as condições sob as quais uma enzima funciona bem, uma variedade de substratos naturais potenciais devem ser ensaiadas quanto à sua degradação, um processo que pode ser seguido utilizando métodos cromatográficos diferentes. A definição das especificidades de substrato para enzimas novas, frequentemente fornece hipóteses para um potencial papel fisiológico destas enzimas, que podem então ser testados experimentalmente. Seguindo essas orientações, fomos capazes de identificar uma fosfolipase C (SMc00171) que degrada fosfatidilcolina para fosfocolina e diacilglicerol, em um passo crucial para a remodelação das membranas na bactéria Sinorhizobium meliloti das condições de limitação de fósforo de crescimento. Para duas previu patatin-como fosfolipases (SMc00930 e SMc01003) do mesmo organismo, que poderia redefinir suas especificidades de substrato e esclarecer que SMc01003 é uma lipase diacilglicerol.
Introdução
Lípidos à base de glicerol como triglicerídeos e fosfolipídios (glicero) constituem importante e, provavelmente, as classes de lípidos mais conhecidos 1. Triacilgliceróis (tags) são as gorduras e óleos, que normalmente funcionam como lipídios de armazenamento e, portanto, como potenciais fontes de energia e de carbono. As tags podem ser degradados por lipases, que são frequentemente segregadas pelo organismo produtor de digerir TAGs externas e torná-los disponíveis como fontes de carbono. Além disso, lipases têm sido amplamente estudados ao longo dos anos devido às suas importantes aplicações biotecnológicas 2.
Devido à sua natureza anfifílica e sua forma quase cilíndrica, propriedades de formação de membrana (glicero) fosfolipídios de exposição e normalmente constituem os principais componentes lipídicos de uma membrana de bicamada 3. Em microorganismos simples, tais como a bactéria Escherichia coli, apenas três variantes principais grupo de cabeça, fosfatidilglicerol (PG), a cardiolipina (CL), e phosphatidylethanolamine (PE) são encontrados, embora se deva ter presente que cada um deles pode ser substituído com um número considerável de diferentes cadeias de acilo gordo na posição sn-1 ou sn posição -2 dando origem a um grande número de diferentes espécies moleculares 4 . Outras bactérias pode ter outros fosfolipídios em adição ou em vez disso. Por exemplo, Sinorhizobium meliloti, uma bactéria do solo, que é capaz de formar uma simbiose nódulo da raiz de fixação de azoto, com a alfafa de leguminosas (Medicago sativa), contém, além de um segundo fosfolípido PE zwitteriónico, fosfatidilcolina (PC) 5. Além disso, os lipídios não contendo fósforo ou glicerol pode ser anfifílico e fazem parte da membrana celular. Por exemplo, em condições de crescimento limitativos de fósforo, em S. meliloti, (glicero) fosfolípidos são em grande parte substituído por lípidos da membrana que não contêm fósforo, ou seja, os sulfolípidos, lípidos ornitina, e diacylglyceryl trimethylhomoserine (DGTS) 6. Em bactérias, DGTS é formado a partir de diacilglicerol (DAG) em uma via de duas etapas 7, mas a fonte para geração de DAG não foi claro. Experimentos pulse-chase sugeriu que o PC pode ser um precursor para DGTS 8 e utilizando a metodologia descrita neste manuscrito foi possível identificar uma fosfolipase C (PLCP, SMc00171) que é formado sob condições limitantes de fósforo e que pode converter PC em DAG e fosfocolina 8.
Em um estudo separado, descobrimos que uma sintetase de acil-CoA (FADD) -deficient mutante de S. meliloti ou de Escherichia coli acumulada ácidos graxos livres ao entrar em fase estacionária de crescimento de 9. Embora estes ácidos gordos pareceu ser derivados de lípidos da membrana, a origem exacta para os ácidos gordos livres ou a enzima (s) libertando-os não eram conhecidos. Mais uma vez, empregando a estratégia delineada neste manuscrito, dois 10 (fosfo) lipases patatina-like (SMc00930 e SMc01003) que contribuiu para a formação de ácidos gordos livres em S. meliloti 11 foram previstos. Surpreendentemente, SMc01003 DAG utilizado como substrato para convertê-lo e, finalmente, monoacilglicerol glicerol e ácidos gordos livres 11. Por conseguinte, é uma lipase SMc01003 DAG (DGLA).
Embora um número de algoritmos de previsão do potencial existe (fosfo) lipases 12,13, a sua função precisa e papel fisiológico normalmente não é conhecido. Aqui destacamos um protocolo, clonar e sobre-expressar lipases (fosfo) previstos ou potenciais. Este manuscrito explica como ensaios de enzimas pode ser desenvolvido e optimizado para o (fosfo) lipase sobre-expresso utilizando substratos cromogénicos artificiais. Nós fornecemos exemplos como com um ensaio enzimático otimizado do real (fosfo) lipase substrato podem ser encontradas e como estas descobertas podem enriquecer a nossa compreensão da fisiologia microbiana.
Protocolo
1. O clone e gene estrutural para sobre-expressar a lipase Previsto
- Usando reacção em cadeia da polimerase (PCR) 14 e oligonucleótidos específicos (Tabela 1) 15, amplificar o gene de interesse (smc01003, smc00930, ou smc00171), previsto para codificar para uma lipase ou fosfolipase, a partir do ADN genómico do organismo hospedeiro (ou seja , S. meliloti).
- Introduzir locais específicos de restrição (com a sequência dos oligonucleótidos concebidos). Digerir o fragmento de DNA amplificado com as enzimas de restrição correspondentes e cloná-lo num vector de expressão, tais como plasmídeos da série pET 16.
- Após verificar a sequência de DNA correcta para o gene clonado, transformar o vector de uma estirpe de expressão, tais como Escherichia coli BL21 (DE3) pLysS 16.
- Preparar uma pré-cultura durante a noite do hospedeiro de expressão E. coli BL21 (DE3) pLYSS, albergando o respectivo vector pET com o gene clonado ou o vector vazio, em 100 ml de frascos de cultura contendo 20 ml de caldo de Luria Bertani (LB) mais os 17 antibióticos necessários. Cultura das células a 30 ° C (ou à temperatura habitual de crescimento da bactéria a partir da qual origina a lipase).
- Usando as pré-culturas durante a noite, inocular 500 ml de meio LB pré-aquecido (mais os antibióticos necessários) em balões de 2 L de cultura para se obter uma densidade óptica inicial a 620 nm (OD 620) = 0,05. Seguir o crescimento das culturas e a uma DO 620 = 0,3, adicionar isopropil-β-D-tiogalactósido (IPTG) até uma concentração final de 100 uM, e incuba-se sob agitação a 30 ° C durante um período de 4 h.
- No final do período de incubação, cada cultura transferir para um tubo de centrífuga de 500 mL e centrifugação a 5000 xg, a 4 ° C durante 30 min. Ressuspender sedimentos celulares bacterianos em 5 ml de tampão de suspensão (por exemplo, SMc00930- e células que expressam SMc01003 em Tris-HCl 50 mM pH 8,0 e as células que expressam SMc00171-em 50 mM de dietanolamina-HCI pH 9,8). Armazenar as suspensões de células a -80 ° C até à sua utilização.
2. Preparar extractos de proteínas livres de células e determinar a concentração de proteína
- Descongelar as suspensões de células bacterianas e armazenar em gelo. Passe suspensões de células três vezes através de uma célula de pressão a frio em 20.000 lb por no 2. Remover as células intactas e os resíduos celulares por centrifugação a 5000 xg durante 30 min a 4 ° C.
- Após centrifugação, preparar alíquotas de 100 e 500 ul do sobrenadante para análise subsequente e armazená-las a -80 ° C até à sua utilização.
- Utilize uma das alíquota de 100 ul para determinar a concentração de proteínas de extractos livres de células distintas por um método de escolha ou como descrito 18.
3. Use substratos artificiais para otimizar EnzymeAtividades de (Fosfo) lipases
- Para uma cobertura inicial de actividades enzimáticas distintas, utilizar substratos artificiais que produzem um produto de cor aquando da hidrólise, tais como p-nitrofenol (p) -np.
- Para os ensaios de enzima já otimizada com substratos de éster de p-nitrofenilo (artificiais descritos para a fosfolipase C PLCP (SMc00171), bem como para as fosfolipases-patatina como preditos SMc00930 e SMc01003), utilizar esquemas de pipetagem descrita na Tabela 2.
- Ao explorar um novo potencial de lipase (fosfo), preparar um primeiro ensaio padrão de enzima contendo Tris-HCl a 50, pH 8,5, NaCl 100 mM, 0,05% de Triton X-100, p-nitrofenilo composto contendo (p-nitrofenil fosfato 0,5 mM , fosfato de bis-p-nitrofenilo, p-nitrofenilo decanoato, palmitato ou p-nitrofenilo), e extracto proteico livre de células (veja 1, 3, 10, 30, 100, 300, e 1000 ug) num volume total de 1 ml em 1 ml cuve plásticoTTES.
NOTA: O uso de pH alcalino (Figura 1) quando se segue a hidrólise do éster p-nitrofenilo num ensaio contínuo. Em alternativa, usar ensaios de ponto de utilização única para uma gama de valores de pH, a adição de NaOH no final do período de incubação para terminar a reacção enzimática e para assegurar que todos os p -np está presente na forma de fenolato. - Seguir o curso de tempo para um aumento da absorvância a 405 nm, devido à formação de p -np, num espectrofotómetro a 30 ° C durante um período de 5 min. Quantificar a formação inicialmente linear de p -np por determinação da inclinação inicial do aumento de absorvência por unidade de tempo.
- Calcular a variação da concentração (Sc) para p -np usando a lei de Lambert-Beer (Aa = ε Sc d) 1.
NOTA: Aa representa a alteração linear da absorvância determinada, ε é o coeficiente de extinção molar, no respectivo comprimento de onda (em unidades de M-1 cm-1), d é o comprimento do caminho de luz (1 cm), e Sc é a mudança de concentração (em unidades de M) a ser determinado.- Considerando-se que o volume de ensaio é de 1 ml, calcular a quantidade de p -np formado.
Observação: o valor = concentração x volume. - Calcula-se a actividade da enzima através da divisão da quantidade de p -np formado pelo tempo em que é formado. Determinar a actividade enzimática específica, dividindo a actividade enzimática pela quantidade de proteína (em mg) que foi responsável pela geração desta actividade.
- Considerando-se que o volume de ensaio é de 1 ml, calcular a quantidade de p -np formado.
- Comparar as alterações de absorção provocadas por extratos de proteína em que um gene candidato (smc00171, smc00930 ou smc01003) tinham sido expressas com extratos que abrigam única um vetor vazio.
NOTA: A fim de continuar com as etapas a seguir, as actividades específicas, causadas por extratos de proteína em que um gene candidato tinha sido expressa, deve ser pelo menos duas vezes ou mminério de que os valores obtidos para as actividades específicas causadas por extractos de proteína única que abrigam um vector vazio. - Para novas experiências, selecione essas condições em que a hidrólise do composto p-nitrofenilo contendo é mínima com extratos livres de células (isto é, o vector vazio) e para o qual a formação mais pronunciado da p -np eo ânion p -nitrophenolate (Figura 1) pode ser observado quando são empregues extractos de proteína, em que um gene candidato tinha sido expressa.
- Após a determinação da actividade da enzima inicial em 3,1, optimizar as condições de ensaio para a enzima respectiva, fazendo variar o pH, o tipo de tampão, tampão de força, as concentrações de NaCl, detergentes, tais como Triton X-100, e a ausência ou a presença de diferentes catiões bivalentes.
- Para diferentes concentrações de cada uma das variáveis, determinar a actividade enzimática específica (ver 3.1.4.2) (o número mais elevado obtido define a condiçãoda actividade enzimática máxima). Utilizar a combinação das condições óptimas encontradas para cada variável para definir um ensaio enzimático optimizado no qual cada uma das variáveis está presente na sua concentração óptima.
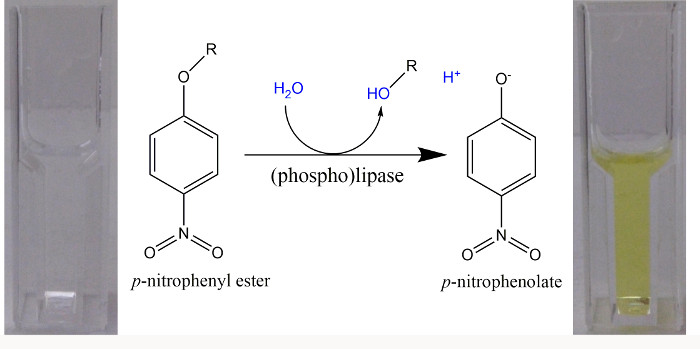
Figura 1. p-nitrofenilo ésteres como substratos artificiais para (fosfo) lipase em um ensaio espectrofotométrico. Após a hidrólise de ésteres de p-nitrofenilo, um ácido (R-OH) e p-nitrofenol (p -np) são formados. Devido ao pKa = 7,2 para a dissociação do fenólico H + a partir de p -np, a um pH> 9,2 mais do que 99% estão na forma p -nitrophenolate amarelo brilhante e um coeficiente de extinção molar de 18.000 M-1 cm - 1 pode ser utilizado num comprimento de onda de 405 nm para a quantificação do livre p -nitrophenolate 22. Quando foram utilizados buffers com um pH de 8,5, a absorvância foi determinada a 400 nm e um coeficiente de extinção molar de 14.500 M-1cm-1 foi empregado 23. Por favor clique aqui para ver uma versão maior desta figura.
NOTA: Depois de ter definido as condições ideais para a atividade da enzima de interesse, embarcar na busca do substrato reais / fisiológica desta lipase. Em princípio, tomar dois, muitas vezes complementares, abordagens para atingir este objetivo, uma abordagem in vivo ou uma abordagem in vitro.
4. In Vivo Identificação da condição fisiológica do substrato de uma lipase
ove_content "> NOTA:. Em uma abordagem in vivo, expressar a lipase de interesse num organismo hospedeiro 8,11 para registrar ao longo do tempo se a expressão da lipase altera o perfil lipídico host's Em outra abordagem in vivo, gerar um mutante deficiente do gene de interesse e 8,11 estudar se o seu perfil de lípidos é diferente do tipo selvagem versão 6,8,11. a fim de obter uma avaliação quantitativa de perfil lipídico de um organismo, um método simples consiste na radiomarcação de compostos celulares , extracção dos lípidos, separando-os por meio de cromatografia, e quantificar os lípidos separados marcados radioactivamente.- Radiomarcação de lipídios.
- Prepara-se uma pré-cultura durante a noite de um organismo de interesse (meliloti E. coli ou S.) em 5 ml de meio de cultura desejada (meio complexo ou meio mínimo definido) e crescer a 30 ° C.
- A partir do pré-cultura, inocular em 20 ml do SAme meio fresco num frasco de cultura de 100 ml para se obter uma DO 620 = 0,3 inicial para a cultura.
- Tomar uma aliquota (1 ml) da cultura em condições estéreis e transferir para um poliestireno de 14 ml estéril tubo de fundo redondo.
- Adicionar 1 uCi de [1- 14 C] acetato de etilo (60 mCi por mmole) para a cultura de 1 ml.
- Incubar a cultura líquida sob agitação a 30 ° C durante um período de 24 h.
- No final do período de incubação, a transferência da cultura para um tubo de microcentrífuga de 1,5 ml e centrifugar a 12,000 xg à temperatura ambiente durante 5 min.
- Ressuspender o sedimento em 100 ul de água. Neste ponto, armazenar a suspensão de células a -20 ° C ou imediatamente continuar com a extracção de lípidos polares (secção 4.2).
- Extracção de lipidos polares.
NOTA: O método descrito aqui segue essencialmente o procedimento descrito por Bligh e Dyer 19.- Para os 100 ul de células aquosa suspension, adicionar 375 ul de solução de metanol: clorofórmio (2: 1; vol / vol).
- Vortex durante 30 segundos e incuba-se durante 5 min à temperatura ambiente.
- Centrifugar 5 min a 12.000 x g à temperatura ambiente.
- Transferir o sobrenadante para um novo tubo de microcentrifugação de 1,5 ml.
- Adicionar 125 ul de clorofórmio e 125 ul de água, vortex 30 seg.
- Centrifugar 1 min a 12.000 x g à temperatura ambiente.
- Transferir a fase inferior de clorofórmio para um tubo fresco e seco com uma corrente de azoto gasoso.
- Dissolve-se lípidos secos em 100 ul de clorofórmio: solução de metanol (1: 1; v / v).
NOTA: neste momento, uma parte alíquota de 5 ul da solução de lípido pode ser quantificada por contagem de cintilação líquida. - Para a análise cromatográfica em camada fina (TLC), secar-se os restantes 95 mL com uma corrente de azoto gasoso e dissolver de novo os lípidos secos em 20 ul de clorofórmio: solução de metanol (1: 1; v / v). Usar uma aliquota de 3 uLpor análise de TLC.
- A separação de lípidos polares, por cromatografia em camada fina (TLC).
NOTA: Dependendo das classes de lípidos a ser analisado, diferentes combinações de fases sólidas e móveis pode ser utilizado para a separação. Aqui uma separação típica de lípidos carregados e polares outro, mais adequados para lípidos polares neutros, utilizando cromatografia em camada fina (HPTLC) folhas de alumínio em gel de sílica de alto desempenho como fase sólida, são descritos.- Separação de lipídios polares cobradas pelo bidimensional TLC (2D-TLC).
- Aplicar uma aliquota de 3 uL de amostra de lípido a um canto de uma folha de alumínio em gel de sílica HPTLC (10 x 10 cm), a 2 cm do bordo da placa.
- Preparar e misturar a fase móvel (140 ml de clorofórmio, 60 ml de metanol, e 10 ml de água) para a separação na primeira dimensão.
- Unte uma câmara de revelação TLC internamente com papel para cromatografia.
NOTA: Este é garantir que a fase de gás da câmaravai ser saturado rapidamente (dentro de 30 min) após a fase móvel para a primeira dimensão foi adicionado à câmara e a câmara foi fechada com uma placa de vidro. - Preparar e misturar a fase móvel (130 ml de clorofórmio, 50 ml de metanol, e 20 ml de ácido acético glacial) para a separação na segunda dimensão e transferência para uma segunda câmara de CCF internamente revestida com o papel de cromatografia e deixar a câmara de saturar.
- Transferir cuidadosamente a folha de alumínio em gel de sílica HPTLC com a amostra de lípido seca para a primeira câmara e desenvolver (ou seja, realizar cromatografia), a placa durante 60 minutos na câmara fechada na primeira dimensão 5.
- Remova a placa da câmara e deixa solventes secar numa câmara de fluxo para 30 min.
- Depois de ligar a placa por 90 graus no que diz respeito ao anterior cromatografia, transferir a folha de alumínio em gel de sílica HPTLC, em que os lípidos foram separadas em uma dimensão, com o second câmara e o desenvolvimento da placa durante 60 minutos na segunda dimensão 5.
- Remover a folha a partir da câmara e permitir que os solventes se secar numa câmara de fluxo durante pelo menos 2 h.
- A separação de lípidos polares neutros.
- Aplicar 3 mL alíquotas de amostras de lípido sobre uma folha de alumínio de gel de sílica HPTLC começando 2 cm dos bordos da placa. Se várias amostras são analisadas numa cromatografia unidimensional, mantenha uma distância de pelo menos 1,5 cm entre os diferentes pontos de aplicação da amostra.
- Preparar e misturar a fase móvel (140 ml de hexano, 60 ml de éter dietílico, e 8 ml de ácido acético) e transferência para uma câmara de desenvolvimento TLC revestidas internamente com papel de cromatografia e cobertos com uma placa de vidro para permitir a saturar câmara (30 min).
- Transferir a folha de alumínio de HPTLC de gel de sílica com as amostras de lípido seca para a câmara e desenvolver a placa durante 30 minutos na câmara fechada.
- Remova a placa da câmarae deixar que os solventes secar numa câmara de fluxo durante 2 h.
- Separação de lipídios polares cobradas pelo bidimensional TLC (2D-TLC).
- Quantificação e visualização de lípidos polares separadas.
- Uma vez que a folha de TLC desenvolvido é seco, incubar isto com uma tela de luminescência fotoestimuláveis (PSL) numa cassete fechado durante 3 dias.
- Expor a tela incubadas a um scanner PSL e adquirir uma imagem virtual de lípidos radiomarcadas foram separadas.
- Realizar quantificação usando software PSL 20.
- Visualização e isolamento de classes de lipídios polares individuais.
- Incubar folha CCF desenvolvida durante 10 min numa câmara de cromatografia na presença de 1 g de cristais de iodo.
NOTA: Separado compostos lipídicos vai dissolver o iodo e aparecem como manchas acastanhadas. - Círculo as manchas com um lápis, compará-los com a mobilidade relativa (Rf) de compostos padrão (ou seja, 1,2-dipalmitoil-sn-glicerol, dipalmitoil-L-α-fosphatidylcholine, DL-α-monopalmitina, ou ácido palmítico), e identificar a classe de lípido que eles possam pertencer.
- Numa hotte, deixar que o iodo se evaporar a partir da folha de TLC.
- Com a ajuda de uma espátula, raspar o gel de sílica contendo o composto de interesse a partir da folha, e extrair o composto a partir do gel de sílica com uma mistura de 100 ul de água e 375 ul de solução de metanol: clorofórmio (2: 1; vol / vol).
- Continue com a extracção de acordo com Bligh e Dyer, conforme descrito (4.2.2 em diante).
- Loja classe de lípido purificado em 100 ul de clorofórmio: solução de metanol (1: 1; v / v) à temperatura de -20 ° C até à sua utilização.
- Incubar folha CCF desenvolvida durante 10 min numa câmara de cromatografia na presença de 1 g de cristais de iodo.
5. Identificação In Vitro da condição fisiológica do substrato de uma lipase
NOTA: Em uma abordagem in vitro, estudar se a lipase de interesse pode converter uma mistura de lípidos isolados ou lipídios puros individuais ao hydrol correspondenteprodutos ysis nas condições definidas como ideal em 3.2.
- Utilize os esquemas de pipetagem para ensaios de enzima conforme a Tabela 3 para fosfolipase específico para PC C SMc00171 (ver 5.2), fosfolipase A (ver 5.3), e DAG lipase SMc01003 (ver 5.4) atividade.
- Determinação da actividade da fosfolipase C específica do PC (Tabela 3).
- Para um tubo de microcentrífuga de 1,5 ml, adicionar 5000 contagens por minuto (cpm) de um total de PC 14 marcado com C e uma solução de Triton X-100.
- Misture e seco sob uma corrente de azoto.
- Adicionar dietanolamina-HCl, tampão de pH 9,8, bem como NaCl e MnCl2 soluções e água bidestilada até se obter um volume final de 99,5 mL. Vortex durante 5 seg.
- Adicionar 0,5 uL de enzima (5 ug de proteína) (isto é, um extracto isento de células que sobre-expresso em SMc00171 está presente) para iniciar a reacção. Misture brevemente.
- Incubar a 30 ° C durante 4 h.
- Parar a reacção pelaadição de 250 ul de metanol e 125 ul de clorofórmio.
- Extraia lipídios como descrito anteriormente (ver 4.2).
- lipídios separados por unidimensionais (1D) -TLC (ver 4.3.2 e 4.4), e analisá-los por imagem PSL.
- Determinação da actividade da fosfolipase A (Tabela 3).
- Para um tubo de microcentrífuga de 1,5 ml, adicionar 5000 cpm totais de 14 fosfolípidos marcados com C e uma solução de Triton X-100.
- Misture e seco sob uma corrente de azoto.
- Para um ensaio final de 100 uL, adicionar Tris-HCl, pH 8,5, solução de NaCl e água. Vortex durante 5 seg.
- Adicionam-se 5 ul de enzima (50 ug de proteína) (isto é, um extracto isento de células que sobre-expresso em SMc00930 ou SMc01003 está presente).
- Incubar a 30 ° C durante 5 h.
- Parar a reacção pela adição de 250 ul de metanol e 125 ul de clorofórmio.
- Extraia lipídios como descrito anteriormente (ver 4.2), separadas-los por CCF-1D usando 130 ml de clorofórmio, 50 ml de metanol, e ácido acético glacial, 20 ml como a fase móvel, e analisá-las por imagiologia PSL.
- Determinação do diacilglicerol atividade lipase (DAG).
- Preparação do DAG 14 C-rotulado.
- Radiomarcador S. culturas meliloti (ver 4.1) e extrair os lípidos polares (ver 4.2), como descrito. S. separada meliloti extractos lipidicos totais por CCF-1D em clorofórmio: metanol: ácido acético (130: 50: 20; v / v), utilizando as condições descritas para a separação na segunda dimensão, em 4.3.1.
- Visualizar PC por coloração com iodo e usar um lápis para marcar a localização de fosfatidilcolina (PC).
- Isolar PC radiomarcado conforme descrito em 4.5.
- Quantificar PC extraído por contagem de cintilação.
NOTA: cerca de 320.000 PC cpm é esperado. - Tratar PC (250.000 cpm) com 0,1 U de fosfolipase C a partir de Clostridium perfringens em mM Tris-HCl, pH 7,2, 0,5 50% De Triton X-100 e 10 mM de CaCl2 durante 2 horas num volume total de 100 ul e parar a reacção pela adição de 250 ul de metanol e 125 ul de clorofórmio.
- Extraia lipídios, como descrito anteriormente, e separada por eles por 1D-TLC (ver 4.3.2).
- Isolar diacilglicerol a partir da placa de sílica e quantificação por contagem de cintilação (como descrito em 4.2)
- Ensaio de lipase de diacilglicerol (Tabela 3).
- Para um tubo de microcentrífuga de 1,5 ml, adicionar 5000 cpm de 14 DAG C-rotulado e uma solução de Triton X-100.
- Misture e seco sob uma corrente de azoto.
- Para um ensaio final de 100 uL, adicionar Tris-HCl (pH 9,0) tampão, uma solução de NaCl e água destilada duas vezes. Vortex durante 5 seg.
- Inicia-se a reacção por adição de 5 ul de enzima (50 ug de proteína do extracto isento de células).
- Incubar a 30 ° C durante 4 h.
- Parar a reacção pela adição de 250 ul de um metanold 125 l de lipídios clorofórmio e extrair como descrito anteriormente (ver 4.2).
- Analisar lípidos polares neutros por 1D-TLC (ver 4.1.3.2) e subsequente imaging PSL.
- Preparação do DAG 14 C-rotulado.
Resultados
Actividade de Fosfolipase específico para PC C SMc00171 com bis-p-nitrofenil fosfato
Extractos isentos de células obtidos a partir de E. coli BL21 (DE3) pLysS x, que tinha smc00171 expressa, foram estudados quanto à sua capacidade para hidrolisar ésteres de fosfato de bis-p-nitrofenilo, utilizando um ensaio espectrofotom...
Discussão
Nos últimos 20 anos, os genomas de muitos organismos já foram seqüenciados e, apesar de uma riqueza de dados seqüência do genoma foi gerado, interpretação funcional está ficando para trás e, portanto, dificulta a nossa compreensão da função do genoma. funções dos genes em genomas são frequentemente atribuídos com base na similaridade com genes de função ou a ocorrência de motivos conservados conhecido. No entanto, a função exacta de um determinado gene, muitas vezes não é conhecido. Especialmente,...
Divulgações
Os autores não têm nada para revelar.
Agradecimentos
Este trabalho foi financiado por doações do Consejo Nacional de Ciencias y Tecnología-México (CONACyT-México) (82614, 153998, 253549 e 178359 em Investigación Científica Básica, bem como 118 em Investigación en Fronteras de la Ciencia) e da Dirección General de Asuntos de pessoal Académico-Universidad Nacional Autónoma de México (UNAM DGAPA-; PAPIIT IN202616, IN203612).
Materiais
| Name | Company | Catalog Number | Comments |
| Chloroform | JT Baker | 9180-03 | TLC analysis & Lipid extraction |
| Methanol | JT Baker | 9070-03 | TLC analysis & Lipid extraction |
| Acetic Acid | JT Baker | 9507-05 | TLC analysis & Lipid extraction |
| Hexanes | JT Baker | 9309-02 | TLC analysis & Lipid extraction |
| Diethylether | Sigma | 32203 | Enzymatic assays |
| bidistilled water | ANY | NA | Enzymatic assays |
| Tris Base | Sigma | T-1503 | Enzymatic assays |
| HCl | Baker | 9535-02 | Enzymatic assays |
| NaCl | Baker | 3624-01 | Enzymatic assays |
| Triton X-100 | Sigma | X-100 | Enzymatic assays |
| LB broth | ANY | NA | Bacterial growth, 10 g tryptone + 5 g yeast extract + 10 g NaCl per liter of bidistilled water |
| tryptone | Becton Dickinson and Company | 211705 | Bacterial growth |
| yeast extract | Becton Dickinson and Company | 212750 | Bacterial growth |
| TY broth | ANY | NA | Bacterial growth, 8 g tryptone + 3 g yeast extract + 66 mg CaCl2·2H2O per liter of bidistilled water |
| CaCl2·2H2O | Baker | 1332-01 | Enzymatic assays |
| isopropyl-β-D-thiogalactoside (IPTG) | Invitrogen | 15529-019 | Bacterial growth |
| Diethanolamine | Sigma | D-8885 | Enzymatic assays |
| MnCl2 | Sigma | 221279 | Enzymatic assays |
| Phospholipase A2 snake venom | Sigma | P0790 | Enzymatic assays |
| Phospholipase C Clostridium perfringens | Sigma | P7633 | Enzymatic assays |
| Bis-p-nitrophenyl phosphate | Sigma | 07422AH | Enzymatic assays |
| p-nitrophenyl stearate | Sigma | N3627 | Enzymatic assays |
| p-nitrophenyl dodecanoate | Sigma | 61716 | Enzymatic assays |
| p-nitrophenyl decanoate | Sigma | N0252 | Enzymatic assays |
| p-nitrophenyl palmitate | Sigma | N2752 | Enzymatic assays |
| p-nitrophenyl butyrate | Sigma | N9876 | Enzymatic assays |
| p-nitrophenyl octanoate | Sigma | 21742 | Enzymatic assays |
| Acetic Acid, sodium salt [1-14C] | Perkin Elmer | NEC084 | Bacterial growth |
| dimethylsulfoxide (DMSO) | JT Baker | 9224-01 | Enzymatic assays |
| Aluminium HPTLC silica gel 60 plates. Silica gel HPTLC plates size 20 x 20 cm, 25 sheets. | Merck | 105547 | TLC analysis & Lipid extraction |
| Spectrometer UV/VIS Lambda 35 | Perkin Elmer | NA | Enzymatic assays |
| Storm 820 Phosphorimager | Molecular Dynamics | NA | Photostimulable Luminescence scanner |
| Multipurpose Scintillation Counter | Beckman Coulter | NA | Radioactivity Quantification |
| French Pressure Cell | ThermoSpectronic | NA | Breakage of cells |
| chromatography paper 3MM Chr | Whatman | 3030917 | TLC analysis |
| Sinorhizobium meliloti 1021our | reference 11 | studied strain | |
| Escherichia coli BL21 (DE3) pLysS Competent cells | Novagen | 69451 | protein expression strain |
| pET9a vector | Novagen | 69431 | protein expression vector |
| pET17b vector | Novagen | 69663 | protein expression vector |
| sterile polystyrene round-bottom tube (14 ml) Falcon | Becton Dickinson | 352057 | radiolabeling of bacterial cultures |
| polypropylene microcentrifuge tubes (1.5 ml) | Eppendorf | 30125.15 | Enzymatic assays |
| 1,2-dipalmitoyl-sn-glycerol | Sigma | D9135 | lipid standard |
| L-α-phosphatidylcholine, dipalmitoyl | Sigma | P6267 | lipid standard |
| DL-α-monopalmitin | Sigma | M1640 | lipid standard |
| palmitic acid | Sigma | P0500 | lipid standard |
Referências
- Nelson, D. L., Cox, M. M. . Lehninger, Principles of Biochemistry. , (2013).
- Jaeger, K. E., Eggert, T. Lipases for biotechnology. Curr. Opin. Biotechnol. 13 (4), 390-397 (2002).
- Dowhan, W., Bogdanov, M., Mileykovskaya, E. Functional roles of lipids in membranes. Biochemistry of Lipids, Lipoproteins and Membranes. , 1-37 (2008).
- Dowhan, W. Molecular basis for membrane phospholipid diversity: why are there so many lipids?. Annu. Rev. Biochem. 66, 199-232 (1997).
- De Rudder, K. E. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti mutants deficient in phospholipid N-methyltransferase still contain phosphatidylcholine. J. Bacteriol. 179, 6921-6928 (1997).
- Geiger, O., Röhrs, V., Weissenmayer, B., Finan, T. M., Thomas-Oates, J. E. The regulator gene phoB mediates phosphate stress-controlled synthesis of the membrane lipid diacylglyceryl-N,N,N-trimethylhomoserine in Rhizobium (Sinorhizobium) meliloti. Mol. Microbiol. 32 (1), 63-73 (1999).
- Klug, R. M., Benning, C. Two enzymes of diacylglyceryl-O-4'-(N,N,N,-trimethyl)homoserine biosynthesis are encoded by btaA and btaB in the purple bacterium Rhodobacter sphaeroides. Proc. Natl. Acad. Sci. USA. 98 (10), 5910-5915 (2001).
- Zavaleta-Pastor, M., et al. Sinorhizobium meliloti phospholipase C required for lipid remodeling duringphosphorus limitation. Proc. Natl. Acad. Sci. USA. 107 (1), 302-307 (2010).
- Pech-Canul, A., et al. FadD is required for utilization of endogenous fatty acids released from membrane lipids. J. Bacteriol. 193 (22), 6295-6304 (2011).
- Banerji, S., Flieger, A. Patatin-like proteins: a new family of lipolytic enzymes present in bacteria?. Microbiology. 150 (Pt 3), 522-525 (2004).
- Sahonero-Canavesi, D. X., et al. Fatty acid-releasing activities in Sinorhizobium meliloti include unusual diacylglycerol lipase. Environ. Microbiol. 17 (9), 3391-3406 (2015).
- Fischer, M., Pleiss, J. The Lipase Engineering Database: a navigation and analysis tool for protein families. Nucl. Acid. Res. 31 (1), 319-321 (2003).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Res. 41 (Database issue), D344-D347 (2013).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. J. Vis. Exp. (63), e3998 (2012).
- Untergasser, A., et al. Primer3 - new capabilities and interfaces. Nucl. Acids Res. 40 (15), e115 (2012).
- Studier, F. W., Rosenberg, A. H., Dunn, J. J., Dubendorff, J. W. Use of T7 RNA polymerase to direct expression of cloned genes. Methods Enzymol. 185, 60-89 (1990).
- Miller, J. H. . Experiments in Molecular Genetics. , (1972).
- Desjardins, P., Hansen, J. B., Allen, M. Microvolume Protein Concentration Determination using the NanoDrop 2000c Spectrophotometer. J. Vis. Exp. (33), e1610 (2009).
- Bligh, E. G., Dyer, W. J. A rapid method for total lipid extraction and purification. Can. J. Biochem. Physiol. 37 (8), 911-917 (1959).
- Molecular Dynamics. . Phosphorimager SI User´s Guide. , (1994).
- Dixon, M., Webb, E. . Enzymes: Third Edition. , (1979).
- Rudolph, A. E., et al. Expression, characterization, and mutagenesis of the Yersinia pestis murine toxin, a phospholipase D superfamily member. J. Biol. Chem. 274 (17), 11824-11831 (1999).
- Kato, S., Yoshimura, T., Hemmi, H., Moriyama, R. Biochemical analysis of a novel lipolytic enzyme YvdO from Bacillus subtilis. Biosci. Biotechnol. Biochem. 74 (4), 701-706 (2010).
- Peppelenbosch, M. P. Kinome profiling. Scientifica (Cairo). , (2012).
- Manafi, M., Kneifel, W., Bascomb, S. Fluorogenic and chromogenic substrates used in bacterial diagnostics. Microbiol. Rev. 55 (3), 335-348 (1991).
- Kuznetsova, E., et al. Enzyme genomics: Application of general enzymatic screens to discover new enzymes. FEMS Microbiol. Rev. 29 (2), 263-279 (2005).
- Scopes, R. K. . Protein Purification, Principles and Practice. , (2010).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim. Biophys. Acta. 1831 (3), 503-513 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados