Method Article
Geração rápida e eficiente de células-tronco pluripotentes Recombinante Humana pela recombinase mediada troca cassete no
Neste Artigo
Resumo
Aqui relatamos um método de edição gene rápida e eficiente com base em RMCE no locus AAVS1 de humanos pluripotentes células estaminais (hPSCs) que melhora a sistemas descritos anteriormente. Usando esta técnica, linhas isogênicas pode ser rápida e confiável gerada para estudos comparativos adequados, facilitar a investigação mediada por transgênese com hPSCs.
Resumo
Mesmo com a revolução das tecnologias de segmentação de genes conduzidos por CRISPR-Cas9, a modificação genética de células-tronco pluripotentes humanas (hPSCs) ainda é demorado. Estudos comparativos que usam linhagens recombinantes com transgenes integrados em loci porto seguro poderia beneficiar de abordagens que usam recombinases direcionados site-specific, como Cre ou FLPe, que são mais rápidos e menos propenso a efeitos off-alvo. Tais métodos têm sido descritos, embora não significativamente gene outperform alvo na maioria dos aspectos. Usando nucleases de dedos de zinco, que anteriormente criou uma linha de células de referência no locus AAVS1 de hPSCs que contém um GFP-higromicina-tk expressar cassete, flanqueado por sequências DRF heterotípicos. Aqui, descrevemos os procedimentos para executar FLPe troca cassete mediada por recombinase (RMCE) usando esta linha. O mestre cA linha ell é transfectada com um vector RMCE dador, que contém um promotor de resistência puromicina, e com FLPe recombinase. Aplicação do programa de selecção tanto positiva (puromicina) e negativo (FIAU) conduz à selecção de RMCE sem integrações aleatórias. RMCE gera linhas transgénicas policlonais pluripotentes totalmente caracterizados em 15 d, com uma eficiência de 100%. Apesar do descrito recentemente limitações do locus AAVS1, a facilidade do sistema abre o caminho para HPSC transgenia em ambientes isogênicos, é necessária para estudos comparativos, e permite telas genéticos semi-alto rendimento para o ganho / perda de análise de funções que seria de outra forma ser altamente demorado.
Introdução
Alvo de recombinação do genoma facilitou os rápidos avanços em muitas áreas de pesquisa. Em camundongos, a sinergia entre a edição genoma e células-tronco tem permitido uma maior compreensão do mecanismo complexo da função dos genes e regulação dos genes. Esse progresso é esperado em células humanas pluripotentes estaminais (hPSCs) bem, embora para muitos anos desde o primeiro isolamento de células estaminais embrionárias humanas (hESCs), e humano mais tarde induziu células-tronco pluripotentes (hiPSCs), edição gene tem constituído um obstáculo técnico . Recentes avanços em engenharia de genoma utilizando nucleases-zinco-alvo dedo nucleases (ZFNs), transcrição nucleases efetoras ativador-like (TALEN), ou cluster regularmente espaçadas curta repete palindr�icas / proteína associada a CRISPR 9 (CRISPR / Cas9) nos permitiram superar estes dificuldades, tornando recombinação alvo de um processo eficiente 1-3.
loci específicos no mgenoma ouse que permitem a expressão do transgene estável, confiável e ubíqua na ausência de efeitos adversos como Rosa26, Hprt1 ou COL1A1, tornaram-se ferramentas essenciais para a realização de estudos genéticos. loci porto seguro permitir a análise comparáveis entre diferentes linhagens de camundongos em contextos isogênicos. Isto não pode ser conseguida utilizando métodos de integração aleatória convencionais, os quais são associados com limitações bem conhecidas como mutagénese por inserção, efeitos dependentes da dose, ou a expressão do transgene variegada. Gene edição facilidade e flexibilidade na loci porto seguro é aumentada através da utilização de recombinases direcionados site-specific como Cre ou flipase (FLPe), que reconhecem especificamente sequências alvo (loxP ou FRT, respectivamente) e catalisam recombinação eficiente entre alvos idênticos. Devido a estas características, edição de gene mediada por recombinase usando sequências loxP ou FRT em loci porto seguro pré-integrado é uma ferramenta comum usada em rato transgênese. Além disso a inserção de cassetes ou excisão mediada entre as sequências alvo idênticas, o uso de locais loxP ou FRT incompatíveis permite a troca de cassete mediada por recombinase (RMCE) 4.
No ser humano, as tentativas para identificar loci portuárias seguras foram realizadas. O HPRT ortóloga rato, apesar de sua associação com a síndrome de perda de função de Lesch-Nyhan, e ROSA26 foram alvo de hPSCs. HPRT foi relatado para silenciar rapidamente a expressão do transgene em ESC e, como ROSA26, sua capacidade de sustentar a expressão do transgene em terminalmente células diferenciadas não foi investigada 5-7. Uma vez que uma mutação nula homozigótica do gene CCR5 parece ser bem tolerada em seres humanos, o seu valor como zona de protecção foi avaliada. CCR5 foi orientada e relatado para manter a expressão do transgene estável em diferentes linhas de células humanas, incluindo CES 8,9. Contudo,esta última não foi comprovado ao nível clonal durante a cultura a longo prazo, e a expressão do transgene ubíqua não foi demonstrada na progenia diferenciada das três camadas germinais. AAVS1, o sítio de integração natural do Adeno Associated Vírus tipo 2 (AAV), foi testada em hESCs assim, por causa da resistência relatado para transgene silenciar 10. Mais tarde, vários grupos utilizado o locus AAVS1 e descrita a expressão do transgene estável no hPSCs indiferenciadas, bem como na sua progénie diferenciada de todas as três camadas germinais, tanto in vitro como in vivo 2,8,11,12. Os resultados recentes, no entanto, matizar estes achados, como o locus AAVS1 foi encontrado para exercer a inibição transgene variável in vitro em hESCs indiferenciadas e em hepatócitos progênie 13.
Estudos adicionais da seleção utilizando abordagens de integração aleatórios e métodos para determinar a integração cópia única destinada a encontrar genolocais de integração de microfone resistentes ao silenciamento do transgene durante a expansão e diferenciação hPSCs 14,15. No geral, até agora, nenhum site genômico foi totalmente validado como um porto seguro em hPSCs e seus descendentes; identificação de um local apropriado para a expressão do transgene estável ubíqua, não só em hPSCs mas também em suas progênies diferenciadas in vitro e in vivo, permanece a ser resolvido. Entre todos os loci estudados e apesar das suas limitações, AAVS1 continua a ser o melhor caracterizados e mais utilizado na investigação sobre células estaminais.
RMCE foi realizado com sucesso em hPSCs em alguns desses loci 6,7,14,16, utilizando principalmente Cre recombinase, mesmo se houver indícios de que FLPe é mais eficiente que Cre 17. Em todos estes casos, uma cassete de resistência à droga positivo foi utilizado para a selecção de colónias recombinantes. Embora bem sucedido, esses procedimentos não constituem um avanço técnico sobre gene-padrãoprocedimentos de edição usando ZFNs, TALENS ou CRISPR / Cas9, como um único processo de selecção de antibiótico não descarta integrações aleatórias e requer triagem colónia para identificar clones corretamente direcionados.
Neste procedimento, que descrevem métodos para realizar RMCE em hPSCs no locus AAVS1 usando uma combinação de positivo (dentro da cassete de entrada) e negativo (dentro da cassete pré-integrado) selecções que permitem a geração de linhas transgénicas policlonais em ± 15 dias com a 100% de eficiência e livre de eventos de integração aleatórios. Portanto, este método representa um progresso além das tecnologias RMCE atualmente descritos em hPSCs.
Protocolo
1. Preparação da linha celular mestre HPSC contendo FRT para transfecção
- Cultura hESC / IPSC linhas celulares mestre sob procedimentos padrão dentro ou fora embrionárias de camundongos Fibroblastos inativados (IMEF) usando células estaminais embrionárias humanas ou meio iPSC livre de alimentador, respectivamente (Tabela 1). Preparar 2 (em alimentadores) ou 1-poços (livre) do alimentador de hPSCs em uma placa de 6 poços para cada condição experimental.
- Observe as culturas, e quando as células atingem 60-70% de confluência, placa resistente aos medicamentos IMEF (iDR4). Resumidamente, os poços de revestimento necessárias de uma placa de 12 poços com 0,5 ml de solução de gelatina a 0,1% e incubar durante 5 min à TA. Placa iDR4s 125.000 por poço e incubar O / N com meio IMEF (Tabela 1).
2. Transfecção HPSC por Nucleofection
- No dia seguinte, a pré-incubar células estaminais embrionárias humanas / IPSC culturas com meio fresco, incluindo 10 uM proteína cinase Rho-associado (ROCHA) inibidor (Y-27632), durante 1 h a 37 ° C. Next, tomar a solução de transfecção hESC da geladeira para pré-aquecer à temperatura ambiente.
- Prepare 1 ml de meio de plaqueamento nucleofection (Tabela 1) por condição nucleofection. Aqui forma IMEF fora das cavidades, lava-se com 1 ml de PBS temperatura ambiente, e adicionar 500 mL de meio de plaqueamento nucleofection. Transferir os outros 500 mL para tubos de 1,5 ml estéreis e armazená-las a 37 ° C até que o chapeamento. NOTA: Uma experiência RMCE típica contém três condições diferentes, com relações moleculares do doador RMCE: vetores pFLPe de 2: 0, 2: 1 e 0: 1.
- Retire hESC / iPSC em uma suspensão de células individuais.
- Tire a mídia, lavá-lo com 2 mL de RT PBS, e adicionar 1 mL de tripsina 0,05% ou Accutase no alimentador ou livre de alimentação culturas HPSC, respectivamente. Incubar durante 7 (tripsina) ou e 2-5 (Accutase) min a 37 ° C. Para o tratamento Accutase - células devem permanecer solto, mas anexado.
- Remova cuidadosamente a placa da incubadora e sem umllowing as células a separar, remover o agente de dissociação por aspiração. Adicionar 1 ml de hESC media e dissociar as células soltas por lavagem suavemente os meios de comunicação sobre a superfície da placa, evitando a formação de espuma. Certifique-se de que as células estão em uma suspensão única célula.
- Coletar as células a 15 ou 50 tubo limpo e estéril mL. Lavar os poços com 1 ml de meio de células estaminais embrionárias humanas e recolher as células no mesmo 15 ou 50 ml tubo. Tomar uma aliquota de 50 uL por contagem (durante o próximo passo), utilizando um dispositivo de contagem de células. Anote o volume de coleta e calcular o número total de células. Girar as células a 300 xg durante 5 min à TA.
- Ressuspender o sedimento de células, a uma suspensão de 10 6 células / mL com PBS utilizando uma pipeta serológica 5 mL e pipetagem suave. Evitar a formação de espuma. Transferir 2 mL por condição experimental (aproximadamente 2 x 10 6 células) para limpar, 15 tubos estéreis mL.
- Enquanto a centrifugação, prépare o RMCE pelos doadores plasmídeos pFLPe mistura em um volume máximo de 10 mL.
- Transferir 2,5 ug de pFLPe (6,5 Kb) a uma estéril tubo limpo, 1,5 mL. Adicionar uma proporção de 2: 1 molecular do vector RMCE doador 13 ao vector pFLPe. Por exemplo, cerca de 10 ug de um vector de 12 Kb RMCE dador.
- Proceda com nucleofection, evitando a formação de espuma em todas as etapas.
NOTA: Alguns protocolos de transfecção HPSC aplicar um passo esgotamento IMEF adicional antes do passo 2.4. No entanto, na prática, IMEF é uma fracção de célula de menor na única suspensão que não altera significativamente a eficiência da transfecção, nem vai criar células recombinantes uma vez que são contaminantes não-proliferativa. Além disso, é essencial proceder-se rapidamente durante o processo de transfecção para aumentar a viabilidade. Por essas razões, o esgotamento IMEF não é aplicado.- Remova a placa iDR4 e os tubos de 1,5 ml com o meio de plaqueamentoda incubadora. Um tubo de cada vez, para fora da pipeta PBS cuidadosamente sem perturbar o sedimento celular. Remover o máximo possível. Ressuspender o sedimento com 100 ul de solução de transfecção por pipetagem suave. Transferir a suspensão de célula para o tubo de 1,5 mL contendo as misturas de plasmídeos e misturar suavemente.
- Transfira a mistura-DNA celular para a tina transfector, prestando atenção para não introduzir bolhas. Introduzir a cuvete no dispositivo Nucleofector e aplicar A13 programa (células cultivadas em alimentadores) ou F16 (free alimentador de).
NOTA: Outros métodos de transfecção ou Nucleofector dispositivos podem exigir a otimização desses programas ou condições de transfecção. - Retomar o cuvete e, utilizando as pipetas de transferência descartáveis disponíveis no kit nucleofection, recolher o seu conteúdo através da aplicação de 0,5 mL de meio de plaqueamento e a aspiração de todo o volume. Realizar esta etapa rapidamente, mas com cuidado e em um movimento.
- Placa gota a gota, no 12-wplaca ell contendo a iDR4 e 500 mL de chapeamento médio. Repita os passos 2.6.1 - 2.6.4 para a próxima condição experimental.
- Coloque a placa de cultura em uma prateleira na incubadora e movê-lo lentamente para trás e para a frente e para os lados para distribuir uniformemente a suspensão de células. Incubar por 24 h antes de a próxima mudança de meio para permitir que as células para recuperar, na presença de 10 uM de inibidor ROCHA (Y-27632).
3. selecção positiva e negativa de células que sofrem RMCE
- meios mudança de cultura por dia (1 ml / poço), e 2-3 d pós-transfecção, começar a selecção com 100 ng / mL de puromicina.
NOTA: As concentrações óptimas de reagentes de selecção positiva e negativa devem ser determinadas experimentalmente para cada linha celular mestre HPSC recém-gerado. Executar uma curva de mortalidade 13, utilizando a linha celular mestre determinar a dose baixa (concentração à qual a toxicidade mínima visual é aparente após 7d de selecção, mas a cultura não é coberto), dose óptima (a menor concentração em que todas as células estão mortas após 7 d de selecção), e dose elevada (concentração que causou toxicidade evidente, matando todas as células após 2-3 d) tanto para a puromicina e fialuridina (FIAU).- Aspirar o meio hESC. Lava-se com 1 mL de PBS. Adicionar meio de células estaminais embrionárias humanas com 100 ng / mL de puromicina.
- Observe o crescimento celular para que a morte e crescimento são equilibrados. Mudança de mídia com puromicina diária, seguindo o passo 3.1.1. Quando a taxa de crescimento ultrapassa a morte celular, aumentar a concentração de puromicina em blocos de 25 - 50 ng / ml até um máximo de 250 ng / mL. Continue a seleção puromicina para 5-7 d.
- 3-4 d após o início da selecção puromicina, por volta do dia 6 pós-transfecção, iniciar a seleção com 0,5 mM UIAF. Alterar a mídia diária e manter UIAF por não mais do que 7 dias contínuos.
4. Expansão e Caracterização de RMCE Lines
- Após a conclusão da selecção, em torno D14 - 15, as colónias resistentes RMCE estão presentes e constituem a nova linha RMCE. Dividir em grandes quantidades em proporção de 1: 2 de acordo com procedimentos padrão (passagem 1, P1).
- Placa 2 poços de uma placa de 12 poços (ou desligar os alimentadores, dependendo das condições de cultura normais da linha de células mestre original). Use um bem para uma maior expansão, armazenamento, ou set-up experimental (nenhuma descrição adicional destes procedimentos é feito neste protocolo), e o outro para caracterização.
- Quando as células estão prontas a partir de P1 para dividir, dissociar o poço para a caracterização de uma suspensão de célula única como descrito em 2.4 e recolher as células num tubo de 15 mL. NOTA: Use as células do HPSC WT (sem modificação genética) e linha de células mestre como controles negativo e positivo, respectivamente, para a caracterização.
- Girar a 300 xg durante 5 min à TA. Ressuspender em 1 ml de PBS e a amostra dividida em duas por citometria de fluxoe análise de DNA 13.
- A citometria de fluxo de análise 13 (300 uL):
- Adicionar soro fetal de vitelo para as células em PBS até uma concentração final de 5%. Transferir as células para um tubo de FACS limpo com um filtro de células e mantê-los em gelo.
NOTA: A viabilidade celular é elevada nestas condições, por isso, é possível, mas não necessário, a utilização de um corante fluorescente para as células não viáveis (por exemplo, 7-AAD ou PI). - Proceder de imediato à análise em um citômetro de fluxo analisador de células e registrar 20.000 - 30.000 eventos. Para a análise, definir as portas acima GFP ou TDT fundo de fluorescência usando a amostra de controlo negativo WT.
- Adicionar soro fetal de vitelo para as células em PBS até uma concentração final de 5%. Transferir as células para um tubo de FACS limpo com um filtro de células e mantê-los em gelo.
- A análise do DNA 13 (700 mL):
- Transferir as células para um tubo limpo de 1,5 mL. Girar a 300 xg durante 5 min à temperatura ambiente e aspirar o sobrenadante. Prossiga para a extração de DNA usando um kit comercial e siga as instruções do fabricante. Armazenar o sedimento seco a -20 ° C durante latanálise er.
- Medir a concentração de ADN.
- Prepare a reacções convencionais de PCR de acordo com as condições na Figura 3 e Tabela 2 13 amostras de corrida de PCR sobre um gel de agarose a 2% contendo gel de mancha de ADN 1x a 150 V durante 15 - 25 min.. Analisar a corrida em um analisador de gel.
Resultados
Usando AAVS1 ZNFs espec�icos, linhas de células mestre completamente caracterizado hESC / IPSC contendo sequências alvo DRF heterotípicos foram geradas e descritas 13. Os FRT contendo linhas de células mestras mantida pluripotência e integridade do genoma após o tratamento ZFN e estavelmente expressa GFP in vitro e in vivo. RMCE é realizada por co-transfecção de linhas celulares com mestre FLPe recombinase e vectores de doadores RMCE (Figura 1A). Vetores RMCE conter sequências DRF idênticos para os da linha de células mestre AAVS1 flanqueando o transgene Figura 1B monitora RMCE usando o constitutivamente expressando TDT pZ:.. F3-P CAGGS tdTPH-F Após transfecção, HPSC formar pequenos grupos de células distribuídas uniformemente ao longo a camada iDR4 MEF, e transfectadas HPSC expressam tanto GFP, bem como transiente TdT (Figura 1B, D 2). As células são deixadas a recuperar durante 2-3d pós-transfecção antes de iniciar a seleção. selecção positiva é primeiro começou a usar uma dose baixa de puromicina para favorecer a inserção dos doadores RMCE. A puromicina é aplicado suavemente no início, devido a morte celular em massa inicial pode levar a células individuais submetidos a recombinação, tornando-os incapazes de sobreviver ao processo. Nos dias seguintes, a concentração de puromicina tem de ser aumentado por etapas até a dose óptima a fim de permitir que as células recombinantes a crescer em pequenas colónias, enquanto que as células morrem nonrecombined gradualmente.
3-4 d após o início da selecção positiva (cerca de 6 D pós-transfecção), selecção negativa é iniciado, a fim de seleccionar apenas para eventos de recombinação FRT-mediadas. RMCE provoca a perda da timidina quinase do gene suicida (TK) e, assim, a sensibilidade para fialuridina (FIAU). Selecção negativa aplicada neste ponto, no qual pequenos grupos de 2-4 GFP - + (células RMCE) / TdT sãocapaz de resistir a concentrações óptimas de FIAU (Figura 1B, D 6), impede a integração aleatória, porque em tais eventos, o gene de TK no locus AAVS1 permanece inalterado. A ocorrência simultânea de FRT-mediada e integração aleatória é altamente improvável, como demonstrado pela ausência de eventos de integração aleatória mais tarde durante a caracterização (Figura 2B).
A GFP RMCE misto - / TdT + colónias continuam a crescer, ao mesmo tempo tornando-se mais homogénea, de modo que por D 9-10 pós-transfecção, algumas colónias RMCE mistos não totalmente seleccionados pode ser encontrado (Figura 1B). GFP - / TdT + RMCE colónias estão presentes apenas quando o dador e RMCE FLPe (2: 1) são utilizados, enquanto RMCE não ocorre na ausência de FLPe (2: 0). Seleção completa com UIAF até o dia 13-15 após a transfecção dá origem a uma GFP homogênea - / TDT + culture. RMCE produz uma média de 12,8 ± 6,8 (n = 6) colónias Puro R / R FIAU em 15 dias 13. A citometria de fluxo caracterização da linha RMCE recém-gerado (as colónias RMCE Puro R / R FIAU combinados numa população de células nonclonal) confirma que 100% das células apresentam o RMCE GFP - / TdT + fenótipo (Figura 2A). Quando são utilizados doadores RMCE sem expressão constitutiva de um repórter fluorescente, a linha Puro R / UIAF R RMCE HPSC resultante é homogeneamente GFP -.
Caracterização de PCR da linha RMCE nonclonal demonstra a troca de cassete completo (sem vestígios da cassete da linha de células de referência pode ser detectado) e que o programa de selecção utilizado gera linhas livre de integração aleatória (tanto do dador RMCE e o FLPe-expressando vetor) (Figura 2B). Estes resultados foram ainda comprovada por sblot outhern e, além disso, nós demonstramos que as linhas RMCE permanecer pluripotentes 13. Isso faz com que ele não é mais necessário realizar tanto a avaliação de todo o genoma de integração aleatória ou o teste de pluripotência durante as experiências RMCE rotina. Em resumo, utilizando este protocolo, HPSC linhas celulares transgénicas no locus AAVS1 livre de integração aleatória pode ser facilmente gerada em 15 d, com uma eficiência de 100% e sem a necessidade de uma extensa caracterização.

Figura 1: Visão esquemática do RMCE (A) Timeline do programa de seleção RMCE.. Esquerda: a linha de células de referência (MCL), que alberga uma cassete flanqueada-FRT (triângulos) que expressam GFP e higromicina-TK (ligados por péptidos de auto-clivagem 2A), é transfectado por nucleofection (NF) com o vector FLPe-expressar e o vector RMCE doador, WHich contém um gene sem promotor resistência puromicina (splicing aceitador - SA- Puro R) e uma cassete experimental variável (X). Direita: nova linha RMCE (RMCEL) obtido após a conclusão da seleção com puromicina (triângulo vermelho) e UIAF (linha azul). (B) RMCE usando um constitutiva TdT-expressando dador e diferentes proporções de DNA dadora e ADN FLPe (0: 1, 2: 1, 2: 0) monitorizada por microscopia de fluorescência (GFP - fluorescência verde, TdT - fluorescência vermelha) em diferentes Os pontos de tempo. D 2 e 6: escala barras representam 100 mm. D 10: barras de escala representam 200 m. Por favor clique aqui para ver uma versão maior desta figura.
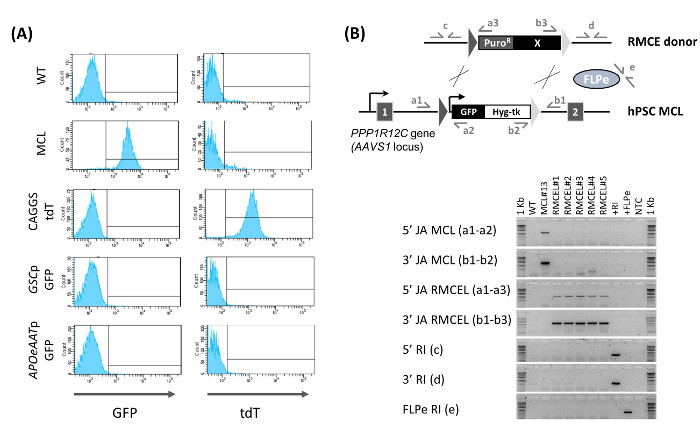
Figura 2:. Caracterização de RMCE Lines (A) Determinação da RMCE porcitometria de fluxo. GFP e expressão TdT é mostrado pela linha WT, linha celular mestre (MCL), e linha RMCE recém-gerado no D 15 pós-nucleofection utilizando um vector constitutivo TdT-expressão (CAGGS TdT), ou doadores com GFP conduzido por um promotor Goosecoid (GSCP GFP, marcador endoderme definitiva) ou um promotor de AAT (APOeAATp GFP, marcador de hepatócitos). (B) A confirmação da RMCE (5 '/ 3' JA RMCEL), ausência de (MCL) cassete da linha de células master (5 '/ 3' JA MCL), ea falta de integração aleatória (5 '/ 3' / FLPe RI ) por PCR utilizando os pares de iniciadores descritos. DNA de WT, MCL, linhas RMCE (1 - 5) foram utilizados, vetor doador RMCE (+ RI), FLPe expressar vector e sem controle de modelo (NTC). Figura modificado a partir de (Ordovas et al., 2015) 13. Por favor clique aqui para ver uma versão maior desta figura.
dentro-page = "1"> 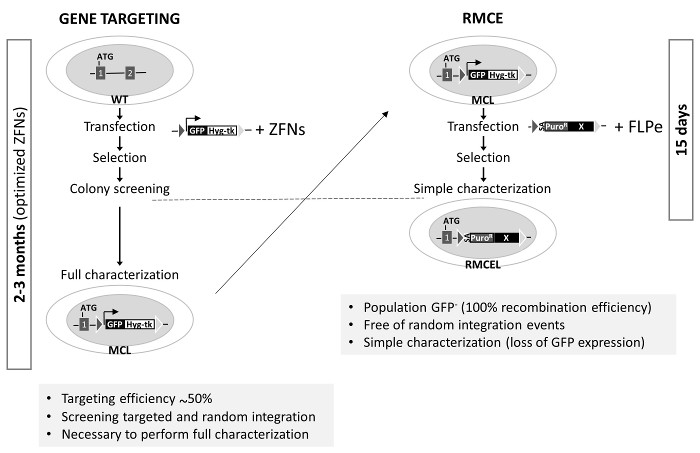
Figura 3. geração da linha celular mestre-RMCE adequado e Comparação de Gene Targeting com RMCE em AAVS1 Esquerda:. Direccionamento de genes utiliza células WT não modificados que são co-transfectadas com um dador (cassete descrita na Figura 1A para a geração da linha celular mestre , MCL) e ZFNs otimizadas específicas. O processo para completar a caracterização completa demora cerca de 3 meses. Direita: RMCE é realizada por co-transfecção do doador com vectores FLPe, e uma linha totalmente caracterizados é gerado em 15 d. Gene visando eficiência no AAVS1 é a partir de estudos anteriores 1,2. Por favor clique aqui para ver uma versão maior desta figura.
Tabela 1. Composição de mídia.
| Ensaio | para a frente | Reverso | amplicon | Ciclo de PCR | ||
| 5'JA MCL | CACTTTGAGCTCTACTGGCTTC | CGTTACTATGGGAACATACGTCA | 1.1 Kb | 95 ºC, 5 '- [95 ºC, 30' '- 68 ºC (-0,5 ºC / ciclo), 1' 30 ''] X15 - [95 ° C, 30 '' - 58 ° C, 30 '' - 72 ° C, 1 '30' '] X25 - 72 ° C, 5' | ||
| 3'JA MCL | TAACTGAAACACGGAAGGAG | AAGGCAGCCTGGTAGACA | 1.4 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 68 ° C (-0,5 ° C / ciclo), 1' 30 ''] X15 - [95 ° C, 30 '' - 58 ° C, 30 '' - 72 ° C, 1 '30' '] X25 - 72 ºC, 5' | ||
| 5'JA RMCEL | CACTTTGAGCTCTACTGGCTTC | CATGTTAGAAGACTTCCTCTGC | 1.1 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 68 ° C (-0,5 ° C / ciclo), 1' 30 ''] X15 - [95 ° C, 30 '' - 58 ° C, 30 '' -72 ºC, 1 '30' '] X25 - 72 ºC, 5' | ||
| 3'JA RMCEL | TTCACTGCATTCTAGTTGTGG | AAGGCAGCCTGGTAGACA | 1,5 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 68 ° C (-0,5 ° C / ciclo), 1' 30 ''] X15 - [95 ° C, 30 '' - 58 ° C, 30 '' - 72 ° C, 1 '30' '] X25 - 72 ºC, 5' | ||
| 5'RI DOADOR | GTACTTTGGGGTTGTCCAG | TTGTAAAACGACGGCCAG | 0,5 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 60 ° C, 30' '- 72 ° C, 30' '] X25 - 72 ° C, 5' | ||
| 3'RI DOADOR | CCTGAGTTCTAACTTTGGCTC | ACACAGGAAACAGCTATGAC | 0,5 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 60 ° C, 30' '- 72 ° C, 30' '] X25 - 72 ° C, 5' | ||
| RI FLPe | CCTAGCTACTTTCATCAATTGTG | GTATGCTTCCTTCAGCACTAC | 0.65 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 60 ° C, 30' '- 72 ° C, 30' '] X25 - 72 ° C, 5' | ||
Tabela 2. Conjuntos de iniciadores utilizados para PCR Genotipagem. Modificado de Ordovas et al., 2015 13.
Discussão
métodos de edição Gene em loci porto seguro continuam a ser uma ferramenta essencial para desenvolver transgenia em hPSCs. Embora o personagem porto seguro de AAVS1 recentemente tem sido questionada por alguns aplicativos 13,18, esse locus permanece atualmente o melhor caracterizada em linhas de células de origem humana. Consciência das suas limitações em hPSCs pode ajudar a obter dados fiáveis. Portanto, AAVS1 ainda deverá ser um local útil, por exemplo, para estudos gain- e perda de função ou expressão indutível / constitutivo de factores que exigem um contexto isogénica dentro e entre os fundos genéticos determinados.
O locus AAVS1 tem sido alvo de muitos grupos usando ZFNs, TALEN ou CRISPR / Cas9 1,2,19. Estas nucleases aumentar significativamente a eficiência de recombinação homóloga no locus de um determinado. No entanto, o processo para rastrear e caracterizar plenamente clones corretamente direcionados, livres de integ aleatórioração e para manter a pluripotência e genoma integridade pode levar até 3 meses (utilizando ferramentas de edição gene Optimized) (Figura 3). Os dois últimos pode ser causada por mutações possíveis gerados pela actividade da nuclease fora do alvo. O sistema RMCE utilizado aqui, no entanto, oferece um método rápido, eficiente, e elegante para atingir este objectivo em apenas duas semanas, uma vez que as sequências alvo de recombinase específica do são pré-integrado dentro do locus de AAVS1. Uso de selecção positiva / negativa e um programa de seleção adequada são os principais fatores que contribuem para a simplificação da edição gene no locus AAVS1 por RMCE.
Caracterização genotípica das novas linhagens transgênicas é significativamente reduzida (sem triagem clonal é necessário), e caracterização associada à atividade nuclease fora do alvo é processado dispensável devido à especificidade de FLPe para FRT. A caracterização também pode ser reduzida em experiências de rotina por RMCE demonstrando aperda completa da expressão de GFP a partir da linha de células de referência (Figura 2A), porque a troca de cassete completo e falta de integração aleatória foram já suficientemente demonstrado por PCR (Figura 2B) e 13 de transferência de Southern. O uso de selecção positiva / negativa também constitui a principal diferença com relatórios anteriores. Vários grupos que anteriormente descritos em RMCE hPSCs, quer na AAVS1 ou outros loci, empregue apenas um único passo de selecção positiva 7,14,16. Isto não constitui uma grande vantagem técnica sobre a edição do gene realizada com nucleases, porque o rastreio colónia tem de ser igualmente realizada para demonstrar a integração e a ausência de integração aleatória correcta ao nível clonal.
A utilização deste sistema RMCE em várias linhas de células estaminais embrionárias humanas / IPSC requer pré-integração do descrito FRT contendo cassete no locus AAVS1 de cada linha independente. No entanto, uma vez gerado,sua rapidez e simplicidade faz com que seja possível desenvolver semi-alto rendimento telas genéticos em configurações isogénicas definidos para as aplicações acima mencionadas que de outra forma seria tecnicamente muito demorado. Além disso, todos os vectores de RMCE são construídas dentro do vector de direccionamento do gene pZ AAVS1 utilizado para segmentar o locus com ZFNs, mas TALENS ou CRISPR / Cas9 também foram relatados. dualidade do vector RMCE é muito útil para gerar múltiplas linhas para um determinado transgene em hPSCs com ou sem FRT. Por exemplo, uma batida demonstrado sucesso em uma tela genética determinado realizada por RMCE seria idealmente devem ser testados em várias linhas HPSC para confirmar os resultados. Na ausência de várias linhas de células mestras RMCE-adequada, directa do gene segmentação do sucesso utilizando nucleases pode ser realizada. A utilidade do caráter dual dos vetores RMCE foi provado pelo nosso grupo 20. Neste estudo, a correcção de genes foi efectuada em paciente derivadoA demência frontotemporal (FTD) iPSCs que carregam uma mutação que causa deficiência de expressão progranulina (PGRN). Gene complementação pela inserção ZFNs-mediadas no locus AAVS1 de uma cassete que restabeleceu os níveis PGRN, corrigiu o fenótipo defeituoso em corticogenesis associada com a presença da mutação. Além disso, uma linha comparável foi gerado por RMCE em hESCs WT para ser utilizado como controlo para a expressão PGRN exógeno.
Na ausência de colónias recombinantes, verificar a eficiência de transfecção do vector RMCE dador. eficiências de transfecção superior a 30% deu os resultados indicados acima. Menor nível de eficiência de transfecção diminuir a eficiência de recombinação. Finalmente, o sistema RMCE foi gerado no locus AAVS1, mas é aplicável a qualquer outro local.
Divulgações
The authors have nothing to disclose.
Agradecimentos
L.O. was funded by IWT/OZM/090838, IACS BPAMER3/08/04, and the Government of Aragon FMI048/08; RB by IWT fellowship SB-121396; M.P. by FWO 1288714N; and R.S. by the Dutch Diabetes Foundation. N.H., J.V., K.C., and Q.C. were supported by IWT fellowships SB-121396, SB-101230, SB-91228, and SB-093228, respectively. Funding to C.M.V. was from FWO G.0667.07, G.0975.11, and KU Leuven (EIW-B4855-EF/05/11, ETH-C1900-PF, EME-C2161-GOA/11/012), IWT-HEPSTEM, BELSPO-IUAP-DEVREPAIR, FP7-HEMIBIO (266777).
Materiais
| Name | Company | Catalog Number | Comments |
| DR4 Mouse Embryonic Fibroblasts (MEF) | AMS bio | GSC-6204G | hPSC culture on feeders |
| CF-1 MEF, mitomycin-C treated | AMS Bio | GSC-6001M | |
| EmbryoMax 0.1% Gelatin Solution | Millipore | ES-006-B | iMEF plating |
| DMEM/F-12, HEPES | Gibco | 31330-038 | |
| KnockOut Serum Replacement | Gibco | 10828-028 | |
| L-Glutamine | Sigma Aldrich | G8540 | For hESC medium |
| L-Glutamine, 200 mM | Gibco | 25030-081 | For iMEF medium |
| 2-Mercaptoethanol, 50 mM | Gibco | 31350-010 | |
| MEM Non-Essential Amino Acids Solution (100x) | Gibco | 11140-035 | |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140122 | |
| Human basic FGF | Peprotech | 100-18C | |
| Y-27632 in Solution | VWR | 688001-500 | |
| DMEM High Glucose | Gibco | 41965-039 | |
| Fetal Bovine Serum (FBS) | Sigma Aldrich | F7524 | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300-054 | hPSC culture on feeders |
| Matrigel, hESC qualified | VWR | 734-1440 | |
| mTeSR1 complete medium kit | Stem cell technologies | 05850 | For Feeder-free culture |
| StemPro Accutase Cell Dissociation Reagent | Gibco | A1110501 | For feeder-free culture |
| Y-27632 in solution | VWR | 56726 | |
| Human Stem Cell Nucleofector Kit 2 | Lonza | VVPH-5022 | |
| Puromycine Dihydrochloride | Gibco | A11138-03 | |
| FIAU (Fialuridine) | ABX | 2910 | |
| Hygromycin B | Sigma Aldrich | H3274 | |
| pFLPe | Modified to from pCAGGS-FLPe (MES4488, Open Biosystems) to remove puromycin | ||
| RMCE donors | - | - | Modified from pZ donor AAVS1 puromycin vector (PZD0020-1KT, Sigma Aldrich)13 |
| 5 mL Round Bottom Polystyrene Test Tube, with Cell Strainer | Falcon | 352235 | |
| PureLink Genomic DNA kit | Invitrogen | K182001 | |
| GoTaq Flexi DNA Polymerase | Promega | M8298 | |
| Agarose | Sigma | A9539 | |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | |
| Nucleofector 2b Device | Lonza (Amaxa) | AAB-1001 | |
| NucleoCounter NC-100 | Chemometec | NC-100 | |
| BD FACS Canto | BD Biosciences | 337175 | |
| NucleoCassette | Chemometec | 941-0002 |
Referências
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nat. Biotechnol. 29 (8), 731-734 (2011).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nat. Biotechnol. 27 (9), 851-857 (2009).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Baer, A., Bode, J. Coping with kinetic and thermodynamic barriers: RMCE, an efficient strategy for the targeted integration of transgenes. Curr. Opin. Biotechnol. 12 (5), 473-480 (2001).
- Sakurai, K., et al. Efficient integration of transgenes into a defined locus in human embryonic stem cells. Nucleic Acids Res. 38 (7), e96(2010).
- Irion, S., et al. Identification and targeting of the ROSA26 locus in human embryonic stem cells. Nat. Biotechnol. 25 (12), 1477-1482 (2007).
- Di Domenico, A. I., Christodoulou, I., Pells, S. C., McWhir, J., Thomson, A. J. Sequential genetic modification of the hprt locus in human ESCs combining gene targeting and recombinase-mediated cassette exchange. Cloning Stem Cells. 10 (2), 217-230 (2008).
- Lombardo, A., et al. Site-specific integration and tailoring of cassette design for sustainable gene transfer. Nat. Methods. 8 (10), 861-869 (2011).
- Lombardo, A., et al. Gene editing in human stem cells using zinc finger nucleases and integrase-defective lentiviral vector delivery. Nat. Biotechnol. 25 (11), 1298-1306 (2007).
- Simth, J. R., et al. Robust , Persistent Transgene Expression in Human Embryonic Stem Cells Is Achieved with AAVS1-Targeted Integration. Stem Cells. 26, 496-504 (2008).
- DeKelver, R. C., et al. Functional genomics, proteomics, and regulatory DNA analysis in isogenic settings using zinc finger nuclease-driven transgenesis into a safe harbor locus in the human genome. Genome Res. 20 (8), 1133-1142 (2010).
- Qian, K., et al. A simple and efficient system for regulating gene expression in human pluripotent stem cells and derivatives. Stem Cells. 32 (5), 1230-1238 (2014).
- Ordovás, L., et al. Efficient Recombinase-Mediated Cassette Exchange in hPSCs to Study the Hepatocyte Lineage Reveals AAVS1 Locus-Mediated Transgene Inhibition. Stem Cell Reports. 5 (5), 918-931 (2015).
- Du, Z. -W., Hu, B. -Y., Ayala, M., Sauer, B., Zhang, S. -C. Cre recombination-mediated cassette exchange for building versatile transgenic human Embryonic Stem Cells lines. Stem Cells. 27, 1032-1041 (2009).
- Papapetrou, E. P., et al. Genomic safe harbors permit high β-globin transgene expression in thalassemia induced pluripotent stem cells. Nat. Biotechnol. 29 (1), 73-78 (2011).
- Ramachandra, C. J., et al. Efficient recombinase-mediated cassette exchange at the AAVS1 locus in human embryonic stem cells using baculoviral vectors. Nucleic Acids Res. 39 (16), e107(2011).
- Takata, Y., Kondo, S., Goda, N., Kanegae, Y., Saito, I. Comparison of efficiency between FLPe and Cre for recombinase-mediated cassette exchange in vitro and in adenovirus vector production. Genes to Cells. 16 (7), 765-777 (2011).
- Mizutani, T., Li, R., Haga, H., Kawabata, K. Transgene integration into the human AAVS1 locus enhances myosin II-dependent contractile force by reducing expression of myosin binding subunit 85. Biochem. Biophys. Res. Commun. 465 (2), 270-274 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Raitano, S., et al. Restoration of progranulin expression rescues cortical neuron generation in an induced pluripotent stem cell model of frontotemporal dementia. Stem Cell Reports. 4 (1), 16-24 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados